È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Identificazione e analisi dei mouse progenitori eritroidi utilizzando il CD71/TER119 Flow-citometria Assay
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Un metodo di citometria a flusso per l'identificazione e l'analisi molecolare di cellule progenitrici murino differenziazione fase-specifici eritroide e precursori, direttamente nel midollo del mouse appena raccolte osseo, milza o del fegato fetale. Il test si basa sulla superficie cellulare marker CD71, Ter119, e le dimensioni delle cellule.
Abstract
Lo studio dell'eritropoiesi si propone di capire come globuli rossi sono formate da progenitori ematopoietici in precedenza e eritroide. In particolare, il tasso di formazione dei globuli rossi è regolata dalla ormone eritropoietina (Epo), la cui sintesi è innescata da ipossia tissutale. Una minaccia a risultati adeguati ossigenazione dei tessuti in un rapido aumento Epo, guidando un aumento della frequenza eritropoietica, un processo noto come la risposta allo stress eritropoietica. Il conseguente aumento del numero dei globuli rossi circolanti aumenta l'ossigeno di consegna dei tessuti. Una risposta efficace dello stress eritropoietica è quindi fondamentale per la sopravvivenza e il recupero da condizioni fisiologiche e patologiche quali l'altitudine elevata, anemia, emorragia, la chemioterapia o il trapianto di cellule staminali.
Il mouse è un modello fondamentale per lo studio della eritropoiesi e la sua risposta allo stress. Eritropoiesi topo definitiva (di tipo adulto) avviene nel fegato fetale tra i giorni embrionale 12,5 e 15,5, nella milza neonatale, e nella milza e nel midollo osseo degli adulti. Metodi classici di identificazione progenitori eritroidi nel tessuto si basano sulla capacità di queste cellule per dare origine a colonie di globuli rossi quando placcato in Epo contenenti semi-solido dei media. Loro progenie precursore eritroidi sono identificate sulla base di criteri morfologici. Nessuno di questi metodi classici consentire l'accesso a un gran numero di differenziazione-stadio-specifiche cellule eritroidi per lo studio molecolare. Qui vi presentiamo un flusso-citometria metodo di identificazione e differenziazione studio-stadio-specifici progenitori eritroidi e precursori, direttamente nel contesto del tessuto del mouse appena isolato. Il test si basa sulla superficie cellulare marker CD71, Ter119, e sul flusso-citometria a parametro 'avanti dispersione', che è una funzione della dimensione della cella. Il CD71/Ter119 test può essere utilizzato per studiare progenitori eritroidi durante la loro risposta allo stress eritropoietica in vivo, per esempio, nei topi anemici o nei topi tenuti in condizioni di scarsità di ossigeno. Può anche essere usata per studiare progenitori eritroidi direttamente nei tessuti di topi adulti geneticamente modificati o embrioni, al fine di valutare il ruolo specifico del pathway molecolare modificata in eritropoiesi.
Protocollo
1. La raccolta dei tessuti
- Preparare provette contenenti 2-5 buffer di colorazione fredda ml (soluzione salina tampone fosfato (PBS) con aggiunta di 0,2% di BSA e glucosio 5mm). Sì che le tubature in ghiaccio prima del raccolto dei tessuti.
- Cull topi in accordo con il protocollo approvato (CO es. 2 inalazione seguita da dislocazione cervicale).
- Prelievo di sangue da puntura cardiaca in EDTA o eparina tubi di raccolta del sangue per analisi successive, ad esempio, di ematocrito, conta dei reticolociti o analisi CBC.
- Raccolta la milza e le ossa, ponendo tessuti da ogni mouse in un tubo separato, preparato al punto 1. Facile accesso a milza è dal lato sinistro. Per il midollo osseo, la raccolta di uno o entrambi i femori. Mantenere il tessuto raccolte sul ghiaccio.
- Se lo si desidera, pesare la milza. I topi sottoposti a una risposta eritropoietica lo stress è probabile che presentino un significativo aumento del peso della milza.
2. Preparazione di cellule della milza
- Utilizzando un pre-umidificata 3 ml stantuffo della siringa, spingere delicatamente la milza, o una parte della milza (idealmente, 0,1 grammi o meno, pari a circa 10 8 cellule) attraverso un colino 40 micron cellula sterile (numero di catalogo Fisherbrand 22363547 o altro) collocato su un tubo da 50 ml. Tenere la provetta in ghiaccio durante questa procedura. Lavare le cellule attraverso il filtro con un totale di 2 ml di tampone colorazione.
- Pipetta delicatamente la sospensione forzata delle cellule per rompere eventuali grumi di piccole dimensioni. Se necessario, ri-deformazione delle cellule.
- Lavare le cellule due volte tramite centrifugazione e ri-sospendere in tampone a freddo.
- Contare le cellule utilizzando un emocitometro. Le rese sono 1-2 x 10 8 cellule / milza. Per le analisi di citometria a flusso, aliquote 1-2000000 cellule per campione, sia in tubi colorazione FACS (BD polistirolo Falcon fondo rotondo tubi, 352008) o U-bottom 96 pozzetti (BD Falcon 353910). Colorazione volume del campione è di 200 microlitri, ad una concentrazione cellulare finale ,5-1 x 10 7 cellule / ml o 1-2 x 10 6 cellule / 200 microlitri del campione.
3. Preparazione di cellule di midollo osseo
- Preparare 1 o 3 ml siringhe con un ago inserito 26G, pre-riempita con tampone colorazione fredda.
- Rimuovere i muscoli collegati al femore in modo da visualizzare l'osso in modo chiaro.
- Utilizzando taglienti forbici chirurgiche, snip fuori entrambe le estremità del femore, il più vicino possibile alle estremità delle ossa. Questo dovrebbe rivelare un piccolo foro alle estremità tagliata, leader nella cavità del midollo osseo, che attraversa la lunghezza del femore.
- Utilizzando il siringhe pre-riempite al punto 1, inserire l'ago attraverso uno di questi fori, e delicatamente a filo il midollo attraverso il buco dall'altra parte, in un tubo.
- Dissociare le cellule lavata pipettando dolce, e filtrare con un colino 40 micron, come per la milza superiori (vedere paragrafo 2.1).
- Lavare le cellule due volte tramite centrifugazione in tampone colorazione fredda
- Contare le cellule e risospendere come nella sezione 2.4. Le rese sono circa 10 7 cellule per femore.
4. Preparazione delle cellule del fegato fetale
- Per preparare topi di sesso femminile a tempo-incinta, istituito topi per l'accoppiamento, la sera, per esaminare le spine vaginale entro le ore 10 del giorno successivo, il giorno in cui viene rilevata la spina vaginale è considerato giorno 0,5. Il personale veterinario sia in grado di assistere i ricercatori che non hanno familiarità con questa tecnica per identificare le donne in gravidanza. Gravidanza può essere determinata anche in peso di monitoraggio del mouse.
- Topi femmina incinte sono scadute abbattuti nei giorni 12,5-14,5 di gravidanza. Le corna uterine vengono rimossi in una Petri-piatto contenente ghiacciata terreno di coltura o di buffer colorazione.
- Gli embrioni sono stati rimossi da ogni dell'utero e del fegato fetale è sezionato. Un microscopio da dissezione è richiesto per il giorno embrionale 12,5 (E12.5) o più giovani.
- Fegato può essere dissociato meccanicamente pipettando nel buffer, e sono trattati singolarmente in piastre a 96 pozzetti, o in pool insieme, a seconda delle esigenze sperimentali.
- Un fegato fetale a E13.5 ha ~ 10 7 cellule. Le cellule vengono lavate due volte in colorazione buffer e risospese a 1-2 x 10 6 cellule / 200 microlitri del campione per l'analisi di citometria di flusso.
5. Colorazione anticorpi per citometria a flusso
- Preparare un anticorpo primario colorazione pre-mix da utilizzare per tutti i campioni, ad eccezione di campioni di controllo, contenente quanto segue:
- ChromePure coniglio IgG (Jackson, 015-000-003), ad una concentrazione finale di 200μg/ml. Controllare la concentrazione magazzino sulla bottiglia (che può variare). Questo è usato per bloccare i recettori Fc in cellule di topo, specie alternative che possono essere utilizzati per questo sono IgG di topo o ratto IgG. Scelta delle specie è determinata dalla potenziale presenza nel protocollo di colorazione di anticorpi diretti contro secondaria primari di ratto / topo / coniglio anticorpi, nel qual caso tali specie non può essere utilizzatod come anticorpi bloccanti. In alternativa, siero di coniglio 5% può inoltre essere utilizzato al posto di IgG purificate. Un'ulteriore alternativa è l'uso di anticorpi monoclonali o frammenti Fab diretto a livello dei recettori Fc mouse. Il protocollo di base di seguito non include alcun anticorpi IgG secondario e quindi di una qualsiasi delle tre specie possono essere utilizzati.
- CD71-FITC, diluito 1:200 (stock 0.5mg/ml, BD-Biosciences, 553266)
- Ter119-PE, diluito 1:200 (stock 0.2mg/ml, BD-Biosciences, 553673)
- Eventuali ulteriori anticorpi diretti a epitopi superficie di interesse, ad esempio, gli anticorpi diretti al Fas o FasL (vedi 1,2). Mescolare la soluzione di anticorpi delicatamente invertendo il tubo di 2-3 volte.
- Aggiungere 200 ml di pre-mix per ogni campione cellulare e risospendere delicatamente le cellule.
- Preparare i campioni di cellule di controllo come segue:
- 'Non colorati': queste cellule vengono lasciate in colorazione tampone e di fornire l'autofluorescenza di sfondo delle celle.
- Controlli 'monocolore': un tale controllo è richiesto per ogni anticorpo / colore utilizzato nel protocollo. Le cellule di questi controlli sono macchiati o con un anticorpo coniugato direttamente primario, o sia con un anticorpo primario e un anticorpo secondario coniugato. Questi controlli vengono utilizzati per correggere sovrapposizione spettrale tra i canali.
- 'Fluorescenza meno uno' (FMO) controlli: un controllo tale è richiesto per ogni anticorpo / colore nel protocollo: le cellule sono colorate con tutti i colori / anticorpi nel protocollo, tranne per il colore / anticorpo per cui questo è il controllo FMO. Il controllo QOR per un particolare canale fornisce lo sfondo vero per quel canale. Può includere non specifici anticorpi dello stesso isotipo e coniugato con lo stesso marchio fluorescenza come l'anticorpo test (controllo isotipico).
- Incubare i campioni ed i controlli rilevanti nel anticorpo primario macchia in ghiaccio per 45 'a 1 ora al buio (messo foglio di copertura in alluminio su ghiaccio secchio).
- Alla fine di incubazione, lavare le cellule con l'aggiunta di 3 ml di tampone colorazione ad ogni provetta e girare per 3 'a 5' a circa 400 xga 4 ° C. Se si utilizza piastre a 96 pozzetti, lavare le cellule tre volte in un volume di 200 l.
- Se del caso, applicare anticorpo secondario macchia. Applicare e lavare come per il primo anticorpo macchia.
- Se del caso, una macchia con annessina V è applicata alla fine di incubazione, utilizzando un buffer Hepes come nelle istruzioni del produttore. Questa macchia è applicata per 15 minuti a temperatura ambiente, o per 1 ora su ghiaccio.
- Le cellule sono nuovamente sospesi per l'analisi di citometria a flusso nella colorazione soluzione contenente una cella-impermeabile colorante del DNA, di escludere le cellule morte. Coloranti del DNA sono disponibili diversi, tra cui ioduro di propidio, 7-amino-actinomicina D (7AAD), o DAPI. La scelta di questi dipende, tra i canali disponibili, dato i canali ripreso per la colorazione degli anticorpi specifici e canali disponibili sul citometro a flusso. 7AAD è ottenuto da BD-Biosciences (559.925) e utilizzato secondo le istruzioni del produttore. Per la colorazione DAPI, fare uno stock di 1mg/ml in dimetilformammide (DMF) da polvere (Roche, Cat # 236276), tenere a -20 ° C e diluire 1:10.000 a 1:15.000 in colorazione buffer.
6. Citometria a flusso ordinamento
- Le cellule sono etichettati con anticorpi anti-CD71, Ter119, marcatori lignaggio, e un colorante vitalità come descritto per analisi di citometria di flusso (sezione 5). Concentrazione delle cellule durante l'etichettatura può essere aumentata a 5 x 10 7 / ml.
- Mescolare un uguale volume di ciascuno dei seguenti anticorpi per rendere il master mix lignaggio:
- FITC Rat anti-topo CD41 MWReg30, BD Pharmingen 553848
- FITC Rat anti-topo CD45R/B220 RA3-6b2, BD Pharmingen 553087
- Hamster FITC anti-topo CD3e 145-2C11, BD Pharmingen 553061
- FITC Rat anti-topo CD11b/Mac-1 M1/70, BD Pharmingen 557396
- FITC Rat anti-topo Ly-6G e Ly-6C (Gr-1) RB6-8C5, BD Pharmingen 553126
- Utilizzare il master mix a 1:80 (Questo è equivalente a diluizione 1:400 di ciascun individuo titoli di anticorpi, che sono tutti 0,5 mg / ml).
- Usa bassa pressione condizioni di ordinamento e ugelli di larghezza. Per l'Aria (BD Biosciences) che usiamo 100 ugelli μ, 20 psi di pressione.
- Collezione buffer: PBS con aggiunta del 20% siero fetale bovino.
- Per controllare la purezza delle popolazioni in ordine, ri-eseguire una piccola aliquota di ogni campione in un buffer che contiene un colorante vitalità (7AAD o simili).
7. Rappresentante dei risultati:
CD71/Ter119 colorazione degli adulti di midollo osseo o milza identifica una sequenza evolutiva di quattro sottoinsiemi, etichettati ProE, EryA, EryB e Eryc (Figura 1) 1. Morfologicamente, questi corrispondono a eritroblasti sempre più maturo. La figura 1 illustra la sequenza di gating in fase di analisi dei dati, che scarta evento molto piccoli (tra cui nuclei, globuli rossi), le cellule aggregati e cellule morte.
Espressione di proteine di superficie cellulare possono essere misurate simultaneamente per ognuno di questi sottoinsiemi, aggiungendo gli anticorpi relativi al tempo stesso Ter119 e CD71 colorazione. La figura 1 mostra un esempio di espressione sulla superficie cellulare del recettore Fas morte 1. Questa misurazione è stata effettuata in topi trattati con Epo, o nei topi di controllo iniettati con soluzione salina. E 'evidente che Epo sopprime l'espressione di Fas nella popolazione EryA in vivo 1.
Espressione di proteine intracellulari o ciclo cellulare lo stato può anche essere misurata per le cellule in ogni sottogruppo. La figura 2 illustra rappresentante analisi del ciclo cellulare delle cellule del midollo osseo appena raccolte. Queste misure richiedono, oltre alla colorazione della superficie delle cellule con CD71 e Ter119, la fissazione e la permeabilizzazione delle cellule per l'etichettatura intracellulare (vedere la sezione di discussione).
Nel fegato fetale, non eritroidi cellule vengono prima esclusi da gating su 'Lin-' cellule che sono negative per CD41, Mac-1, Gr-1, B220 e CD3 (Figura 3). Le cellule rimanenti sono suddivisi in 6 sottogruppi, S0 a S5. Il modello preciso di cellule nel fegato fetale dipende dall'età embrionale (vedere la sezione di discussione). Un rappresentante di analisi del ciclo cellulare del sottoinsieme S3 in E13.5 fegato fetale viene visualizzato (Figura 4).
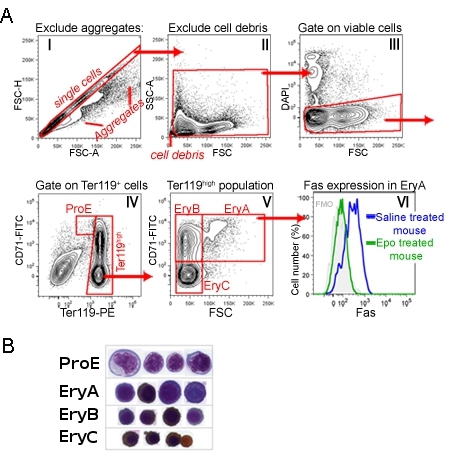
Figura 1 CD71/Ter119 sottoinsiemi eritroide nella milza del mouse strategia Gating A.:.. Le cellule della milza sono stati elaborati ed etichettato con anticorpi diretti a CD71, Ter119 e Fas. Questa figura mostra la strategia di analisi che segue la fase di acquisizione dati. Istogramma I mostra tutti gli eventi acquisiti. Il cancello diagonale rappresenta gli eventi che possono essere singole cellule, escluse le doppiette o più grandi aggregati. Le cellule in questa porta verranno analizzati nella istogramma II. Eventi qui molto piccoli, nuclei probabile o detriti, sono esclusi. Le cellule gated sono mostrati in istogramma III, in cui sono esclusi DAPI cellule positive, che sono probabilmente cellule apoptotiche membrana permeabile, da ulteriori analisi. Istogramma IV mostra la popolazione risultante di cellule della milza vitali. Il cancello ProE contiene CD71 alto Ter119 cellule intermedie. Ter119 cellule alte sono ulteriormente analizzati in istogramma V. Qui le cellule CD71 elevati sono suddivisi in meno maturi, grandi eritroblasti 'EryA' (CD71 alto Ter119 alta FSC alto) e più piccoli, più maturi 'EryB' eritroblasti (CD71 alto Ter119 alto FSC basso) . Il sottoinsieme eritroblasti più maturi sono Eryc (CD71 bassa Ter119 alta FSC basso). VI istogramma mostra espressione sulla superficie cellulare Fas, in particolare nel sottogruppo EryA, nei topi, nello stato basale (iniettati con soluzione fisiologica), e topi iniettati con una singola dose di Epo. Colorazione con anticorpi Fas è stata effettuata in contemporanea con la colorazione CD71/Ter119. B. Cytospin preparazioni di cellule ordinate da ciascuno dei sottoinsiemi indicati. Le cellule sono state colorate con Giemsa e con diaminobenzidina, quest'ultimo genera una macchia marrone con l'emoglobina. Dati Cytospin è stato originariamente pubblicato nel Liu et al. Sangue. 1 luglio 2006, 108 (1) :123-33. Epub Mar 2006 9.
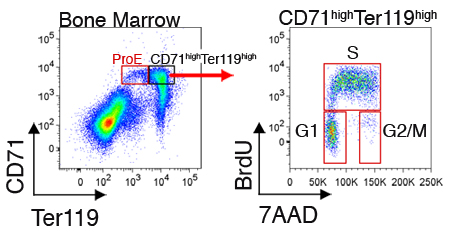
Figura 2. Analisi del ciclo cellulare del CD71 alta Ter119 eritroblasti alto nel midollo osseo dei topi. I topi sono stati iniettati per via intraperitoneale con BrdU, e della milza o del midollo osseo sono state raccolte 30 a 60 minuti dopo. Le cellule sono state fissate e permeabilizzate e oltre ad essere colorate per CD71 e Ter119, sono state colorate per incorporazione di BrdU nel loro DNA la replica con un anticorpo monoclonale diretto a BrdU (protocollo di fissazione, permeabilizzazione e BrdU-colorazione è stata secondo le istruzioni del produttore). BrdU-positive le cellule sono in fase S del ciclo. Cellule interfase sono BrdU-negativi e possono essere risolti in fasi G1 o G2 / M, usando il DNA colorante 7AAD.
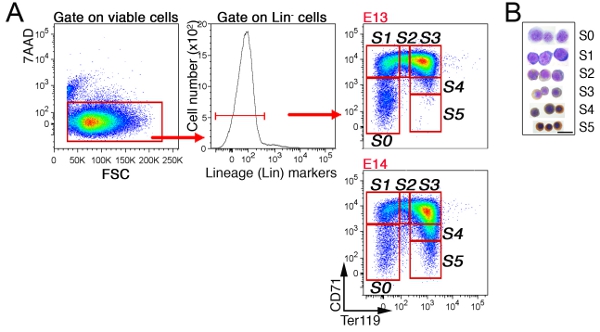
Figura 3 CD71/Ter119 sottoinsiemi eritroide nel fegato fetale topo strategia Gating A.:.. Cellule del fegato fetale sono stati etichettati per CD71, Ter119, e un cocktail di anticorpi coniugati con fluoresceina direttamente i non eritroide marcatori lignaggio ('Lin'). Cellule vitali (7AAD-negativo) sono stati analizzati per l'espressione Lin, e Lin-cellule sono ulteriormente suddivisi in S0 a S5 sottoinsiemi eritroidi. Giovane, E13 fegato fetale è composto da eritroblasti meno mature, dimostra l'assenza di cellule mature in S4/S5 sottoinsiemi. B. Cytospin preparazioni di cellule ordinate da ciascuno dei sottoinsiemi indicati. Le cellule sono state colorate con Giemsa e con diaminobenzidina, quest'ultimo genera una macchia marrone con l'emoglobina. Dati Cytospin originariamente published in Pop et al, PLoS Biol 8 (9):. e1000484. doi: 10.1371/journal.pbio.1000484.
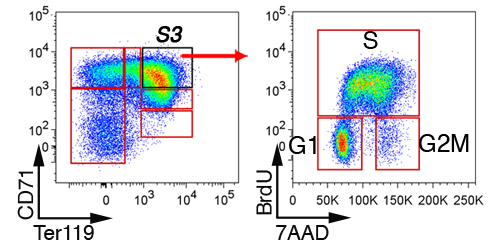
Figura 4. Analisi del ciclo cellulare di sottoinsiemi fegato fetale eritroide. Femmine di topo gravide sono stati iniettati con BrdU e fegato fetale sono state raccolte 30 a 60 minuti dopo, fissa, permeabilizzate e colorate con anticorpi anti-CD71, Ter119 e BrdU. Ciclo cellulare delle cellule stato S3 è mostrato.
Discussione
Il flusso-citometria metodologia permette indagine simultanea di tutte le funzioni cellulari che possono essere rilevati con una fluorescenza coniugato anticorpo specifico o ligando, tra cui marcatori di superficie cellulare, espressione di proteine, la sopravvivenza cellulare, segnalazione cellulare utilizzando anticorpi fosfo-specifici 3 e lo stato del ciclo cellulare. Queste misurazioni possono essere effettuate in ciascuna di un certo numero di differenziazione fase sottoinsiemi specifici, nel contesto di...
Divulgazioni
Gli esperimenti sugli animali sono stati eseguiti in conformità con le linee guida e dei regolamenti previsti dalla University of Massachusetts Medical School IACUC commissione.
Riconoscimenti
Ringraziamo il UMass nucleo citometria a flusso: Richard Konz, Ted Giehl, Barbara Gosselin, Yuehua Gu e Tammy Krupoch. Questo lavoro è stato finanziato dal NIH / NHLBI RO1 HL084168 (MS) e NIH CA T32-130807 (JRS). Risorse di base sostenuto dalla Endocrinologia Diabetes Research Center concedere DK32520 sono stati utilizzati anche.
Materiali
| Name | Company | Catalog Number | Comments |
| Nome del reagente | Azienda | Numero di catalogo | |
|---|---|---|---|
| Fas-biotina | BD Pharmingen | 554256 | |
| Streptavidina-APC | Molecular Probes | S868 | |
| 40 micron filtro cellule sterili | Fisherbrand | 22363547 | |
| Polistirolo a fondo rotondo tubi per la colorazione FACS | BD Falcon | 352008 | |
| U-bottom 96 pozzetti | BD Falcon | 353910 | |
| ChromePure coniglio IgG | Jackson ImmunoResearch | 015-000-003 | |
| CD71-FITC (stock 0.5mg/ml) | BD-Biosciences | 553266 | |
| Ter119-PE (stock 0.2mg/ml) | BD-Biosciences | 553673 | |
| 7AAD | BD-Biosciences | 559925 | |
| DAPI polvere | Roche | 236276 | |
| FITC Rat anti-topo CD41 MWReg30 | BD Pharmingen | 553848 | |
| FITC Rat anti-topo CD45R/B220 RA3-6b2 | BD Pharmingen | 553087 | |
| FITC Rat anti-topo CD411b/Mac-1 M1/70 | BD Pharmingen | 557396 | |
| FITC Rat anti-topo Ly-6G e Ly-6C (Gr-1) RB6-8C5 | BD Pharmingen | 553126 | |
| Hamster FITC anti-topo CD3e 145-2C11 | BD Pharmingen | 553061 | |
| APC BrdU flusso kit | BD Pharmingen | 557892 | |
| Annessina V-biotina | BD Pharmingen | 556418 |
Riferimenti
- Liu, Y. Suppression of Fas-FasL coexpression by erythropoietin mediates erythroblast expansion during the erythropoietic stress response in. 108, 123-133 (2006).
- Socolovsky, M. Negative Autoregulation by FAS Mediates Robust Fetal Erythropoiesis. PLoS Biol. 5, e252-e252 (2007).
- Krutzik, P. O., Hale, M. B., Nolan, G. P. Characterization of the murine immunological signaling network with phosphospecific flow cytometry. J Immunol. 175, 2366-2373 (2005).
- Socolovsky, M. Ineffective erythropoiesis in Stat5a(-/-)5b(-/-) mice due to decreased survival of early erythroblasts. Blood. 98, 3261-3273 (2001).
- Guihard, S. The MAPK ERK1 is a negative regulator of the adult steady-state splenic erythropoiesis. Blood. 115, 3686-3694 (2010).
- Yu, X. An erythroid chaperone that facilitates folding of alpha-globin subunits for hemoglobin synthesis. J Clin Invest. 117, 1856-1865 (2007).
- Chen, M. L. Erythroid dysplasia, megaloblastic anemia, and impaired lymphopoiesis arising from mitochondrial dysfunction. Blood. 114, 4045-4053 (2009).
- Chen, K. Resolving the distinct stages in erythroid differentiation based on dynamic changes in membrane protein expression during erythropoiesis. Proc Natl Acad Sci U S A. 106, 17413-17418 (2009).
- McGrath, K. E., Bushnell, T. P., Palis, J. Multispectral imaging of hematopoietic cells: where flow meets morphology. J Immunol Methods. 336, 91-97 (2008).
- Pop, R. A key commitment step in erythropoiesis is synchronized with the cell cycle clock through mutual inhibition between PU.1 and S-phase progression. PLoS Biol. 8, (2010).
- Borsook, H., Lingrel, J. B., Scaro, J. L., Millette, R. L. Synthesis of haemoglobin in relation to the maturation of erythroid cells. Nature. 196, 347-350 (1962).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon