È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Isolamento dei progenitori dei megacariociti di topo
In questo articolo
Erratum Notice
Riepilogo
Questo metodo descrive la purificazione mediante citometria a flusso di MEP e MKp da topi femori, tibie e ossa pelviche.
Abstract
I megacariociti del midollo osseo sono grandi cellule poliploidi che assicurano la produzione di piastrine nel sangue. Derivano da cellule staminali ematopoietiche attraverso la megacariopoiesi. Le fasi finali di questo processo sono complesse e coinvolgono classicamente i progenitori bipotenti megacariociti-eritrociti (MEP) e i progenitori unipotenti dei megacariociti (MKp). Queste popolazioni precedono la formazione di megacariociti in buona fede e, come tali, il loro isolamento e caratterizzazione potrebbero consentire l'analisi robusta e imparziale della formazione di megacariociti. Questo protocollo presenta in dettaglio la procedura per raccogliere cellule ematopoietiche dal midollo osseo di topo, l'arricchimento dei progenitori ematopoietici attraverso l'esaurimento magnetico e infine una strategia di selezione cellulare che produce popolazioni MEP e MKp altamente purificate. In primo luogo, le cellule del midollo osseo vengono raccolte dal femore, dalla tibia e anche dalla cresta iliaca, un osso che contiene un numero elevato di progenitori ematopoietici. L'uso di ossa della cresta iliaca aumenta drasticamente il numero totale di cellule ottenute per topo e contribuisce quindi a un uso più etico degli animali. Un esaurimento del lignaggio magnetico è stato ottimizzato utilizzando perle magnetiche a 450 nm che consentono una selezione cellulare molto efficiente mediante citometria a flusso. Infine, il protocollo presenta la strategia di etichettatura e gating per la cernita delle due popolazioni progenitrici di megacariociti altamente purificate: MEP (Lin-Sca-1-c-Kit+CD16/32-CD150+CD9dim) e MKp (Lin- Sca-1-c-Kit+CD16/32-CD150+CD9bright ). Questa tecnica è facile da implementare e fornisce materiale cellulare sufficiente per eseguire i) caratterizzazione molecolare per una conoscenza più approfondita della loro identità e biologia, ii) saggi di differenziazione in vitro, che forniranno una migliore comprensione dei meccanismi di maturazione dei megacariociti, o iii) modelli in vitro di interazione con il loro microambiente.
Introduzione
Le piastrine del sangue sono prodotte dai megacariociti. Queste grandi cellule poliploidi si trovano nel midollo osseo e come per tutte le cellule del sangue sono derivate da cellule staminali ematopoietiche (HSC)1. La via classica di produzione dei megacariociti nel midollo osseo ha origine da HSC e prevede la generazione di diversi progenitori che limitano progressivamente il loro potenziale di differenziazione2. Il primo progenitore che firma l'impegno per la linea megacariocitica è il Megakaryocyte-Erythrocyte Progenitor (MEP), un progenitore bipotente in grado di produrre sia cellule eritroidi che megacariociti3,4,5. Il MEP produce quindi un progenitore/precursore unipotente (MKp) che si differenzierà in un megacariocita maturo in grado di produrre piastrine. I meccanismi coinvolti nella generazione di questi progenitori, così come la loro differenziazione e maturazione in megacariociti sono complessi e solo parzialmente compresi. Inoltre, l'eterogeneità della popolazione MEP in termini di potenziale di differenziazione e il livello di impegno intrinseco di queste cellule non sono ancora chiari. Per decifrare questi processi, è essenziale ottenere (o avere accesso a) popolazioni purificate di MEP e MKp per analisi molecolari fini e monocellulari.
Diversi studi hanno dimostrato particolari combinazioni di marcatori di superficie cellulare per l'identificazione di progenitori impegnati nel lignaggio megacariocitico nel topo6,7,8. Da questi è stato ideato un metodo che consente la purificazione di MEP e MKp dai topi. Questo metodo è stato ottimizzato per ottenere cellule in numero e qualità adeguati per un gran numero di saggi. Con considerazioni etiche in mente, e al fine di ridurre al minimo il numero di animali coinvolti negli esperimenti, abbiamo indotto a raccogliere il midollo osseo dal femore e dalla tibia, e anche dalla cresta iliaca. Questo osso contiene un'alta frequenza e numero di progenitori ematopoietici ed è il più delle volte danneggiato durante la raccolta delle ossa lunghe. Presentato qui è un metodo dettagliato per la raccolta affidabile di questo osso.
Il secondo criterio di ottimizzazione è quello di produrre popolazioni cellulari altamente purificate. Fluorescent Activated Cell Sorting (FACS) è un metodo di scelta al fine di ottenere popolazioni purificate di cellule di interesse. Tuttavia, i bassi rendimenti vengono raggiunti quando la popolazione cellulare di interesse è molto rara. Sono quindi necessarie procedure di arricchimento. In questo protocollo, è stata optata per una procedura di selezione negativa utilizzando perle magnetiche.
Access restricted. Please log in or start a trial to view this content.
Protocollo
I protocolli che coinvolgono animali sono stati eseguiti in conformità con il Comitato CREMEAS per l'etica degli esperimenti sugli animali dell'Università di Strasburgo (Comité Régional d'Ethique en Matière d'Expérimentation Animale Strasbourg). Numero di permesso: E67-482-10).
1. Raccolta delle ossa del mouse
- Sacrificare l'animale nel rispetto delle linee guida istituzionali.
NOTA: I dati presentati in questo manoscritto sono stati ottenuti da topi C57Bl/6 di età da 8 a 12 settimane. Il numero di cellule ottenute e la frequenza delle popolazioni citate possono variare con l'età e il ceppo di topo. - Spruzzare il corpo con il 70% di etanolo.
- Usando le forbici, fai un'incisione di 0,5-1 cm della pelle perpendicolare alla colonna vertebrale e strappa la pelle intorno a tutto il corpo. Tirare giù la pelle dalla parte inferiore del corpo e rimuovere la pelle.
- Posiziona l'animale sul pad di dissezione, a faccia in giù. Individua le ossa pelviche facendo scorrere le dita lungo la colonna vertebrale esposta dall'alto verso il basso. Per localizzare la cresta iliaca, identificare la piccola protuberanza nella regione lombare vicino agli arti posteriori (la regione anterosuperiora dell'osso pelvico). La Figura 1A,B presenta una rappresentazione schematica dell'anatomia del topo.
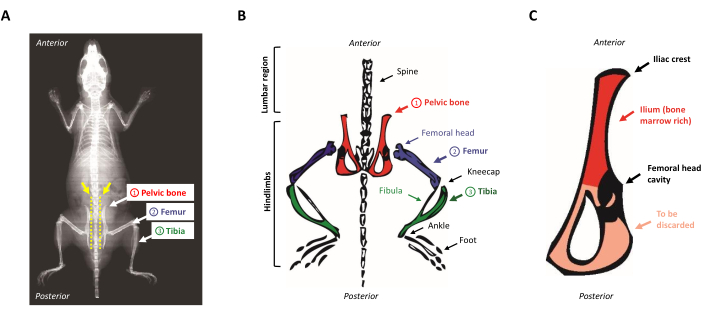
Figura 1: Anatomia del topo. ( A )Radiografiadel topo che mostra le ossa degli arti posteriori. Si noti lo spazio tra l'osso pelvico e la colonna vertebrale (freccia gialla), dove le forbici devono essere inserite per separare correttamente gli arti posteriori dal corpo del topo (linea tratteggiata gialla). (B) Rappresentazione schematica delle ossa ricche di midollo osseo di interesse. Le ossa pelviche sono raffigurate in rosso, i femori in viola e le tibie in verde. (C) Rappresentazione schematica dell'osso pelvico del topo. L'ileo corrisponde alla parte ricca di midollo dell'osso pelvico ed è evidenziata in rosso. Fare clic qui per visualizzare una versione più grande di questa figura.
- Posizionare le forbici parallele alla colonna vertebrale contro le vertebre e vicino alla protuberanza della cresta iliaca. Procedere a tagliare i muscoli lungo il lato della colonna vertebrale sopra l'osso pelvico facendo scorrere le forbici lungo le vertebre fino alla coda.
NOTA: Questa prima sezione di muscoli può essere eseguita anche utilizzando una lama di bisturi. - Posizionare le forbici parallelamente alla colonna vertebrale e procedere al taglio tra le vertebre e la cresta iliaca, come indicato dalla linea tratteggiata gialla in Figura 1A. Assicurati di rimanere il più vicino possibile alle vertebre. Tagliare i muscoli rimanenti per staccare l'arto dal corpo.
NOTA: ci dovrebbe essere poca o nessuna resistenza. - Ripeti dall'altra parte per staccare il secondo arto.
- Trasferire gli arti su una superficie pulita e scartare il resto del corpo nel rispetto delle linee guida istituzionali.
- Esporre le ossa pelviche, femorali e tibiali rimuovendo il più possibile il tessuto circostante con la pinna e il bisturi.
- Procedere con cura a dislocare la testa del femore dall'osso pelvico tenendo l'estremità distale del femore con la pinna mentre si tagliano delicatamente i muscoli attorno all'articolazione con i bisturi. Muovere le ossa per facilitare la lussazione.
- Raschiare via il muscolo rimanente dall'osso pelvico e tagliare con un bisturi nel mezzo della cavità che teneva la testa del femore. L'ileo viene mantenuto in quanto ricco di progenitori ematopoietici mentre il lato triangolare molto sottile dell'osso viene scartato, come mostrato in Figura 1C.
- Rimuovere i tessuti residui intorno all'ileo con il bisturi e posizionare l'osso pulito in PBS sterile integrato con siero per vitello neonato al 2% (PBS-2% NBCS).
- Usando le forbici, tagliare il piede dalla gamba alla caviglia.
- Tenere la parte inferiore della tibia con la pinca e raschiare il muscolo verso il ginocchio. Scartare le fibule e tagliare l'altopiano tibiale con il bisturi. Mettere la tibia in PBS-2% NBCS sterile.
- Rimuovere i tessuti residui intorno al femore con bisturi.
- Tenere la parte superiore del femore con una pinca; posizionare la lama del bisturi alla base della rotula. Applicare una forza verso la rotula parallela al femore fino al distacco della rotula. Posizionare il femore in PBS-2% NBCS sterile. La rimozione della rotula fornisce un accesso pulito per l'inserimento dell'ago per il lavaggio del midollo.
2. Esaurimento magnetico delle cellule positive al lignaggio
- In un armadio a flusso laminare, trasferire le ossa in una capsula di Petri sterile riempita con PBS-2% NBCS sterile.
- Con un bisturi tagliato la testa dei femori.
- Riempire una siringa da 1 mL con PBS-2%NBCS sterile e attaccare un ago da 21 G all'uscita.
- Riempire un tubo di polipropilene da 5 ml con 2 mL di PBS-2% NBCS sterile.
- Tenere il femore con la pinca; inserire delicatamente l'ago nella scanalatura sinistra dopo la rimozione della rotula. Applicare la rotazione sull'ago durante l'inserimento per evitare di tappare l'ago. Assicurarsi che l'ago sia completamente inserito nell'osso fino alla smussatura.
- Trasferire l'osso con l'ago nel tubo contenente 2 ml di PBS-2%NBCS. Erogare e aspirare il PBS-2% NBCS dalla siringa fino a quando l'osso non è limpido.
- Rimuovere l'ago dal femore e inserirlo nel foro sul lato opposto dove si trovava la testa del femore. Erogare e aspirare nuovamente il tampone e scartare l'osso.
- Per la cresta iliaca e la tibia, tenere l'osso con la pinca; inserire delicatamente l'ago nel lato aperto. Applicare la rotazione sull'ago durante l'inserimento per evitare di tappare l'ago. Assicurarsi che l'ago sia completamente inserito nell'osso fino alla smussatura. Trasferire l'osso con l'ago nel tubo contenente 2 ml di PBS-2%NBCS. Erogare e aspirare il PBS-2% NBCS dalla siringa fino a quando l'osso non è limpido. Scartare le ossa.
NOTA: le ossa di un massimo di tre topi possono essere lavate nello stesso tubo. Mettere in comune le sospensioni cellulari. - Passare la sospensione cellulare in pool attraverso un tappo del filtro cellulare da 40 μm posto su un tubo sterile di polistirene da 5 ml.
- Procedere al conteggio delle celle.
NOTA: il conteggio delle cellule può essere eseguito con qualsiasi emocitometro, utilizzando Trypan Blue per la valutazione della vitalità o con qualsiasi contatore di celle automatizzato. Un topo produce in genere 105 ± 7 x 106 celle. - Prendere da parte 100 μL della sospensione cellulare come midollo osseo totale, aggiungere 500 μL di PBS-2% NBCS e salvarlo sul ghiaccio per la procedura di colorazione.
- Pellet la sospensione filtrata per centrifugazione a 400 x g per 5 min a 4 °C ed eliminare il surnatante.
NOTA: I globuli rossi possono essere lussati riconsegendo il pellet in soluzione di lisi appena preparata (1/10° in dH2O). Incubare per 5 minuti fino a quando la sospensione diventa chiara e rosso vivo e aggiungere 10 volumi di PBS sterile. Procedere al lavaggio delle cellule in PBS-2%NBCS mediante centrifugazione a 400 x g per 5 minuti a 4 °C. Fare attenzione quando si rimuove il surnatante poiché il pellet cellulare è molto allentato. Eseguire un secondo lavaggio con PBS-2%NBCS mediante centrifugazione a 400 x g per 5 min a 4 °C e procedere al passaggio 2.13. - Risuspendare il pellet cellulare in un cocktail di anticorpi primari appena preparato con un rapporto di 100 μL per 1 x 107 cellule. Incubare su ghiaccio per 30-45 min.
| Anticorpo | Diluizione |
| Gr-1-biotina | 1:500 |
| B220-biotina | 1:500 |
| Mac-1-biotina | 1:500 |
| CD3-biotina | 1:500 |
| CD4-biotina | 1:500 |
| CD5-biotina | 1:500 |
| CD8-biotina | 1:500 |
| TER119-biotina | 1:1000 |
| CD127-biotina | 1:500 |
Tabella 1.
- Prendere da parte 10 μL della sospensione cellulare in un tubo sterile di polistirene da 5 mL etichettato Frazione di Lin-Pos. Aggiungere 90 μL di PBS-2%NBCS e salvarlo sul ghiaccio per la procedura di colorazione.
- Procedere a lavare le cellule due volte con PBS-2%NBCS sterile mediante centrifugazione a 400 x g per 5 min a 4 °C. Assicurarsi di eseguire l'ultimo lavaggio in un tubo sterile da 5 ml di polipropilene.
- Durante le fasi di lavaggio, preparare le perle per l'esaurimento magnetico.
- Risussospendare le perle nel flaconcino vorticosamente per 30 s.
- Trasferire un volume di perline corrispondente a due perline per cella bersaglio in un tubo di polipropilene da 5 mL.
- Lavare le perle due volte con PBS-2%NBCS posizionando il tubo sul magnete e rimuovendo il tampone di lavaggio utilizzando una pipetta Pasteur sterile in vetro.
- Spese di sospensione delle perle in 500 μL di PBS-2NBCS sterile.
- Risuspenare il pellet di celle etichettate in 250 μL di perle e mescolare delicatamente per 5 minuti sul ghiaccio. Aggiungere 2 ml di PBS-2%NBCS sterile e mescolare delicatamente. Non agitare il tubo.
- Posizionare il tubo sul magnete per 2 minuti.
- Procedere alla raccolta della frazione non magnetica con una pipetta Pasteur sterile in vetro e aggiungerla sui restanti 250 μL di perle magnetiche. Sigillare il tubo con parafilm.
- Posizionare il tubo su un rullo tubiero per 20 minuti a 4 °C.
- Aggiungere 2 ml di PBS-2%NBCS sterile e mescolare delicatamente. Non agitare il tubo.
- Posizionare il tubo nel magnete per 2 minuti.
- Procedere alla raccolta della frazione non magnetica in un tubo sterile di polipropilene da 5 ml etichettato Frazione Lin-Neg con una pipetta Pasteur sterile in vetro.
- Pellet le celle mediante centrifugazione a 400 x g per 5 min a 4 °C e rimuovere il surnatante.
- Spese di sospensione delle celle non magnetiche in 500 μL di PBS-2%NBCS sterile.
- Procedere al conteggio delle celle.
NOTA: un mouse produce in genere 3,9 ± 1,1 x 106 celle. La colorazione tipica del lignaggio pre e post esaurimento è presentata nella Figura 2B.
3. Selezione cellulare dei progenitori dei megacariociti mediante citometria a flusso
- Prendi i tubi etichettati Midollo osseo totale, Frazione lin-Pos e Frazione Lin-Neg.
- Procedere a dividere equamente il contenuto del midollo osseo totale del tubo in sei tubi sterili da 5 mL di polistirene. Etichettare i tubi con i numeri 1-6.
- Procedere all'etichettatura del tubo Lin-Pos Fraction con il numero 7.
- Procedere a dividere il contenuto della frazione Lin-Neg del tubo come segue.
- Trasferire 50 μL in un tubo sterile di polistirene da 5 mL contenente 250 μL di PBS-2%NBCS sterile. Quindi, dividere equamente il suo contenuto in 3 tubi sterili da 5 ml di polistirolo. Etichetta questi tubi con i numeri 8-10.
- I restanti 450 μL di sospensione della cella di frazione di Lin-Neg corrispondono al tubo con il numero 11.
- Aggiungere gli anticorpi alle provette come descritto nella Tabella 2.
| Tubo | Etichetta | Cocktail di anticorpi |
| Midollo osseo totale | ||
| 1 | Controllo non macchiato | |
| 2 | Controllo singolo macchiato | CD45-FITC (1/200) |
| 3 | Controllo singolo macchiato | CD45-PE (1/200) |
| 4 | Controllo singolo macchiato | TER119-APC (1/200) |
| 5 | Controllo singolo macchiato | CD45-PECy7 (1/200) |
| 6 | Controllo singolo macchiato | CD45-APC-Cy7 biotina (1/200) |
| Frazione di Lin-Pos | ||
| 7 | Controllo singolo macchiato | Controllo singolo macchiato. Streptavicina-APC-Cy7 (1/500) |
| Frazione di Lin-Neg | ||
| 8 | Controllo FMO FITC | c-kit-APC (1/200) + Sca-1-PE (1/200) + CD16/32-PE (1/200) + CD150-PECy7 (1/200) + Streptavitina-APC-Cy7 (1/500) |
| 9 | Controllo FMO PE | CD9-FITC (1/200) + c-kit-APC (1/200) + CD150-PECy7 (1/200) + Streptavidin-APC-Cy7 (1/500) |
| 10 | Controllo FMO PECy7 | CD9-FITC (1/200) + c-kit-APC (1/200) + Sca-1-PE (1/200) + CD16/32-PE (1/200) + Streptavitina-APC-Cy7 (1/500) |
| 11 | Tubo positivo per lo smistamento | CD9-FITC (1/200) + c-kit-APC (1/200) + Sca-1-PE (1/200) + CD16/32-PE (1/200) + CD150-PECy7 (1/200) + Streptavitina-APC-Cy7 (1/500) |
Tabella 2.
- Incubare sul ghiaccio per 30-45 minuti al buio.
- Lavare le cellule con PBS-2%NBCS sterile mediante centrifugazione a 400 x g per 5 minuti a 4 °C.
- Risuspendare i pellet di cella come segue.
- Per i tubi da 1 a 10, riconsegregiare il pellet in 300 μL di PBS-2%NBCS sterile integrato con 7AAD (2,5 μg/mL finale) (PBS-7AAD).
ATTENZIONE: 7AAD è un intercanale del DNA e deve quindi essere maneggiato con appositi DPI (guanti). - Per il tubo 11, ripresa del pellet in PBS-7AAD sterile ad una concentrazione massima di 5 x 106 celle per mL e un volume minimo di 1 mL.
- Per i tubi da 1 a 10, riconsegregiare il pellet in 300 μL di PBS-2%NBCS sterile integrato con 7AAD (2,5 μg/mL finale) (PBS-7AAD).
- Preparare due tubi di raccolta in polipropilene etichettati MEP e MKp contenenti 2 ml di PBS-2%NBCS.
NOTA: in alternativa, le cellule possono essere raccolte in terreno di coltura o tampone di lisi cellulare a seconda della successiva applicazione per le cellule ordinate. L'uso di tubi di polistirolo non è raccomandato a causa di possibili interferenze con le goccioline cariche contenenti le cellule di interesse. - Tenere tutti i tubi sul ghiaccio al buio.
- Procedere alla configurazione del cell sorter.
- Utilizzare i tubi 1-7 per impostare la tensione e la compensazione, i tubi 7-10 per determinare le porte di smistamento per le popolazioni cellulari di interesse e il tubo 11 per lo smistamento cellulare.
- I primi passi della strategia di gating mirano a escludere doppietti e cellule morte dall'analisi, come descritto nella Figura 3. Identifica le singole celle vitali e visualizza il dot plot SSC-vs Lin-APC-Cy7 per confermare l'efficienza dell'esaurimento del lignaggio. Dalle celle Lin- un gate è impostato per selezionare le celle positive per c-kit e negative o dim per Sca-1 e CD16/32. Un dot plot di espressione CD9 vs CD150 per le celle selezionate consente l'identificazione di quattro popolazioni.
NOTA: le celle MEP e MKp sono entrambe positive per CD150. È possibile definire tre livelli di espressione per CD9 (neg, dim e high). MKp esprime un alto livello di CD9 e MEP express CD9 ad un livello intermedio di intensità di fluorescenza. La popolazione MEP corrisponde a Lin- c-Kit+ Sca-1-CD16/32-/dim CD150+ CD9dim e la popolazione MKp corrisponde a Lin- c-Kit+ Sca-1-CD16/32-/dim CD150+ CD9bright. La discriminazione tra le popolazioni CD9 high e CD9 dim per le cellule CD150 positive è impostata in base al livello massimo di espressione di CD9 nella popolazione CD150 negativa. Un mouse produce in genere 5,3 ± 0,6 x 103 MKp e 27,2 ± 2,4 x 103 MEP.
Access restricted. Please log in or start a trial to view this content.
Risultati
L'analisi fenotipica delle cellule identificate come MEP e MKp è stata eseguita mediante citometria a flusso. Le cellule sono state etichettate con anticorpi coniugati a fluorescenza contro CD41a e CD42c, marcatori classici delle linee megacariocitiche e piastriniche. Entrambi i marcatori sono stati espressi dalle cellule della popolazione MKp mentre questi marcatori non sono ancora stati rilevati sulla superficie delle cellule della popolazione MEP (Figura 4Ai,4Aii). La po...
Access restricted. Please log in or start a trial to view this content.
Discussione
Il metodo descritto in questo articolo consente l'estrazione e la purificazione di MEP e MKp di topo. Un parametro importante nell'ottimizzazione del protocollo è stato quello di ottenere un numero sufficiente di cellule che sarebbe stato compatibile con la maggior parte dei saggi molecolari e cellulari. La pratica generale della raccolta delle ossa di topo per l'estrazione delle cellule ematopoietiche di solito consiste nella raccolta sia dei femori che delle tibie di ciascun topo. L'osso pelvico, un'altra fonte di mat...
Access restricted. Please log in or start a trial to view this content.
Divulgazioni
Gli autori non dichiarano interessi finanziari concorrenti.
Riconoscimenti
Gli autori desiderano ringraziare Monique Freund, Catherine Ziessel e Ketty per l'assistenza tecnica. Questo lavoro è stato sostenuto da ARMESA (Association de Recherche et Développement en Médecine et Santé Publique), e da Grant ANR-17-CE14-0001-01 a Henri.de la. Salle.
Access restricted. Please log in or start a trial to view this content.
Materiali
| Name | Company | Catalog Number | Comments |
| 21-gauge needles | BD Microlance | 301155 | |
| 7AAD | Sigma-Aldrich | A9400 | |
| Antibody Gr-1-biotin | eBioscience | 13-5931-85 | Magnetic depletion |
| Antibody B220-biotin | eBioscience | 13-0452-85 | Magnetic depletion |
| Antibody Mac-1-biotin | eBioscience | 13-0112-85 | Magnetic depletion |
| Antibody CD3e-biotin | eBioscience | 13-0031-85 | Magnetic depletion |
| Antibody CD4-biotin | eBioscience | 13-9766-82 | Magnetic depletion |
| Antibody CD5-biotin | eBioscience | 13-0051-85 | Magnetic depletion |
| Antibody CD8a-biotin | eBioscience | 13-0081-85 | Magnetic depletion |
| Antibody TER119-biotin | eBioscience | 13-5921-85 | Magnetic depletion |
| Antibody CD127-biotin | eBioscience | 13-1271-85 | Magnetic depletion |
| Antibody CD45-FITC | eBioscience | 11-0451-85 | Cell sorting |
| Antibody CD45-PE | eBioscience | 12-0451-83 | Cell sorting |
| Antibody TER119-APC | eBioscience | 17-5921-83 | Cell sorting |
| Antibody CD45-PECy7 | eBioscience | 25-0451-82 | Cell sorting |
| Antibody CD45-biotin | eBioscience | 13-0451-85 | Cell sorting |
| Antibody CD9-FITC | eBioscience | 11-0091-82 | Cell sorting |
| Antibody c-kit-APC | eBioscience | 17-1171-83 | Cell sorting |
| Antibody Sca-1-PE | eBioscience | 12-5981-83 | Cell sorting |
| Antibody CD16/32-PE | eBioscience | 12-0161-83 | Cell sorting |
| Antibody CD150-PECy7 | eBioscience | 25-1502-82 | Cell sorting |
| Culture medium StemSpan-SFEM | Stemcell technologies | #09650 | |
| Dissection pad | Fisher Scientific | 10452395 | |
| DPBS | Life Technologies | 14190-094 | |
| Ethanol | vWR Chemicals | 83813.360 | |
| Forceps | Euronexia | P-120-AS | |
| Glass pasteur pipette | Dutscher | 42011 | |
| Magnet : DynaMag-5 | Thermo Fisher Scientific | 12303D | |
| Magnetic beads: Dynabeads Sheep Anti-Rat IgG | Thermo Fisher Scientific | 11035 | |
| Megacult | Stemcell technologies | #04970 | |
| MethoCult SF M3436 | Stemcell technologies | #03436 | |
| Newborn Calf Serum | Dutscher | 50750-500 | |
| Red Cell Lysis solution | BD Bioscience | 555899 | |
| Scalpels | Fisher Scientific | 12308009 | |
| Scissors | Euronexia | C-165-ASB | |
| Sterile 1 mL syringes | BD Bioscience | 303172 | |
| Sterile 15mL tubes | Sarstedt | 62.554.502 | |
| Sterile 5mL polypropylene tubes | Falcon | 352063 | |
| Sterile 5mL polystyrene tubes | Falcon | 352054 | |
| Sterile tubes with 70µm cell strainer cap | Falcon | 352235 | |
| Sterile petri dish | Falcon | 353003 | |
| Streptavidin-APC-Cy7 | BD Biosciences | 554063 | Cell sorting |
| Tube roller | Benchmark Scientific | R3005 |
Riferimenti
- Kaushansky, K. Historical review: megakaryopoiesis and thrombopoiesis. Blood. 111 (3), 981-986 (2008).
- Akashi, K., Traver, D., Miyamoto, T., Weissman, I. L. A clonogenic common myeloid progenitor that gives rise to all myeloid lineages. Nature. 404 (6774), 193-197 (2000).
- Debili, N., et al. Characterization of a bipotent erythro-megakaryocytic progenitor in human bone marrow. Blood. 88 (4), 1284-1296 (1996).
- Forsberg, E. C., Serwold, T., Kogan, S., Weissman, I. L., Passegué, E. New evidence supporting megakaryocyte-erythrocyte potential of flk2/flt3+ multipotent hematopoietic progenitors. Cell. 126 (2), 415-426 (2006).
- Vannucchi, A. M., et al. Identification and characterization of a bipotent (erythroid and megakaryocytic) cell precursor from the spleen of phenylhydrazine-treated mice. Blood. 95 (8), 2559-2568 (2000).
- Pronk, C. J., et al. Elucidation of the phenotypic, functional, and molecular topography of a myeloerythroid progenitor cell hierarchy. Cell Stem Cell. 1 (4), 428-442 (2007).
- Nakorn, T. N., Miyamoto, T., Weissman, I. L. Characterization of mouse clonogenic megakaryocyte progenitors. Proceedings of the National Academy of Sciences of the United States of America. 100 (1), 205-210 (2003).
- Ng, A. P., et al. Characterization of thrombopoietin (TPO)-responsive progenitor cells in adult mouse bone marrow with in vivo megakaryocyte and erythroid potential. Proceedings of the National Academy of Sciences of the United States of America. 109 (7), 2364-2369 (2012).
- Strassel, C., et al. Hirudin and heparin enable efficient megakaryocyte differentiation of mouse bone marrow progenitors. Experimental Cell Research. 318 (1), 25-32 (2012).
- Brouard, N., et al. A unique microenvironment in the developing liver supports the expansion of megakaryocyte progenitors. Blood Advances. 1 (21), 1854-1866 (2017).
- Boscher, J., Gachet, C., Lanza, F., Léon, C. Megakaryocyte culture in 3D methylcellulose-based hydrogel to improve cell maturation and study the impact of stiffness and confinement. Journal of Visualized Experiments:JOVE. , (2021).
- Sanjuan-Pla, A., et al. Platelet-biased stem cells reside at the apex of the haematopoietic stem-cell hierarchy. Nature. 502 (7470), 232-236 (2013).
- Haas, S., et al. Inflammation-driven fast-track differentiation of HSCs into the megakaryocytic lineage. Experimental Hematology. 42 (8), 14(2014).
- Shin, J. Y., Hu, W., Naramura, M., Park, C. Y. High c-Kit expression identifies hematopoietic stem cells with impaired self-renewal and megakaryocytic bias. The Journal of Experimental Medicine. 211 (2), 217-231 (2014).
Access restricted. Please log in or start a trial to view this content.
Erratum
Formal Correction: Erratum: Isolation of Mouse Megakaryocyte Progenitors
Posted by JoVE Editors on 7/28/2021. Citeable Link.
An erratum was issued for: Isolation of Mouse Megakaryocyte Progenitors. A figure was updated.
Figure 2 was updated from:
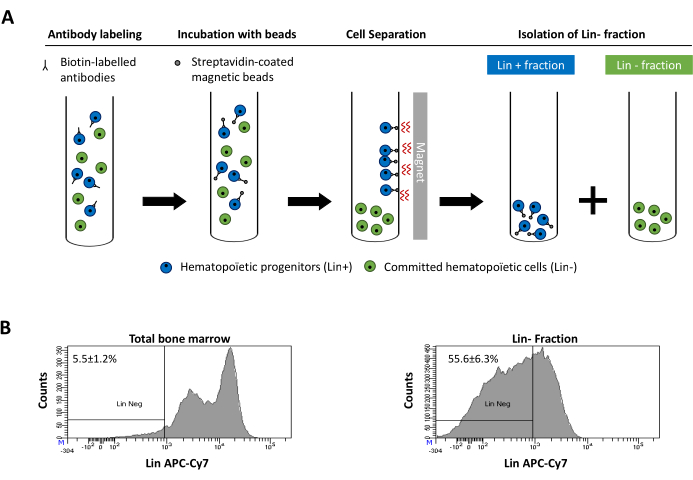
Figure 2: Magnetic depletion of lineage committed (Lin) cells. (A) Schematic representation of the magnetic depletion protocol. First, unsorted bone marrow cells are labeled with the biotin-conjugated rat anti-mouse antibody cocktail. Cells are then incubated with anti-rat Ig coated magnetic beads and subsequently subjected to the magnetic depletion using a strong magnet. The magnet will retain the labeled magnetic Lin+ fraction against the tube walls, while the unlabeled non-magnetic Lin- negative fraction will be collected in a new tube. (B) Lineage committed cells can be identified using fluorescent conjugated streptavidin. Typical analysis of the lineage expression in cells prior to magnetic depletion (total bone marrow) and after magnetic depletion (Lin- Fraction) N = 21. Please click here to view a larger version of this figure.
to:
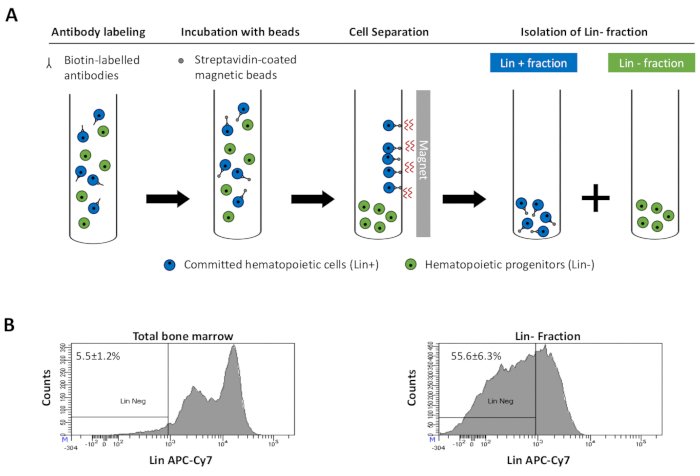
Figure 2: Magnetic depletion of lineage committed (Lin) cells. (A) Schematic representation of the magnetic depletion protocol. First, unsorted bone marrow cells are labeled with the biotin-conjugated rat anti-mouse antibody cocktail. Cells are then incubated with anti-rat Ig coated magnetic beads and subsequently subjected to the magnetic depletion using a strong magnet. The magnet will retain the labeled magnetic Lin+ fraction against the tube walls, while the unlabeled non-magnetic Lin- negative fraction will be collected in a new tube. (B) Lineage committed cells can be identified using fluorescent conjugated streptavidin. Typical analysis of the lineage expression in cells prior to magnetic depletion (total bone marrow) and after magnetic depletion (Lin- Fraction) N = 21. Please click here to view a larger version of this figure.
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon