このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
ライブ顕微鏡イメージング下での3Dヒドロゲルの制御株
要約
提示された方法は、ライブ共焦点顕微鏡を可能にしながら、シリコーンゴムに埋め込まれた3Dソフトヒドロゲルの単軸ストレッチを含む。繊維のアライメントと同様に、外部および内部ヒドロゲル株の特性評価が実証されている。開発された装置およびプロトコルはさまざまなひずみのレジームに対する細胞の応答を査定できる。
要約
外力は組織形成、発達、維持において重要な要素です。これらの力の効果は、多くの場合、特殊なin vitroストレッチ法を使用して研究されます。様々な利用可能なシステムは、ソフトハイドロゲルを歪める3D技術のアクセシビリティながら、2D基板ベースのストレッチャーを使用し、より制限されています。ここでは、サンプルキャリアとして弾性シリコーンストリップを用いて、その周囲から軟質ヒドロゲルの外部ストレッチを可能にする方法について説明する。このプロトコルで利用される伸張システムは3Dプリント部品と低コストのエレクトロニクスから構成され、他のラボで簡単かつ容易に複製できます。実験プロセスは、シリコーンストリップの中心での切り抜きに厚い(>100 μm)ソフトフィブリンヒドロゲル(約100 Paの弾性率)を重合することで始まります。シリコーンゲルの構造は、印刷された伸張装置に取り付けられ、共焦点顕微鏡の段階に置かれる。ライブ顕微鏡では、ストレッチデバイスが活性化され、ゲルは様々なストレッチマグニチュードで画像化されます。その後、画像処理を使用して得られたゲル変形を定量化し、ゲルの3D厚さ(Z軸)全体で比較的均質な歪みと繊維のアライメントを実証します。この方法の利点は、その場合の顕微鏡で実行しながら、3Dで非常に柔らかいヒドロゲルを歪める能力、およびユーザーのニーズに応じてサンプルの形状とサイズを操作する自由が含まれます。さらに、適切な適応により、この方法は、他のタイプのヒドロゲル(例えば、コラーゲン、ポリアクリルアミドまたはポリエチレングリコール)を伸ばすために使用することができ、よりバイオミメティックな3D条件下での外力に対する細胞および組織応答の分析を可能にすることができる。
概要
機械的な力に対する組織応答は、遺伝子発現1、細胞分化2、および組織改修3を含む幅広い生物学的機能の不可欠な部分である。また、繊維アライメントや高密度化などの細胞外マトリックス(ECM)における力誘発変化は、細胞の挙動および組織形成4、5、6に影響を及ぼす可能性がある。ECMの繊維状メッシュ構造は、非線形弾性、非アフィン変形、塑性変形7、8、9、10、11、12などの興味深い機械的特性を有する。これらの特性は、細胞とその周囲の微小環境が外部の機械的力13,14に応答する方法に影響を与える。ECMと組織が機械的な力にどのように反応するかを理解することで、組織工学の分野や、より正確な計算モデルと理論モデルの開発が進むのが可能になります。
サンプルを機械的に伸ばす最も一般的な方法は、細胞の挙動への影響を探るために細胞を含む2D基質に焦点を当てています。これらは、例えば、ポリジメチルシロキサン(PDMS)基質に株を適用し、ストレッチ方向15、16、17、18、19に関連して細胞の再配向角度を分析することを含む。しかし、外部ストレッチに対する3D細胞埋め込みヒドロゲルの反応を調べる方法は、組織微小環境をより密接に模倣する状況であり、より限定的である。3D行列20と比較して細胞が2D基質上で異なる動作をするため、3Dストレッチ法への進歩は特に重要である。これらの行動には、細胞の再調整、タンパク質発現レベル、および移行パターン21、22、23が含まれる。
3Dサンプルストレッチを可能にする方法とデバイスは、市販の24、25、26、27、28と実験室研究29のために開発されたものの両方を含む。これらの方法は、分解性シリコーンチューブ30、マルチウェルチャンバー31、クランプ26、32、バイオリアクター11、33、カンチレバー34、35、36、および磁石37、38を使用する。いくつかの技術は、ゲル5の2つの単一点から針を引っ張るなどして、3Dヒドロゲルを局所的に変形させるストレッチを生成し、他の技術はゲル16の大部分の全体の変形を可能にする。さらに、これらのシステムのほとんどは、Z方向の歪みフィールドに関する限られた情報で、X-Y平面のひずみ場の分析に焦点を当てています。さらに、これらのデバイスのほんの一握りは、その画像化において顕微鏡的に可能である。この場所での主な課題は、高倍率イメージング(例えば、共焦点顕微鏡)は、対物レンズから試料までの数百ミクロンの限られた作業距離です。ストレッチ中にライブイメージングを可能にするデバイスは、Z軸の歪みの均一性を犠牲にするか、または他の実験室39、40で再現することは比較的複雑で困難である。
3Dヒドロゲルを伸ばすためにこのアプローチは、ライブ共焦点顕微鏡の間に静的または周期的な単軸歪みを可能にする。伸張装置(「スマートサイクリック単軸ストレッチャー -SCyUS」と呼ばれる)は、3Dプリント部品と低コストのハードウェアを使用して構築され、他のラボで簡単に再現できます。装置に取り付けられているのは、その中心に幾何学的な切り抜きを有する市販のシリコーンゴムである。ヒドロゲル成分は、切り出しを充填するために重合される。重合の間、フィブリンまたはコラーゲンなどの生物学的ヒドロゲルは、切り抜きの内壁に自然に付着する。SCyUSを用いて、シリコーンストリップは単軸に伸ばされ、制御された株を埋め込まれた3Dヒドロゲル41に移す。
このシステムは他の既存の方法と比較して特徴および利点の独特な組合せを可能にする。まず、このシステムは、厚い3Dソフトヒドロゲル(厚さ>100 μm、<1 kPa剛性)の単軸ストレッチを周囲から可能にし、ハイドロゲル全体で Z-均質変形を行います。これらのヒドロゲルは柔らかすぎて、従来の引張技術でつかんで引き伸ばされる。第二に、3Dプリンティングは研究者が容易に利用でき、設計に使用されるエレクトロニクスは低コストであるため、ストレッチングデバイスは他のラボで容易に複製することができます。第三に、そしておそらく最も魅力的な特徴は、シリコーンストリップの切り抜きの幾何学およびサイズは容易に操作でき、調整可能なひずみの勾配および境界条件、そして、サンプルの様々な量の使用を可能にする、数マイクロリットルまで。
提示された議定書は、生共焦点顕微鏡下で単軸ストレッチによって進行された0.5mm厚いシリコーンゴムストリップの直径~2mmのディスクにフィブリンゲルを成形して構成する。以下では、幾何学的切り抜きに作用する株を測定および分析するための実験手順、ヒドロゲルで開発された内部株、ならびに種々のストレッチ操作後の繊維アライメントを得る実験手順について詳しく説明する。最後に、ヒドロゲルに細胞を埋め込み、制御された外部ストレッチに曝す可能性について議論する。
Access restricted. Please log in or start a trial to view this content.
プロトコル
1. 溶液準備(事前に実施)
- フィブリノーゲンラベリング
注: ラベル付け手順は、フィブリンゲルの変形を解析する場合にのみ必要です。細胞実験のために、標識されていないゲルを使用することができる。- 10 mg/mL スクシニミジルエステル蛍光色素(DMSOに溶解)の38 μLを、15 mg/mLフィブリノーゲン溶液(モル比5:1)を50mL遠心チューブに加え、室温で1時間シェーカーに置きます。その後、チューブを遠心分離機に3分間、800 x g( 室温)で入れます。
- 前工程から上清を濾過し、デキストランゲル樹脂(材料表)を詰めた脱塩カラムを通して、未反応染料を分離し、42 を以下のステップに従って分離する。
- フィブリノーゲンバッファーの25 mLでカラムを事前洗浄してください。
- ステップ1.1.1から標識フィブリノーゲンをゆっくりと列に注入し、フィルターに入る気泡がないことを確認します。溶出した溶液の最初の〜0.3mL(かすかな着色された液体の4〜6滴)を捨てます。次に、精製溶液の次の1.0〜1.5 mLを収集します(より具体的な詳細については、メーカーのプロトコルに従ってください)。
- シリンジ駆動フィルター(0.22-0.45 μm)を使用して得られた精製溶液を殺菌して、濾過プロセスを完了します。
- カラムを洗浄してリサイクルするには、20 mLのフィブリノーゲンバッファーで洗浄し、20%エタノールの25 mLで保管します。
- 溶出後、得られた精製された標識フィブリノーゲンを、所望の伸ばしたゲルの数に応じて、〜7〜50μLの小さなアリコートに分ける。直径2mmの円ゲルを伸ばしてそれぞれ約3.5μLのフィブリノーゲン(1ゲルあたり2.5 μL+ピペット誤差に1 μL)を使用します。
- アリコートを-20°Cの冷凍庫に保管してください。彼らは約1年まで使用することができます(解凍して再び凍結することはお勧めしません)。
- このプロトコルの残りの部分については、精製された標識フィブリノーゲンの約7 μLを、ステップ4まで冷蔵庫(4°C)に保管してください。このボリュームは、2つの伸伸されたゲルを作成することを目的としています(ゲルあたり2.5 μLが必要で、サンプル調製のエラーを考慮するために1μLの追加ボリュームが使用されます)。
注:このフィルタリング手順は、通常、最初の15 mg/mLフィブリノーゲン溶液を約10mg/mLの最終濃度に希釈します。希釈係数は、製造業者のプロトコルで規定されているフィブリノーゲンの初期体積および濃度に依存する。
- 7 μLのトロンビン溶液(トロンビンバッファーを使用して希釈して2単位/mL、 材料表)を調製し、ステップ4まで冷蔵庫(4°C)に保管します。この容積は2つの伸びたゲルの切り抜きを充満することを意図している。
注: 内部歪み分析を行うには、直径 1 μm の蛍光球形ビーズ (水中の懸濁液 [2% 固体] と 2 mM NaN3)をトロンビン溶液に追加する必要があります (40x の目標に対しては 1:25 v/v % の比率を推奨します)。ビーズは、細胞の存在または不在のいずれかで、内部歪み測定が望まれる場合にのみ含める必要があります。
2. シリコンストリップの準備
- 厚さ0.5mmのシリコーンゴムを取り出し、ストリップの中央に直径2mm の穴をあけた15 x 80 mm 2ストリップに切ります(図1)。可能であれば、高精度のためのプログラム可能なレーザーカッターを使用してください。プログラム可能な機械が利用できない場合は、はさみはストリップの輪郭を切断するのに十分であり、小さい穴あけは中心の切り抜きのために十分である。
注:市販のシリコーンゴムは、通常、両面にラップで購入されます。可能であれば、シリコーンの両側にオリジナルのプラスチックカバーを保持します。以前の実験でシリコーンストリップを再利用する場合は、トリプシンで0.5時間処理し、0.2 M NaOHに0.5時間浸し、70%エタノールに1時間浸します。使用前に乾燥させてください。 - 少なくとも20×30 mm2の寸法を持つシールフィルム(疎水性)層を準備し、シリコーンストリップよりも広いので、幾何学的な切り抜き全体にシールを形成できるようにします。
- 70%エタノールで10cm皿を洗い、その後、非リンティング繊細なタスクワイプ(無菌と非無菌の両方の実験のために)で拭いて乾燥させます。このステップは、シールフィルム層がプレートに貼り付き、調製プロセス中にサンプルの動きを制限することができるので重要です。
- 洗浄された10cm皿にシールフィルム層を入れ、各皿に2つのストリップを並べて配置するのに十分なスペースがある(図2A)。
- シリコンストリップの片側からラップを取り外し、露出した側をシールフィルム層に置き、切り抜きがシールフィルム層で完全に囲まれるようにする(図2B)。次に、汚れ切り材の周囲をシールするために、きれいな手袋をした指を使用して、シリコーンをシールフィルム層に対して軽く押し付けます。
注:シリコーンとシールフィルムの間にエアポケットがないことを確認してください(特にカットアウトの周り)。これを行うには、皿の底面を調べます(図2C)。
- シリコンストリップの片側からラップを取り外し、露出した側をシールフィルム層に置き、切り抜きがシールフィルム層で完全に囲まれるようにする(図2B)。次に、汚れ切り材の周囲をシールするために、きれいな手袋をした指を使用して、シリコーンをシールフィルム層に対して軽く押し付けます。
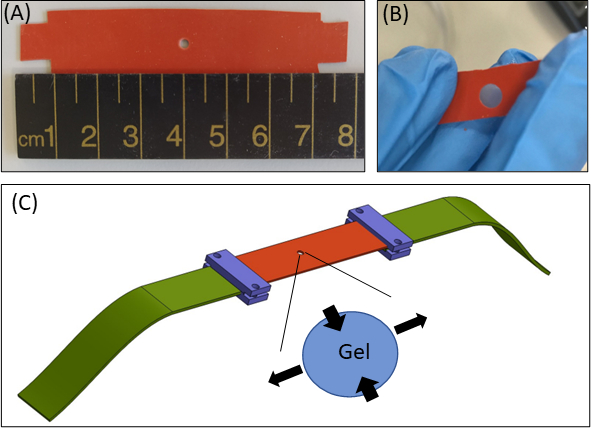
図1:ヒドロゲルの緊張アプローチ。(A)15 × 80 mm2シリコーンストリップで、ストリップの中央に直径2mmの切り出しを有する(B)フィブリンゲルが埋め込まれた円形の切り抜きのあるシリコーンストリップ。例証的な目的のために、シリコーンの切り抜きは、実際の実験(C)シリコーンストリップ(オレンジ)、円形ゲル(中間にカットアウト)、およびシリコーンを延伸装置に接続する布伸長剤(緑色)を用いた延伸アプローチの概略図よりも大きい。ゲルの拡大面積は、シリコーンの単軸延伸に応答して、ゲルの変形を示す。簡略のため、ゲルの厚さ(Z軸)に沿った圧縮は図に示されていません。図1B&1Cは、この図のより大きなバージョンを表示するには、ここをクリックしてくださいロイトブラットRibaららから適応されています。

図2:ゲル重合前のシール膜層上にシリコーンストリップを適切に配置した例。(A)10cm皿に2つのシールフィルム層を配置する(B)シーリングフィルム層上のシリコーンストリップの配置(C)皿の底面図、シリコーンとシールフィルム層の間のエアシールを表示する。左: シールフィルム層の適切なシールは、エアポケットなしでカットアウトの周りのシリコーンストリップに。右: シールフィルム層の不適切なシールは、切り抜きの端の周りにエアポケットを持つシリコンストリップカットアウト。これはシリコーンの下のヒドロゲルの部品の漏出につながる。赤い矢印は、エアポケットが形成された領域を指しています。この図の大きなバージョンを表示するには、ここをクリックしてください。
3. 細胞を用いてトロンビンを準備する
注:このステップは、細胞をヒドロゲルに埋め込む必要がある場合、および生物学的フード(材料表)の無菌条件下でのみ実行します。
- 殺菌:細胞実験の前日に、シリコーンストリップとシールフィルム層を一晩70%エタノールに入れ、両側で30分間UV殺菌を行います(10cmの皿がまだ無菌でない場合は、70%エタノール洗浄後30分間もUV光下で殺菌する必要があります)。プロトコルで利用される紫外線システムは生物フードに組み込まれる1つである。
注:オートクレーブ滅菌サイクル(140°C)は、最大260°Cに耐性があるため、シリコーンストリップで行うことができます。 - 細胞数を測定して細胞濃度を決定し、遠心分離機を行い、1.5 mL遠心分離管内のトロンビン(2単位/mL)の7μLで細胞ペレットを再中断します。トロンビンの細胞濃度は800細胞/μLを推奨します。使用するまで細胞を冷やしてください(細胞に損傷を与えないように30分以上は過ぎない)。
4. フィブリンゲルの重合
- 2ユニット/mLトロンビン&10 mg/mLラベル付きフィブリノーゲン溶液を冷蔵庫から取り出し(ステップ1で準備)し、アクセス可能な氷の上に置きます。
注:溶液は、重合反応キネティクスを遅くするために、混合プロセスの前に冷たく保たれています。これにより、タンパク質のより均質な混合が可能になります。 - 皿(es)をセットアップ(ステップ2)で、2.5μLの標識フィブリノーゲンとピペットをシリコーンカットアウト(シールフィルム層を底面に取り付けた状態)に均一に抽出し、カットアウトの全円周がフィブリノーゲンと接触するようにします。エアポケットや気泡が溶液のどこにも形成されるように注意して、切り抜きの下端(シールフィルム層とシリコーンの間の界面)に特に注意を払ってください。
- すぐに2.5 μLのトロンビン(細胞/ビーズの有無にかかわらず)を取り、切り出しでフィブリノーゲン溶液に直接ピペットします(5 μLフィブリンの最終体積に達する)。その後、2つの溶液を慎重に上下に10回ピペット処理して素早く混ぜます。混合プロセス中に、チップをボリューム全体に移動して、可能な限り均質なソリューションを作成します。
- 非常に少量のリン酸緩衝塩(PBS)を加えて、重合プロセス中にヒドロゲルが乾燥しないように、各皿の端に沿って、細胞実験用の細胞媒体、 材料表を加えます。PBS/細胞媒体とサンプルとの間に接触がないことを確認します。
- 皿を覆い、37°Cのインキュベーターに30分間置きます。
注:必要なインキュベーション時間は、ゲルの体積に依存します。より大きなボリュームを使用する場合、インキュベーション時間が長くなります。 - インキュベーターから皿を取り出し、PBS/細胞培地を皿に加え、ゲルシリコンコンストラクト全体を水没させる。
- 慎重にシーリングフィルム層がストリップに付着したままであることを確認するために皿から一度に1つずつサンプルの構造を持ち上げます。シリコーンの一方の端からもう一方の端にそっとピーリングして、シリコーンからシール膜層をゆっくりと取り外します(図3)。応力集中が存在するカットアウトに近い領域から引き出すことは避けてください(これは主に非円形ジオメトリにとって重要です)。サンプルを損傷するため、カットアウトとの接触は避けてください。
- ストリップが皿に浮かんでいるようなPBS/セル媒体で皿に戻します。その後、各サンプルの状態を定性的に評価するために、標準的な細胞培養顕微鏡に皿を持って行きます。ゲルは均一で、切り抜き全体を通して連続的でなければならず、気泡は存在しなくてはなりません。 図 4 をガイドとして使用して、さらに分析するための最適なサンプルを選択します。

図3:シリコーンストリップの底面からシール膜層を適切に除去する。除去プロセスは、ヒドロゲルがカットアウトの内壁との接着を引き裂いたり壊したりしないようにゆっくりと行う必要があります。白い矢印は、除去の方向を示しています。 この図の大きなバージョンを表示するには、ここをクリックしてください。

図4:シリコーンカットアウト中のフィブリンゲルの顕微鏡観察。(A)適切に重合したフィブリンゲルの2つの例。切り出しの端に対するゲルの相対的な均質性と完全接着性に注意してください(B)サンプル重合不良の2つの例。上: 多くの気泡と、左下に形成された凝集体に注意してください。下: 切り抜きエッジからのゲルの引き裂きと、切り抜きの左下領域の凝集体に注目してください。スケールバー= 300 μmこの図の大きなバージョンを表示するには、ここをクリックしてください。
5. SCyUS デバイスでのサンプルの読み込み
- 槽にPBS/細胞媒体(図5)を充填し、サンプルジェルを上部に持つシリコーンストリップを置き、両端が浴槽の両側に座るようにします。浴はゲルの乾燥を避けるためのものです。クランプを配置し、(紫色)ファブリック ストリップ(緑色)と一緒に締め付け、すべてのピースが中央に切り抜きされた直線ストリップを形成するようにします(図5)。
- SCyUSデバイスを取り出し、アルミニウム液井戸と22mm x 40mmの長方形のガラスカバースリップ(No. 1または1.5)を取り付けます(図6Aiii)。密封材(例えば、真空グリース)を使用してウェルの底部にカバースリップを取り付け、漏れることなく液体で満たされるようにします。
- PBS/細胞培地の約1〜2 mLでウェルを充填し、ストリップ+ファブリック+ゲル構造(図6B)をデバイスに入れる。布製ストリップ(2)をブラケット(1)にクランプして、カットアウト+ゲル(5)が図のように中央に収まるように、ピンダウンインサート(4)をデバイスに慎重に挿入し、所定の位置にロックします。
- 次に、他のファブリック側をスピンドルに挿入し(サーボモーターを取り付けずに)、スピンドルにロックします(図6C)。
- 取り付けられたサンプルを持つSCyUSデバイスを顕微鏡の段階に挿入します(図6C)。USBケーブルでマイクロコントローラ(材料表)をコンピュータに接続し、サーボモーターをマイクロコントローラに接続します。コンピュータの SCyUS コントロール モジュールを開きます。サンプルは、共焦点顕微鏡の下でゲルの厚さと繊維の均質性の妥当性を確認するためにイメージングの準備が整いました。
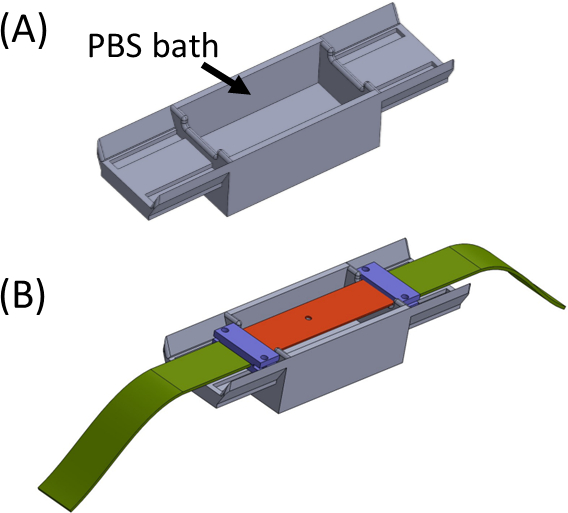
図5:(A)PBS浴を含むジグ(3D印刷)(B)は、ブラケットの正しいインラインアタッチメント(紫色)およびゲルの乾燥を防止するために、ジグ上にストリップ配置。この図は、ロイトブラット・リバらの許可を得て変更されていますが、この図の大きなバージョンを表示するには、ここをクリックしてください。
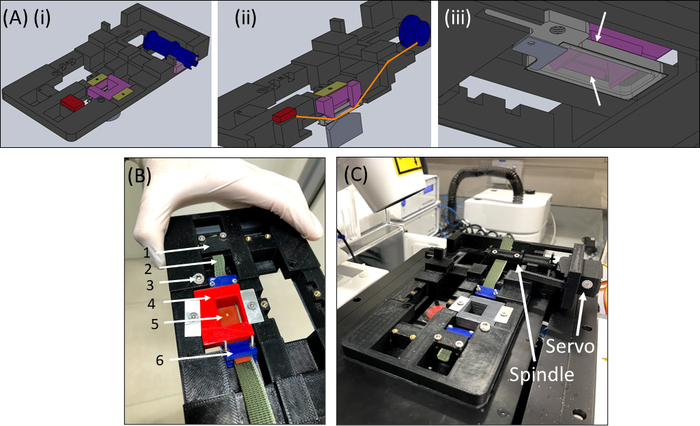
図 6: SCyUS ストレッチ デバイス(A) SCyUS の主要部分の CAD モデルのいくつかのビュー:サーボ(青)に接続されたスピンドル、固定アンカー(赤)、シリコーンストリップをピン留めする挿入(紫色)、およびインサートが立ち上がるのを防ぐフィクサー(黄緑色)。システムの上面図(Ai)、ストリップの経路を示すシステムのカットビュー(Aii)、ガラスカバースリップを備えたアルミニウム液の下面(Aiii)を示す。液体井戸は主要なねじに合うねじの回転と上下に動かすことができる。アルミニウムウェルの上向きの動きは紫色のインサートの側面翼によって制限され、白い矢印(B)実際のシステム:(1)静的アンカー(2)緑色の伸縮不可能な生地(3)アルミニウム液体ウェル高さコントロール用ネジ(4)赤いピンダウンインサート(5)円形の切り抜き(6)青い接続クランプを持つシリコーンストリップ(サーボモーターとスピンドルは矢印で示されています。この図は、ロイトブラット・リバらの許可を得て変更されていますが、この図の大きなバージョンを表示するには、ここをクリックしてください。
6. サンプリングに適したゲルを確保する
- 共焦点顕微鏡(材料表)を使用して、低倍率(10×)、低解像度(約1.4 μm x 1.4 μmピクセルサイズ)の共焦点Z-スタック(≤10 Zスライス、ステップサイズ約10μ) m)488/543/561のレーザーを用いてゲル全体のタイル画像で、シリコーンの厚さ全体の幾何学的切り抜きの周囲に均質性と接着性を調べる(図7A-B)。この Zスタック イメージを、次の手順のマップとして使用します。
- 低解像度のライブイメージングを使用して、ゲルをスキャンし、切り抜きの内壁への完全な接着が涙や泡なしで明らかな最も低いZ位置を決定し、顕微鏡のZ位置(Zl)に注意してください。その円周を通してシリコーンに対するゲルの完全な接着を決定するには、ゲルとシリコーンストリップの蛍光ラベル(暗い背景)の界面を顕微鏡下でスキャンする(図7C)。
- ゲル内に連続性がなくなるまで Z方向に上に移動し 、Z位置(Zu)に注意してください。
- Z方向の上限(Zu)を下限から引きます(Zl)。これはサンプルの参照厚さ (Zo):

Zoが 100 μm ≥場合、ゲルは分析に満足できるものと見なされます。なお、シリコーンカットアウトの厚さは約500μmですが、カットアウトにおけるゲル重合は通常、ゲル厚が小さくなります。100 μm は、シリコーンの切り抜きからゲルを抜いたり裂けたりすることなく、安定した延伸プロセスを確保するための最小推奨厚です。
注: 異なる XY 位置では、ゲルの厚さが異なる場合があります。このプロトコルのセクションはゲルの最小厚さを測定し、ゲルの品質を決定し、伸張に十分かどうかを示すことを可能にします。さらに、ゲルの中心を見つけることは、静的または動的かどうか、後伸縮に戻るための基準点を提供します。

図7:ゲル均質性。タイル画像は、コンフォーカル顕微鏡ソフトウェア(材料表)(A)不適切なトロンビンとフィブリノーゲン混合前重合による比較的不均一な繊維密度を有するフィブリンゲルサンプルの単一ステッチタイルZスライス画像を使用して撮影およびステッチされた。このゲルは、比較的均質な繊維密度を有するフィブリンゲルサンプルの単一ステッチタイルZスライス画像を、信頼性の高い分析(B)を提供しない。これは、ストレッチ実験に適したゲルです。画像A&Bのスケールバーは200μm(C)蛍光標識ゲル(赤)とシリコーン(黒の背景)の間の界面のズームインです。スケールバー = 100 μm.この図の大きなバージョンを表示するには、ここをクリックしてください。
7. SCyUS操作、ストレッチ&イメージング
- これで、サンプルが満足のいく品質であると判断され、SCyUS デバイスに適切に設定された後、サンプルの前にストレッチされた位置を決定します。これは、共焦点顕微鏡下で のライブ イメージング(ステップ6.2と同様)を使用することによって達成される。
- サーボモーターが ゼロサーボPosに移動 ボタンをクリックしてゼロ位置にあることを確認し( 図8A をクリックし、 図8B がゼロであることを確認してください)、図 6Cに示すように伸伸ばされたデバイスに取り付けます。
注:サンプルに余分な張力を入れないように、このステップをゆっくりと慎重に行ってください。 - サンプルを撮像しながら、カットアウトの右側が動くのが観察されるまで+1ボタンをクリックして、モータを時計回りに1度(図8C)ずつ動かします。次に、-1ボタンをクリックして、移動(図8D)を逆にして、最後から2番目のステップの位置に戻します。これにより、サンプルが最小の張力下にあることが確認されます。[最小サーボ位置の設定]ボタン(図8E)をクリックして、基準位置を設定します。「最小サーボ位置へ移動」ボタン(図8F)をクリックすると、いつでも参照位置に戻ります。
注: この手順では、エラーを最小限に抑えるために高倍率の目的 (≥40×) を使用することをお勧めします。 - 高倍率(40×)、高解像度(0.2 μm×0.2 μmピクセルサイズ)、ゲル領域全体の単一のZスライスタイル画像をキャプチャします。これは、後処理解析の参照画像として使用されます。ゲル厚の中央に単一のZスライス画像を取り込むことをお勧めします(Eq. 1からZoを使用して)、ストレッチ後にほぼ同じZ位置に戻ることを可能にします。また、ゲル領域全体の高解像度タイル画像にかなりの時間がかかることを考慮する(〜20〜30分)。
- これで、サンプルは静的ストレッチの準備が整いました。GUI で一度に 1 度 (図 8C)を進めることにより、サーボモーターを目的の伸縮大に合わせて調整します (これをゆっくりと 1 度/秒で実行)。
- サーボモーターが ゼロサーボPosに移動 ボタンをクリックしてゼロ位置にあることを確認し( 図8A をクリックし、 図8B がゼロであることを確認してください)、図 6Cに示すように伸伸ばされたデバイスに取り付けます。
- 解析が望まれるストレッチの大きさごとに、後処理解析のためにゲル領域全体の単一のZスライス高解像度タイル画像をキャプチャします。ステップ6.2と同様に、ゲル(赤)とシリコーン(暗い背景)の界面をスキャンして、ゲルが周囲を通してシリコーンから切り離れていないことを確認し、以前のストレッチの大きさからの接着の変化を探します。
注: モータのアクティブ化中に、ライブイメージングを使用して、X-Yのゲル位置を追従します(低解像度、低倍率の設定)。ゲルは、ゲルの底部が上昇するZ-ポアソン効果を経験するため、顕微鏡のZ位置は、各ストレッチの大きさに対するゲル厚の近似中心に調整する必要があります。これは、ストレッチの大きさごとにZo (Eq. 1) を再計算することで実現できます。Z方向のストレッチは比較的均質であるため、ゲルの正確な中心深さを見つけることは重要ではありません。
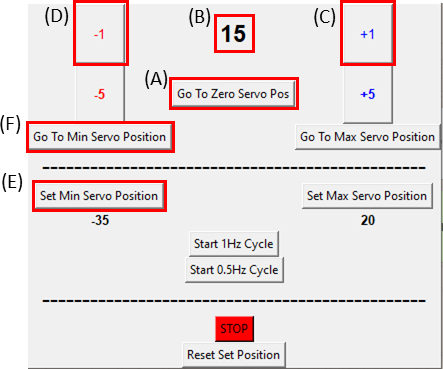
図 8: SCyUS コントロール モジュールの GUI。(A) モーターの位置 (度)値の範囲は-90°から90°(B)'最小サーボ位置を設定します。このボタンは、ゼロサーボ位置(C)'プラス1°'ボタンとは異なる新しい基準位置を設定するための、サーボモーターを時計回りに1度移動(D)'マイナス1°'ボタンがサーボモーターを1度動かす、事前設定された最小位置を可能にします 反時計回り(E)'ゼロ位置に移動'ボタンは、サーボモータの位置を0°に設定します([A]はゼロに設定されます)(F)'最小サーボ位置に移動'ボタンは、ユーザー定義の'最小'位置にサーボモーターを移動します。この図の大きなバージョンを表示するには、ここをクリックしてください。
8. 後処理外部歪み測定
- 切り抜き境界の有効な歪さを測定するには、Y軸の中心にある伸縮方向(X-軸)のエッジからエッジまでの長さを測定する(図9A)。
- 画像処理ソフトウェア (ImageJ FIJI43)にプリストレッチ画像をアップロードし、中心にある穴の軸方向の長さ ( ) として定義されるエッジからエッジまでの最大距離
 を測定します。
を測定します。 - 上から下までの最大距離を垂直距離 ( ) として定義
 します。
します。 - すべてのストレッチ間隔画像に対してこのプロセスを繰り返し、
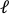
 切り取り周辺の軸方向()と垂直な距離(図9A、下)を計算し、次の計算を実行してカットアウトエッジの歪を見つけます。
切り取り周辺の軸方向()と垂直な距離(図9A、下)を計算し、次の計算を実行してカットアウトエッジの歪を見つけます。
- 画像処理ソフトウェア (ImageJ FIJI43)にプリストレッチ画像をアップロードし、中心にある穴の軸方向の長さ ( ) として定義されるエッジからエッジまでの最大距離
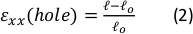

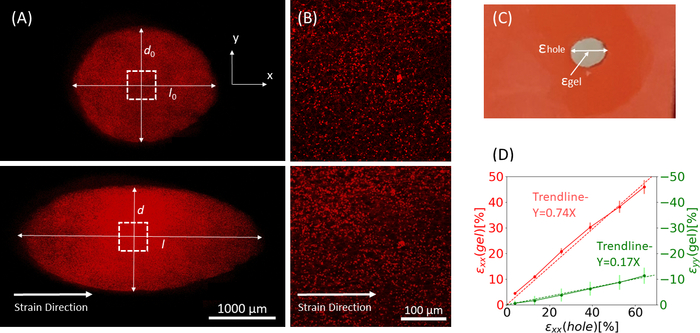
図9:シリコーンストリップの外部延伸によるゲル株。(A)伸伸していないフィブリンゲル(上)のX-Y断面、及びε穴の塗布後=64%のx方向(下)に沿った歪み。ゲルは蛍光ビーズと埋め込まれている。ε穴の計算に使用されるdとlの関連する長さは、画像(B)で示された破線正方形の画像(C)この研究で考慮された歪みの種類の図で示されています:ε穴は、その最大直径での切り抜きの軸ひずみです。 εゲルは、ゲルの中心にある軸方向歪み(ビーズ凝集位置で測定)(D)ε穴とεゲルの間にxx方向(赤線)とyy方向(緑色の線)の両方で直線的な関係が見つかった。この図は、ロイトブラット・リバらの許可を得て適合しており、この図の大きなバージョンを表示するには、ここをクリックしてください。
9. 繊維配向解析
- 繊維アラインメントの定量化を使用して、繊維状ゲルの構造応答をストレッチの大きさの増加に特徴付けます。ImageJ FIJI ソフトウェア (NIH)43に高解像度の画像をアップロードし、OrientationJ (EPFL)44モジュールを使用して分析します (設定: ガウスグラデーションと 3 ピクセルウィンドウ、図 10)。
- 方向ヒストグラムの 2D ネマティック順序パラメータ(NOP)を計算します:45
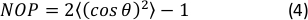
注: NOP = 1 の値は軸方向(角度ゼロ)に沿った完全な位置合わせを示し、NOP = 0 は等方性を示します。方向角度 θは、画像解析を通じて得られた歪み軸(x軸)に対する繊維角度で、OrientationJ ドキュメントで正確に定義されています。44
- 方向ヒストグラムの 2D ネマティック順序パラメータ(NOP)を計算します:45

図 10: フィジー ImageJ ソフトウェアを使用した繊維配向解析(A) 'OrientationJ' が見つかる 'プラグイン' プルダウンメニューの場所を示す矢印付きの ImageJ のメインメニュー。'OrientationJ'の拡張メニューの下で、[オリエンテーションJディストリビューション]オプション(B)オリエンテーションJのディストリビューションモジュールをクリックします。「ローカルウィンドウσ」を3ピクセルに設定し、「グラデーション」を「ガウス」に設定します。次に「実行」ボタン(赤い矢印)を押します。 この図の大きなバージョンを表示するには、ここをクリックしてください。
10. 手動内部ゲルひずみ分析
- ビーズが埋め込まれたヒドロゲルの高倍率のイメージングを行いながら、各ストレッチの大きさの後に同じ場所に戻すために、容易に認識できる特徴(例えばビーズの集合体)を持つ関心領域(ROI)を手動で見つけます。
注: Z 方向の圧縮(ポアソン効果)は、ストレッチが増加するにつれてビード密度の増加につながる可能性があるため、ビーズの凝集体を十分に大きく選択して、明確に識別できるようにすることをお勧めします。このプロトコルは、フィブリンゲルの中央領域の分析を必要としますが、任意の領域を選択することができます。 - ストレッチ前の位置(ステップ 6)で、選択した ROI の高解像度 Zスタック イメージをキャプチャします。各ストレッチ間隔が終了したら、同じ ROI に戻り、画像キャプチャプロセスを繰り返します。
- イメージを取り出し、ImageJ にインポートします。ROI では、表示される各ビードの集約の X-Y ピクセル位置を記録します。記録したデータをスプレッドシートに転送します。
- 集計の各ペア間の距離を測定し、参照画像内の同じペアの距離と比較して 、X 方向と Y 方向の歪みの計算を可能にします。
注:(静的な画像キャプチャの代わりに)ゲルが伸びている間に連続的なリアルタイムムービーが記録される場合、前述の46,47のように、デジタル画像または体積相関(DIC/DVC)法で歪みの自動分析を行うことができます。ただし、この設定では、Z-スタックがX-Y平面内だけでなく、ポアソン効果(圧縮)によるZ方向でも移動するため、DIC/DVC自動解析は難しいので、録画されたムービーの間にかなりのドリフトを占めています。
Access restricted. Please log in or start a trial to view this content.
結果
3Dフィブリンヒドロゲルを運ぶシリコーンストリップに適用される大きさの増加の静的ストレッチからの代表的なデータは、1μmの蛍光ビーズで埋め込まれた、図9に示されている。この分析は、シリコンストレッチが切り抜きの幾何学的変化およびゲル内で発達した株に及ぼす影響を示しています。ゲル全体のZ-スタック画像を使用して、元の円形状の切り?...
Access restricted. Please log in or start a trial to view this content.
ディスカッション
本明細書に提示される方法とプロトコルは、主にRoitblat Ribaらの以前の研究に基づいており、41 ここに SCyUS デバイスの完全なコンピュータ支援設計(CAD)、Pythonおよびマイクロコントローラコードが含まれています。
既存のアプローチよりも提示された方法の主な利点は、非常に柔らかい3Dヒドロゲル(〜100 Paの弾性率)をその円周から、 およびラ?...
Access restricted. Please log in or start a trial to view this content.
開示事項
著者らは開示するものは何もない。
謝辞
ここに含まれるいくつかの数字は、著作権クリアランスセンターの許可によって適応されています: スプリンガーネイチャー, 生物医学工学年報.生きた顕微鏡イメージングを可能にしながら、均一なZ軸株で3Dヒドロゲルを緊張させ、A.ロイトブラット・リバ、S.ナタン、A.コレル、H.ラシュキン、O.チャイチェヤン、A.レスマン、著作権©(2019)。
https://doi.org/10.1007/s10439-019-02426-7
Access restricted. Please log in or start a trial to view this content.
資料
| Name | Company | Catalog Number | Comments |
| Alexa Fluor 546 carboxylic acid, succinimidyl ester | Invitrogen | A20002 | |
| Cell Medium (DMEM High Glucose) | Biological Industries | 01-052-1A | Add 10% FBS, 1% PNS, 1% L-Glutamine, 1% Sodium Pyruvate |
| Cover Slip #1.5 | Bar-Naor Ltd. | BN72204-30 | 22×40 mm |
| DIMETHYL SULPHOXIDE 99.5% GC DMSO | Sigma-Aldrich Inc. | D-5879-500 ML | |
| Dulbecco's Phosphate-Buffered Saline | Biological Industries | 02-023-1A | |
| EVICEL Fibrin Sealant (Human) | Omrix Biopharmaceuticals | 3902 | Fibrinogen: 70 mg/mL, Thrombin: 800-1200 IU/mL |
| Fibrinogen Buffer | N/A | Recipe for 1L: 7g NaCl, 2.94g trisodium citrate dihydrate, 9g glycine, 20g arginine hydrochloride & 0.15g calcium chloride dihydrate. Bring final volume to 1L with PuW (pH 7.0-7.2) | |
| Fluorescent micro-beads FluoSpheres (1 µm) | Invitrogen | F8820 | Orange (540/560) Provided as suspension (2% solids) in water plus 2 mM sodium azide |
| High-Temperature Silicone Rubber | McMaster-Carr | 3788T41 | 580 µm-thick E = 1.5 Mpa Poisson Ratio = 0.48 Tensile Strength = 4.8 MPa Upper limit of stretch = +300% engineering strain |
| HiTrap desalting column 5 mL (Sephadex G-25 packed) | GE Healthcare | 17-1408-01 | |
| HIVAC-G High Vacuum Sealing Compound | Shin-Etsu Chemical Co., Ltd. | HIVAC-G 100 | |
| ImageJ FIJI software39 | National Institute of Health, Bethesda, MD | Version 1.8.0_112 | |
| Microcontroller (Adruino Uno + Adafruit Motorshield v2.3) | Arduino/Adafruit | Arduino-DK001/Adafruit-1438 | |
| MicroVL 21R Centrifuge | Thermo Scientific | 75002470 | |
| Parafilm | Bemis | PM-996 | |
| Primovert Light Microscope | Carl Zeiss Suzhou Co., Ltd. | 491206-0011-000 | |
| SCyUS CAD (Solidworks) | Dassault Systèmes | N/A | |
| SCyUS Code37 | N/A | N/A | |
| Servomotor - TowerPro SG-5010 | Adafruit | 155 | |
| SL 16R Centrifuge | Thermo Scientific | 75004030 | For 50 mL tubes |
| Sterile 10 cm non-culture plates | Corning | 430167 | |
| Thrombin buffer | N/A | Recipe for 1L: 20g mannitol, 8.77g NaCl, 2.72g sodium acetate trihydrate, 24 mL 25% Human Serum Albumin, 5.88g calcium chloride. Bring final volume to 1L with PuW (pH 7.0) | |
| Trypsin EDTA Solution B (0.25%), EDTA (0.05%) | Biological Industries | 03-052-1B | |
| USB Cable (Type B Male to Type A Male) | N/A | N/A | |
| Zeiss LSM 880 Confocal Microscope | Carl Zeiss AG | 2811000417 | |
| ZEN 2.3 SP1 FP3 (black) | Carl Zeiss AG | Release Version 14.0.0.0 |
参考文献
- Bleuel, J., Zaucke, V., Bruggemann, G. P., Niehoff, A. Effects of cyclic tensile strain on chondrocyte metabolism: a systematic review. PLoS ONE. 10, 0119816(2015).
- Pennisi, C. P., Olesen, C. G., de Zee, M., Rasmussen, J., Zachar, V. Uniaxial cyclic strain drives assembly and differentiation of skeletal myocytes. Tissue Engineering Part A. 17, 2543-2550 (2011).
- Grodzinsky, A. J., Levenston, M. E., Jin, M., Frank, E. H. Cartilage Tissue Remodeling in Response to Mechanical Forces. Annual Review of Biomedical Engineering. 2 (1), 691-713 (2000).
- Munster, S., et al. Strain history dependence of the nonlinear stress response of fibrin and collagen networks. Proceedings of the National Academy of Sciences of the USA. 110, 12197-12202 (2013).
- Vader, D., Kabla, A., Weitz, D., Mahadevan, L. Strain-induced alignment in collagen gels. PLoS ONE. 4, 5902(2009).
- Badylak, S. F. The extracellular matrix as a scaffold for tissue reconstruction. Seminars in Cell & Developmental Biology. 13 (5), 377-383 (2002).
- Natan, S., Koren, Y., Shelah, O., Goren, S., Lesman, A. Molecular Biology of the Cell. 31 (14), 1474-1485 (2020).
- Ban, E., et al. Mechanisms of Plastic Deformation in Collagen Networks Induced by Cellular Forces. Biophysical Journal. 114 (2), 450-461 (2018).
- Kim, J., et al. Stress-induced plasticity of dynamic collagen networks. Nature Communications. 8, 842(2017).
- Storm, C., Pastore, J. J., MacKintosh, F. C., Lubensky, T. C., Janmey, P. A. Nonlinear elasticity in biological gels. Nature. 435, 191-194 (2005).
- Wen, Q., Basu, A., Janmey, P. A., Yodh, A. G. Non-affine deformations in polymer hydrogels. Soft Matter. 8, 8039-8049 (2012).
- Muiznieks, L. D., Keeley, F. W. Molecular assembly and mechanical properties of the extracellular matrix: A fibrous protein perspective. Biochimica et Biophysica Acta. 1832, 866-875 (2012).
- Brown, A. E. X., Litvinov, R. I., Discher, D. E., Purohit, P. K., Weisel, J. W. Multiscale mechanics of fibrin polymer: gel stretching with protein unfolding and loss of water. Science. 325, 741-744 (2009).
- Carroll, S. F., Buckley, C. T., Kelly, D. J. Cyclic tensile strain can play a role in directing both intramembranous and endochondral ossification of mesenchymal stem cells. Frontiers in Bioengineering and Biotechnology. 5, 73(2017).
- Livne, A., Bouchbinder, E., Geiger, B. Cell reorientation under cyclic stretching. Nature Communications. 5, 3938(2014).
- Wang, L., et al. Patterning cellular alignment through stretching hydrogels with programmable strain gradients. ACS Applied Materials & Interfaces. 7, 15088-15097 (2015).
- Xu, G. K., Feng, X. Q., Gao, H. Orientations of Cells on Compliant Substrates under Biaxial Stretches: A Theoretical Study. Biophysical Journal. 114 (3), 701-710 (2017).
- Chagnon-Lessard, S., Jean-Ruel, H., Godin, M., Pelling, A. E. Cellular orientation is guided by strain gradients. Integrative Biology (United Kingdom). 9 (7), 607-618 (2013).
- Lu, J., et al. Cell orientation gradients on an inverse opal substrate. ACS Applied Materials & Interfaces. 7 (19), 10091-10095 (2015).
- Baker, B. M., Chen, C. S. Deconstructing the third dimension - 3D culture microenvironments alter cellular cues. Journal of Cell Science. 125, 3015-3024 (2012).
- Bono, N., et al. Unraveling the role of mechanical stimulation on smooth muscle cells: a comparative study between 2D and 3D models. Biotechnology and Bioengineering. 113, 2254-2263 (2016).
- Pampaloni, F., Reynaud, E. G., Stelzer, E. H. K. The third dimension bridges the gap between cell culture and live tissue. Nature Reviews Molecular Cell Biology. 8, 839-845 (2007).
- Riehl, B. D., Park, J. H., Kwon, I. K., Lim, J. Y. Mechanical stretching for tissue engineering: two-dimensional and three-dimensional constructs. Tissue Engineering Part B: Reviews. 18, 288-300 (2012).
- Flexcell. Linear Tissue Train Culture Plate. Flexcell. , (2019).
- Flexcell. Tissue Train. Flexcell. , (2019).
- CellScale. MCT6 Stretcher. CellScale. , (2019).
- STREX. STB-150. STREX. , (2019).
- STREX. Stretch Chambers. STREX. , (2019).
- Kamble, H., Barton, M. J., Jun, M., Park, S., Nguyen, N. T. Cell stretching devices as research tools: engineering and biological considerations. Lab on a Chip. 16, 3193-3203 (2016).
- Weidenhamer, N. K., Tranquillo, R. T. Influence of cyclic mechanical stretch and tissue constraints on cellular and collagen alignment in fibroblast-derived cell sheets. Tissue Engineering Part C: Methods. 19, 386-395 (2013).
- Yung, Y. C., Vandenburgh, H., Mooney, D. J. Cellular strain assessment tool (CSAT): precision-controlled cyclic uniaxial tensile loading. Journal of Biomechanics. 42, 178-182 (2009).
- Chen, K., et al. Role of boundary conditions in determining cell alignment in response to stretch. Proceedings of the National Academy of Sciences of the USA. 115, 986-991 (2018).
- Heher, P., et al. A novel bioreactor for the generation of highly aligned 3D skeletal muscle-like constructs through orientation of fibrin via application of static strain. Acta Biomaterialia. 24, 251-265 (2015).
- Foolen, J., Deshpande, V. S., Kanters, F. M. W., Baaijens, F. P. T. The influence of matrix integrity on stress-fiber remodeling in 3D. Biomaterials. 33, 7508-7518 (2012).
- Walker, M., Godin, M., Pelling, A. E. A vacuum-actuated microtissue stretcher for long-term exposure to oscillatory strain within a 3D matrix. Biomedical Microdevices. 20, 43(2018).
- Zhao, R. G., Boudou, T., Wang, W. G., Chen, C. S., Reich, D. H. Decoupling cell and matrix mechanics in engineered microtissues using magnetically actuated microcantilevers. Advanced Materials. 25, 1699-1705 (2013).
- Li, Y. H., et al. Magnetically actuated cell-laden micro-scale hydrogels for probing strain-induced cell responses in three dimensions. NPG Asia Materials. 8, 238(2016).
- Li, Y. H., et al. An approach to quantifying 3D responses of cells to extreme strain. Scientific Reports. 6, 19550(2016).
- Humphrey, J. D., et al. A theoretically-motivated biaxial tissue culture system with intravital microscopy. Biomechanics and Modeling in Mechanobiology. 7, 323-334 (2008).
- Niklason, L. E., et al. Enabling tools for engineering collagenous tissues integrating bioreactors, intravital imaging, and biomechanical modeling. Proceedings of the National Academy of Sciences of the USA. 107, 3335-3339 (2010).
- Roitblat Riba, A., et al. Straining 3D hydrogels with uniform z-axis strains while enabling live microscopy imaging. Annals of Biomedical Engineering. , (2019).
- Gomez, D., Natan, S., Shokef, Y., Lesman, A. Mechanical interaction between cells facilitates molecular transport. Advanced Biosystems. 3 (12), 1900192(2019).
- Schindelin, J., et al. Fiji: an open- source platform for biological-image analysis. Nature Methods. 9, 676-682 (2012).
- EPFL Switzerland. OrientationJ plug in. EPFL Switzerland. , (2019).
- Goren, S., Koren, Y., Xu, X., Lesman, A. Elastic anisotropy governs the decay of cell-induced displacements. Biophysical Journal. 118 (5), 1152-1164 (2019).
- Notbohm, J., Lesman, A., Tirrell, D. A., Ravichandran, G. Quantifying cell-induced matrix deformation in three dimensions based on imaging matrix fibers. Integrative Biology. 7 (10), 1186-1195 (2015).
- Lesman, A., Notbohm, J., Tirrell, D. A., Ravichandran, G. Contractile forces regulate cell division in three-dimensional environments. Journal of Cell Biology. 205 (2), 155-162 (2014).
- Cha, C. Y., et al. Tailoring Hydrogel Adhesion to Polydimethylsiloxane Substrates Using Polysaccharide Glue. Angewandte Chemie International Edition. 52, 6949-6952 (2019).
- Wirthl, D., et al. Instant tough bonding of hydrogels for soft machines and electronics. Science Advances. 3, (2017).
- Juarez-Moreno, J. A., Avila-Ortega, A., Oliva, A. I., Aviles, F., Cauich-Rodriguez, J. V. Effect of wettability and surface roughness on the adhesion properties of collagen on PDMS films treated by capacitively coupled oxygen plasma. Applied Surface Science. 349, 763-773 (2015).
- Kim, H. T., Jeong, O. C. PDMS surface modification using atmospheric pressure plasma. Microelectronic Engineering. 88, 2281-2285 (2011).
- Prasad, B. R., et al. Controlling cellular activity by manipulating silicone surface roughness. Colloids and Surfaces. 78, 237-242 (2010).
Access restricted. Please log in or start a trial to view this content.
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved