Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Kontrollierte Dehnung von 3D-Hydrogelen unter Live-Mikroskopie-Bildgebung
In diesem Artikel
Zusammenfassung
Die vorgestellte Methode beinhaltet das einachsige Dehnen von 3D-Weichhydrogelen, die in Silikonkautschuk eingebettet sind, während eine konfokale Live-Mikroskopie ermöglicht wird. Die Charakterisierung der externen und internen Hydrogelstämme sowie die Faserausrichtung werden demonstriert. Das entwickelte Gerät und Protokoll kann die Reaktion von Zellen auf verschiedene Belastungsregime beurteilen.
Zusammenfassung
Äußere Kräfte sind ein wichtiger Faktor bei der Bildung, Entwicklung und Erhaltung von Gewebe. Die Auswirkungen dieser Kräfte werden oft mit spezialisierten In-vitro-Dehnungsmethoden untersucht. Verschiedene verfügbare Systeme verwenden 2D-Substrat-basierte Keilrahmen, während die Zugänglichkeit von 3D-Techniken zum Belasten weicher Hydrogele eingeschränkter ist. Hier beschreiben wir eine Methode, die das äußere Dehnen von weichen Hydrogelen aus ihrem Umfang ermöglicht, wobei ein elastischer Silikonstreifen als Probenträger verwendet wird. Das in diesem Protokoll verwendete Stretching-System besteht aus 3D-gedruckten Teilen und kostengünstiger Elektronik, so dass es einfach und leicht in anderen Labors repliziert werden kann. Der experimentelle Prozess beginnt mit der Polymerisation dicker (>100 μm) weicher Fibrinhydrogele (Elastizitätsmodul von ~100 Pa) in einem Ausschnitt in der Mitte eines Silikonstreifens. Silikon-Gel-Konstrukte werden dann an der bedruckten Dehnvorrichtung befestigt und auf den konfokalen Mikroskopstand gelegt. Unter Live-Mikroskopie wird das Dehnungsgerät aktiviert und die Gele werden in verschiedenen Dehnungsgrößen abgebildet. Die Bildverarbeitung wird dann verwendet, um die resultierenden Gelverformungen zu quantifizieren und relativ homogeneDehnungen und Faserausrichtung über die gesamte 3D-Dicke des Gels (Z-Achse) zu demonstrieren. Zu den Vorteilen dieser Methode gehören die Fähigkeit, extrem weiche Hydrogele während der In-situ-Mikroskopie in 3D zu belasten, und die Freiheit, die Geometrie und Größe der Probe nach den Bedürfnissen des Benutzers zu manipulieren. Darüber hinaus kann diese Methode bei richtiger Anpassung verwendet werden, um andere Arten von Hydrogelen (z. B. Kollagen, Polyacrylamid oder Polyethylenglykol) zu dehnen und die Analyse der Reaktion von Zellen und Geweben auf äußere Kräfte unter biomimetischen 3D-Bedingungen zu ermöglichen.
Einleitung
Die Reaktion des Gewebes auf mechanische Kräfte ist ein integraler Bestandteil einer Vielzahl biologischer Funktionen, einschließlich Genexpression1, Zelldifferenzierung2und Gewebeumbau3. Darüber hinaus können kraftinduzierte Veränderungen in der extrazellulären Matrix (ECM) wie Faserausrichtung und Verdichtung das Zellverhalten und die Gewebebildung beeinflussen4,5,6. Die faserige Netzstruktur des ECM hat faszinierende mechanische Eigenschaften, wie nichtlineare Elastizität, nicht-affine Verformung und plastische Verformungen7,8,9,10,11,12. Diese Eigenschaften beeinflussen, wie Zellen und ihre umgebende Mikroumgebung auf äußere mechanische Kräfte reagieren13,14. Zu verstehen, wie das ECM und Gewebe auf mechanische Kräfte reagieren, wird Fortschritte auf dem Gebiet des Tissue Engineering und bei der Entwicklung genauerer rechnerischer und theoretischer Modelle ermöglichen.
Die gebräuchlichsten Methoden zum mechanischen Dehnen von Proben haben sich auf zellbeladene 2D-Substrate konzentriert, um die Auswirkungen auf das Zellverhalten zu untersuchen. Dazu gehören beispielsweise das Aufbringen von Dehnungen auf Polydimethylsiloxan (PDMS)-Substrate und die Analyse von Zellreorrichtungswinkeln in Bezug auf die Streckrichtung15,16,17,18,19. Methoden, die die Reaktion von 3D-zelleingebetteten Hydrogelen auf externe Dehnungen untersuchen, eine Situation, die die Mikroumgebung des Gewebes genauer nachahmt, sind jedoch begrenzter. Fortschritte in Richtung 3D-Stretching-Methoden sind von besonderer Bedeutung, da sich Zellen auf 2D-Substraten im Vergleich zu 3D-Matrizen anders verhalten20. Zu diesen Verhaltensweisen gehören zelluläre Neuausrichtung, Proteinexpressionsniveaus und Migrationsmuster21,22,23.
Methoden und Geräte, die eine 3D-Probendehnung ermöglichen, umfassen sowohl kommerziell erhältliche24,25,26,27,28 als auch solche, die für die Laborforschung entwickelt wurden29. Diese Verfahren verwenden aufwähfstbare Silikonschläuche30,Mehrbrunnenkammern31,Klemmen26,32, Bioreaktoren11,33, Ausleger34,35,36und Magnete37,38. Einige Techniken erzeugen Dehnungen, die lokal 3D-Hydrogele verformen, zum Beispiel durch Ziehen von Nadeln von zwei einzelnen Punkten im Gel5,während andere eine Verformung des gesamten Volumens des Gels16ermöglichen. Darüber hinaus konzentrieren sich die meisten dieser Systeme auf die Analyse des Dehnungsfeldes in der X-Y-Ebene, mit begrenzten Informationen über das Dehnungsfeld in Z-Richtung. Darüber hinaus sind nur eine Handvoll dieser Geräte in der Lage, mikroskopische In-situ-Bildgebung zu ermöglichen. Die größte Herausforderung bei der in situ hochvergrößerenden Bildgebung (z. B. konfokales Mikroskop) ist der begrenzte Arbeitsabstand von einigen hundert Mikrometern von der Objektivlinse zur Probe. Geräte, die Live-Bildgebung während der Dehnung ermöglichen, opfern die Gleichmäßigkeit der Dehnung in der Z-Achseoder sind relativ komplex und in anderen Labors schwer zu reproduzieren39,40.
Dieser Ansatz zum Dehnen von 3D-Hydrogelen ermöglicht statische oder zyklische einachsige Belastungen während der konfokalen Live-Mikroskopie. Das Dehnungsgerät (als "Smart Cyclic Uniaxial Stretcher – SCyUS" bezeichnet) besteht aus 3D-gedruckten Teilen und kostengünstiger Hardware, was eine einfache Reproduktion in anderen Labors ermöglicht. An dem Gerät ist ein handelsübliches Silikonkautschuk mit einem geometrischen Ausschnitt in der Mitte befestigt. Hydrogel-Komponenten werden polymerisiert, um den Ausschnitt zu füllen. Während der Polymerisation haften biologische Hydrogele wie Fibrin oder Kollagen auf natürliche Weise an den Innenwänden des Ausschnitts. Mit dem SCyUS wird der Silikonstreifen unoxidal gedehnt und kontrollierte Stämme auf das eingebettete 3D-Hydrogel41übertragen.
Dieses System ermöglicht eine einzigartige Kombination von Funktionen und Vorteilen im Vergleich zu anderen bestehenden Methoden. Erstens ermöglicht das System das einachsige Dehnen dicker 3D-Weichhydrogele (>100 μm dick, <1 kPa Steifigkeit) aus ihrer Peripherie mit Z-homogenerVerformung im gesamten Hydrogel. Diese Hydrogele sind zu weich, um mit herkömmlichen Zugtechniken gegriffen und gedehnt zu werden. Zweitens kann das Stretching-Gerät leicht in anderen Labors repliziert werden, da der 3D-Druck für Forscher leicht verfügbar ist und die im Design verwendete Elektronik kostengünstig ist. Drittens, und vielleicht das attraktivste Merkmal, kann die Geometrie und die Größe des Ausschnitts im Silikonstreifen leicht manipuliert werden, was abstimmbare Dehnungsgradienten und Randbedingungen sowie die Verwendung einer Vielzahl von Probenvolumina bis auf wenige Mikroliter ermöglicht.
Das vorgestellte Protokoll besteht darin, Fibringel in ~ 2 mm Durchmesser Scheiben in 0,5 mm dicken Silikonkautschukstreifen zu formen, die durch einachsige Dehnung unter live konfokale Mikroskopie durchgeführt werden. Im Folgenden werden die experimentellen Verfahren zur Messung und Analyse der auf den geometrischen Ausschnitt einwirkenden Dehnungen, die im Hydrogel entwickelten inneren Dehnungen sowie die daraus resultierende Faserausrichtung nach verschiedenen Dehnungsmanipulationen ausführlich erläutert. Schließlich wird die Möglichkeit diskutiert, Zellen in das Hydrogel einzubetten und sie einer kontrollierten äußeren Dehnung auszusetzen.
Access restricted. Please log in or start a trial to view this content.
Protokoll
1. Lösungsvorbereitung (im Voraus durchzuführen)
- Fibrinogen-Markierung
HINWEIS: Der Markierungsschritt ist nur erforderlich, wenn die Analyse der Verformung des Fibringels gewünscht wird. Für zelluläre Experimente ist es möglich, ein unmarkiertes Gel zu verwenden.- 38 μL 10 mg/ml Succinimidylester-Fluoreszenzfarbstoff (gelöst in DMSO) zu 1,5 ml 15 mg/ml Fibrinogenlösung (Molverhältnis 5:1) in ein 50-ml-Zentrifugenröhrchen geben und für 1 Stunde bei Raumtemperatur auf einen Shaker legen. Anschließend legen Sie das Röhrchen für 3 Minuten bei 800 x g (Raumtemperatur) in die Zentrifuge.
- Filtern Sie den Überstand aus dem vorherigen Schritt durch eine Entsalzungssäule, die mit Dextrangelharz (Table of Materials) verpackt ist, um den nicht umgesetzten Farbstoff zu trennen,42, indem Sie diese Schritte ausführen.
- Waschen Sie die Säule mit 25 ml Fibrinogenpuffer vor.
- Injizieren Sie das markierte Fibrinogen aus Schritt 1.1.1 langsam in die Säule und stellen Sie sicher, dass keine Blasen in den Filter gelangen. Verwerfen Sie die ersten ~ 0,3 ml eluierte Lösung (4-6 Tropfen schwach gefärbte Flüssigkeit). Sammeln Sie dann die folgenden 1,0-1,5 ml gereinigte Lösung (folgen Sie dem Protokoll des Herstellers für genauere Details).
- Beenden Sie den Filtrationsprozess, indem Sie die resultierende gereinigte Lösung mit einem spritzengetriebenen Filter (0,22-0,45 μm) sterilisieren.
- Um die Säule zu reinigen und zu recyceln, waschen Sie mit 20 ml Fibrinogenpuffer und lagern Sie sie dann in 25 ml 20% Ethanol.
- Nach der Elution teilen Sie das resultierende gereinigte markierte Fibrinogen in kleine Aliquoten von ~ 7-50 μL, abhängig von der gewünschten Anzahl von gedehnten Gelen. Bereiten Sie für jedes gedehnte Kreisgel mit einem Durchmesser von 2 mm etwa 3,5 μL Fibrinogen vor (2,5 μL werden pro Gel + 1 μL für Pipettierfehler verwendet).
- Lagern Sie die Aliquoten in einem -20 °C Gefrierschrank. Sie können bis zu etwa einem Jahr verwendet werden (es wird nicht empfohlen, aufzutauen und wieder einzufrieren).
- Für den Rest dieses Protokolls halten Sie etwa 7 μL des gereinigten markierten Fibrinogens bis Schritt 4 im Kühlschrank (4 °C). Dieses Volumen ist für die Herstellung von zwei gedehnten Gelen vorgesehen (2,5 μL werden pro Gel benötigt, und ein zusätzliches Volumen von 1 μL wird verwendet, um Fehler bei der Probenvorbereitung zu berücksichtigen).
HINWEIS: Dieses Filterverfahren verdünnt typischerweise die anfängliche 15 mg / ml Fibrinogenlösung auf eine Endkonzentration von etwa 10 mg / ml. Der Verdünnungsfaktor hängt vom Anfangsvolumen und der Konzentration von Fibrinogen ab, wie im Protokoll des Herstellers angegeben.
- 7 μL Thrombinlösung vorbereiten (mit Thrombinpuffer auf 2 Einheiten/ml verdünnen, Materialtabelle)und bis Schritt 4 im Kühlschrank (4 °C) aufbewahren. Dieses Volumen soll die Ausschnitte von zwei gestreckten Gelen füllen.
HINWEIS: Zur Durchführung einer internen Dehnungsanalyse sollten fluoreszierende kugelförmige Perlen mit einem Durchmesser von 1 μm (gekauft als Suspension [2% Feststoffe] in Wasser plus 2 mM NaN3)zur Thrombinlösung hinzugefügt werden (ein Verhältnis von 1:25 v/v % Perle:Thrombin wird für ein 40-faches Ziel empfohlen). Perlen sollten nur eingeschlossen werden, wenn interne Dehnungsmessungen gewünscht werden, entweder in Gegenwart oder Abwesenheit von Zellen.
2. Silikonstreifenvorbereitung
- Holen Sie den 0,5 mm dicken Silikonkautschuk und schneiden Sie ihn in 15 x 80 mm2 Streifen mit einem Loch mit 2 mm Durchmesser in der Mitte des Streifens (Abbildung 1). Wenn möglich, verwenden Sie einen programmierbaren Laserschneider für hohe Präzision. Wenn keine programmierbaren Maschinen zur Verfügung stehen, reicht eine Schere zum Schneiden des Bandumrisses aus und ein kleiner Locher ist für den Mittelausschnitt ausreichend.
HINWEIS: Kommerzieller Silikonkautschuk wird normalerweise mit Beidseitig mit Plastikfolie gekauft. Bewahren Sie nach Möglichkeit eine originale Kunststoffabdeckung auf beiden Seiten des Silikons auf. Wenn Sie Silikonstreifen aus einem früheren Experiment wiederverwenden, behandeln Sie sie 0,5 Stunden lang mit Trypsin, tränken Sie 0,5 Stunden lang 0,2 M NaOH ein und weichen Sie sie dann 1 Stunde lang in 70% Ethanol ein. Lassen Sie sie vor Gebrauch trocknen. - Bereiten Sie Dichtfilmschichten (hydrophobe) Schichten mit Abmessungen von mindestens 20 × 30 mm2vor, damit sie breiter als der Silikonstreifen sind und sich so eine Dichtung über den gesamten geometrischen Ausschnitt bilden kann.
- Waschen Sie eine 10-cm-Schüssel mit 70% Ethanol und wischen und trocknen Sie sie dann mit nicht linting empfindlichen Aufgabentüchern ab (sowohl für sterile als auch für nicht sterile Experimente). Dieser Schritt ist wichtig, da er es den Siegelfilmschichten ermöglicht, besser an der Platte zu haften und die Bewegung der Probe während des Vorbereitungsprozesses einzuschränken.
- Legen Sie die Siegelfolienschichten in die gewaschene 10-cm-Schüssel, damit genügend Platz vorhanden ist, um zwei Streifen nebeneinander in jede Schüssel zu legen (Abbildung 2A).
- Entfernen Sie die Plastikfolie von einer Seite des Silikonstreifens und legen Sie die freiliegende Seite auf die Siegelfolienschicht, so dass der Ausschnitt vollständig von der Siegelfolienschicht umgeben ist (Abbildung 2B). Drücken Sie dann vorsichtig auf das Silikon gegen die Dichtfilmschicht, um den Bereich um den Ausschnitt mit sauberen behandschuhten Fingern zu versiegeln.
HINWEIS: Stellen Sie sicher, dass sich keine Lufteinschlüsse zwischen dem Silikon und der Dichtfolie befinden, insbesondere um den Ausschnitt herum. Untersuchen Sie dazu die Unterseite der Schüssel (Abbildung 2C).
- Entfernen Sie die Plastikfolie von einer Seite des Silikonstreifens und legen Sie die freiliegende Seite auf die Siegelfolienschicht, so dass der Ausschnitt vollständig von der Siegelfolienschicht umgeben ist (Abbildung 2B). Drücken Sie dann vorsichtig auf das Silikon gegen die Dichtfilmschicht, um den Bereich um den Ausschnitt mit sauberen behandschuhten Fingern zu versiegeln.
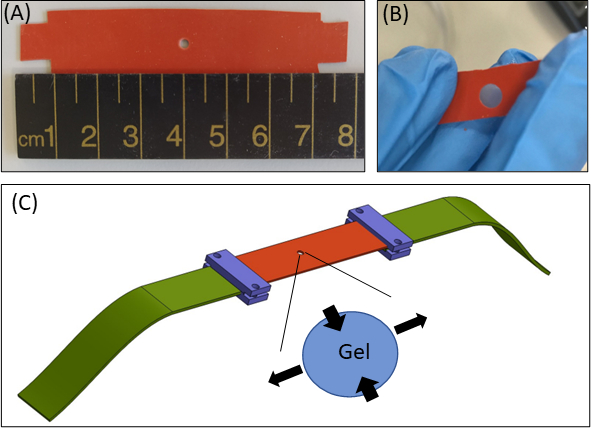
Abbildung 1: Hydrogel-Belastungsansatz. (A) 15 × 80 mm2 Silikonstreifen mit einem Ausschnitt von 2 mm Durchmesser in der Mitte des Streifens (B) Ein Silikonstreifen mit einem kreisförmigen Ausschnitt mit eingebettetem Fibringel. Zur Veranschaulichung ist der Ausschnitt im Silikon größer als in den eigentlichen Experimenten (C) Schematische Darstellung des Dehnansatzes mit dem Silikonstreifen (orange), kreisförmigem Gel (Ausschnitt in der Mitte) und Stoffverlängerern (grün), die das Silikon mit der Dehnvorrichtung verbinden. Vergrößerte Fläche des Gels zeigt die Verformung des Gels als Reaktion auf einachsige Dehnung des Silikons an. Der Einfachheit halber ist die Kompressionentlang der Dicke des Gels (Z-Achse) in der Abbildung nicht dargestellt. Die Abbildungen 1B & 1C wurden von Roitblat Riba et al.41adaptiertBitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 2: Beispiel für die korrekte Platzierung eines Silikonstreifens auf einer Dichtfilmschicht vor der Gelpolymerisation. (A) Platzierung von zwei Dichtfilmschichten in einer 10 cm großen Schüssel (B) Platzierung von Silikonstreifen auf den Dichtfilmschichten (C) Unteransicht der Schüssel, die die Luftdichtung zwischen dem Silikon und der Siegelfolienschicht zeigt. Links: Ordnungsgemäße Abdichtung der Dichtfilmschicht mit dem Silikonstreifen um den Ausschnitt ohne Lufteinschlüsse. Rechts: Unsachgemäße Abdichtung der Dichtfilmschicht zum Silikonstreifenausschnitt mit Lufteinschlüssen am Rand des Ausschnitts. Dies führt zu einem Auslaufen der Hydrogelkomponenten unter dem Silikon. Der rote Pfeil zeigt auf einen Bereich, in dem sich eine Lufttasche gebildet hat. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
3. Thrombin mit Zellen zubereiten
HINWEIS: Führen Sie diesen Schritt nur durch, wenn das Einbetten von Zellen in das Hydrogel gewünscht ist, und unter sterilen Bedingungen in einer biologischen Haube (Materialtabelle).
- Sterilisation: Am Tag vor dem Zellexperiment die Silikonstreifen und Siegelfilmschichten über Nacht in 70% Ethanol legen und dann 30 Minuten lang auf jeder Seite UV-Sterilisation durchführen (wenn das 10 cm Geschirr nicht bereits steril ist, sollte es auch nach einer 70% igen Ethanolwäsche 30 Minuten lang unter UV-Licht sterilisiert werden). Das im Protokoll verwendete UV-System ist dasjenige, das in die biologische Haube eingebaut ist.
HINWEIS: Alternativ kann ein Autoklaven-Sterilisationszyklus (140 °C) an den Silikonstreifen durchgeführt werden, da sie bis zu 260 °C beständig sind. - Führen Sie eine Zellzählung durch, um die Zellkonzentration zu bestimmen, und zentrifugieren und suspendieren Sie dann das Zellpellet mit 7 μL Thrombin (2 Einheiten / ml) in einem 1,5 ml Zentrifugenröhrchen. Wir empfehlen eine Zellkonzentration von 800 Zellen/μL Thrombin. Halten Sie die Zellen bis zum Gebrauch gekühlt (überschreiten Sie nicht mehr als eine halbe Stunde, um eine Schädigung der Zellen zu vermeiden).
4. Polymerisation von Fibringelen
- Holen Sie die 2 Unit/ ml Thrombin & 10 mg / ml markierte Fibrinogenlösungen aus dem Kühlschrank (vorbereitet in Schritt 1) und legen Sie sie auf Eis, wo sie zugänglich sind.
HINWEIS: Lösungen werden vor dem Mischprozess kalt gehalten, um die Kinetik der Polymerisationsreaktion zu verlangsamen. Dies ermöglicht eine homogenere Mischung der Proteine. - Extrahieren Sie bei der Einrichtung der Schüssel(n) (Schritt 2) 2,5 μL markiertes Fibrinogen und pipetieren Sie es gleichmäßig in den Silikonausschnitt (wobei die Siegelfilmschicht an der Unterseite befestigt ist), so dass der gesamte Umfang des Ausschnitts mit Fibrinogen in Kontakt kommt. Achten Sie darauf, dass sich nirgendwo in der Lösung Lufteinschlüsse oder Blasen bilden, wobei Sie besonders auf die unteren Kanten des Ausschnitts (die Schnittstelle zwischen der Dichtungsfolienschicht und silikon) achten.
- Nehmen Sie sofort 2,5 μL Thrombin (mit oder ohne Zellen/Perlen) und pipetieren Sie es direkt in die Fibrinogenlösung im Ausschnitt (erreichen Sie das Endvolumen von 5 μL Fibrin). Mischen Sie dann schnell die beiden Lösungen, indem Sie sorgfältig ~ 10 Mal auf und ab pipettieren. Bewegen Sie während des Mischvorgangs die Spitze um das gesamte Volumen, um eine möglichst homogene Lösung zu erhalten.
- Fügen Sie eine sehr kleine Menge Phosphatpuffersalzlösung (PBS) [alternativ Zellmedium für Zellexperimente, Materialtabelle]entlang des Randes jeder Schüssel hinzu, damit das Hydrogel während des Polymerisationsprozesses nicht austrocknet. Stellen Sie sicher, dass kein Kontakt zwischen dem PBS/Zellmedium und den Proben besteht, da dies die Probe beschädigt.
- Decken Sie die Schüssel(n) ab und stellen Sie sie für 30 Minuten bei 37 °C in den Inkubator.
HINWEIS: Die erforderliche Inkubationszeit ist abhängig vom Volumen des Gels. Werden größere Volumina verwendet, sollte sich die Inkubationszeit erhöhen. - Entfernen Sie die Schüssel(n) aus dem Inkubator und fügen Sie PBS / Zellmedium in die Schüssel hinzu, wobei das gesamte Gel-Silikon-Konstrukt untergetaucht wird.
- Heben Sie die Probenkonstrukte vorsichtig einzeln aus der Schüssel und stellen Sie sicher, dass die Siegelfilmschicht auf dem Streifen haftet. Lösen Sie die Dichtfilmschicht langsam vom Silikon, indem Sie vorsichtig von einem Ende des Silikons zum anderen rühren (Abbildung 3). Vermeiden Sie das Ziehen aus Bereichen in der Nähe des Ausschnitts, in denen Spannungskonzentrationen vorhanden sein können (dies ist hauptsächlich für nicht kreisförmige Geometrien wichtig). Vermeiden Sie jeglichen Kontakt mit dem Ausschnitt, da er die Probe beschädigt.
- Legen Sie den Streifen mit PBS / Zellmedium wieder in die Schüssel, so dass der Streifen in der Schüssel schwimmt. Dann nehmen Sie die Schüssel(n) zu einem Standard-Zellkulturmikroskop, um den Zustand jeder Probe qualitativ zu beurteilen. Gele müssen gleichmäßig und durchgehend im gesamten Ausschnitt sein und keine Blasen sollten vorhanden sein. Wählen Sie anhand von Abbildung 4 die besten Proben für die weitere Analyse aus.

Abbildung 3: Ordnungsgemäßes Entfernen der Dichtfilmschicht vom Boden des Silikonstreifens. Der Entfernungsprozess sollte langsam durchgeführt werden, damit das Hydrogel seine Haftung an den Innenwänden des Ausschnitts nicht reißt oder bricht. Der weiße Pfeil zeigt die Entfernungsrichtung an. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 4: Mikroskopische Beobachtung von Fibringelen im Silikonausschnitt. (A) Zwei Beispiele für ein richtig polymerisiertes Fibringel. Beachten Sie die relative Homogenität des Gels und die vollständige Haftung an den Rändern des Ausschnitts (B) Zwei Beispiele für Probenpolymerisationsfehler. Oben: Beachten Sie die vielen Blasen und die Aggregate, die sich unten links bilden. Unten: Beachten Sie das Reißen des Gels von den Ausschnittkanten und den Aggregaten im unteren linken Bereich des Ausschnitts. Maßstabsleiste = 300 μm Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
5. Probenladen auf das SCyUS-Gerät
- Füllen Sie das Bad mit PBS/Zellmedium (Abbildung 5) und legen Sie den Silikonstreifen, der das Probengel trägt, über die Oberseite, so dass die Enden auf jeder Seite des Bades sitzen. Das Bad soll ein Austrocknen des Gels vermeiden. Legen und festziehen Sie die Klemmen (lila) zusammen mit den Stoffstreifen (grün), so dass alle Teile zu einem geraden Streifen mit dem Ausschnitt in der Mitte verbunden sind (Abbildung 5).
- Rufen Sie das SCyUS-Gerät ab, und befestigen Sie die Aluminiumflüssigkeitsbohrung und den rechteckigen Glasdeckel von 22 mm x 40 mm (Nr. 1 oder 1,5)(Abbildung 6Aiii). Befestigen Sie den Deckelschlupf mit Dichtungsmaterial (z. B. Vakuumfett) an der Unterseite des Brunnens, damit der Brunnen ohne Leckage mit Flüssigkeit gefüllt werden kann.
- Füllen Sie den Brunnen mit ~ 1-2 ml PBS / Zellmedium und legen Sie den Streifen + Stoff + Gelkonstrukt (Abbildung 6B) in das Gerät. Klemmen Sie den Stoffstreifen (2) in die Halterung (1), so dass sich der Ausschnitt + Gel (5) wie gezeigt in der Mitte befindet, legen Sie dann vorsichtig den Pin-Down-Einsatz (4) in das Gerät und verriegeln Sie ihn.
- Als nächstes setzen Sie die andere Gewebeseite in die Spindel ein (ohne den Servomotor zu befestigen) und verriegeln Sie sie in der Spindel (Abbildung 6C).
- Setzen Sie das SCyUS-Gerät mit der angeschlossenen Probe in die Stufe des Mikroskops ein (Abbildung 6C). Verbinden Sie den Mikrocontroller (Table of Materials) über ein USB-Kabel mit dem Computer und verbinden Sie den Servomotor mit dem Mikrocontroller. Öffnen Sie das SCyUS-Steuermodul auf dem Computer. Die Probe ist nun bereit für die Bildgebung, um die Angemessenheit der Dicke des Gels und der Faserhomogenität unter dem konfokalen Mikroskop zu überprüfen.
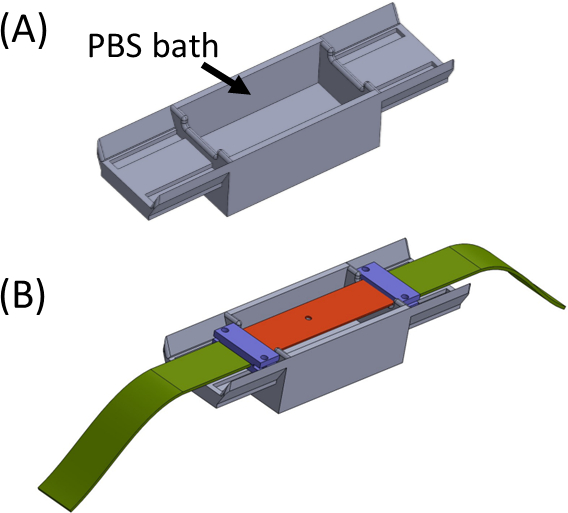
Abbildung 5: (A) Vorrichtung, die ein PBS-Bad enthält (3D-gedruckt) (B) Streifenplatzierung auf der Vorrichtung, um eine ordnungsgemäße Inline-Befestigung der Halterungen (in lila) zu gewährleisten und das Trocknen des Gels zu verhindern. Diese Zahl wurde mit Genehmigung von Roitblat Riba et al.41geändertBitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
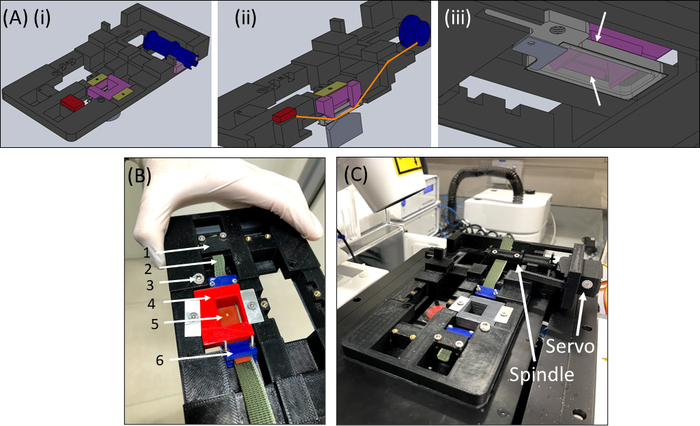
Abbildung 6: SCyUS-Stretching-Gerät. (A) Mehrere Ansichten eines CAD-Modells der Hauptteile des SCyUS: Spindel, die mit dem Servo verbunden ist (blau), statischer Anker (rot), Einsatz, der den Silikonstreifen festnagelt (lila) und Fixierer, die verhindern, dass der Einsatz aufsteigt (gelb-grün). Eine Oberansicht des Systems (Ai), eine Schnittansicht des Systems (Aii), die den Weg des Streifens (orange Linie) zeigt, und eine Unteransicht (Aiii) des Aluminiumflüssigkeitsbrunnens mit einem Glasdeckel. Der Flüssigkeitsbrunnen kann mit der Drehung einer Schraube, die in das Hauptgewinde einpasst, auf und ab bewegt werden. Die Aufwärtsbewegung der Aluminiumbohrung wird durch die Seitenflügel des violetten Einsatzes begrenzt, wie die weißen Pfeile zeigen (B) Das eigentliche System: (1) statischer Anker (2) grünes nicht dehnbares Gewebe ( 3 )Schraubezur Höhenkontrolle des Aluminiumflüssigkeitsbrunnens (4) roter Pin-Down-Einsatz (5) ein Silikonstreifen mit einem kreisförmigen Ausschnitt (6) blaue Verbindungsklemmen (C) Das Auf einem konfokalen Mikroskop platzierte Stretchsystem. Der Servomotor und die Spindel sind mit Pfeilen dargestellt. Diese Zahl wurde mit Genehmigung von Roitblat Riba et al.41geändertBitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
6. Stellen Sie sicher, dass ein ausreichendes Gel für die Probenahme geeignet ist
- Mit dem konfokalen Mikroskop (Table of Materials) wird ein konfokaler Z-Stack (≤10× Z-Scheiben mit einer Schrittgröße von ca. 10 μm ausreichend) mit niedriger Vergrößerung (~ 1,4 μm Pixelgröße) des gesamten Gels mit Lasern von 488/543/561 (≤10 Z-Scheiben mit einer Schrittgröße von ca. 10 μm ist ausreichend) Fliesenbild des gesamten Gels untersucht, um homogene Und Haftung am Umfang des geometrischen Ausschnitts während der gesamten Dicke des Silikons zu untersuchen ( Abbildung7A-B). Verwenden Sie dieses Z-Stack-Bild als Karte für die folgenden Schritte.
- Scannen Sie mit Hilfe der hochauflösenden Live-Bildgebung das Gel und bestimmen Sie die niedrigste Z-Position,bei der die volle Haftung an den Innenwänden des Ausschnitts ohne Risse oder Blasen sichtbar ist, und notieren Sie die Z-Positiondes Mikroskops ( Zl). Um die vollständige Haftung des Gels am Silikon während seines gesamten Umfangs zu bestimmen, scannen Sie die Grenzfläche der fluoreszierenden Markierung des Gels und des Silikonstreifens (dunkler Hintergrund) unter dem Mikroskop (Abbildung 7C).
- Bewegen Sie sich in Z-Richtungnach oben, bis das Gel keine Kontinuität mehr besteht, und beachten Sie die Z-Position(Zu):
- Subtrahieren Sie die obere Grenze (Zu) der Z-Richtungvon der unteren Grenze (Zl). Dies ist die Referenzdicke der Probe (Zo):

Wenn Zo ≥ 100 μm, gilt das Gel als zufriedenstellend für die Analyse. Beachten Sie, dass die Dicke des Silikonausschnitts etwa 500 μm beträgt, aber die Gelpolymerisation im Ausschnitt führt typischerweise zu einer geringeren Geldicke. 100 μm ist die empfohlene Mindestdicke, um einen stabilen Dehnungsprozess zu gewährleisten, ohne Reiße oder Ablösung des Gels vom Silikonausschnitt.
HINWEIS: An verschiedenen XY-Stellen kann die Dicke des Gels variieren. Dieser Abschnitt des Protokolls misst die minimale Dicke des Gels, so dass wir die Gelqualität bestimmen und angeben können, ob es zum Dehnen ausreicht. Darüber hinaus bietet das Finden der Mitte des Gels einen Bezugspunkt, um zum Post-Stretching zurückzukehren, egal ob statisch oder dynamisch.

Abbildung 7: Gelhomogenität. Kachelbilder wurden mit der konfokalen Mikroskopsoftware (Table of Materials) (A) aufgenommen und genäht Ein einzelnes genähtes Kachel-Z-Slice-Bild einer Fibringelprobe mit relativ inhomogener Faserdichte aufgrund unsachgemäßer Thrombin- und Fibrinogenmischung vor polymerisation. Dieses Gel liefert keine zuverlässige Analyse (B) Ein einzelnes genähtes Kachel-Z-Scheibenbild einer Fibringelprobe mit relativ homogener Faserdichte. Dies ist ein akzeptables Gel für Dehnungsexperimente. Maßstabsleiste für Bilder A & B ist 200 μm (C) Zoomen Sie die Grenzfläche zwischen dem fluoreszierend markierten Gel (rot) und dem Silikon (schwarzer Hintergrund) an. Maßstabsleiste = 100 μm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
7. SCyUS-Betrieb, Stretching & Imaging
- Nachdem nun festgestellt wurde, dass die Probe von zufriedenstellender Qualität ist und ordnungsgemäß auf das SCyUS-Gerät eingestellt ist, bestimmen Sie die vordehnte Position der Probe. Dies wird durch live-Bildgebung unter dem konfokalen Mikroskop erreicht (ähnlich wie Bei schritt 6.2).
- Stellen Sie sicher, dass sich der Servomotor in seiner Nullposition befindet, indem Sie auf die Schaltfläche Go To Zero Servo Pos klicken (Klicken Sie auf Abbildung 8A und stellen Sie sicher, dass in Abbildung 8B Null angezeigt wird), und befestigen Sie ihn an der Dehnungsvorrichtung , wie in Abbildung 6Cdargestellt.
HINWEIS: Führen Sie diesen Schritt langsam und vorsichtig aus, um die Probe nicht übermäßig zu belasten. - Bewegen Sie den Motor bei der Abbildung der Probe um ein Grad(Abbildung 8C)im Uhrzeigersinn, indem Sie auf die +1-Taste klicken, bis sich die rechte Seite des Ausschnitts bewegt. Kehren Sie dann die Bewegung (Abbildung 8D) zurück in die vorletzte Schrittposition, indem Sie auf die Schaltfläche -1 klicken. Dadurch wird überprüft, ob die Probe unter minimaler Spannung steht. Klicken Sie auf die Schaltfläche Min. Servoposition einstellen (Abbildung 8E), um die Referenzposition festzulegen. Es ist jederzeit möglich, zur Referenzposition zurückzukehren, indem Sie auf die Schaltfläche Go To Min Servo Position klicken ( Abbildung8F).
HINWEIS: Es wird empfohlen, für diesen Schritt ein Objektiv mit hoher Vergrößerung (≥40×) zu verwenden, um Fehler zu minimieren. - Erfassen Sie ein bild mit hoher Vergrößerung (40×), hoher Auflösung (~ 0,2 μm × 0,2 μm Pixelgröße), einem einzelnen Z-Slice-Kachelbild des gesamten Gelbereichs. Dieses wird als Referenzbild für die Nachbearbeitungsanalyse verwendet. Es wird empfohlen, ein einzelnes Z-Slice-Bild in der Mitte der Geldicke aufzunehmen (mit Zo von Eq. 1),dies ermöglicht die Rückkehr in ungefähr die gleiche Z-Positionnach dem Dehnen. Berücksichtigen Sie auch, dass hochauflösende Kachelbilder des gesamten Gelbereichs viel Zeit in Anspruch nehmen (~ 20-30 min).
- Nun ist die Probe bereit für die statische Dehnung. Stellen Sie den Servomotor auf die gewünschte Dehnungsgröße ein, indem Sie einen Grad(Abbildung 8C)gleichzeitig in der GUI vorrücken (führen Sie dies langsam durch, etwa 1 Grad / Sekunde).
- Stellen Sie sicher, dass sich der Servomotor in seiner Nullposition befindet, indem Sie auf die Schaltfläche Go To Zero Servo Pos klicken (Klicken Sie auf Abbildung 8A und stellen Sie sicher, dass in Abbildung 8B Null angezeigt wird), und befestigen Sie ihn an der Dehnungsvorrichtung , wie in Abbildung 6Cdargestellt.
- Erfassen Sie bei jeder Dehnungsgröße, bei der die Analyse gewünscht wird, ein einzelnes hochauflösendes Z-Slice-Kachelbild des gesamten Gelbereichs für die Nachbearbeitungsanalyse. Überprüfen Sie ähnlich wie bei Schritt 6.2, ob sich das Gel während seines gesamten Umfangs nicht vom Silikon gelöst hat, indem Sie die Grenzfläche zwischen dem Gel (rot) und dem Silikon (dunkler Hintergrund) scannen und nach Änderungen der Haftung gegenüber der vorherigen Dehnungsgröße suchen.
HINWEIS: Verwenden Sie während der Aktivierung des Motors Live-Bildgebung, um die Gelposition in X-Y zu verfolgen (mit Einstellungen mit niedriger Auflösung und geringer Vergrößerung). Das Gel erfährt einen Z-Poisson-Effekt, bei dem der Boden des Gels ansteigt, daher sollte die Z-Positiondes Mikroskops auch für jede Dehnungsgröße auf den ungefähren Mittelpunkt der Geldicke eingestellt werden. Dies kann durch Neuberechnung von Zo (Eq. 1) für jede Dehnungsgröße erreicht werden. Da die Dehnung in Z-Richtungrelativ homogen ist, ist es nicht kritisch, die genaue Mittentiefe des Gels zu finden.
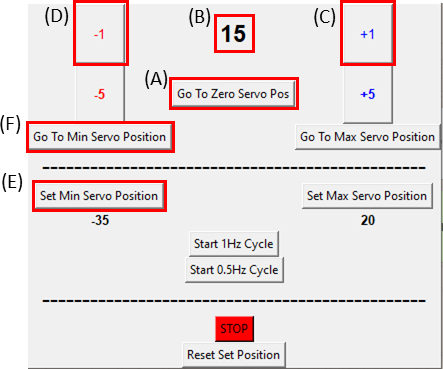
Abbildung 8: GUI für das SCyUS-Steuermodul. (A) Position des Motors in Grad. Der Wert reicht von -90° bis 90° (B) 'Minimale Servoposition einstellen'. Diese Taste ermöglicht eine voreingestellte Mindestposition, um eine neue Referenzposition einzustellen, die sich von der Taste Zero Servo Position (C) "Plus 1 °" unterscheidet, bewegt den Servomotor um einen Grad im Uhrzeigersinn (D) "Minus 1 °" Taste bewegt den Servomotor um ein Grad gegen den Uhrzeigersinn (E) "Zur Nullposition gehen" Taste setzt die Servomotorposition auf 0 ° ([A] wird auf Null gesetzt) (F) "Zur minimalen Servoposition gehen" Taste bewegt den Servomotor in die benutzerdefinierte "Min" Position. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
8. Nachbearbeitung externer Dehnungsmessungen
- Um die effektiven Dehnungen der Ausschnittgrenzen zu messen, messen Sie dieKantenlängen in der Dehnungsrichtung ( X-Achse) in der Mitte der Y-Achse(Abbildung 9A).
- Laden Sie das Pre-Stretch-Bild in die Bildverarbeitungssoftware (ImageJ FIJI43) hoch und messen Sie den größten Kanten-zu-Kanten-Abstand, der als axiale Länge der Bohrung ( ) in der Mitte definiert
 ist.
ist. - Definieren Sie den größten Abstand von oben nach unten als senkrechten Abstand (
 ).
). - Wiederholen Sie diesen Vorgang für alle Dehnungsintervallbilder, berechnen Sie die axialen (
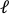 ) und senkrechten (
) und senkrechten (  ) Abstände der ausgeschnittenen Peripherie (Abbildung 9A, unten) und führen Sie dann die folgenden Berechnungen durch, um die Dehnungen der Ausschneidkanten zu ermitteln:
) Abstände der ausgeschnittenen Peripherie (Abbildung 9A, unten) und führen Sie dann die folgenden Berechnungen durch, um die Dehnungen der Ausschneidkanten zu ermitteln:
- Laden Sie das Pre-Stretch-Bild in die Bildverarbeitungssoftware (ImageJ FIJI43) hoch und messen Sie den größten Kanten-zu-Kanten-Abstand, der als axiale Länge der Bohrung ( ) in der Mitte definiert
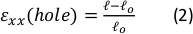

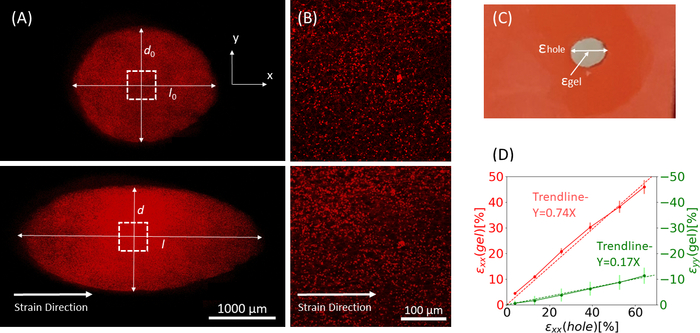
Abbildung 9: Gelbelastungen durch äußere Dehnung des Silikonstreifens. (A) X-Y-Querschnitt eines undehnten Fibringels (oben) und nach Dem Auftragen εLoch = 64% Dehnung entlang der x-Richtung (unten). Das Gel ist mit fluoreszierenden Perlen eingebettet. Die relevanten Längen von d und l, die für die Berechnung εLochs verwendet werden, sind in den Bildern angegeben (B) Zoom-in-Bilder der gestrichelten quadratischen Fläche, die in A (C) markiert ist Illustration der in dieser Studie berücksichtigten Dehnungstypen: εLoch ist die axiale Dehnung des Ausschnitts bei seinem maximalen Durchmesser, und εGel ist die axiale Dehnung in der Mitte des Gels (gemessen an den Perlenaggregatpositionen) (D) Es wurde eine lineare Beziehung zwischen εLoch und εGel in xx-Richtung (rote Linie) und yy-Richtung (grüne Linie) gefunden. Diese Abbildung wurde mit Genehmigung von Roitblat Riba et al.41angepasstBitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
9. Analyse der Faserorientierung
- Verwenden Sie die Quantifizierung der Faserausrichtung, um die strukturelle Reaktion des faserigen Gels auf zunehmende Dehnungsgrößen zu charakterisieren. Laden Sie hochauflösende Bilder in die ImageJ FIJI-Software (NIH)43 hoch und analysieren Sie sie dann mit dem Modul OrientationJ (EPFL)44 (Einstellungen: Gaußscher Farbverlauf und 3-Pixel-Fenster, Abbildung 10).
- Berechnen Sie den 2D Nematic Order Parameter (NOP) des Orientierungs histogramms wie:45
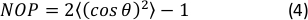
HINWEIS: Ein Wert von NOP = 1 zeigt die perfekte Ausrichtung entlang der axialen Richtung (Winkel Null) und NOP = 0 die Isotropie an. Der Orientierungswinkel θist der Faserwinkel in Bezug auf die Dehnungsachse (x-Achse), der durch Bildanalyse erhalten und in der OrientationJ-Dokumentation genau definiert ist. 44
- Berechnen Sie den 2D Nematic Order Parameter (NOP) des Orientierungs histogramms wie:45

Abbildung 10: Analyse der Faserorientierung mit der SOFTWARE FIJI ImageJ. (A) Hauptmenü von ImageJ mit einem Pfeil, der die Position des Pulldown-Menüs "Plugins" angibt, in dem sich "OrientationJ" befindet. Klicken Sie im erweiterten Menü von 'OrientationJ' auf die Option 'OrientationJ Distribution' (B) OrientationJ's Distribution Modul. Setzen Sie 'Lokales Fenster σ' auf 3 Pixel und 'Gradient' auf 'Gaussian'. Drücken Sie dann die Schaltfläche "Ausführen" (roter Pfeil). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
10. Manuelle interne Gelstammanalyse
- Während Sie live hochvergrößerungsreiche Bildgebung eines Hydrogels mit eingebetteten Perlen durchführen, lokalisieren Sie manuell eine Region von Interesse (ROI) mit leicht erkennbaren Merkmalen (z. B. Aggregate von Perlen), um nach jeder Dehnungsgröße an die gleiche Stelle zurückzukehren.
HINWEIS: Die Kompression in Z-Richtung (Poisson-Effekt) kann zu einer Erhöhung der Perlendichte führen, wenn die Dehnung zunimmt, daher empfehlen wir, Perlenaggregate zu wählen, die groß genug sind, damit sie klar identifizierbar sind. Dieses Protokoll erfordert die Analyse der zentralen Region des Fibringels, obwohl jede Region ausgewählt werden kann. - Erfassen Sie in der Pre-Stretch-Position (Schritt 6) ein hochauflösendes Z-Stack-Bild des ausgewählten ROI. Kehren Sie nach jedem gewünschten Dehnungsintervall zum gleichen ROI zurück und wiederholen Sie den Bilderfassungsvorgang.
- Nehmen Sie die Bilder und importieren Sie sie in ImageJ. Notieren Sie im ROI die X-Y-Pixelposition jedes sichtbaren Perlenaggregats. Übertragen Sie die aufgezeichneten Daten in eine Tabelle.
- Messen Sie die Abstände zwischen jedem Aggregatepaar und vergleichen Sie sie mit den Abständen der gleichen Paare im Referenzbild, um die Berechnung von Dehnungen in X- und Y-Richtung zu ermöglichen.
HINWEIS: Wenn ein kontinuierlicher Echtzeitfilm aufgezeichnet wird, während das Gel gestreckt wird (anstelle einer statischen Bildaufnahme), kann eine automatische Analyse von Dehnungen mit digitalen Bild- oder Volumenkorrelationsmethoden (DIC / DVC) durchgeführt werden, wie zuvor gezeigt46,47. Es ist jedoch zu beachten, dass die automatische DIC/DVC-Analyse in dieser Einstellung eine Herausforderung darstellt, da sich der Z-Stackaufgrund des Poisson-Effekts (Kompression) nicht nur in der X-Y-Ebene, sondern auch in Z-Richtungbewegt, was zu erheblichen Drifts während des aufgenommenen Films führt.
Access restricted. Please log in or start a trial to view this content.
Ergebnisse
Repräsentative Daten von statischen Dehnungen zunehmender Größenordnungen, die auf den Silikonstreifen mit einem 3D-Fibrinhydrogel aufgebracht werden, eingebettet mit 1 μm fluoreszierenden Perlen, sind in Abbildung 9 dargestellt. Die Analyse zeigt die Wirkung von Silikondehnung auf geometrische Veränderungen des Ausschnitts sowie die entwickelten Dehnungen innerhalb des Gels. Z-Stapelbilderdes gesamten Gels werden verwendet, um die Verformung des ursprünglichen kreisf...
Access restricted. Please log in or start a trial to view this content.
Diskussion
Die hier vorgestellte Methode und das Protokoll basieren weitgehend auf unserer früheren Studie von Roitblat Riba et al.41 Wir schließen hier die vollständigen Computer-Aided Design (CAD), Python- und Mikrocontroller-Codes des SCyUS-Geräts ein.
Zu den wesentlichen Vorteilen der vorgestellten Methode gegenüber bestehenden Ansätzen gehört die Möglichkeit, sehr weiche 3D-Hydrogele (Elastizitätsmodul von ~100 Pa) aus ihrem Umfang und unter konfokale Li...
Access restricted. Please log in or start a trial to view this content.
Offenlegungen
Die Autoren haben nichts preiszugeben.
Danksagungen
Einige hier enthaltene Figuren wurden mit Genehmigung des Copyright Clearance Center angepasst: Springer Nature, Annals of Biomedical Engineering. Straining von 3D-Hydrogelen mit gleichmäßigen Z-Achsen-Dehnungen bei gleichzeitiger Ermöglichung von Live-Mikroskopie-Bildgebung, A. Roitblat Riba, S. Natan, A. Kolel, H. Rushkin, O. Tchaicheeyan, A. Lesman, Copyright© (2019).
https://doi.org/10.1007/s10439-019-02426-7
Access restricted. Please log in or start a trial to view this content.
Materialien
| Name | Company | Catalog Number | Comments |
| Alexa Fluor 546 carboxylic acid, succinimidyl ester | Invitrogen | A20002 | |
| Cell Medium (DMEM High Glucose) | Biological Industries | 01-052-1A | Add 10% FBS, 1% PNS, 1% L-Glutamine, 1% Sodium Pyruvate |
| Cover Slip #1.5 | Bar-Naor Ltd. | BN72204-30 | 22×40 mm |
| DIMETHYL SULPHOXIDE 99.5% GC DMSO | Sigma-Aldrich Inc. | D-5879-500 ML | |
| Dulbecco's Phosphate-Buffered Saline | Biological Industries | 02-023-1A | |
| EVICEL Fibrin Sealant (Human) | Omrix Biopharmaceuticals | 3902 | Fibrinogen: 70 mg/mL, Thrombin: 800-1200 IU/mL |
| Fibrinogen Buffer | N/A | Recipe for 1L: 7g NaCl, 2.94g trisodium citrate dihydrate, 9g glycine, 20g arginine hydrochloride & 0.15g calcium chloride dihydrate. Bring final volume to 1L with PuW (pH 7.0-7.2) | |
| Fluorescent micro-beads FluoSpheres (1 µm) | Invitrogen | F8820 | Orange (540/560) Provided as suspension (2% solids) in water plus 2 mM sodium azide |
| High-Temperature Silicone Rubber | McMaster-Carr | 3788T41 | 580 µm-thick E = 1.5 Mpa Poisson Ratio = 0.48 Tensile Strength = 4.8 MPa Upper limit of stretch = +300% engineering strain |
| HiTrap desalting column 5 mL (Sephadex G-25 packed) | GE Healthcare | 17-1408-01 | |
| HIVAC-G High Vacuum Sealing Compound | Shin-Etsu Chemical Co., Ltd. | HIVAC-G 100 | |
| ImageJ FIJI software39 | National Institute of Health, Bethesda, MD | Version 1.8.0_112 | |
| Microcontroller (Adruino Uno + Adafruit Motorshield v2.3) | Arduino/Adafruit | Arduino-DK001/Adafruit-1438 | |
| MicroVL 21R Centrifuge | Thermo Scientific | 75002470 | |
| Parafilm | Bemis | PM-996 | |
| Primovert Light Microscope | Carl Zeiss Suzhou Co., Ltd. | 491206-0011-000 | |
| SCyUS CAD (Solidworks) | Dassault Systèmes | N/A | |
| SCyUS Code37 | N/A | N/A | |
| Servomotor - TowerPro SG-5010 | Adafruit | 155 | |
| SL 16R Centrifuge | Thermo Scientific | 75004030 | For 50 mL tubes |
| Sterile 10 cm non-culture plates | Corning | 430167 | |
| Thrombin buffer | N/A | Recipe for 1L: 20g mannitol, 8.77g NaCl, 2.72g sodium acetate trihydrate, 24 mL 25% Human Serum Albumin, 5.88g calcium chloride. Bring final volume to 1L with PuW (pH 7.0) | |
| Trypsin EDTA Solution B (0.25%), EDTA (0.05%) | Biological Industries | 03-052-1B | |
| USB Cable (Type B Male to Type A Male) | N/A | N/A | |
| Zeiss LSM 880 Confocal Microscope | Carl Zeiss AG | 2811000417 | |
| ZEN 2.3 SP1 FP3 (black) | Carl Zeiss AG | Release Version 14.0.0.0 |
Referenzen
- Bleuel, J., Zaucke, V., Bruggemann, G. P., Niehoff, A. Effects of cyclic tensile strain on chondrocyte metabolism: a systematic review. PLoS ONE. 10, 0119816(2015).
- Pennisi, C. P., Olesen, C. G., de Zee, M., Rasmussen, J., Zachar, V. Uniaxial cyclic strain drives assembly and differentiation of skeletal myocytes. Tissue Engineering Part A. 17, 2543-2550 (2011).
- Grodzinsky, A. J., Levenston, M. E., Jin, M., Frank, E. H. Cartilage Tissue Remodeling in Response to Mechanical Forces. Annual Review of Biomedical Engineering. 2 (1), 691-713 (2000).
- Munster, S., et al. Strain history dependence of the nonlinear stress response of fibrin and collagen networks. Proceedings of the National Academy of Sciences of the USA. 110, 12197-12202 (2013).
- Vader, D., Kabla, A., Weitz, D., Mahadevan, L. Strain-induced alignment in collagen gels. PLoS ONE. 4, 5902(2009).
- Badylak, S. F. The extracellular matrix as a scaffold for tissue reconstruction. Seminars in Cell & Developmental Biology. 13 (5), 377-383 (2002).
- Natan, S., Koren, Y., Shelah, O., Goren, S., Lesman, A. Molecular Biology of the Cell. 31 (14), 1474-1485 (2020).
- Ban, E., et al. Mechanisms of Plastic Deformation in Collagen Networks Induced by Cellular Forces. Biophysical Journal. 114 (2), 450-461 (2018).
- Kim, J., et al. Stress-induced plasticity of dynamic collagen networks. Nature Communications. 8, 842(2017).
- Storm, C., Pastore, J. J., MacKintosh, F. C., Lubensky, T. C., Janmey, P. A. Nonlinear elasticity in biological gels. Nature. 435, 191-194 (2005).
- Wen, Q., Basu, A., Janmey, P. A., Yodh, A. G. Non-affine deformations in polymer hydrogels. Soft Matter. 8, 8039-8049 (2012).
- Muiznieks, L. D., Keeley, F. W. Molecular assembly and mechanical properties of the extracellular matrix: A fibrous protein perspective. Biochimica et Biophysica Acta. 1832, 866-875 (2012).
- Brown, A. E. X., Litvinov, R. I., Discher, D. E., Purohit, P. K., Weisel, J. W. Multiscale mechanics of fibrin polymer: gel stretching with protein unfolding and loss of water. Science. 325, 741-744 (2009).
- Carroll, S. F., Buckley, C. T., Kelly, D. J. Cyclic tensile strain can play a role in directing both intramembranous and endochondral ossification of mesenchymal stem cells. Frontiers in Bioengineering and Biotechnology. 5, 73(2017).
- Livne, A., Bouchbinder, E., Geiger, B. Cell reorientation under cyclic stretching. Nature Communications. 5, 3938(2014).
- Wang, L., et al. Patterning cellular alignment through stretching hydrogels with programmable strain gradients. ACS Applied Materials & Interfaces. 7, 15088-15097 (2015).
- Xu, G. K., Feng, X. Q., Gao, H. Orientations of Cells on Compliant Substrates under Biaxial Stretches: A Theoretical Study. Biophysical Journal. 114 (3), 701-710 (2017).
- Chagnon-Lessard, S., Jean-Ruel, H., Godin, M., Pelling, A. E. Cellular orientation is guided by strain gradients. Integrative Biology (United Kingdom). 9 (7), 607-618 (2013).
- Lu, J., et al. Cell orientation gradients on an inverse opal substrate. ACS Applied Materials & Interfaces. 7 (19), 10091-10095 (2015).
- Baker, B. M., Chen, C. S. Deconstructing the third dimension - 3D culture microenvironments alter cellular cues. Journal of Cell Science. 125, 3015-3024 (2012).
- Bono, N., et al. Unraveling the role of mechanical stimulation on smooth muscle cells: a comparative study between 2D and 3D models. Biotechnology and Bioengineering. 113, 2254-2263 (2016).
- Pampaloni, F., Reynaud, E. G., Stelzer, E. H. K. The third dimension bridges the gap between cell culture and live tissue. Nature Reviews Molecular Cell Biology. 8, 839-845 (2007).
- Riehl, B. D., Park, J. H., Kwon, I. K., Lim, J. Y. Mechanical stretching for tissue engineering: two-dimensional and three-dimensional constructs. Tissue Engineering Part B: Reviews. 18, 288-300 (2012).
- Flexcell. Linear Tissue Train Culture Plate. Flexcell. , (2019).
- Flexcell. Tissue Train. Flexcell. , (2019).
- CellScale. MCT6 Stretcher. CellScale. , (2019).
- STREX. STB-150. STREX. , (2019).
- STREX. Stretch Chambers. STREX. , (2019).
- Kamble, H., Barton, M. J., Jun, M., Park, S., Nguyen, N. T. Cell stretching devices as research tools: engineering and biological considerations. Lab on a Chip. 16, 3193-3203 (2016).
- Weidenhamer, N. K., Tranquillo, R. T. Influence of cyclic mechanical stretch and tissue constraints on cellular and collagen alignment in fibroblast-derived cell sheets. Tissue Engineering Part C: Methods. 19, 386-395 (2013).
- Yung, Y. C., Vandenburgh, H., Mooney, D. J. Cellular strain assessment tool (CSAT): precision-controlled cyclic uniaxial tensile loading. Journal of Biomechanics. 42, 178-182 (2009).
- Chen, K., et al. Role of boundary conditions in determining cell alignment in response to stretch. Proceedings of the National Academy of Sciences of the USA. 115, 986-991 (2018).
- Heher, P., et al. A novel bioreactor for the generation of highly aligned 3D skeletal muscle-like constructs through orientation of fibrin via application of static strain. Acta Biomaterialia. 24, 251-265 (2015).
- Foolen, J., Deshpande, V. S., Kanters, F. M. W., Baaijens, F. P. T. The influence of matrix integrity on stress-fiber remodeling in 3D. Biomaterials. 33, 7508-7518 (2012).
- Walker, M., Godin, M., Pelling, A. E. A vacuum-actuated microtissue stretcher for long-term exposure to oscillatory strain within a 3D matrix. Biomedical Microdevices. 20, 43(2018).
- Zhao, R. G., Boudou, T., Wang, W. G., Chen, C. S., Reich, D. H. Decoupling cell and matrix mechanics in engineered microtissues using magnetically actuated microcantilevers. Advanced Materials. 25, 1699-1705 (2013).
- Li, Y. H., et al. Magnetically actuated cell-laden micro-scale hydrogels for probing strain-induced cell responses in three dimensions. NPG Asia Materials. 8, 238(2016).
- Li, Y. H., et al. An approach to quantifying 3D responses of cells to extreme strain. Scientific Reports. 6, 19550(2016).
- Humphrey, J. D., et al. A theoretically-motivated biaxial tissue culture system with intravital microscopy. Biomechanics and Modeling in Mechanobiology. 7, 323-334 (2008).
- Niklason, L. E., et al. Enabling tools for engineering collagenous tissues integrating bioreactors, intravital imaging, and biomechanical modeling. Proceedings of the National Academy of Sciences of the USA. 107, 3335-3339 (2010).
- Roitblat Riba, A., et al. Straining 3D hydrogels with uniform z-axis strains while enabling live microscopy imaging. Annals of Biomedical Engineering. , (2019).
- Gomez, D., Natan, S., Shokef, Y., Lesman, A. Mechanical interaction between cells facilitates molecular transport. Advanced Biosystems. 3 (12), 1900192(2019).
- Schindelin, J., et al. Fiji: an open- source platform for biological-image analysis. Nature Methods. 9, 676-682 (2012).
- EPFL Switzerland. OrientationJ plug in. EPFL Switzerland. , (2019).
- Goren, S., Koren, Y., Xu, X., Lesman, A. Elastic anisotropy governs the decay of cell-induced displacements. Biophysical Journal. 118 (5), 1152-1164 (2019).
- Notbohm, J., Lesman, A., Tirrell, D. A., Ravichandran, G. Quantifying cell-induced matrix deformation in three dimensions based on imaging matrix fibers. Integrative Biology. 7 (10), 1186-1195 (2015).
- Lesman, A., Notbohm, J., Tirrell, D. A., Ravichandran, G. Contractile forces regulate cell division in three-dimensional environments. Journal of Cell Biology. 205 (2), 155-162 (2014).
- Cha, C. Y., et al. Tailoring Hydrogel Adhesion to Polydimethylsiloxane Substrates Using Polysaccharide Glue. Angewandte Chemie International Edition. 52, 6949-6952 (2019).
- Wirthl, D., et al. Instant tough bonding of hydrogels for soft machines and electronics. Science Advances. 3, (2017).
- Juarez-Moreno, J. A., Avila-Ortega, A., Oliva, A. I., Aviles, F., Cauich-Rodriguez, J. V. Effect of wettability and surface roughness on the adhesion properties of collagen on PDMS films treated by capacitively coupled oxygen plasma. Applied Surface Science. 349, 763-773 (2015).
- Kim, H. T., Jeong, O. C. PDMS surface modification using atmospheric pressure plasma. Microelectronic Engineering. 88, 2281-2285 (2011).
- Prasad, B. R., et al. Controlling cellular activity by manipulating silicone surface roughness. Colloids and Surfaces. 78, 237-242 (2010).
Access restricted. Please log in or start a trial to view this content.
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten