É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Metabolômica Direcionada Baseada em Cromatografia Gasosa e Espectrometria de Massas de Amostras de Corais Duros
Neste Artigo
Resumo
Aqui, apresentamos a extração e preparação de metabólitos polares e semipolares de um holobionte de coral, bem como tecido hospedeiro coral separado e frações celulares de Symbiodiniaceae, para análise por cromatografia gasosa acoplada à espectrometria de massas.
Resumo
Abordagens baseadas em cromatografia gasosa e espectrometria de massas (GC-MS) provaram ser poderosas para elucidar a base metabólica da simbiose cnidário-dinoflagelado e como os corais respondem ao estresse (isto é, durante o branqueamento induzido pela temperatura). O perfil de metabólitos em estado estacionário do holobionte do coral, que compreende o hospedeiro cnidário e seus micróbios associados (Symbiodiniaceae e outros protistas, bactérias, arqueias, fungos e vírus), tem sido aplicado com sucesso sob condições ambientais e de estresse para caracterizar o estado metabólico holístico do coral.
No entanto, para responder às questões que envolvem as interações simbióticas, é necessário analisar os perfis de metabólitos do hospedeiro coral e seus simbiontes algais de forma independente, o que só pode ser alcançado pela separação física e isolamento dos tecidos, seguido de extração e análise independentes. Embora a aplicação da metabolômica seja relativamente nova no campo dos corais, os esforços sustentados de grupos de pesquisa resultaram no desenvolvimento de métodos robustos para analisar metabólitos em corais, incluindo a separação do tecido hospedeiro dos corais e dos simbiontes algais.
Este artigo apresenta um guia passo-a-passo para a separação de holobiontes e a extração de metabólitos para análise de CG-EM, incluindo os principais passos de otimização para consideração. Demonstramos como, uma vez analisado independentemente, o perfil combinado de metabólitos das duas frações (coral e Symbiodiniaceae) é semelhante ao perfil do todo (holobionte), mas separando os tecidos, também podemos obter informações importantes sobre o metabolismo e as interações entre os dois parceiros que não podem ser obtidas do todo isoladamente.
Introdução
Metabólitos representam os produtos finais dos processos celulares, e a metabolômica - o estudo do conjunto de metabólitos produzidos por um determinado organismo ou ecossistema - pode fornecer uma medida direta do funcionamento do organismo1. Isso é particularmente crítico para explorar ecossistemas, interações simbióticas e ferramentas de restauração, já que o objetivo da maioria das estratégias de manejo é preservar (ou restaurar) funções específicas de serviços ecossistêmicos2. Os recifes de coral são um ecossistema aquático que demonstra o valor potencial da metabolômica para elucidar interações simbióticas e ligar as respostas fisiológicas dos corais aos impactos em nível de comunidade e ecossistema3. A aplicação de cromatografia gasosa de alto rendimento-espectrometria de massas (GC-MS) é especialmente valorizada devido à sua capacidade de analisar rapidamente uma ampla gama de classes de metabólitos simultaneamente com alta seletividade e sensibilidade, fornecer rápida identificação de compostos quando bibliotecas espectrais estão disponíveis e fornecer um alto nível de reprodutibilidade e precisão, com um custo relativamente baixo por amostra.
Os corais são holobiontes constituídos pelo animal coral, endossimbiontes dinoflagelados fotossintéticos (família: Symbiodiniaceae4) e um microbioma complexo 5,6. De modo geral, a aptidão do holobionte é mantida principalmente através da troca de pequenas moléculas e elementos para apoiar o funcionamento metabólico de cada membro 7,8,9,10. As abordagens metabolômicas têm se mostrado especialmente poderosas para elucidar as bases metabólicas da especificidade da simbiose9,11, a resposta clareadora ao estresse térmico 7,8,12,13, as respostas à doença 14, as respostas à exposição à poluição 15, a fotoaclimatação 16 e a sinalização química 17 em corais, além de auxiliar na descoberta de biomarcadores 18,19. Além disso, a metabolômica pode fornecer confirmação valiosa das conclusões inferidas a partir de técnicas baseadas em DNA eRNA9,20. Há, portanto, considerável potencial para o uso de metabolômicas para avaliar a saúde recifal e desenvolver ferramentas para a conservação recifal3, como por meio da detecção de biomarcadores metabólicos de estresse18,19 e para examinar o potencial de estratégias de manejo ativo, como subsídios nutricionais21.
Separar as células hospedeiras e simbiontes e analisar seus perfis metabólicos de forma independente, em vez de juntos como o holobionte, pode fornecer mais informações sobre as interações do parceiro, estados fisiológicos e metabólicos independentes e potenciais mecanismos moleculares de adaptação 11,12,22,23,24. Sem separar o coral e Symbiodiniaceae, é quase impossível elucidar a contribuição e o metabolismo de corais e/ou Symbiodiniaceae de forma independente, exceto com reconstrução complexa do genoma e modelagem metabólica25, mas isso ainda não foi aplicado à simbiose coral-dinoflagelado. Além disso, tentar extrair informações sobre o metabolismo individual do hospedeiro ou simbionte algal do perfil de metabólitos do holobionte pode levar a interpretações errôneas.
Por exemplo, até recentemente, acreditava-se que a presença de ácidos graxos poli-insaturados C18:3n-6, C18:4n-3 e C16 em extratos de tecidos de coral e holobionte era derivada do simbionte algal, pois os corais eram assumidos como não possuindo as desaturases ωx essenciais para a produção de ácidos graxos ômega-3 (ω3); no entanto, evidências genômicas recentes sugerem que múltiplos cnidários têm a capacidade de produzir PUFA ω3 de novo e biossintetizar PUFA ω3 de cadeia longa26. A combinação de GC-MS com marcação isotópica estável (por exemplo, 13 C-bicarbonato, NaH 13CO 3) pode ser usada para rastrear o destino do carbono fotossinteticamente fixado através de redes metabólicas holobiont de coral sob condições de controle e em resposta a estressores externos27,28. No entanto, um passo crítico no rastreamento do destino de 13 C é a separação do tecido coral das células de algas - só então a presença de um composto marcado com 13C na fração hospedeira do coral pode ser inequivocamente atribuída como um metabólito derivado de Symbiodiniaceae translocado para o coral ou um produto a jusante de um composto marcado translocado. Essa técnica demonstrou seu poder ao desafiar a suposição de longa data de que o glicerol é a forma primária na qual a fotossíntese é translocada do simbionte para o hospedeiro29, bem como elucidar como o fluxo nutricional entre parceiros muda durante o clareamento27,28 e em resposta a espécies incompatíveis de Symbiodiniaceae11.
Embora a decisão de separar tecidos seja impulsionada principalmente pela questão de pesquisa, a praticidade, a confiabilidade e os potenciais impactos metabólicos dessa abordagem são importantes a serem considerados. Aqui, nós fornecemos métodos detalhados e demonstrados para a extração de metabólitos do holobionte, bem como as frações separadas do hospedeiro e do simbionte. Nós comparamos os perfis de metabólitos do hospedeiro e simbionte independentemente e como esses perfis se comparam com o perfil de metabólitos holobiontes.
Protocolo
NOTA: O planejamento experimental, a coleta e o armazenamento das amostras foram descritos em detalhes em outra publicação 2,30,31. A aprovação da licença para a recolha de corais selvagens deve ser obtida antes da recolha e experimentação. As amostras foram coletadas de colônias de Montipora mollis (cor verde-morfo) importadas da Batavia Coral Farms (Geraldton, WA), originalmente coletadas de um recife ao largo das Ilhas Abrohlos (Austrália Ocidental; 28°52'43.3"S 114°00'17.0"E) a uma profundidade de 1 m sob a Licença de Aquicultura AQ1643. Antes da amostragem, as colônias foram mantidas em um aquário de 800 L a 35 PSU, sob luz azul e branca a 150 μmol de fótons·m−2·s−1, e a 25 °C ± 0,5 °C por 3 meses. Os fragmentos de coral (~5 cm2, N = 6) foram congelados em nitrogênio líquido e armazenados a −80 °C até o processamento.
1. Preparação das soluções e equipamentos de extração
- Pelo menos 1 dia antes da remoção do tecido de coral, prepare as soluções e equipamentos de extração.
- Pré-resfriar água ultrapura em copos limpos e sem detergente a 4 °C.
- Misture metanol de grau LC a 100% com uma concentração final de 10 μg·mL−1 do(s) padrão(s) interno(s) apropriado(s) (por exemplo, 13C6 sorbitol).
- Crie uma solução de extração de metanol a 50% usando metade de metanol 100% LC e metade de água ultrapura. Conservar ambas as soluções de metanol a -20 °C.
NOTA: Para ajudar a evitar a degradação dos metabolitos, recomenda-se realizar as etapas de processamento da amostra em lotes de cinco fragmentos de coral de cada vez, com um adicional biológico (somente água) em branco (total de amostras N = 6). Uma vez que cada amostra de coral tenha sido separada em duas frações (tecido hospedeiro coral, doravante "Hospedeiro", e células microalgais, doravante "Simbionte"), o número total de amostras em um lote de processamento será de 12.
2. Extinção do metabolismo dos corais
NOTA: O planejamento experimental, a coleta e o armazenamento das amostras foram descritos em detalhes em outra publicação 2,30,31. No entanto, deve-se notar que o tempo necessário para extinguir o metabolismo (ou seja, o tempo entre a coleta e a preservação das amostras de coral) é crítico para capturar a resposta original30. Preservar a amostra o mais rápido possível após a coleta para evitar alterações na composição do metabólito devido à degradação da amostra ou respostas fisiológicas não alvo32.
- Coloque o fragmento de coral em um saco de coleta de amostra estéril e escorra o máximo possível qualquer excesso de água do mar. Submergir a amostra em azoto líquido durante um mínimo de 30 s. Mover as amostras o mais rapidamente possível para um congelador de -80 °C para armazenamento.
NOTA: As amostras podem ser congeladas a -80°C em recipientes bloqueados à luz até o processamento, evitando ciclos de congelamento-descongelamento.
3. Remoção do tecido coral do esqueleto
NOTA: As amostras devem ser mantidas sempre em gelo (4 °C) para garantir que estão simultaneamente na forma líquida, evitando o metabolismo contínuo.
- Coloque um saco de coleta de amostras limpo e estéril no gelo para que o saco fique estável e aberto em cima do gelo em um poço raso, mas não fique submerso no gelo. Adicionar 10 ml de água ultrapura fria (4 °C) ao saco.
NOTA: Isso ajudará a evitar o congelamento-descongelamento repetido do fragmento de coral devido ao ar frio pressurizado e ao gelo circundante. - Selecione um fragmento de coral com pinça esterilizada e lave com água ultrapura fria (4 °C) usando uma pipeta Pasteur estéril até que nenhum resíduo de água do mar permaneça. Submergir o fragmento de coral enxaguado no saco contendo os 10 mL de água ultrapura.
NOTA: Este enxágue é fundamental para remover quaisquer sais residuais que possam interferir com a análise a jusante. Evitar o contacto das mãos com a água ou o fragmento de coral através do saco para manter a amostra a 4 °C. - Colocar uma ponta de pipeta estéril de 1 mL sobre a extremidade de uma pistola de ar com fita adesiva elétrica, com ~5 mm cortados na extremidade da ponta (Figura 1A).
- Aponte a pistola de ar para o fragmento de coral com o saco semi-selado e o fluxo de ar em baixo-médio para remover suavemente o tecido, incentivando um movimento circular da água sobre o fragmento de coral.
- Após ~3 min, ou quando todo o tecido parece ter sido removido do esqueleto, desligue o ar e remova o aerógrafo. Sele completamente o saco.
- Esprema todo o tecido de coral removido para um canto inferior do saco. Corte o canto oposto e despeje suavemente o conteúdo do saco em um tubo de 15 mL sobre gelo.
4. Homogeneização opcional
NOTA: Algumas espécies de corais são mais viscosas do que outras, o que significa que a escovação de ar removerá o tecido em touceiras em vez de em uma lama. Se aglomerados de tecido forem visíveis no homogeneizado escovado a ar, uma etapa de homogeneização a 4 °C pode ser adicionada para todas as amostras.
- Limpe um homogeneizador mecânico de dente de serra duas vezes com 4 °C de metanol a 70% e, finalmente, com água ultrapura a 4 °C.
- Homogeneizar a amostra de coral em um tubo de 15 mL por ~1 min até que a amostra esteja totalmente homogeneizada e nenhum aglomerado seja visível.
- Limpar o homogeneizador como no passo 4.1 entre cada amostra. Mantenha o tempo de homogeneização consistente em todas as amostras.
5. Coleta de amostras para normalização
- Coletar uma alíquota de 1.000 μL do tecido homogeneizado para contagem de células de Symbiodiniaceae, análise do conteúdo de proteína do tecido do hospedeiro coral e estimativa da clorofila a . Conservar a -20 °C até estar pronto a analisar (secção 10).
6. Separação opcional de células de Symbiodiniaceae com tecido hospedeiro de coral
- Centrifugar o homogeneizado de coral a 2.500 × g por 5 min a 4 °C usando uma centrífuga refrigerada.
NOTA: Esta velocidade é ideal para separar as células Symbiodiniaceae mais pesadas, mantendo suas paredes celulares intactas, do tecido hospedeiro, que está suspenso no sobrenadante. - Retire o sobrenadante que contém o material hospedeiro e coloque em um novo tubo de 15 mL.
NOTA: Os lipídios do tecido hospedeiro tipicamente formam uma estreita camada rosa/branca no topo das células simbiontes. Essa camada pode ser coletada juntamente com o sobrenadante solúvel do hospedeiro por pipetagem (Figura 1B). - Vórtice vigorosamente o anfitrião por exatamente 1 min. Manter a amostra de pellet de alga e a amostra sobrenadante do hospedeiro no gelo.
- Adicionar 2 ml de água ultrapura a 4 °C ao pellet de algas. Vórtice vigorosamente por exatos 2 min para ressuspender a pelota.
NOTA: Se fragmentos individuais de 1 cm não foram coletados da colônia de corais para genotipagem de Symbiodiniaceae, uma alíquota de 200 μL da suspensão celular de Symbiodiniaceae pode ser coletada aqui, preservada na solução tampão de DNA preferida e armazenada conforme descrito em Thurber et al.30 para genotipagem de Symbiodiniaceae (por exemplo, de acordo com González-Pech et al.12). - Repita as etapas 6.1-6.4 mais uma vez.
NOTA: A separação confiável do hospedeiro e do simbionte depende da biomassa e das espécies de corais, pois algumas espécies podem ser mais viscosas do que outras. Recomenda-se um mínimo de três etapas de lavagem, mas isso pode ser aumentado dependendo do sucesso da separação. Repita as etapas de lavagem 4.7-4.9 até que nenhuma célula Symbiodiniaceae possa ser vista na parte inferior da fração hospedeira e até que a fração Symbiodiniaceae esteja visivelmente livre de material hospedeiro (por exemplo, nenhuma camada branca na parte superior) (Figura 1). - Retire o sobrenadante que contém o material hospedeiro e coloque em um novo tubo de 15 mL.
- Reter o pellet simbionte no tubo de 15 mL.
7. Secagem da amostra
- Congelar o homogeneizado holobionto ou ambas as frações separadas do hospedeiro e Symbiodiniaceae, a -80 °C por ~120 min. Liofilizar as amostras durante a noite com um vácuo de 0,01 mbar a -85 °C.
NOTA: Para evitar a perda de amostra durante a liofilização, recomenda-se usar uma tampa cortada de outro tubo estéril, ou parafilme estéril, com um orifício de ~2 mm perfurado cuidadosamente usando uma agulha estéril de 25 G. - Quando seco, usando uma balança de laboratório, pesar um dos seguintes: 1) 25 mg do holobionte; 2) 15 mg da fração simbionte; ou 3) 30 mg do tecido hospedeiro de cada amostra em tubos de microcentrífuga separados e livres de plastificante de 2 mL.
NOTA: Etapa crítica: A otimização da biomassa para extração é essencial para garantir que o GC-MS não seja sobrecarregado, garantindo sinal suficiente. O material de coral seco é muito estático. Para evitar a perda de amostras, use dispositivos antiestáticos para eliminar cargas eletrostáticas das amostras e dos recipientes de pesagem. Uma alternativa simples e econômica é colocar uma folha de secadora de roupa sob o tubo de amostra. O pellet simbionte seco pode ser cortado até o peso desejado usando uma lâmina estéril.
8. Extrações de metabólitos intracelulares
- Extração de metabólito intracelular de holobionte liofilizado:
- Adicionar 400 μL de metanol 100% frio (-20 °C) com padrão(s) interno(s) (IS; 13ºC6 sorbitol e/ou 13C5-15 N valina, a 10μM) para cada tubo.
- Adicione um pequeno número de 710-1.180 μm de contas de vidro lavadas com ácido (~10 mg) a cada amostra. Colocar num moinho de contas a 50 Hz durante 3 minutos num moinho de contas pré-refrigerado (-20 °C).
- Adicionar mais 600 μL de metanol 100% frio (-20 °C) com ISs (13 C6 sorbitol e/ou 13C 5-15 N valina, a 10μM) a cada tubo.
- Vórtice para misturar por 1 min. Colocar numa coqueteleira rotisserie a 4 °C durante 30 min.
- Extração de metabólitos intracelulares de células de Symbiodiniaceae liofilizadas separadas:
- Adicionar 200 μL de metanol 100% frio (−20 °C) com ISs (13 C6 sorbitol e/ou 13C5-15 N valina, a 10μM) ao material seco de Symbiodiniaceae.
- Adicione um pequeno número de 710-1.180 μm de contas de vidro lavadas com ácido (~10 mg). Colocar num moinho de contas a 50 Hz durante 3 minutos num moinho de contas pré-refrigerado (-20 °C).
- Adicione mais 800 μL de metanol 100% frio (-20 °C) com ISs e vórtice por 30 s.
- Extração de metabólito intracelular do tecido do hospedeiro liofilizado separado:
- Adicionar 1 ml de metanol 100% frio (−20 °C) LC contendo ISs (13 C6 sorbitol e/ou 13C 5-15 N valina, a 10μM) ao material hospedeiro seco.
- Vórtice para misturar por 20 s. Colocar num suporte de tubo flutuante num banho de sonicação regulado a 4 °C durante 30 minutos.
9. Purificação do extrato de metabolito
- Centrifugar as amostras (holobionte/hospedeiro/simbionte) a 3.000 × g por 30 min a 4 °C.
- Transfira todo o sobrenadante para um novo tubo de microcentrífuga de 2 mL, tomando cuidado para não perturbar a pastilha de detritos celulares.
NOTA: Estes são os extratos semipolares. Estes podem ser mantidos no gelo temporariamente, mas armazenados a longo prazo a -80 °C no escuro. - Aos restos celulares restantes, adicionar 1.000 μL de metanol 50% frio (-20 °C). Vórtice vigorosamente por 1 min para ressuspender.
- Centrifugar as amostras a 3.000 × g durante 30 min a 4 °C.
- Coletar e agrupar o sobrenadante (extratos polares) com os extratos semipolares da mesma amostra.
NOTA: Os restos celulares podem ser armazenados a -80 °C e utilizados para a normalização do teor de proteínas (secção 11). - Centrifugar os extratos agrupados a 16.100 g por 15 min para remover todos os precipitados e mover o sobrenadante para um novo tubo de microcentrífuga livre de plastificante (2 mL).
NOTA: Os extractos de amostra podem ser armazenados a -80 °C no escuro. - Quando estiver pronto para analisar, alíquota 50 μL de cada extrato em uma pastilha de vidro. Concentrar durante 30 min a 30 °C utilizando um concentrador a vácuo. Repetir mais quatro vezes (para 250 μL de extracto seco total).
NOTA: As amostras secas podem ser armazenadas à temperatura ambiente em condições de dessecante até análise.
10. Derivatização de metabólitos
NOTA : Um processo de derivatização on-line de duas etapas é usado para a metoximação e trimetilsililação dos metabólitos polares.
- Adicionar 25 μL de cloridrato de metoxiamina (30 mg/ml em piridina) a cada amostra.
- Agitar a 37 °C num agitador orbital regulado a 750 rpm durante 2 horas.
- Adicionar 25 μL de N,O-bis (trimetilsilil)trifluoroacetamida + trimetilclorossilano a cada amostra.
- Agitar novamente a 37 °C e 750 rpm por 1 h.
- Deixar as amostras equilibrarem-se à temperatura ambiente durante 1 h antes de injetar 1 μL numa proporção de divisão de 1:10 no CG.
11. Análise por cromatografia gasosa acoplada à espectrometria de massas
NOTA: O espectrômetro de massa deve ser ajustado de acordo com as recomendações do fabricante usando tris-(perfluorobutil)-amina (CF43).
- Use hélio de pureza ultra-alta como gás transportador a uma taxa de fluxo constante da coluna de 1 mL/min.
- Use uma coluna DB-5 de 30 m com espessura de filme de 1 μm e diâmetro interno de 0,25 mm.
- Programa de forno GC
- Ajuste a temperatura de entrada para 280 °C.
- Comece com a injeção com uma temperatura de forno de 100 °C e segure por 4 min.
- Aumente a temperatura em 10 °C/min para 320 °C e, em seguida, segure por 11 min.
- Parâmetros do espectrômetro de massa
- Ajuste a linha de transferência MS para 280 °C e ajuste a fonte de íons para 200 °C.
- Use argônio como gás da célula de colisão para gerar o íon do produto MRM (monitoramento de reação múltipla).
- Alcance a detecção de metabólitos em relação a uma biblioteca MRM segmentada no tempo contendo alvos MRM.
12. Contagem de células de Symbiodiniaceae, análise do conteúdo de proteínas do tecido do hospedeiro coral e estimativa da clorofila a
- Contagem de células de Symbiodiniaceae:
- Pegue uma alíquota do homogeneizado de tecido coral.
- Centrifugar as amostras a 2.000 × g para pellet das algas.
- Retire o sobrenadante de ~200 μL do pellet de algas e coloque em um novo tubo de microcentrífuga.
NOTA: Esta será a amostra de proteína que será usada para normalizar os dados; armazená-lo a -20 °C antes de analisar, se necessário. - Ressuspenda o pellet de algas em 1 mL de água do mar filtrada pipetando suavemente para cima e para baixo. Se necessário, diluir a suspensão de algas para facilitar a contagem celular.
- Realizar uma contagem de células usando um hemocitômetro sob um microscópio de luz, adicionando 10 μL a uma das câmaras. Complete 8-10 contagens por amostra.
NOTA: Métodos alternativos para contar as células algais também podem ser aplicados quando disponíveis (por exemplo, citometria de fluxo, microscopia confocal de alto rendimento). - Calcular a concentração das células simbiontes (mL−1), tendo em conta quaisquer factores de diluição utilizados.
- Ensaio para o teor de proteína
- Quantificar o conteúdo proteico da amostra (por exemplo, através do ensaio colorimétrico de Bradford, como descrito inicialmente por Bradford et al.33, ou do ensaio de Lowry34,35, cujo protocolo já foi descrito para cnidários em outros estudos 36).
- Extração de clorofila a
- Use um pellet de células de ~200, 000 células, congelado ou fresco.
- Transferir cada pellet de alga para 2 mL de dimetilformamida (DMF) em um frasco para cintilação de vidro e incubar no escuro a 4 °C por 48 h.
NOTA: O DMF é tóxico e cancerígeno, pelo que a preparação da amostra deve ser concluída sob um exaustor de fumos o mais escuro possível e sobre gelo. Se houver < 200.000 células, use menos DMF. - Centrifugar por 3 min a 16.000 × g.
- Transfira 200 mL para uma placa de poço UV-96 para medições fotométricas. Execute cada amostra em triplicado com DMF como o branco.
- Medir a absorbância nos comprimentos de onda (E) 663,8 nm, 646 nm e 750 nm. Subtrair a absorbância a 750 nm da absorbância em ambos os outros comprimentos de onda.
NOTA: A medição a 750 nm corrige qualquer espalhamento ou turbidez na amostra. - Calcular a concentração de clorofila a (μg/ml) utilizando a equação (1):
Concentração de Chl a (μg/mL) = (12,00 × E 663,8) - (3,11 × E646,8) (1)
13. Quantificação da biomassa celular após extrações de metabólitos para normalização
NOTA: Existem duas opções para a quantificação da biomassa celular descritas a seguir: a quantificação da proteína relacionada à biomassa utilizando um método colorimétrico de Bradford modificado e a medição do peso seco dos detritos celulares. Qualquer um dos métodos é apropriado para uso, pois ambos oferecem quantificação precisa da biomassa celular.
- Conteúdo proteico dos restos celulares
- Ressuspender os restos celulares congelados com 1 mL de NaOH 0,2 M e incubar as amostras a 98 °C por 20 min.
- Resfriar as amostras no gelo por ~10 min e centrifugar a 3.000 × g por 5 min à temperatura ambiente.
- Quantificar o conteúdo proteico da amostra (por exemplo, através do ensaio colorimétrico de Bradford, conforme descrito inicialmente por Bradford et al.33 e modificado por Smart et al.37).
- Medição do peso seco dos detritos celulares
- Ressuspender os restos celulares da extração do metabólito intracelular em água bidestilada (~10 mL).
- Filtrar a solução a vácuo utilizando um filtro de membrana pré-pesado (poro de 0,22 μm, 47 mm).
- Lave os tubos contendo biomassa duas vezes com água ultrapura para garantir a transferência completa da biomassa para o filtro de membrana.
- Retire o filtro de membrana que contém a biomassa e seque-o usando um forno de micro-ondas (baixa potência; ~250 W por 20 min).
- Guarde o papel de filtro em um exsicador durante a noite. Registar o peso seco do papel de filtro e calcular o peso seco da biomassa subtraindo o peso do filtro de membrana seca (utilizando um filtro de membrana seco limpo seco juntamente com o filtro de amostra) do peso total.
14. Análise dos dados
- Analise alvos de metabólitos usando bancos de dados de metabólitos onde cada destino é composto por um quantificador e qualificador MRM.
- Inspecione visualmente os alvos de metabólitos detectados e integre-os manualmente, conforme necessário.
- Use uma área de pico do metabólito para calcular a abundância relativa de cada amostra para cada grupo. Os valores são corrigidos em branco e normalizados para a área de pico padrão interno da amostra e, em seguida, para a amostra do conteúdo de proteína de restos celulares de acordo com Smart et al.37.
- Eliminar metabolitos com um desvio padrão relativo superior a 35% em todos os grupos de tratamento (N = 23 metabolitos).
- Transforme os dados (por exemplo, raiz cúbica) e centralize-os na média; confirmam uma distribuição normal e homogeneidade da variância.
- Realizar a análise dos dados (ANOVA e construção do mapa de calor; por exemplo, utilizando https://www.metaboanalyst.ca)38. Agrupar as amostras para examinar a variabilidade dentro do tratamento usando os pacotes "cluster", "factoextra" e "klustR". Calcule a estatística gap (um método para determinar o número ótimo de clusters39) usando a função "clusGap" em R e gráficos usando o pacote R "tidyverse". Realizar PERMANOVAs para examinar a significância na separação entre os perfis de metabólitos de tratamento (por exemplo, em Primer).
Resultados
Todos os dados produzidos durante este trabalho estão disponíveis nas informações complementares.
Separação simbionte hospedeiro
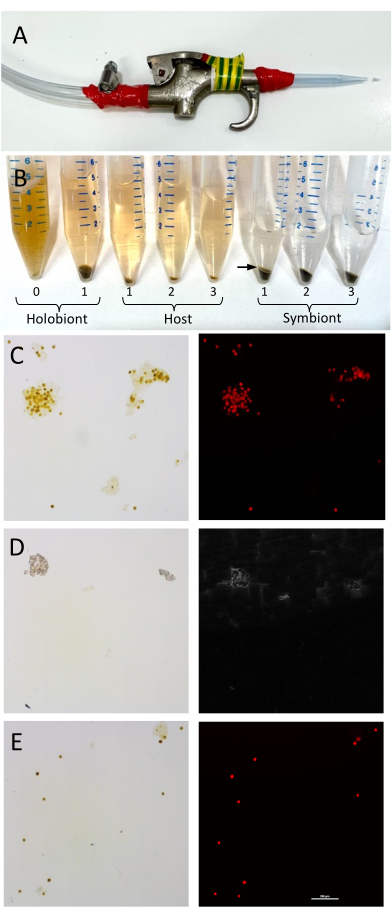
Figura 1: Configuração e validação da separação dos tecidos hospedeiros dos corais e das células de Symbiodiniaceae. (A) A
Discussão
A separação do hospedeiro e do simbionte é fácil e rapidamente alcançável através de centrifugação simples, e os resultados aqui mostram que a separação das frações pode fornecer informações valiosas indicativas de contribuições específicas dos membros do holobionte, o que pode contribuir para a análise funcional da saúde dos corais. Em corais adultos, a síntese lipídica é realizada primariamente pelo simbionte algal residente 40, que fornece lipídios (por exemplo, triacilgliceróis e fos...
Divulgações
Os autores não têm conflitos de interesse a declarar.
Agradecimentos
J.L.M. foi apoiado por uma bolsa de pesquisa do Chanceler da UTS.
Materiais
| Name | Company | Catalog Number | Comments |
| 100% LC-grade methanol | Merck | 439193 | LC grade essential |
| 2 mL microcentrifuge tubes, PP | Eppendorf | 30121880 | Polypropylene provides high resistance to chemicals, mechanical stress and temperature extremes |
| 2030 Shimadzu gas chromatograph | Shimadzu | GC-2030 | |
| 710-1180 µm acid-washed glass beads | Merck | G1152 | This size is optimal for breaking the Symbiodiniaceae cells |
| AOC-6000 Plus Multifunctional autosampler | Shimadzu | AOC6000 | |
| Bradford reagent | Merck | B6916 | Any protein colourimetric reagent is acceptable |
| Compressed air gun | Ozito | 6270636 | Similar design acceptable. Having a fitting to fit a 1 mL tip over is critical. |
| DB-5 column with 0.25 mm internal diameter column and 1 µm film thickness | Agilent | 122-5013 | |
| DMF | Merck | RTC000098 | |
| D-Sorbitol-6-13C and/or 13C5–15N Valine | Merck | 605514/ 600148 | Either or both internal standards can be added to the methanol. |
| Flat bottom 96-well plate | Merck | CLS3614 | |
| Glass scintillation vials | Merck | V7130 | 20 mL, with non-plastic seal |
| Immunoglogin G | Merck | 56834 | if not availbe, Bovine Serum Albumin is acceptable |
| Primer | v4 | ||
| R | v4.1.2 | ||
| Shimadzu LabSolutions Insight software | v3.6 | ||
| Sodium Hydroxide | Merck | S5881 | Pellets to make 1 M solution |
| tidyverse | v1.3.1 | R package | |
| TissueLyser LT | Qiagen | 85600 | Or similar |
| TQ8050NX triple quadrupole mass spectrometer | Shimadzu | GCMS-TQ8050 NX | |
| UV-96 well plate | Greiner | M3812 | |
| Whirl-Pak sample bag | Merck | WPB01018WA | Sample collection bag; Size: big enough to house a ~5 cm coral fragment, but not too big that the water is too spread |
Referências
- Bundy, J. G., Davey, M. P., Viant, M. R. Environmental metabolomics: A critical review and future perspectives. Metabolomics. 5 (1), 3-21 (2008).
- Matthews, J. L., Beale, D. J., Hillyer, K. E., Warden, A. C., Jones, O. A. H., et al. The metabolic significance of symbiont community composition in the coral-algal symbiosis. Applied Environmental Metabolomics. , 211-229 (2022).
- Lawson, C. A., van Oppen, M. J. H., Aranda Lastra, M., et al. Informing coral reef conservation through metabolomic approaches. Coral Reef Conservation and Restoration in the Omics Age. Coral Reefs of the World. , 179-202 (2022).
- LaJeunesse, T. C., et al. Systematic revision of Symbiodiniaceae highlights the antiquity and diversity of coral endosymbionts. Current Biology. 28 (16), 2570-2580 (2018).
- Rohwer, F., Seguritan, V., Azam, F., Knowlton, N. Diversity and distribution of coral-associated bacteria. Marine Ecology Progress Series. 243, 1-10 (2002).
- Maire, J., et al. Intracellular bacteria are common and taxonomically diverse in cultured and in hospite algal endosymbionts of coral reefs. The ISME Journal. 15 (7), 2028-2042 (2021).
- Hillyer, K. E., et al. Metabolite profiling of symbiont and host during thermal stress and bleaching in the coral Acropora aspera. Coral Reefs. 36, 105-118 (2016).
- Hillyer, K. E., Tumanov, S., Villas-Bôas, S., Davy, S. K. Metabolite profiling of symbiont and host during thermal stress and bleaching in a model cnidarian-dinoflagellate symbiosis. Journal of Experimental Biology. 219 (4), 516-527 (2016).
- Matthews, J. L., et al. Optimal nutrient exchange and immune responses operate in partner specificity in the cnidarian-dinoflagellate symbiosis. Proceedings of the National Academy of Sciences of the United States of America. 114 (50), 13194-13199 (2017).
- Rosset, S. L., et al. The molecular language of the cnidarian-dinoflagellate symbiosis. Trends in Microbiology. 29 (4), 320-333 (2020).
- Matthews, J. L., et al. Partner switching and metabolic flux in a model cnidarian-dinoflagellate symbiosis. Royal Society. 285 (1892), 20182336 (2018).
- González-Pech, R. A., et al. Physiological factors facilitating the persistence of Pocillopora aliciae and Plesiastrea versipora in temperate reefs of south-eastern Australia under ocean warming. Coral Reefs. 41, 1239-1253 (2022).
- Williams, A., et al. Metabolomic shifts associated with heat stress in coral holobionts. Science Advances. 7 (1), (2021).
- Deutsch, J. M., et al. Metabolomics of healthy and stony coral tissue loss disease affected Montastraea cavernosa corals. Frontiers in Marine Science. 8, 1421 (2021).
- Stien, D., et al. A unique approach to monitor stress in coral exposed to emerging pollutants. Scientific Reports. 10 (1), 9601 (2020).
- Lohr, K. E., et al. Resolving coral photoacclimation dynamics through coupled photophysiological and metabolomic profiling. Journal of Experimental Biology. 222 (8), (2019).
- Jorissen, H., et al. Coral larval settlement preferences linked to crustose coralline algae with distinct chemical and microbial signatures. Scientific Reports. 11 (1), 14610 (2021).
- Roach, T. N., Dilworth, J., Jones, A. D., Quinn, R. A., Drury, C. Metabolomic signatures of coral bleaching history. Nature Ecology & Evolution. 5 (4), 495-503 (2021).
- Parkinson, J. E., et al. Molecular tools for coral reef restoration: Beyond biomarker discovery. Conservation Letters. 13 (1), 12687 (2020).
- Jiang, J., et al. How Symbiodiniaceae meets the challenges of life during coral bleaching. Coral Reefs. 40, 1339-1353 (2021).
- Guerra, F. D., Attia, M. F., Whitehead, D. C., Alexis, F. Nanotechnology for environmental remediation: materials and applications. Molecules. 23 (7), 1760 (2018).
- Matthews, J. L., et al. Metabolite pools of the reef building coral Montipora capitata are unaffected by Symbiodiniaceae community composition. Coral Reefs. 39, 1727-1737 (2020).
- Papina, M., Meziane, T., van Woesik, R. Symbiotic zooxanthellae provide the host-coral Montipora digitata with polyunsaturated fatty acids. Comparative Biochemistry and Physiology Part B: Biochemistry and Molecular Biology. 135 (3), 533-537 (2003).
- Kellogg, R., Patton, J. Lipid droplets, medium of energy exchange in the symbiotic anemone Condylactis gigantea: A model coral polyp. Marine Biology. 75, 137-149 (1983).
- Ankrah, N. Y., Chouaia, B., Douglas, A. E. The cost of metabolic interactions in symbioses between insects and bacteria with reduced genomes. mBio. 9 (5), e01433 (2018).
- Kabeya, N., et al. Genes for de novo biosynthesis of omega-3 polyunsaturated fatty acids are widespread in animals. Science Advances. 4 (5), (2018).
- Hillyer, K. E., Dias, D., Lutz, A., Roessner, U., Davy, S. K. 13C metabolomics reveals widespread change in carbon fate during coral bleaching. Metabolomics. 14 (1), 12 (2018).
- Hillyer, K. E., Dias, D. A., Lutz, A., Roessner, U., Davy, S. K. Mapping carbon fate during bleaching in a model cnidarian symbiosis: the application of 13C metabolomics. New Phytologist. 214 (4), 1551-1562 (2017).
- Burriesci, M. S., Raab, T. K., Pringle, J. R. Evidence that glucose is the major transferred metabolite in dinoflagellate-cnidarian symbiosis. Journal of Experimental Biology. 215 (19), 3467-3477 (2012).
- Thurber, R. V., et al. Unified methods in collecting, preserving, and archiving coral bleaching and restoration specimens to increase sample utility and interdisciplinary collaboration. PeerJ. 10, 14176 (2022).
- Grottoli, A. G., et al. Increasing comparability among coral bleaching experiments. Ecological Applications. 31 (4), 02262 (2020).
- Mushtaq, M. Y., Choi, Y. H., Verpoorte, R., Wilson, E. G. Extraction for metabolomics: access to the metabolome. Phytochemical Analysis. 25 (4), 291-306 (2014).
- Bradford, M. M. A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding. Analytical Biochemistry. 72 (1), 248-254 (1976).
- Peterson, G. L., et al. A simplification of the protein assay method of Lowry et al. which is more generally applicable. Analytical Biochemistry. 83 (2), 346-356 (1977).
- Lowry, O. H., Rosebrough, N. J., Farr, A. L., Randall, R. J. Protein measurement with the Folin phenol reagent. Journal of Biological Chemistry. 193 (1), 265-275 (1951).
- Zamer, W. E., Shick, J. M., Tapley, D. W. Protein measurement and energetic considerations: Comparisons of biochemical and stoichiometric methods using bovine serum albumin and protein isolated from sea anemones. Limnology and Oceanography. 34 (1), 256-263 (1989).
- Smart, K. F., Aggio, R. B., Van Houtte, J. R., Villas-Boas, S. G. Analytical platform for metabolome analysis of microbial cells using methyl chloroformate derivatization followed by gas chromatography-mass spectrometry. Nature Protocols. 5 (10), 1709-1729 (2010).
- Pang, Z., et al. Using MetaboAnalyst 5.0 for LC-HRMS spectra processing, multi-omics integration and covariate adjustment of global metabolomics data. Nature Protocols. 17 (8), 1735-1761 (2022).
- Tibshirani, R., Walther, G., Hastie, T. Estimating the number of clusters in a data set via the gap statistic. Journal of the Royal Statistical Society: Series B (Statistical Methodology). 63 (2), 411-423 (2001).
- Chen, W. -. N., et al. Diel rhythmicity of lipid-body formation in a coral-Symbiodinium endosymbiosis). Coral Reefs. 31 (2), 521-534 (2012).
- Imbs, A. Fatty acids and other lipids of corals: composition, distribution, and biosynthesis. Russian Journal of Marine Biology. 39 (3), 153-168 (2013).
- Rosset, S., et al. Lipidome analysis of Symbiodiniaceae reveals possible mechanisms of heat stress tolerance in reef coral symbionts. Coral Reefs. 38 (6), 1241-1253 (2019).
- Carreón-Palau, L., Parrish, C. C., Del Angel-Rodriguez, J. A., Perez-Espana, H. Seasonal shifts in fatty acids and sterols in sponges, corals, and bivalves, in a southern Gulf of Mexico coral reef under river influence. Coral Reefs. 40 (2), 571-593 (2021).
- Imbs, A. B., Dang, L. T. Seasonal dynamics of fatty acid biomarkers in the soft coral Sinularia flexibilis, a common species of Indo-Pacific coral reefs. Biochemical Systematics and Ecology. 96, 104246 (2021).
- Oku, H., Yamashiro, H., Onaga, K., Sakai, K., Iwasaki, H. Seasonal changes in the content and composition of lipids in the coral Goniastrea aspera. Coral Reefs. 22 (1), 83-85 (2003).
- Weis, V. M. Cell biology of coral symbiosis: foundational study can inform solutions to the coral reef crisis. Integrative and Comparative Biology. 59 (4), 845-855 (2019).
- Oakley, C., Davy, S., van Oppen, M., Lough, J. Cell biology of coral bleaching. Coral Bleaching. , 189-211 (2018).
- Lu, W., et al. Metabolite measurement: Pitfalls to avoid and practices to follow. Annual Review of Biochemistry. 86, 277-304 (2017).
- Lawson, C. A., et al. Heat stress decreases the diversity, abundance and functional potential of coral gas emissions. Global Change Biology. 27 (4), 879-891 (2021).
- Olander, A., et al. Comparative volatilomics of coral endosymbionts from one-and comprehensive two-dimensional gas chromatography approaches. Marine Biology. 168 (5), 76 (2021).
- Wuerz, M., et al. Symbiosis induces unique volatile profiles in the model cnidarian Aiptasia. Journal of Experimental Biology. 225 (19), (2022).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados