Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Зонды натяжения ДНК для картирования переходных сил рецептора пиконьютона иммунными клетками
В этой статье
Резюме
В этой статье описывается подробный протокол использования датчиков натяжения на основе ДНК для визуализации рецепторных сил, приложенных иммунными клетками. Этот подход может отображать рецепторные силы >4,7 пН в режиме реального времени и может интегрировать силы с течением времени.
Аннотация
Механические силы, передаваемые на стыке между двумя соседними клетками и на стыке между клетками и внеклеточным матриксом, имеют решающее значение для регуляции многих процессов, начиная от развития и заканчивая иммунологией. Поэтому разработка инструментов для изучения этих сил на молекулярном уровне имеет решающее значение. Наша группа разработала набор датчиков молекулярного натяжения для количественной оценки и визуализации сил, генерируемых клетками и передаваемых конкретным лигандам. Наиболее чувствительный класс датчиков молекулярного натяжения состоит из шпилек со стволовыми петлями нуклеиновых кислот. Эти датчики используют пары флуорофор-гаситель, чтобы сообщать о механическом расширении и разворачивании шпилек ДНК под действием силы. Одна из проблем, связанных с датчиками натяжения шпилек ДНК, заключается в том, что они обратимы с быстрым складыванием шпильки после прекращения натяжения, и, таким образом, переходные силы трудно зарегистрировать. В этой статье мы описываем протоколы подготовки датчиков натяжения ДНК, которые можно «заблокировать» и предотвратить повторное складывание, чтобы обеспечить «хранение» механической информации. Это позволяет регистрировать высокопереходные пиконетонные силы, которые впоследствии могут быть «стерты» добавлением комплементарных нуклеиновых кислот, снимающих блокировку. Эта способность переключаться между картированием напряжения в реальном времени и механическим хранением информации выявляет слабые, кратковременные и менее обильные силы, которые обычно используются Т-клетками как часть их иммунных функций.
Введение
Иммунные клетки защищаются от патогенов и раковых клеток, непрерывно ползая и сканируя поверхности клеток-мишеней на наличие антигенов, усеивая их поверхность 1,2. Распознавание антигена инициируется при связывании между Т-клеточным рецептором (TCR) и комплексом пептидно-главной гистосовместимости MHC (pMHC), экспрессируемым на поверхности клеток-мишеней. Поскольку распознавание TCR-pMHC происходит на стыке между двумя подвижными клетками, долгое время подозревалось, что он испытывает механические силы. Более того, это привело к механосенсорной модели активации TCR, которая предполагает, что силы TCR способствуют его функции 3,4. Чтобы понять, когда, где и как механические силы способствуют функционированию Т-клеток, необходимо разработать инструменты для визуализации молекулярных сил, передаваемых Т-клетками. Традиционно для исследования клеточных сил5,6 используются такие методы, как тракционно-силовая микроскопия (TFM) и микропиллярные матрицы. Однако силовая чувствительность TFM и микростолбовых матриц находится в масштабе наноньютона (nN) и, таким образом, часто недостаточна для изучения молекулярных пиконьютонных (pN) сил, передаваемых клеточными рецепторами7. Чтобы улучшить силу и пространственное разрешение для обнаружения, наша лаборатория впервые разработала зонды молекулярного натяжения, которые первоначально были синтезированы с использованием полимеров полиэтиленгликоля (ПЭГ)7. Зонды молекулярного натяжения состоят из выдвижной молекулярной «пружины» (ПЭГ, белок, ДНК), окруженной флуорофором и гасителем, и закреплены на поверхности. Силы, приложенные к концу зонда, приводят к его расширению, разделяя флуорофор и гаситель, и, таким образом, генерируя сильный сигнал флуоресценции (рис. 1А)8,9,10.
За последнее десятилетие мы разработали библиотеку различных классов зондов молекулярного натяжения с пружинными элементами, изготовленными из нуклеиновых кислот11, белков10 и полимеров8. Среди них датчики натяжения на основе ДНК обеспечивают самое высокое соотношение сигнал/шум и наибольшую чувствительность к силе, которая легко настраивается от нескольких пН до ~20 пН11. Мы использовали эти зонды натяжения ДНК в реальном времени для изучения молекулярных сил, генерируемых многими различными типами клеток, включая фибробласты, раковые клетки, тромбоциты и иммунные клетки11,12,13. В этой рукописи будут описаны протоколы синтеза и сборки зондов натяжения ДНК на поверхности для картирования молекулярных рецепторных сил с разрешением силы pN с использованием обычного флуоресцентного микроскопа. В то время как текущая процедура включает химические модификации нуклеиновой кислоты для введения флуоресцентного репортера (рис. 1B), важно отметить, что многие этапы модификации и очистки могут быть переданы на аутсорсинг компаниям, занимающимся синтезом ДНК. Таким образом, технология зондов натяжения ДНК проста и доступна для более широких сообществ клеточной биологии и механобиологии.
Вкратце, для сборки датчиков натяжения ДНК шпилька ДНК гибридизуется с флуоресцентной цепью лиганда на одном плече и якорной нитью гасителя на другом плече, а затем иммобилизуется на стеклянной подложке (рис. 1C, натяжение в реальном времени). При отсутствии механической силы шпилька закрывается, и таким образом флуоресценция гасится. Однако, когда приложенная механическая сила больше, чем F1/2 (сила в равновесии, которая приводит к 50% вероятности разворачивания), шпилька механически плавится, и генерируется флуоресцентный сигнал.
Основываясь на датчике натяжения ДНК в реальном времени, мы также описываем протоколы для отображения накопленных сил, что особенно полезно для изучения взаимодействия между рецепторами на иммунных клетках и их естественным лигандом. Это связано с тем, что иммунные рецепторы часто демонстрируют короткоживущие связи 3,14. Накопленные силы визуализируются с помощью «запирающей» цепи, которая преимущественно связывается с открытыми шпильками ДНК и позволяет хранить флуоресцентные сигналы, связанные с механическими событиями вытягивания (рис. 1C, заблокированное натяжение). Фиксирующая прядь предназначена для связывания загадочного места связывания, который подвергается воздействию при механически индуцированном плавлении шпильки, и блокировки шпильки в открытом состоянии путем блокировки повторного складывания шпильки, тем самым сохраняя сигнал натяжения и генерируя накопленную карту натяжения. Кроме того, блокирующая цепь имеет восьминуклеотидную опору, которая обеспечивает реакцию смещения цепи, опосредованную опорой на носок, с ее полным дополнением, «разблокирующей» нитью. С добавлением отпирающей пряди связанная фиксирующая прядь снимается с конструкции шпильки, стирая сохраненный сигнал натяжения и сбрасывая шпильку обратно в состояние реального времени.
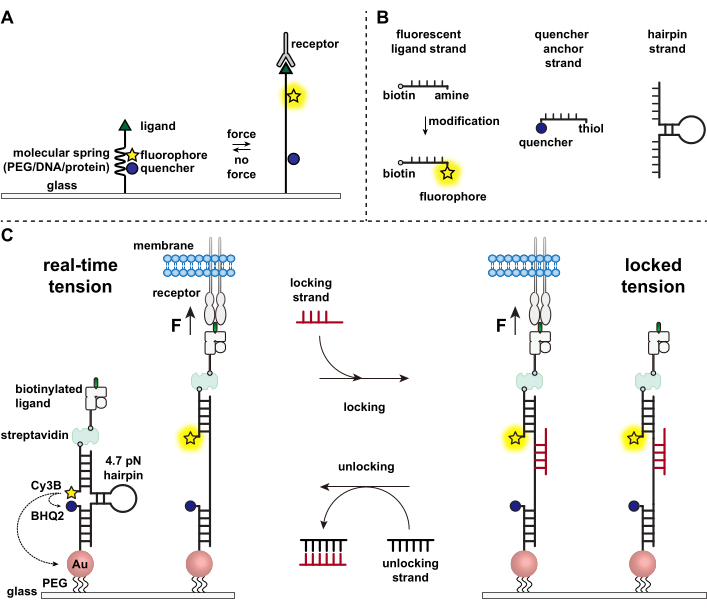
Рисунок 1: Схема современных датчиков молекулярного натяжения. (A) Общая конструкция датчика молекулярного натяжения в реальном времени, (B) Нити для конструкции датчика натяжения на основе ДНК и (C) спроектированные датчики натяжения на основе ДНК и их переключение между состоянием реального времени и заблокированным состоянием. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
Основной протокол состоит из четырех основных разделов - подготовка олигонуклеотидов, подготовка поверхности, визуализация и анализ данных. Этот протокол был успешно продемонстрирован нашей лабораторией и другими в наивных и активированных OT-1 CD8+ Т-клетках, OT-II CD4+ клетках, а также гибридомах и может применяться для опроса различных рецепторов иммунных клеток, включая рецептор Т-клеток, рецептор запрограммированной клеточной смерти (PD1) и силы антигена 1, связанного с функцией лимфоцитов (LFA-1). OT-1 CD8+ наивные Т-клетки используются в качестве примера клеточной линии в этой статье.
протокол
Трансгенные мыши OT-1 размещены в Отделе ресурсов животных в Университете Эмори. Все эксперименты были одобрены и выполнены в соответствии с протоколом Институционального комитета по уходу за животными и их использованию (IACUC).
1. Олигонуклеотидный препарат
- Растворите ДНК цепи лиганда в воде (удельное сопротивление 18,2 МОм, используемое во всем протоколе). Закрутите и раскрутите раствор с помощью настольной центрифуги. Отрегулируйте объем воды таким образом, чтобы конечная концентрация составляла 1 мМ. Проверьте концентрацию с помощью нанокапельного спектрофотометра для измерения поглощения на длине волны 260 нм и определения конечной концентрации на основе коэффициента экстинкции олигонуклеотида.
ПРИМЕЧАНИЕ: Цепь лиганда имеет модификацию на каждом конце, 5'-амин и 3'-биотин, для конъюгирования с флуорофором и представления биотинилированного лиганда. Аминогруппа в цепи лиганда должна быть конъюгирована с флуорофором. Краситель Cy3B используется для этого сопряжения из-за его высокой яркости и фотостабильности, но он обычно не предлагается в продаже и требует собственного сопряжения. Соответственно, в следующем разделе описывается конъюгация между аминами и эфирными красителями NHS. Для конечных пользователей, которые не имеют доступа к средствам или ресурсам для модификации нуклеиновых кислот, модифицированные нуклеиновые кислоты можно приобрести у поставщиков услуг по синтезу ДНК, которые предлагают яркие и фотостабильные красители, такие как семейство красителей Alexa и Atto. - Приготовьте 10 растворов PBS и 1 M NaHCO3 . Смешайте 10 мкл 1 мМ раствора цепи аминового лиганда (10 нмоль) с 10 мкл 10x PBS, 10 мкл 1 М NaHCO3 и 60 мкл H2O. Растворите 50 мкг сложного эфира Cy3B NHS в 10 мкл ДМСО непосредственно перед использованием и добавьте в смесь до общего объема реакции 100 мкл. Добавьте сложный эфир Cy3B NHS в последнюю очередь. Дайте вступать в реакцию при комнатной температуре в течение 1 часа или 4 °C в течение ночи.
- Подготовьте фиксирующую цепь Atto647N, конъюгируя цепь блокировки амина с эфиром Atto647N NHS. Приготовьте 10 растворов PBS и 1 M NaHCO3 . Смешайте 10 мкл раствора нити блокировки аминов 1 мМ (10 нмоль) с 10 мкл 10x PBS, 10 мкл 1 М NaHCO3 и 60 мкл H2O. Растворите 50 мкг сложного эфира Atto647N NHS в 10 мкл ДМСО непосредственно перед использованием и добавьте в смесь до общего объема реакции 100 мкл. Добавьте сложный эфир Atto647N NHS в последнюю очередь. Дайте вступать в реакцию при комнатной температуре в течение 1 часа или 4 °C в течение ночи.
- После реакций удаляют побочные продукты, излишки красителя и соли с помощью обессоливающей гель-фильтрации P2. Разбавьте реакционную смесь H2O до общего объема 300 мкл, который подходит для последующей стадии очистки ВЭЖХ. Добавьте 650 мкл гидратированного геля P2 в центробежное устройство и вращайте при 18 000 x g в течение 1 мин. Удалите жидкость в нижней части устройства, добавьте реакционную смесь в колонку, содержащую гель P2, открутите вниз при 18 000 x g в течение 1 мин и соберите реакционную смесь на дне устройства.
ПРИМЕЧАНИЕ: Гель P2 следует увлажнить не менее чем за 4 ч до использования с H2O. - Очистите обессоленную реакционную смесь с помощью ВЭЖХ с помощью колонки C18, предназначенной для очистки олигонуклеотидов, с растворителем A: 0,1 М TEAA в H2O и B: ACN в качестве подвижной фазы для линейного градиентного элюирования 10-100% B в течение 50 мин при скорости потока 0,5 мл / мин. Впрыскивают обессоленную реакционную смесь в обратную фазу ВЭЖХ с контуром впрыска 500 мкл для очистки. Соберите продукт, который имеет пик поглощения для ДНК (260 нм) и пик поглощения для флуорофора (560 нм для Cy3B и 647 нм для Atto647N), и высушите их в вакуумном центробежном концентраторе в течение ночи (см. рис. 2A).
- Восстановите высушенный олигокраситель в 100 мкл воды. Определите концентрацию цепи лиганда Cy3B и цепи блокировки Atto647N с помощью спектрофотометра nanodrop. Убедитесь, что соотношение маркировки красителя близко к 1:1. При необходимости при определении концентрации олигонуклеотидов скорректируйте поглощение красителя 260 нм.
- Валидируйте очищенный продукт с помощью MALDI-TOF-MS, используя 3-HPA в качестве субстрата в 50% ACN/H2O с 0,1% TFA и 5 мг/мл цитрата аммония, используя 0,5 мкл продукта в концентрации 1-5 мкМ для подготовки образцов MALDI-TOF-MS. Пример масс-спектра можно найти на рисунке 2B.
- Растворите прядь шпильки и анкерную прядь гасителя в воде и убедитесь, что концентрация исходных растворов составляет от 50 до 100 мкМ.
ПРИМЕЧАНИЕ: Прядь шпильки не модифицирована и может быть синтезирована непосредственно у поставщика. Анкерная прядь имеет тиоловую анкерную группу и гаситель BHQ2 и может быть синтезирована непосредственно по индивидуальному заказу у поставщика. - Аликвотируйте все олигонуклеотиды. Для краткосрочного использования и хранения храните эти олигонуклеотиды при температуре 4 °C. Для длительного хранения заморозьте и храните при температуре -20 °C. К этому моменту все олигонуклеотиды готовы к сборке зонда натяжения ДНК.
ПРИМЕЧАНИЕ: Повторяющиеся циклы замораживания-оттаивания не являются проблемой для олигонуклеотидов.
2. Подготовка поверхности
ПРИМЕЧАНИЕ: Подготовка подложек для натяжения шпилек ДНК занимает два дня. Датчик натяжения шпильки ДНК будет функционализирован на стеклянных покровных стеклах.
- День 1
- Поместите 25-миллиметровые покровные стекла на стойку из политетрафторэтилена в стакане объемом 50 мл. Каждая стойка вмещает до 8 покровных листов. Промойте покровные стекла, погрузив их в воду три раза.
- Добавьте 40 мл раствора этанола в соотношении 1:1 (v:v), смешанного с водой, в стакан, содержащий стойку и покровные стекла, и запечатайте стакан парафиновой пленкой.
- Обработайте стакан ультразвуком в течение 15 минут в ультразвуковом очистителе (рабочая частота 35 кГц), чтобы очистить покровные стекла. После обработки ультразвуком слейте жидкость и промойте стакан со стойкой и покровными стеклами в нем водой не менее 6 раз, чтобы удалить оставшийся органический растворитель.
- Приготовьте свежий раствор Пираньи, смешав серную кислоту и перекись водорода в соотношении 3:1. Чтобы приготовить 40 мл раствора пираньи, сначала добавьте 30 мл серной кислоты в чистый стакан объемом 50 мл, а затем медленно добавьте 10 мл H 2 O2. Раствор Piranha быстро нагревается и пузырится при добавлении H 2 O2. Аккуратно перемешайте пиранью с помощью конца стеклянной пипетки.
- Затем перенесите стойку, в которой хранятся покровные стекла, в стакан с аккуратно перемешанным раствором пираньи для травления (рис. 3А). Дайте раствору Piranha гидроксилировать и очистите покровные стекла в течение 30 минут при комнатной температуре. После травления «Пираньи» перенесите стойку с помощью стального или политетрафторэтиленового пинцета в чистый стакан объемом 50 мл с водой и снова промойте водой не менее 6 раз.
ВНИМАНИЕ: Большое количество органических веществ может бурно реагировать с раствором пираньи и вызывать взрыв. Будьте осторожны и всегда работайте с раствором Piranha в вытяжном шкафу. Обязательно наденьте лабораторный халат, перчатки и защитные очки. Никогда не храните свежий раствор пираньи в закрытой таре.
ПРИМЕЧАНИЕ: Соотношение перекиси водорода и серной кислоты должно поддерживаться ниже 1:2 (v:v) и никогда не должно превышать 1:1. Погружая стойку с покровными стеклами в раствор Пираньи, медленно и осторожно помещайте их в раствор. Не выбрасывайте раствор сразу после протравливания, так как он еще активен и горячий. Оставьте его в стакане на ночь, прежде чем вылить в контейнер для кислотных отходов. - Погрузите стойку, удерживающую покровные стекла, в стакан объемом 50 мл с 40 мл этанола для удаления воды. Выбросьте этанол и повторите 3 раза, чтобы убедиться, что вода была удалена.
- Затем погрузите стойку в 3% аминопропилтриэтоксисилан (APTES) (v/v) в 40 мл этанола для реакции с -OH на покровных стеклах в течение 1 ч при комнатной температуре (рис. 3B).
ПРИМЕЧАНИЕ: Этанол можно заменить ацетоном. - Промойте поверхности 6 раз, погрузив их в 40 мл этанола, затем высушите в духовке при 80 ° C в течение 20 минут. После охлаждения высушенные аминомодифицированные покровные стекла хранят при температуре -20 °C для будущего использования (до 6 месяцев).
- Нижнюю внутреннюю сторону пластиковых чашек Петри диаметром 10 см накройте парафиновой пленкой. Парафиновая пленка предотвращает скольжение покровных стекол внутри чашки Петри и помогает сохранить раствор для следующих этапов функционализации на покровных стеклах. Поместите охлажденные аминомодифицированные покровные стекла в чашки Петри. Сторона, подлежащая функционализации, должна быть обращена вверх.
- Чтобы модифицировать аминные группы на покровных стеклах, добавьте 300 мкл 0,5% липоевой кислоты PEG NHS (LA-PEG-SC) и 2,5% mPEG NHS (mPEG-SC) в 0,1 М NaHCO3 на каждое покровное стекло и инкубируйте в течение 1 ч при комнатной температуре (рис. 3C). На каждое 25-миллиметровое покровное стекло следует взвесить 1,5 мг LA-PEG-SC и 7,5 мг mPEG-SC. Растворяйте реагенты NHS непосредственно перед добавлением на поверхности, так как они имеют короткий период полураспада (~ 10 мин) в водном растворе при комнатной температуре. После реакции промойте поверхности 3 раза водой.
ПРИМЕЧАНИЕ: Реакцию NHS можно проводить при 4 ° C в течение ночи. Реагенты NHS имеют более длительный период полураспада перед гидролизом при 4 ° C, что составляет около 4-6 часов. Это приведет к трехдневной процедуре подготовки поверхности. - Добавьте 100 мкл 0,1 М NaHCO3 , содержащего 1 мг/мл сульфо-NHS ацетата, к набору «сэндвич-стекла» (два покровных стекла, обращенных друг к другу, с реакционным буфером между ними). Дайте пассивации произойти в течение не менее 30 минут. Для экономии реагента этот этап может быть выполнен с 50 мкл 1 мг / мл сульфо-ацетата NHS. После пассивации трижды смойте водой.
- Добавьте 0,5 мл наночастиц золота (AuNP, 8,8 нм, дубильная кислота, 0,05 мг/мл) в каждое покровное стекло и инкубируйте в течение 30 минут при комнатной температуре (рис. 3D). Чтобы сохранить реагент, этот шаг можно выполнить, также зажав два покровных стекла. Убедитесь, что в системе нет солей из предыдущих шагов, чтобы избежать агрегации наночастиц золота. Не оставляйте покровные стекла сохнуть после этого шага.
- Между тем, предварительно гибридизируйте шпильку 4,7 pN, цепь лиганда Cy3B и якорную цепь BHQ2, которые образуют зонды натяжения ДНК, конструируются в соотношении 1,1: 1: 1 в 1 M NaCl при 300 нМ в пробирке для ПЦР. Отжигают пряди, нагревая раствор до 95 °C в течение 5 мин, затем постепенно охлаждают, снижая температуру до 20 °C в течение 30 мин в термоциклере.
- Промойте покровные стекла водой три раза после 30 минут инкубации с наночастицами золота. Добавьте дополнительную якорную цепь BHQ2 (из запаса 100 мкМ) в отожженный раствор ДНК, чтобы получить соотношение между якорной цепью BHQ2 и цепью лиганда Cy3B 10:1. На этом этапе раствор ДНК должен содержать 300 нМ конструкции натяжного зонда и 2,7 мкМ цепи BHQ2. Добавьте 100 мкл на два покровных стекла, чтобы получился «сэндвич» (рис. 3E).
- Осторожно поместите влажный лабораторный шарик в чашку Петри (подальше от покровных стекол) и запечатайте чашку парафиновой пленкой, чтобы предотвратить высыхание раствора. Накройте блюдо фольгой и выдерживайте при температуре 4 °C в течение ночи.
- День 2
- Смойте излишки щупов с покровных стекол с помощью 1x PBS. Проверьте качество поверхности зонда на натяжение ДНК под эпифлуоресцентным микроскопом.
- Приготовьте 40 мкг/мл стрептавидина в 1x PBS и инкубируйте на покровных стеклах в течение 30 мин при комнатной температуре (рис. 3F). Обычно для покровного стекла диаметром 25 мм достаточно 100 мкл. После инкубации промыть PBS 3 раза, чтобы смыть лишнее количество стрептавидина.
- Приготовьте 40 мкг/мл биотинилированного антитела/лиганда в 1x PBS. Добавьте 50-100 мкл на бутерброд и выдерживайте в течение 30 минут при комнатной температуре (рис. 3G). Промойте PBS три раза после инкубации, чтобы смыть избыточное количество биотинилированных антител / лиганда.
- Тщательно соберите чистые камеры визуализации с поверхностями. Поверхности могут быть легко растресканы при затягивании камер. Добавьте 0,5-1 мл сбалансированного солевого раствора Хэнка (HBSS) в камеры визуализации и держите их готовыми к визуализации с помощью клеток (рис. 3H).
3. Визуализация клеточных рецепторных сил
- Подготовьте иммунные клетки, представляющие интерес для HBSS, в дозе 1-2 x 106 клеток/мл.
ПРИМЕЧАНИЕ: В качестве примера в этой статье используются наивные клетки OT-1 CD8+. Очистите наивные Т-клетки OT-1 CD8+ a из селезенки принесенных в жертву мышей с помощью набора для выделения CD8+ Т-клеток мыши MACS с сепаратором MACS в соответствии с инструкциями производителя. Изолируйте и обогащайте CD8 + Т-клетки, удаляя любые Т-клетки, отличные от CD8 +, с которыми связан коктейль магнитных истощающих антител. Ресуспендируют очищенные OT-1 CD8+ наивные Т-клетки в HBSS в дозе 2 x 106 клеток/мл и хранят на льду перед использованием. - Проверьте качество поверхности зонда натяжения шпильки ДНК под флуоресцентным микроскопом (объектив 100x) для контроля качества перед добавлением лигандов или гальванических клеток. Изображение и количественная оценка средней интенсивности фона в канале Cy3B поверхности зонда натяжения шпильки ДНК по крайней мере из 5 различных положений и 3 реплик. Обеспечьте согласованность условий получения изображений, чтобы это значение можно было использовать в качестве надежного маркера качества поверхности и плотности зонда (рис. 4C).
ПРИМЕЧАНИЕ: Количественно определите количество нитей ДНК на наночастицу золота и количество наночастиц золота намкм 2 в первые несколько раз подготовки поверхности в соответствии с литературой12, что может быть использовано в качестве еще одного надежного маркера качества поверхности. - Пластина ~ 4 x 10 4 - 10 x 104 клетки на каждом натяжном зонде ДНК функционализирует покровное стекло и позволяет им прикрепляться и распространяться в течение ~ 15 мин при комнатной температуре.
- По мере того, как клетки наносятся на натяжные зонды ДНК и начинают распространяться, визуализируйте флуоресцентные сигналы, которые генерируются в канале Cy3B со 100-кратным объективом (рис. 3I).
- После того, как клетки начнут вырабатывать сигнал натяжения в реальном времени на поверхности натяжного зонда ДНК в канале Cy3B, получите изображения в каналах Cy3B и Atto647N (микроскопия TIRF дает лучшее соотношение сигнал/шум, чем эпифлуоресценция). Затем добавьте цепь Atto647N в камеры визуализации в конечной концентрации 200 нМ для механически селективной гибридизации.
- После 10 минут инкубации быстро и осторожно удалите буфер, содержащий флуоресцентную фиксирующую нить Atto647N, и замените ее свежими сбалансированными солями Хэнка. Снова получите изображение в каналах Cy3B и Atto647N и определите коэффициент корреляции Пирсона с помощью программного обеспеченияFiji 15.
- В момент, представляющий интерес для исследования, введите нефлуоресцентную фиксирующую нить в клетки в камере визуализации для хранения сигнала натяжения. Приготовьте фиксирующую нить (100 мкМ) и добавьте в клетки в конечной концентрации 1 мкМ. Аккуратно перемешайте пипеткой. Продолжительность блокировки может варьироваться, но рекомендуемое время составляет 10 минут.
- Получение покадровых видеороликов или конечных изображений в эпифлуоресценции как для качественного картирования напряжения, так и для количественного анализа по мере необходимости (рис. 3J и рис. 5).
ПРИМЕЧАНИЕ: Если требуется измерение натяжения в несколько моментов времени, инициируйте стирание сохраненных сигналов натяжения, добавив разблокирующую прядь. Чтобы избежать чрезмерного промывания, более высокая конечная концентрация разблокирующей нити при 2 мкМ используется для инициирования реакции смещения цепи, опосредованной захватом пальца, с фиксирующей нитью в течение 3 мин, которая стирает сохраненные сигналы (рис. 3J). Аккуратно смойте излишки олигонуклеотидов с помощью HBSS. Поверхность натяжного зонда шпильки ДНК и клетки готовы к следующему раунду хранения и картирования натяжения. Разблокировка сигналов напряжения не нужна, если только один момент времени представляет интерес для исследования.
4. Анализ данных
ПРИМЕЧАНИЕ: Анализ изображений выполняется с использованием программного обеспечения Fiji, а количественный анализ выполняется с использованием программного обеспечения для анализа.
- Исправьте любой дрейф во время получения изображения с помощью команды « Исправить 3D-дрейф » в разделе «Регистрация » в меню «Плагины ».
- Удалите фон камеры изображения с помощью команды « Вычитать » в меню «Процесс ».
- Определите коэффициент корреляции Пирсона с функцией «Колокализация» в меню «Анализ».
- Усредните и вычтите фон флуоресценции, создаваемый неоткрытыми зондами, из трех разных локальных фоновых областей. Рисуйте рентабельность инвестиций ячеек на изображениях с вычитанием фона или изображениях RICM (отражение, интерференционно-контрастная микроскопия) с помощью инструмента « Выделение изображения J от руки ». Измерьте любую метрику, представляющую интерес для ROI, например, интегральную интенсивность флуоресценции и занятость напряжения, используя инструмент «Измерение» в меню «Анализ» (рис. 6).
- Экспортируйте измерения для количественного анализа с помощью программного обеспечения для анализа.
- Стройте график данных с помощью любого программного обеспечения для анализа.
Результаты
Здесь мы показываем репрезентативные изображения контроля качества поверхности (рис. 4). Высококачественная поверхность должна иметь чистый фон в канале RICM (рис. 4B) и равномерную интенсивность флуоресценции в канале Cy3B (рис. 4C). При одинак?...
Обсуждение
С помощью подробных процедур, представленных здесь, можно подготовить субстраты зонда натяжения шпильки ДНК для картирования и количественной оценки напряжения рецепторов, продуцируемого иммунными клетками. Когда клетки наносятся на подложку зонда натяжения шпильки ДНК, они приземл...
Раскрытие информации
Авторы заявляют об отсутствии конфликта интересов.
Благодарности
Эта работа была поддержана грантами NIH R01GM131099, NIH R01GM124472 и NSF CAREER 1350829. Мы благодарим Центр тетрамеров NIH за лиганды pMHC. Это исследование было частично поддержано Emory Comprehensive Glycomics Core.
Материалы
| Name | Company | Catalog Number | Comments |
| 3-hydroxypicolinic acid (3-HPA) | Sigma | 56197 | maldi-TOF-MS matrix |
| mPEG-SC | Biochempeg | MF001023-2K | surface prep |
| (3-Aminopropyl)triethoxysilane | Acros | AC430941000 | surface prep |
| 10x Red blood cell lysis buffer | Biolegend | 00-4333-57 | buffer |
| 8.8 nm gold nanoparticles, tannic acid | Nanocomposix | customized order | surface prep |
| Atto647N NHS ester | Sigma | 18373-1MG-F | fluorophore, oligo prep |
| Attofluor Cell Chamber, for microscopy | Thermo Fisher Scientific | A7816 | imaging |
| BD Syringes only with Luer-Lok | BD bioscience | 309657 | cells |
| biotinylated anti-mouse CD3e | ebioscience | 13-0031-82 | antibody/ligand |
| Biotinylated pMHC ovalbumin (SIINFEKL) | NIH Tetramer Core Facility at Emory University | NA | antibody/ligand |
| bovine serum albumin | Sigma | 735078001 | block non-specific interactions |
| Cell strainers | Biologix | 15-1100 | cells |
| Coverslip Mini-Rack, teflon | Thermo Fisher Scientific | C14784 | surface prep |
| Cy3B NHS ester | GE Healthcare | PA63101 | fluorophore, oligo prep |
| Dulbecco's phosphate-buffered saline (DPBS) | Corning | 21-031-CM | buffer |
| ethanol | Sigma | 459836 | surface prep |
| Hank’s balanced salts (HBSS) | Sigma | H8264 | buffer |
| hydrogen peroxide | Sigma | H1009 | surface prep |
| LA-PEG-SC | Biochempeg | HE039023-3.4K | surface prep |
| Midi MACS (LS) startup kit | Miltenyi Biotec | 130-042-301 | cells |
| mouse CD8+ T cell isolation kit | Miltenyi Biotec | 130-104-075 | cells |
| Nanosep MF centrifugal devices | Pall laboratory | ODM02C35 | oligo prep |
| No. 2 round glass coverslips | VWR | 48382-085 | surface prep |
| NTA-SAM | Dojindo Molecular Technologies | N475-10 | surface prep |
| P2 gel | Bio-rad | 1504118 | oligo prep |
| sufuric acid | EMD Millipore Corporation | SX1244-6 | surface prep |
| Sulfo-NHS acetate | Thermo Fisher Scientific | 26777 | surface prep |
| Equipment | |||
| Agilent AdvanceBio Oligonucleotide C18 column, 4.6 x 150 mm, 2.7 μm | 653950-702 | oligonucleotide preparation | |
| Barnstead Nanopure water purifying system | Thermo Fisher | water | |
| CFI Apo 100× NA 1.49 objective | Nikon | Microscopy | |
| Cy5 cube | CHROMA | Microscopy | |
| evolve electron multiplying charge coupled device (EMCCD) | Photometrics | Microscopy | |
| High-performance liquid chromatography | Agilent 1100 | oligonucleotide preparation | |
| Intensilight epifluorescence source | Nikon | Microscopy | |
| Matrix-assisted laser desorption/ionization time-of-flight mass spectrometer (MALDI-TOF-MS) | Voyager STR | oligonucleotide preparation | |
| Nanodrop 2000 UV-Vis Spectrophotometer | Thermo Fisher | oligonucleotide preparation | |
| Nikon Eclipse Ti inverted microscope | Nikon | Microscopy | |
| Nikon Perfect Focus System | Nikon | Microscopy | |
| NIS Elements software | Nikon | Microscopy | |
| quad band TIRF 405/488/561/647 cube | CHROMA | Microscopy | |
| RICM cube | CHROMA | Microscopy | |
| TIRF launcher with 488 nm (50 mW), 561 nm (50 mW), and 640 nm | Coherent | Microscopy | |
| TRITC cube | CHROMA | Microscopy | |
| oligo name | 5' modification / 3' modification | sequence (5' to 3') | Use |
| 15mer amine locking strand | 5' modification: no modification 3' modification: /3AmMO/ | AAA AAA CAT TTA TAC CCT ACC TA | locking real-time tension signal |
| 15mer Atto647N locking strand | 5' modification: Atto647N 3' modification: /3AmMO/ | AAA AAA CAT TTA TAC CCT ACC TA | locking real-time tension signal |
| 15mer non-fluoresccent locking strand | 5' modification: no modification 3' modification: no modification | A AAA AAC ATT TAT AC | locking real-time tension signal for quantitative analysis |
| 4.7 pN hairpin strand | 5' modification: no modification 3' modification: no modification | GTGAAATACCGCACAGATGCGT TTGTATAAATGTTTTTTTCATTTAT ACTTTAAGAGCGCCACGTAGCC CAGC | hairpin probe |
| amine ligand strand | 5' modification: /5AmMC6/ 3' modification: /3Bio/ | CGCATCTGTGCG GTA TTT CAC TTT | hairpin probe |
| BHQ2 anchor strand | 5' modification: /5ThiolMC6-D/ 3' modification: /3BHQ_2/ | TTTGCTGGGCTACGTGGCGCTCTT | hairpin probe |
| Cy3B ligand strand | 5' modification: Cy3B 3' modification: /3Bio/ | CGCATCTGTGCG GTA TTT CAC TTT | hairpin probe |
| unlocking strand | 5' modification: no modification 3' modification: no modification | TAG GTA GGG TAT AAA TGT TTT TTT C | unlocking accumulated tension signal |
Ссылки
- Dustin, M. L. T-cell activation through immunological synapses and kinapses. Immunological Reviews. 221 (1), 77-89 (2008).
- Spillane, K. M., Tolar, P. B cell antigen extraction is regulated by physical properties of antigen-presenting cells. Journal of Cell Biology. 216 (1), 217-230 (2017).
- Feng, Y., et al. Mechanosensing drives acuity of αβ T-cell recognition. Proceedings of the National Academy of Sciences. 114 (39), 8204-8213 (2017).
- Hong, J., et al. A TCR mechanotransduction signaling loop induces negative selection in the thymus. Nature Immunology. 19 (12), 1379-1390 (2018).
- Basu, R., et al. Cytotoxic T cells use mechanical force to potentiate target cell killing. Cell. 165 (1), 100-110 (2016).
- Bashour, K. T., et al. CD28 and CD3 have complementary roles in T-cell traction forces. Proceedings of the National Academy of Sciences. 111 (6), 2241-2246 (2014).
- Ma, V. P. Y., Salaita, K. DNA nanotechnology as an emerging tool to study mechanotransduction in living systems. Small. 15 (26), 1900961 (2019).
- Liu, Y., Yehl, K., Narui, Y., Salaita, K. Tension sensing nanoparticles for mechano-imaging at the living/nonliving interface. Journal of the American Chemical Society. 135 (14), 5320-5323 (2013).
- Glazier, R., et al. DNA mechanotechnology reveals that integrin receptors apply pN forces in podosomes on fluid substrates. Nature Communications. 10 (1), 1-13 (2019).
- Galior, K., Liu, Y., Yehl, K., Vivek, S., Salaita, K. Titin-based nanoparticle tension sensors map high-magnitude integrin forces within focal adhesions. Nano Letters. 16 (1), 341-348 (2016).
- Zhang, Y., Ge, C., Zhu, C., Salaita, K. DNA-based digital tension probes reveal integrin forces during early cell adhesion. Nature Communications. 5, 5167 (2014).
- Liu, Y., et al. DNA-based nanoparticle tension sensors reveal that T-cell receptors transmit defined pN forces to their antigens for enhanced fidelity. Proceedings of the National Academy of Sciences. 113 (20), 5610-5615 (2016).
- Zhang, Y., et al. Platelet integrins exhibit anisotropic mechanosensing and harness piconewton forces to mediate platelet aggregation. Proceedings of the National Academy of Sciences. 115 (2), 325-330 (2018).
- Huang, J., et al. The kinetics of two-dimensional TCR and pMHC interactions determine T-cell responsiveness. Nature. 464 (7290), 932-936 (2010).
- Ma, R., et al. DNA probes that store mechanical information reveal transient piconewton forces applied by T cells. Proceedings of the National Academy of Sciences. 116 (34), 16949-16954 (2019).
- Hui, E., et al. T cell costimulatory receptor CD28 is a primary target for PD-1-mediated inhibition. Science. 355 (6332), 1428-1433 (2017).
- Whitley, K. D., Comstock, M. J., Chemla, Y. R. Elasticity of the transition state for oligonucleotide hybridization. Nucleic Acids Research. 45 (2), 547-555 (2016).
- Brockman, J. M., et al. Live-cell super-resolved PAINT imaging of piconewton cellular traction forces. Nature Methods. 17 (10), 1018-1024 (2020).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеThis article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены