È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Sonde di tensione del DNA per mappare le forze transitorie del recettore di Piconewton da parte delle cellule immunitarie
In questo articolo
Riepilogo
Questo documento descrive un protocollo dettagliato per l'utilizzo di sonde di tensione basate sul DNA per visualizzare le forze recettoriali applicate dalle cellule immunitarie. Questo approccio può mappare le forze recettoriali >4.7pN in tempo reale e può integrare le forze nel tempo.
Abstract
Le forze meccaniche trasmesse alla giunzione tra due cellule vicine e alla giunzione tra le cellule e la matrice extracellulare sono fondamentali per regolare molti processi che vanno dallo sviluppo all'immunologia. Pertanto, lo sviluppo di strumenti per studiare queste forze su scala molecolare è fondamentale. Il nostro gruppo ha sviluppato una suite di sensori di tensione molecolare per quantificare e visualizzare le forze generate dalle cellule e trasmesse a ligandi specifici. La classe più sensibile di sensori di tensione molecolare è costituita da forcine ad anello stelo di acido nucleico. Questi sensori utilizzano coppie fluoroforo-quencher per segnalare l'estensione meccanica e lo sviluppo delle forcine del DNA sotto forza. Una sfida con i sensori di tensione a forcina del DNA è che sono reversibili con un rapido ripiegamento della forcina al termine della tensione e quindi le forze transitorie sono difficili da registrare. In questo articolo, descriviamo i protocolli per la preparazione di sensori di tensione del DNA che possono essere "bloccati" e impediti di ripiegarsi per consentire la "memorizzazione" di informazioni meccaniche. Ciò consente la registrazione di forze piconewton altamente transitorie, che possono essere successivamente "cancellate" dall'aggiunta di acidi nucleici complementari che rimuovono la serratura. Questa capacità di alternare tra la mappatura della tensione in tempo reale e la memorizzazione di informazioni meccaniche rivela forze deboli, di breve durata e meno abbondanti, che sono comunemente impiegate dalle cellule T come parte delle loro funzioni immunitarie.
Introduzione
Le cellule immunitarie difendono dagli agenti patogeni e dalle cellule tumorali strisciando e scansionando continuamente le superfici delle cellule bersaglio alla ricerca di antigeni, costellando la loro superficie 1,2. Il riconoscimento dell'antigene viene avviato al momento del legame tra il recettore delle cellule T (TCR) e il complesso di istocompatibilità peptide-maggiore MHC (pMHC) espresso sulla superficie delle cellule bersaglio. Poiché il riconoscimento TCR-pMHC avviene alla giunzione tra due cellule mobili, è stato a lungo sospettato di sperimentare forze meccaniche. Inoltre, questo ha portato al modello meccanosensore di attivazione TCR, che suggerisce che le forze TCR contribuiscono alla sua funzione 3,4. Per capire quando, dove e come le forze meccaniche contribuiscono alla funzione delle cellule T, è imperativo sviluppare strumenti per visualizzare le forze molecolari trasmesse dalle cellule T. Tradizionalmente, metodi come la microscopia della forza di trazione (TFM) e gli array di micropilastri sono usati per studiare le forze cellulari 5,6. Tuttavia, la sensibilità alla forza degli array di TFM e micropilastri è alla scala nanonewton (nN) e quindi è spesso insufficiente per studiare le forze molecolari di piconewton (pN) trasmesse dai recettori cellulari7. Per migliorare la forza e la risoluzione spaziale per il rilevamento, il nostro laboratorio ha aperto la strada allo sviluppo di sonde di tensione molecolare, che sono state inizialmente sintetizzate utilizzando polimeri di polietilenglicole (PEG)7. Le sonde di tensione molecolare sono costituite da una "molla" molecolare estensibile (PEG, proteina, DNA) affiancata da un fluoroforo e un quencher e sono ancorate su una superficie. Le forze applicate al terminale della sonda portano alla sua estensione, separando il fluoroforo e il quencher, e generando così un forte segnale di fluorescenza (Figura 1A)8,9,10.
Negli ultimi dieci anni abbiamo sviluppato una libreria di diverse classi di sonde di tensione molecolari con elementi a molla costituiti da acidi nucleici11, proteine10 e polimeri8. Tra questi, le sonde di tensione basate sul DNA forniscono il più alto rapporto segnale/rumore e la massima sensibilità alla forza, che è facilmente sintonizzata da pochi pN fino a ~20 pN11. Abbiamo utilizzato queste sonde di tensione del DNA in tempo reale per studiare le forze molecolari generate da molti diversi tipi di cellule, tra cui fibroblasti, cellule tumorali, piastrine e cellule immunitarie11,12,13. Questo manoscritto descriverà i protocolli per sintetizzare e assemblare sonde di tensione del DNA su una superficie per mappare le forze dei recettori molecolari con risoluzione della forza pN utilizzando un microscopio a fluorescenza convenzionale. Mentre l'attuale procedura include modifiche chimiche all'acido nucleico per introdurre il reporter fluorescente (Figura 1B), è importante notare che molte delle fasi di modifica e purificazione possono essere esternalizzate a società di sintesi del DNA personalizzate. Pertanto, la tecnologia delle sonde di tensione del DNA è facile e accessibile alle più ampie comunità di biologia cellulare e meccanobiologia.
In breve, per assemblare i sensori di tensione del DNA, una forcina di DNA viene ibridata con un filamento di ligando fluorescente su un braccio e un filamento di ancoraggio di quencher sull'altro braccio e quindi immobilizzato su un substrato di vetro (Figura 1C, tensione in tempo reale). In assenza di forza meccanica, la forcina viene chiusa e quindi la fluorescenza viene spenta. Tuttavia, quando la forza meccanica applicata è maggiore di F1/2 (la forza all'equilibrio che porta a una probabilità del 50% di dispiegarsi), la forcina si scioglie meccanicamente e viene generato un segnale fluorescente.
Basandoci sul sensore di tensione del DNA in tempo reale, descriviamo anche i protocolli per mappare le forze accumulate, che è particolarmente utile per studiare le interazioni tra i recettori sulle cellule immunitarie e il loro ligando naturale. Questo perché i recettori immunitari spesso mostrano legami di breve durata 3,14. Le forze accumulate vengono visualizzate utilizzando un filamento di "bloccaggio" che si lega preferenzialmente alle forcine di DNA aperte e consente la memorizzazione di segnali di fluorescenza associati a eventi di trazione meccanica (Figura 1C, tensione bloccata). Il filo di bloccaggio è progettato per legare un sito di legame criptico che viene esposto alla fusione indotta meccanicamente della forcina e bloccare la forcina nello stato aperto bloccando la ripiegatura della forcina, memorizzando così il segnale di tensione e generando una mappa di tensione accumulata. Inoltre, il filo di bloccaggio è progettato con un appiglio a otto nucleotidi, che consente una reazione di spostamento del filamento mediata dalla presa con il suo complemento completo, il filamento "sbloccante". Con l'aggiunta del trefolo di sblocco, il filo di bloccaggio legato viene rimosso dal costrutto a forcina, cancellando il segnale di tensione memorizzato e ripristinando la forcina allo stato in tempo reale.
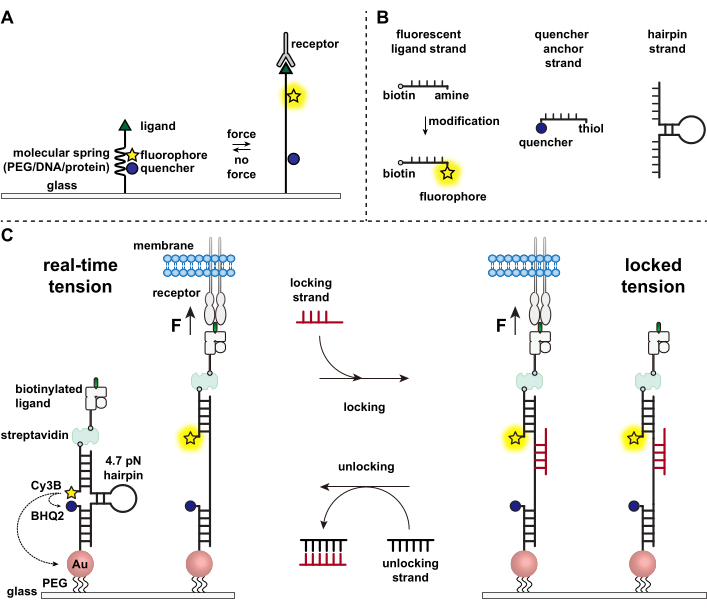
Figura 1: Schema delle sonde di tensione molecolari all'avanguardia . (A) Progettazione generale della sonda di tensione molecolare in tempo reale, (B) Filamenti per il costrutto della sonda di tensione basata sul DNA e (C) sonde di tensione basate sul DNA ingegnerizzate e loro commutazione tra stato in tempo reale e stato bloccato. Fare clic qui per visualizzare una versione ingrandita di questa figura.
Il protocollo principale consiste di quattro sezioni principali: preparazione degli oligonucleotidi, preparazione della superficie, imaging e analisi dei dati. Questo protocollo è stato dimostrato con successo dal nostro laboratorio e da altri in cellule T CD8+ OT-1 naïve e attivate, cellule CD4+ OT-II e ibridomi e può essere applicato per interrogare diversi recettori delle cellule immunitarie tra cui il recettore delle cellule T, il recettore della morte cellulare programmata (PD1) e le forze dell'antigene 1 associato alla funzione linfocitaria (LFA-1). Le cellule T CD8 + naïve OT-1 sono usate come linea cellulare di esempio in questo articolo.
Protocollo
I topi transgenici OT-1 sono ospitati presso la Division of Animal Resources Facility della Emory University. Tutti gli esperimenti sono stati approvati ed eseguiti nell'ambito del protocollo Institutional Animal Care and Use Committee (IACUC).
1. Preparazione di oligonucleotidi
- Sciogliere il DNA del filamento del ligando in acqua (resistività 18,2 MΩ, utilizzata in tutto il protocollo). Vortice e far girare la soluzione con una centrifuga da tavolo. Sintonizzare il volume d'acqua in modo tale che la concentrazione finale sia di 1 mM. Convalidare la concentrazione utilizzando uno spettrofotometro a nanogoccia per misurare l'assorbanza a 260 nm e determinare la concentrazione finale in base al coefficiente di estinzione dell'oligonucleotide.
NOTA: Il filamento del ligando ha una modifica ad ogni terminale, 5' ammina e 3' biotina, per coniugarsi con il fluoroforo e presentare il ligando biotinilato. Il gruppo amminico nel filamento del ligando deve essere coniugato con un fluoroforo. Il colorante Cy3B viene utilizzato per questa coniugazione a causa della sua elevata luminosità e fotostabilità, ma non è generalmente offerto commercialmente e richiede una coniugazione interna. Di conseguenza, la sezione seguente descrive la coniugazione tra ammine e coloranti esteri del NHS. Per gli utenti finali che non hanno accesso a strutture o risorse per la modifica dell'acido nucleico, gli acidi nucleici modificati possono invece essere acquistati da fornitori di sintesi del DNA personalizzati che offrono coloranti luminosi e fotostabili, come la famiglia di coloranti Alexa e Atto. - Preparare 10 soluzioni PBS e 1 M NaHCO3 . Mescolare 10 μL della soluzione di filamento di legante amminico 1 mM (10 nmol) con 10 μL di 10x PBS, 10 μL di 1 M NaHCO3 e 60 μL di H2O. Sciogliere 50 μg di estere Cy3B NHS in 10 μL di DMSO immediatamente prima dell'uso e aggiungere alla miscela per un volume totale di reazione di 100 μL. Aggiungere l'estere Cy3B NHS per ultimo. Lasciare reagire a temperatura ambiente per 1 ora o 4 °C durante la notte.
- Preparare il filo di bloccaggio Atto647N coniugando il filo di bloccaggio dell'ammina con l'estere Atto647N NHS. Preparare 10x PBS e 1 M soluzione NaHCO3 . Mescolare 10 μL della soluzione del filamento di bloccaggio dell'ammina da 1 mM (10 nmol) con 10 μL di 10x PBS, 10 μL di 1 M NaHCO3 e 60 μL di H2O. Sciogliere 50 μg di estere NHS Atto647N in 10 μL di DMSO immediatamente prima dell'uso e aggiungere alla miscela per un volume di reazione totale di 100 μL. Aggiungere l'estere NHS Atto647N per ultimo. Lasciare reagire a temperatura ambiente per 1 ora o 4 °C durante la notte.
- Dopo le reazioni, rimuovere i sottoprodotti, il colorante in eccesso e i sali mediante filtrazione del gel di desalinizzazione P2. Diluire la miscela di reazione con H2O fino a un volume totale di 300 μL, che è appropriato per la successiva fase di purificazione HPLC. Aggiungere 650 μL di gel P2 idratato a un dispositivo centrifugo e centrifugare a 18.000 x g per 1 minuto. Rimuovere il liquido nella parte inferiore del dispositivo, aggiungere la miscela di reazione alla colonna contenente gel P2, ruotare verso il basso a 18.000 x g per 1 minuto e raccogliere la miscela di reazione nella parte inferiore del dispositivo.
NOTA: P2 gel deve essere idratato almeno 4 ore prima dell'uso con H2O. - Purificare la miscela di reazione dissalata con HPLC utilizzando una colonna C18 designata per la purificazione oligonucleotidica, con solvente A: 0,1 M TEAA in H2O e B: ACN come fase mobile per un'eluizione lineare del gradiente 10-100% B su 50 min ad una portata di 0,5 mL/min. Iniettare la miscela di reazione dissalata in HPLC in fase inversa con un circuito di iniezione da 500 μL per la purificazione. Raccogliere il prodotto che ha un picco di assorbanza per il DNA (260 nm) e un picco di assorbanza per il fluoroforo (560 nm per Cy3B e 647 nm per Atto647N) ed essiccarlo in un concentratore centrifugo sotto vuoto durante la notte (vedere Figura 2A).
- Ricostituire il prodotto oligocolorante essiccato in 100 μL di acqua. Determinare la concentrazione del filamento del ligando Cy3B e del filamento di bloccaggio Atto647N con lo spettrofotometro a nanogoccia. Assicurarsi che il rapporto di etichettatura del colorante sia vicino a 1:1. Correggere l'assorbanza di 260 nm del colorante, se necessario, quando si determina la concentrazione di oligonucleotidi.
- Convalidare il prodotto purificato con MALDI-TOF-MS utilizzando 3-HPA come substrato in ACN/H 2O al 50% con TFA allo 0,1% e 5 mg/ml di citrato ammonico utilizzando 0,5 μL di prodotto a 1-5 μM per la preparazione del campione MALDI-TOF-MS. Un esempio di spettro di massa può essere trovato nella Figura 2B.
- Sciogliere il filo a forcina e il filo di ancoraggio di spegnimento in acqua e assicurarsi che la concentrazione delle soluzioni madre sia compresa tra 50 e 100 μM.
NOTA: il filo a forcina non è modificato e può essere sintetizzato direttamente da un fornitore. Il filo di ancoraggio ha un gruppo di ancoraggio tiolo e un quencher BHQ2 e può essere sintetizzato direttamente su misura da un fornitore. - Aliquot tutti gli oligonucleotidi. Per l'uso e la conservazione a breve termine, conservare questi oligonucleotidi a 4 °C. Per una conservazione a lungo termine, congelarli e mantenerli a -20 °C. A questo punto, tutti gli oligonucleotidi sono pronti per l'assemblaggio della sonda di tensione del DNA.
NOTA: cicli ripetuti di congelamento-disgelo non sono problematici per gli oligonucleotidi.
2. Preparazione della superficie
NOTA: La preparazione dei substrati della sonda di tensione a forcina del DNA richiede due giorni. La sonda di tensione della forcina del DNA sarà funzionalizzata su vetrini di vetro.
- Giorno 1
- Posizionare i vetrini da 25 mm su una griglia in politetrafluoroetilene in un becher da 50 ml. Ogni rack può contenere fino a 8 copertine. Risciacquare i coperchi immergendoli in acqua tre volte.
- Aggiungere 40 mL di una soluzione di etanolo con rapporto 1:1 (v:v) miscelata con acqua al becher contenente la cremagliera e i vetrini di copertura e sigillare il becher usando una pellicola di paraffina.
- Sonicare il becher per 15 minuti in un pulitore ad ultrasuoni (frequenza operativa 35 KHz) per pulire i vetrini di copertura. Dopo la sonicazione, scartare il liquido e sciacquare il becher con il rack e coprirlo con acqua almeno 6 volte per rimuovere qualsiasi solvente organico rimanente.
- Preparare la soluzione fresca di Piranha mescolando acido solforico e perossido di idrogeno nel rapporto di 3: 1. Per preparare 40 ml di soluzione di Piranha, aggiungere prima 30 ml di acido solforico in un becher pulito da 50 ml e poi aggiungere lentamente 10 ml di H 2 O2. La soluzione di Piranha si riscalda rapidamente e bolle dopo l'aggiuntadi H 2 O2. Mescolare delicatamente il piranha usando l'estremità di una pipetta di vetro.
- Quindi, trasferire il rack che contiene i vetrini di copertura sul becher contenente la soluzione di Piranha delicatamente miscelata per l'incisione (Figura 3A). Lasciare idrossilare la soluzione di Piranha e pulire i vetrini per 30 minuti a temperatura ambiente. Dopo l'incisione Piranha, trasferire il rack con pinzette in acciaio o politetrafluoroetilene in un becher pulito da 50 ml con acqua e risciacquare nuovamente con acqua almeno 6 volte.
ATTENZIONE: Grandi quantità di sostanze organiche potrebbero reagire vigorosamente con la soluzione di Piranha e causare esplosioni. Fare attenzione e lavorare sempre con la soluzione Piranha in una cappa aspirante. Assicurati di indossare un camice, guanti e occhiali di sicurezza. Non conservare mai la soluzione fresca di Piranha in un contenitore sigillato.
NOTA: Il rapporto tra perossido di idrogeno e acido solforico deve essere mantenuto al di sotto di 1:2 (v:v) e non deve mai superare 1:1. Quando si immerge il rack con vetrini di copertura nella soluzione di Piranha, posizionarli nella soluzione lentamente e con attenzione. Non gettare la soluzione immediatamente dopo l'incisione, poiché è ancora attiva e calda. Lasciarlo nel becher per una notte prima di versarlo nel contenitore dei rifiuti acidi. - Immergere il rack contenente i vetrini di copertura in un becher da 50 ml con 40 mL di etanolo per rimuovere l'acqua. Scartare l'etanolo e ripetere 3 volte per assicurarsi che l'acqua sia stata rimossa.
- Quindi immergere il rack in amminopropiltrietossisilano al 3% (APTES) (v/v) in 40 ml di etanolo per reagire con il -OH sui vetrini di copertura per 1 ora a temperatura ambiente (Figura 3B).
NOTA: L'etanolo può essere sostituito dall'acetone. - Risciacquare le superfici 6 volte immergendole in 40 mL di etanolo, quindi asciugare in forno a 80 °C per 20 minuti. Dopo il raffreddamento, conservare i vetrini essiccati modificati con ammine a -20 °C per un uso futuro (fino a 6 mesi).
- Coprire il lato inferiore interno di piastre di Petri di plastica di 10 cm di diametro con pellicola di paraffina. Il film di paraffina impedisce ai vetrini di scivolare all'interno della piastra di Petri e aiuta a mantenere la soluzione per le fasi successive di funzionalizzazione sui vetrini di copertura. Posizionare i coprivetrini raffreddati modificati con ammine nelle piastre di Petri. Il lato da funzionalizzare dovrebbe essere rivolto verso l'alto.
- Per modificare i gruppi amminici sui vetrini, aggiungere 300 μL di acido lipoico 0,5% p/v PEG NHS (LA-PEG-SC) e 2,5% p/v mPEG NHS (mPEG-SC) in 0,1 M NaHCO3 su ciascun vetrino e incubare per 1 ora a temperatura ambiente (Figura 3C). Per ogni coprislip da 25 mm, pesare 1,5 mg di LA-PEG-SC e 7,5 mg di mPEG-SC. Sciogliere i reagenti NHS immediatamente prima di aggiungerli alle superfici, poiché hanno una breve emivita (~10 min) in soluzione acquosa a temperatura ambiente. Dopo la reazione, sciacquare le superfici 3 volte con acqua.
NOTA: La reazione NHS può essere eseguita a 4 °C durante la notte. I reagenti del NHS hanno un'emivita più lunga prima dell'idrolisi a 4 °C, che è di circa 4-6 ore. Ciò si tradurrà in una procedura di preparazione della superficie di tre giorni. - Aggiungere 100 μL di 0,1 M NaHCO3 contenente 1 mg/mL di solfo-NHS acetato a una serie di vetrini "sandwich" (due vetrini rivolti l'uno verso l'altro con tampone di reazione in mezzo). Lasciare che la passivazione avvenga per almeno 30 minuti. Per risparmiare reagente, questo passaggio potrebbe essere fatto con 50 μL di 1 mg / mL di solfo-NHS acetato. Risciacquare con acqua tre volte dopo la passivazione.
- Aggiungere 0,5 ml di nanoparticelle d'oro (AuNP, 8,8 nm, acido tannico, 0,05 mg/ml) a ciascun coprivetrino e incubare per 30 minuti a temperatura ambiente (Figura 3D). Per salvare il reagente, questo passaggio può essere fatto inserendo anche due vetrini di copertura. Assicurarsi che non siano presenti sali nel sistema dai passaggi precedenti per evitare l'aggregazione di nanoparticelle d'oro. Non lasciare asciugare i coprivetrini dopo questo passaggio.
- Nel frattempo, pre-ibridare la forcina da 4,7 pN, il filamento di ligando Cy3B e il filamento di ancoraggio BHQ2 che formano il costrutto delle sonde di tensione del DNA con un rapporto di 1,1: 1: 1 in 1 M NaCl a 300 nM in un tubo PCR. Ricottura dei fili riscaldando la soluzione fino a 95 °C per 5 minuti, quindi raffreddare gradualmente diminuendo la temperatura a 20 °C in 30 minuti in un termociclatore.
- Risciacquare i coperchi con acqua tre volte dopo 30 minuti di incubazione con nanoparticelle d'oro. Aggiungere ulteriore filamento di ancoraggio BHQ2 (da 100 μM di stock) alla soluzione di DNA ricotto per ottenere il rapporto tra il filamento di ancoraggio BHQ2 e il filamento di ligando Cy3B 10:1. A questo punto, la soluzione di DNA dovrebbe contenere 300 nM di costrutto di sonda di tensione e 2,7 μM di filamento BHQ2. Aggiungere 100 μL per due vetrini per fare il "sandwich" (Figura 3E).
- Posizionare con cautela una sfera di tessuto da laboratorio bagnata nella capsula di Petri (lontano da vetrini) e sigillare la capsula con pellicola di paraffina per evitare che la soluzione si secchi. Coprire il piatto con un foglio di alluminio e incubare a 4 °C per una notte.
- Giorno 2
- Lavare via le sonde in eccesso dai vetrini con 1x PBS. Verificare la qualità della superficie della sonda di tensione del DNA al microscopio epifluorescente.
- Preparare 40 μg/mL di streptavidina in 1x PBS e incubare su vetrini per 30 minuti a temperatura ambiente (Figura 3F). Di solito, 100 μL sono sufficienti per un coprislip da 25 mm. Risciacquare con PBS 3 volte dopo l'incubazione per lavare via la quantità in eccesso di streptavidina.
- Preparare 40 μg/mL di anticorpo/ligando biotinilato in 1x PBS. Aggiungere 50-100 μL per panino e incubare per 30 minuti a temperatura ambiente (Figura 3G). Risciacquare con PBS tre volte dopo l'incubazione per lavare via la quantità in eccesso di anticorpo/ligando biotinilato.
- Assemblare accuratamente le camere di imaging pulite con le superfici. Le superfici possono essere facilmente incrinate quando si stringono le camere. Aggiungere 0,5-1 mL di soluzione salina bilanciata di Hank (HBSS) alle camere di imaging e tenerle pronte per l'imaging con le cellule (Figura 3H).
3. Imaging delle forze del recettore cellulare
- Preparare le cellule immunitarie di interesse in HBSS a 1-2 x 106 cellule / ml.
NOTA: le cellule naïve CD8+ OT-1 sono usate come esempio in questo documento. Purificare le cellule T naïve OT-1 CD8+ a dalla milza dei topi sacrificati utilizzando il kit di isolamento delle cellule T CD8+ del mouse MACS con un separatore MACS seguendo le istruzioni del produttore. Isolare e arricchire le cellule T CD8+ rimuovendo tutte le cellule T non CD8+ a cui è legato il cocktail di anticorpi che impoveriscono magneticamente. Risospendere le cellule T ot-1 CD8+ naïve purificate in HBSS a 2 x 106 cellule/ml e conservare in ghiaccio prima dell'uso. - Controllare la qualità della superficie della sonda di tensione a forcina del DNA al microscopio a fluorescenza (obiettivo 100x) per il controllo di qualità prima di aggiungere ligandi o cellule di placcatura. Immagine e quantificazione dell'intensità media di fondo nel canale Cy3B della superficie di una sonda di tensione a forcina di DNA da almeno 5 posizioni diverse e 3 repliche. Mantenere coerenti le condizioni di acquisizione dell'imaging in modo che questo valore possa essere utilizzato come indicatore affidabile della qualità della superficie e della densità della sonda (Figura 4C).
NOTA: Quantificare il numero di filamenti di DNA per nanoparticella d'oro e il numero di nanoparticelle d'oro per μm2 le prime volte di preparazione della superficie secondo la letteratura12, che può essere utilizzato come un altro indicatore affidabile della qualità della superficie. - Piastra ~ 4 x 10 4 - 10 x 104 cellule su ogni sonda di tensione del DNA funzionalizzata coprislip e consentire loro di attaccare e diffondere per ~ 15 minuti a temperatura ambiente.
- Quando le cellule vengono placcate sulle sonde di tensione della forcina del DNA e iniziano a diffondersi, visualizzare i segnali di fluorescenza generati nel canale Cy3B con l'obiettivo 100x (Figura 3I).
- Dopo che le cellule iniziano a produrre un segnale di tensione in tempo reale sulla superficie della sonda di tensione a forcina del DNA nel canale Cy3B, acquisire immagini in entrambi i canali Cy3B e Atto647N (la microscopia TIRF fornisce un migliore rapporto segnale-rumore rispetto all'epifluorescenza). Successivamente, aggiungere il filamento Atto647N alle camere di imaging ad una concentrazione finale di 200 nM per l'ibridazione meccanicamente selettiva.
- Dopo 10 minuti di incubazione, rimuovere rapidamente e delicatamente il tampone contenente il filo di bloccaggio fluorescente Atto647N e sostituirlo con sali freschi bilanciati di Hank. Immagine in entrambi i canali Cy3B e Atto647N di nuovo e determinare il coefficiente di correlazione di Pearson con il software Fiji15.
- Nel momento di interesse per l'indagine, introdurre un filamento di bloccaggio non fluorescente nelle celle nella camera di imaging per memorizzare il segnale di tensione. Preparare il filo di bloccaggio (100 μM) e aggiungere alle celle ad una concentrazione finale di 1 μM. Pipetta delicatamente per miscelare. La durata del blocco può variare ma 10 minuti è il tempo consigliato.
- Acquisire filmati time-lapse o immagini end-point in epifluorescenza sia per la mappatura qualitativa della tensione che per l'analisi quantitativa, se necessario (Figura 3J e Figura 5).
NOTA: se si desidera misurare la tensione in più punti temporali, avviare la cancellazione dei segnali di tensione memorizzati mediante l'aggiunta di un trefolo di sblocco. Per evitare un risciacquo eccessivo, viene utilizzata una maggiore concentrazione finale del filo di sblocco a 2 μM per avviare una reazione di spostamento del filamento mediata dalla punta con il filo di bloccaggio per 3 minuti, che cancella i segnali memorizzati (Figura 3J). Risciacquare delicatamente gli oligonucleotidi in eccesso con HBSS. La superficie della sonda di tensione della forcina del DNA e le cellule sono pronte per un altro giro di memorizzazione e mappatura della tensione. Lo sblocco dei segnali di tensione non è necessario se solo un punto temporale è di interesse nello studio.
4. Analisi dei dati
NOTA: l'analisi delle immagini viene eseguita utilizzando il software Fiji e l'analisi quantitativa viene eseguita utilizzando il software di analisi.
- Correggi qualsiasi deriva durante l'acquisizione dell'immagine con il comando Correggi deriva 3D in Registrazione nel menu Plugin .
- Rimuovete lo sfondo della fotocamera dell'immagine con il comando Sottrai nel menu Elabora .
- Determinare il coefficiente di correlazione di Pearson con la funzione di colocalizzazione nel menu Analizza .
- Media e sottrazione del fondo di fluorescenza prodotto dalle sonde non aperte da tre diverse regioni di fondo locali. Disegna ROI delle celle su immagini sottratte dallo sfondo o immagini RICM (microscopia a contrasto di interferenza di riflessione) con lo strumento Selezioni a mano libera di Image J. Misurare qualsiasi metrica di interesse dei ROI, ad esempio l'intensità di fluorescenza integrata e l'occupazione della tensione utilizzando lo strumento Misura nel menu Analizza (Figura 6).
- Esporta le misure per l'analisi quantitativa con il software di analisi.
- Traccia i dati con qualsiasi software di analisi.
Risultati
Qui mostriamo immagini rappresentative del controllo della qualità della superficie (Figura 4). Una superficie di alta qualità dovrebbe avere uno sfondo pulito nel canale RICM (Figura 4B) e un'intensità di fluorescenza uniforme nel canale Cy3B (Figura 4C). Con le stesse apparecchiature di imaging e identiche condizioni di acquisizione dell'imaging a fluorescenza, l'intensità della fluorescenza di fondo dovrebbe essere coerente e...
Discussione
Con le procedure dettagliate fornite qui, è possibile preparare substrati di sonda di tensione a forcina di DNA per mappare e quantificare la tensione del recettore prodotta dalle cellule immunitarie. Quando le cellule vengono placcate sul substrato della sonda di tensione della forcina del DNA, atterrano, si attaccano e si diffondono mentre i recettori percepiscono i ligandi sia chimicamente che meccanicamente, l'ultimo dei quali viene rilevato dalle nostre sonde. Tuttavia, in alcuni casi le cellule potrebbero non rius...
Divulgazioni
Gli autori non dichiarano alcun conflitto di interessi.
Riconoscimenti
Questo lavoro è stato supportato da NIH Grants R01GM131099, NIH R01GM124472 e NSF CAREER 1350829. Ringraziamo il NIH Tetramer Facility per i ligandi pMHC. Questo studio è stato supportato, in parte, dall'Emory Comprehensive Glycomics Core.
Materiali
| Name | Company | Catalog Number | Comments |
| 3-hydroxypicolinic acid (3-HPA) | Sigma | 56197 | maldi-TOF-MS matrix |
| mPEG-SC | Biochempeg | MF001023-2K | surface prep |
| (3-Aminopropyl)triethoxysilane | Acros | AC430941000 | surface prep |
| 10x Red blood cell lysis buffer | Biolegend | 00-4333-57 | buffer |
| 8.8 nm gold nanoparticles, tannic acid | Nanocomposix | customized order | surface prep |
| Atto647N NHS ester | Sigma | 18373-1MG-F | fluorophore, oligo prep |
| Attofluor Cell Chamber, for microscopy | Thermo Fisher Scientific | A7816 | imaging |
| BD Syringes only with Luer-Lok | BD bioscience | 309657 | cells |
| biotinylated anti-mouse CD3e | ebioscience | 13-0031-82 | antibody/ligand |
| Biotinylated pMHC ovalbumin (SIINFEKL) | NIH Tetramer Core Facility at Emory University | NA | antibody/ligand |
| bovine serum albumin | Sigma | 735078001 | block non-specific interactions |
| Cell strainers | Biologix | 15-1100 | cells |
| Coverslip Mini-Rack, teflon | Thermo Fisher Scientific | C14784 | surface prep |
| Cy3B NHS ester | GE Healthcare | PA63101 | fluorophore, oligo prep |
| Dulbecco's phosphate-buffered saline (DPBS) | Corning | 21-031-CM | buffer |
| ethanol | Sigma | 459836 | surface prep |
| Hank’s balanced salts (HBSS) | Sigma | H8264 | buffer |
| hydrogen peroxide | Sigma | H1009 | surface prep |
| LA-PEG-SC | Biochempeg | HE039023-3.4K | surface prep |
| Midi MACS (LS) startup kit | Miltenyi Biotec | 130-042-301 | cells |
| mouse CD8+ T cell isolation kit | Miltenyi Biotec | 130-104-075 | cells |
| Nanosep MF centrifugal devices | Pall laboratory | ODM02C35 | oligo prep |
| No. 2 round glass coverslips | VWR | 48382-085 | surface prep |
| NTA-SAM | Dojindo Molecular Technologies | N475-10 | surface prep |
| P2 gel | Bio-rad | 1504118 | oligo prep |
| sufuric acid | EMD Millipore Corporation | SX1244-6 | surface prep |
| Sulfo-NHS acetate | Thermo Fisher Scientific | 26777 | surface prep |
| Equipment | |||
| Agilent AdvanceBio Oligonucleotide C18 column, 4.6 x 150 mm, 2.7 μm | 653950-702 | oligonucleotide preparation | |
| Barnstead Nanopure water purifying system | Thermo Fisher | water | |
| CFI Apo 100× NA 1.49 objective | Nikon | Microscopy | |
| Cy5 cube | CHROMA | Microscopy | |
| evolve electron multiplying charge coupled device (EMCCD) | Photometrics | Microscopy | |
| High-performance liquid chromatography | Agilent 1100 | oligonucleotide preparation | |
| Intensilight epifluorescence source | Nikon | Microscopy | |
| Matrix-assisted laser desorption/ionization time-of-flight mass spectrometer (MALDI-TOF-MS) | Voyager STR | oligonucleotide preparation | |
| Nanodrop 2000 UV-Vis Spectrophotometer | Thermo Fisher | oligonucleotide preparation | |
| Nikon Eclipse Ti inverted microscope | Nikon | Microscopy | |
| Nikon Perfect Focus System | Nikon | Microscopy | |
| NIS Elements software | Nikon | Microscopy | |
| quad band TIRF 405/488/561/647 cube | CHROMA | Microscopy | |
| RICM cube | CHROMA | Microscopy | |
| TIRF launcher with 488 nm (50 mW), 561 nm (50 mW), and 640 nm | Coherent | Microscopy | |
| TRITC cube | CHROMA | Microscopy | |
| oligo name | 5' modification / 3' modification | sequence (5' to 3') | Use |
| 15mer amine locking strand | 5' modification: no modification 3' modification: /3AmMO/ | AAA AAA CAT TTA TAC CCT ACC TA | locking real-time tension signal |
| 15mer Atto647N locking strand | 5' modification: Atto647N 3' modification: /3AmMO/ | AAA AAA CAT TTA TAC CCT ACC TA | locking real-time tension signal |
| 15mer non-fluoresccent locking strand | 5' modification: no modification 3' modification: no modification | A AAA AAC ATT TAT AC | locking real-time tension signal for quantitative analysis |
| 4.7 pN hairpin strand | 5' modification: no modification 3' modification: no modification | GTGAAATACCGCACAGATGCGT TTGTATAAATGTTTTTTTCATTTAT ACTTTAAGAGCGCCACGTAGCC CAGC | hairpin probe |
| amine ligand strand | 5' modification: /5AmMC6/ 3' modification: /3Bio/ | CGCATCTGTGCG GTA TTT CAC TTT | hairpin probe |
| BHQ2 anchor strand | 5' modification: /5ThiolMC6-D/ 3' modification: /3BHQ_2/ | TTTGCTGGGCTACGTGGCGCTCTT | hairpin probe |
| Cy3B ligand strand | 5' modification: Cy3B 3' modification: /3Bio/ | CGCATCTGTGCG GTA TTT CAC TTT | hairpin probe |
| unlocking strand | 5' modification: no modification 3' modification: no modification | TAG GTA GGG TAT AAA TGT TTT TTT C | unlocking accumulated tension signal |
Riferimenti
- Dustin, M. L. T-cell activation through immunological synapses and kinapses. Immunological Reviews. 221 (1), 77-89 (2008).
- Spillane, K. M., Tolar, P. B cell antigen extraction is regulated by physical properties of antigen-presenting cells. Journal of Cell Biology. 216 (1), 217-230 (2017).
- Feng, Y., et al. Mechanosensing drives acuity of αβ T-cell recognition. Proceedings of the National Academy of Sciences. 114 (39), 8204-8213 (2017).
- Hong, J., et al. A TCR mechanotransduction signaling loop induces negative selection in the thymus. Nature Immunology. 19 (12), 1379-1390 (2018).
- Basu, R., et al. Cytotoxic T cells use mechanical force to potentiate target cell killing. Cell. 165 (1), 100-110 (2016).
- Bashour, K. T., et al. CD28 and CD3 have complementary roles in T-cell traction forces. Proceedings of the National Academy of Sciences. 111 (6), 2241-2246 (2014).
- Ma, V. P. Y., Salaita, K. DNA nanotechnology as an emerging tool to study mechanotransduction in living systems. Small. 15 (26), 1900961 (2019).
- Liu, Y., Yehl, K., Narui, Y., Salaita, K. Tension sensing nanoparticles for mechano-imaging at the living/nonliving interface. Journal of the American Chemical Society. 135 (14), 5320-5323 (2013).
- Glazier, R., et al. DNA mechanotechnology reveals that integrin receptors apply pN forces in podosomes on fluid substrates. Nature Communications. 10 (1), 1-13 (2019).
- Galior, K., Liu, Y., Yehl, K., Vivek, S., Salaita, K. Titin-based nanoparticle tension sensors map high-magnitude integrin forces within focal adhesions. Nano Letters. 16 (1), 341-348 (2016).
- Zhang, Y., Ge, C., Zhu, C., Salaita, K. DNA-based digital tension probes reveal integrin forces during early cell adhesion. Nature Communications. 5, 5167 (2014).
- Liu, Y., et al. DNA-based nanoparticle tension sensors reveal that T-cell receptors transmit defined pN forces to their antigens for enhanced fidelity. Proceedings of the National Academy of Sciences. 113 (20), 5610-5615 (2016).
- Zhang, Y., et al. Platelet integrins exhibit anisotropic mechanosensing and harness piconewton forces to mediate platelet aggregation. Proceedings of the National Academy of Sciences. 115 (2), 325-330 (2018).
- Huang, J., et al. The kinetics of two-dimensional TCR and pMHC interactions determine T-cell responsiveness. Nature. 464 (7290), 932-936 (2010).
- Ma, R., et al. DNA probes that store mechanical information reveal transient piconewton forces applied by T cells. Proceedings of the National Academy of Sciences. 116 (34), 16949-16954 (2019).
- Hui, E., et al. T cell costimulatory receptor CD28 is a primary target for PD-1-mediated inhibition. Science. 355 (6332), 1428-1433 (2017).
- Whitley, K. D., Comstock, M. J., Chemla, Y. R. Elasticity of the transition state for oligonucleotide hybridization. Nucleic Acids Research. 45 (2), 547-555 (2016).
- Brockman, J. M., et al. Live-cell super-resolved PAINT imaging of piconewton cellular traction forces. Nature Methods. 17 (10), 1018-1024 (2020).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon