Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
DNA-Spannungssonden zur Kartierung der transienten Piconewton-Rezeptor-Kräfte durch Immunzellen
In diesem Artikel
Zusammenfassung
In dieser Arbeit wird ein detailliertes Protokoll für die Verwendung von DNA-basierten Spannungssonden beschrieben, um die von Immunzellen ausgeübten Rezeptorkräfte abzubilden. Dieser Ansatz kann Rezeptorkräfte >4,7 pN in Echtzeit abbilden und Kräfte über die Zeit integrieren.
Zusammenfassung
Mechanische Kräfte, die an der Verbindungsstelle zwischen zwei benachbarten Zellen und an der Verbindungsstelle zwischen Zellen und der extrazellulären Matrix übertragen werden, sind entscheidend für die Regulierung vieler Prozesse, die von der Entwicklung bis zur Immunologie reichen. Daher ist es von entscheidender Bedeutung, die Werkzeuge zu entwickeln, um diese Kräfte auf molekularer Ebene zu untersuchen. Unsere Gruppe entwickelte eine Reihe von molekularen Spannungssensoren, um die von Zellen erzeugten und auf bestimmte Liganden übertragenen Kräfte zu quantifizieren und zu visualisieren. Die empfindlichste Klasse von molekularen Spannungssensoren besteht aus Nukleinsäure-Stammschleifen-Haarnadeln. Diese Sensoren verwenden Fluorophor-Quencher-Paare, um die mechanische Dehnung und Entfaltung von DNA-Haarnadeln unter Krafteinwirkung zu melden. Eine Herausforderung bei DNA-Hairpin-Spannungssensoren besteht darin, dass sie reversibel sind, wobei die Hairpin-Spannung beim Beenden der Spannung schnell zurückgefaltet werden kann und somit transiente Kräfte schwer zu erfassen sind. In diesem Artikel beschreiben wir die Protokolle zur Herstellung von DNA-Spannungssensoren, die "verriegelt" und am erneuten Falten gehindert werden können, um das "Speichern" mechanischer Informationen zu ermöglichen. Dies ermöglicht die Aufzeichnung von hochtransienten Piconewton-Kräften, die anschließend durch die Zugabe komplementärer Nukleinsäuren, die die Sperre entfernen, "gelöscht" werden können. Diese Fähigkeit, zwischen Echtzeit-Spannungsmapping und mechanischer Informationsspeicherung umzuschalten, offenbart schwache, kurzlebige und weniger reichlich vorhandene Kräfte, die von T-Zellen üblicherweise als Teil ihrer Immunfunktionen eingesetzt werden.
Einleitung
Immunzellen wehren sich gegen Krankheitserreger und Krebszellen, indem sie kontinuierlich die Oberflächen von Zielzellen nach Antigenen durchsuchen und deren Oberfläche abtasten 1,2. Die Antigenerkennung wird durch die Bindung zwischen dem T-Zell-Rezeptor (TCR) und dem Peptid-Major-Histokompatibilitätskomplex MHC (pMHC) eingeleitet, der auf der Oberfläche der Zielzellen exprimiert wird. Da die TCR-pMHC-Erkennung an der Verbindungsstelle zwischen zwei beweglichen Zellen erfolgt, steht sie seit langem im Verdacht, mechanische Kräfte zu erfahren. Darüber hinaus führte dies zum Mechanosensormodell der TCR-Aktivierung, das darauf hindeutet, dass TCR-Kräfte zu seiner Funktion beitragen 3,4. Um zu verstehen, wann, wo und wie mechanische Kräfte zur Funktion von T-Zellen beitragen, ist es unerlässlich, Werkzeuge zu entwickeln, um die von T-Zellen übertragenen molekularen Kräfte sichtbar zu machen. Traditionell werden Methoden wie Zugkraftmikroskopie (TFM) und Mikrosäulen-Arrays verwendet, um zelluläre Kräfte zu untersuchen 5,6. Die Kraftempfindlichkeit von TFM- und Mikrosäulen-Arrays liegt jedoch im Nanonewton (nN)-Bereich und reicht daher oft nicht aus, um die molekularen Piconewton (pN)-Kräfte zu untersuchen, die von Zellrezeptoren übertragenwerden 7. Um die Kraft und die räumliche Auflösung für die Detektion zu verbessern, leistete unser Labor Pionierarbeit bei der Entwicklung von molekularen Spannungssonden, die ursprünglich mit Polyethylenglykol (PEG)-Polymeren synthetisiert wurden7. Molekulare Spannungssonden bestehen aus einer ausziehbaren molekularen "Feder" (PEG, Protein, DNA), die von einem Fluorophor und Quencher flankiert wird und auf einer Oberfläche verankert ist. Kräfte, die auf den Endpunkt der Sonde ausgeübt werden, führen zu ihrer Verlängerung, trennen Fluorophor und Quencher und erzeugen so ein starkes Fluoreszenzsignal (Abbildung 1A)8,9,10.
In den letzten zehn Jahren haben wir eine Bibliothek verschiedener Klassen von molekularen Zugsonden mit Federelementen aus Nukleinsäuren11, Proteinen10 und Polymeren8 entwickelt. Unter diesen bieten die DNA-basierten Zugsonden das höchste Signal-Rausch-Verhältnis und die größte Kraftempfindlichkeit, die leicht von wenigen pN bis zu ~20 pN11 eingestellt werden kann. Wir haben diese Echtzeit-DNA-Spannungssonden verwendet, um die molekularen Kräfte zu untersuchen, die von vielen verschiedenen Zelltypen erzeugt werden, darunter Fibroblasten, Krebszellen, Blutplättchen und Immunzellen11,12,13. In diesem Manuskript werden Protokolle zur Synthese und Montage von DNA-Zugsonden auf einer Oberfläche beschrieben, um molekulare Rezeptorkräfte mit pN-Kraftauflösung unter Verwendung eines konventionellen Fluoreszenzmikroskops abzubilden. Während das derzeitige Verfahren chemische Modifikationen der Nukleinsäure umfasst, um den fluoreszierenden Reporter einzuführen (Abbildung 1B), ist es wichtig zu beachten, dass viele der Modifikations- und Aufreinigungsschritte an kundenspezifische DNA-Syntheseunternehmen ausgelagert werden können. Daher ist die Technologie der DNA-Spannungssonden einfach und für die breitere Gemeinschaft der Zellbiologie und Mechanobiologie zugänglich.
Kurz gesagt, um DNA-Spannungssensoren zusammenzubauen, wird eine DNA-Haarnadel mit einem fluoreszierenden Ligandenstrang auf einem Arm und einem Quencher-Ankerstrang auf dem anderen Arm hybridisiert und dann auf einem Glassubstrat immobilisiert (Abbildung 1C, Echtzeitspannung). In Abwesenheit einer mechanischen Kraft wird die Haarnadel geschlossen und somit die Fluoreszenz gelöscht. Wenn jedoch die ausgeübte mechanische Kraft größer als F1/2 ist (die Kraft im Gleichgewicht, die zu einer 50%igen Wahrscheinlichkeit der Entfaltung führt), schmilzt die Haarnadel mechanisch und es wird ein fluoreszierendes Signal erzeugt.
Aufbauend auf dem Echtzeit-DNA-Spannungssensor beschreiben wir auch Protokolle zur Kartierung akkumulierter Kräfte, was besonders nützlich ist, um Interaktionen zwischen Rezeptoren auf Immunzellen und ihrem natürlichen Liganden zu untersuchen. Dies liegt daran, dass Immunrezeptoren oft kurzlebige Bindungen aufweisen 3,14. Die akkumulierten Kräfte werden mit einem "verriegelnden" Strang abgebildet, der bevorzugt an offene DNA-Haarnadeln bindet und die Speicherung von Fluoreszenzsignalen ermöglicht, die mit mechanischen Zugereignissen verbunden sind (Abbildung 1C, verriegelte Spannung). Der Verriegelungsstrang ist so konzipiert, dass er eine kryptische Bindungsstelle bindet, die beim mechanisch induzierten Schmelzen der Haarnadel freigelegt wird, und die Haarnadel im offenen Zustand sperrt, indem er die Rückfaltung der Haarnadel blockiert, wodurch das Spannungssignal gespeichert und eine akkumulierte Spannungskarte erzeugt wird. Darüber hinaus ist der Locking-Strang mit einem Acht-Nukleotid-Zehengriff ausgelegt, der eine Zehengriff-vermittelte Strangverdrängungsreaktion mit seinem vollständigen Komplement, dem "entsperrenden" Strang, ermöglicht. Durch das Hinzufügen des Entriegelungsstrangs wird der gebundene Verriegelungsstrang vom Hairpin-Konstrukt abgestreift, wodurch das gespeicherte Spannungssignal gelöscht und der Hairpin wieder in den Echtzeitzustand zurückgesetzt wird.
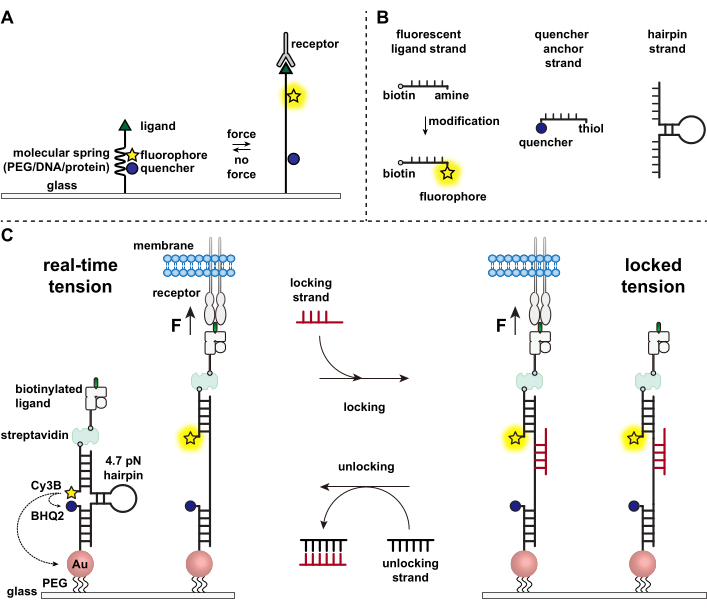
Abbildung 1: Schema der hochmodernen molekularen Spannungssonden. (A) Allgemeines Design einer Echtzeit-Molekularspannungssonde, (B) Stränge für das DNA-basierte Spannungssondenkonstrukt und (C) DNA-basierte Spannungssonden und deren Umschalten zwischen Echtzeitzustand und verriegeltem Zustand. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
Das Hauptprotokoll besteht aus vier Hauptabschnitten - Oligonukleotidpräparation, Oberflächenpräparation, Bildgebung und Datenanalyse. Dieses Protokoll wurde von unserem Labor und anderen erfolgreich in naiven und aktivierten OT-1 CD8+ T-Zellen, OT-II CD4+ Zellen sowie Hybridomen demonstriert und kann angewendet werden, um verschiedene Immunzellrezeptoren zu untersuchen, einschließlich des T-Zell-Rezeptors, des programmierten Zelltodrezeptors (PD1) und der Lymphozytenfunktions-assoziierten Antigen-1-Kräfte (LFA-1). OT-1 CD8+ naive T-Zellen werden in dieser Arbeit als Beispielzelllinie verwendet.
Protokoll
Die transgenen OT-1-Mäuse sind in der Division of Animal Resources Facility der Emory University untergebracht. Alle Experimente wurden nach dem Protokoll des Institutional Animal Care and Use Committee (IACUC) genehmigt und durchgeführt.
1. Herstellung von Oligonukleotiden
- Lösen Sie die DNA des Ligandenstrangs in Wasser auf (18,2 MΩ Widerstand, der im gesamten Protokoll verwendet wird). Vortexieren und schleudern Sie die Lösung mit einer Tischzentrifuge herunter. Stellen Sie das Wasservolumen so ein, dass die Endkonzentration 1 mM beträgt. Validieren Sie die Konzentration mit einem Nanotropfen-Spektralphotometer, um die Absorption bei 260 nm zu messen und die Endkonzentration basierend auf dem Extinktionskoeffizienten des Oligonukleotids zu bestimmen.
ANMERKUNG: Der Ligandenstrang weist an jedem Ende eine Modifikation auf, 5'-Amin und 3'-Biotin, um mit dem Fluorophor zu konjugieren und den biotinylierten Liganden zu präsentieren. Die Amingruppe im Ligandenstrang muss mit einem Fluorophor konjugiert werden. Cy3B-Farbstoff wird aufgrund seiner hohen Helligkeit und Photostabilität für diese Konjugation verwendet, wird jedoch in der Regel nicht kommerziell angeboten und erfordert eine hauseigene Konjugation. Dementsprechend wird im folgenden Abschnitt die Konjugation zwischen Aminen und NHS-Esterfarbstoffen beschrieben. Für Endbenutzer, die keinen Zugang zu Einrichtungen oder Ressourcen für die Nukleinsäuremodifikation haben, können modifizierte Nukleinsäuren stattdessen von Anbietern kundenspezifischer DNA-Synthese erworben werden, die helle und photostabile Farbstoffe anbieten, wie z. B. die Farbstofffamilie Alexa und Atto. - Bereiten Sie 10x PBS- und 1 M NaHCO3-Lösungen vor. Mischen Sie 10 μl der 1 mM Amin-Liganden-Stranglösung (10 nmol) mit 10 μl 10x PBS, 10 μl 1 MNaHCO3 und 60 μlH2O. Lösen Sie unmittelbar vor Gebrauch 50 μg Cy3B NHS-Ester in 10 μl DMSO auf und fügen Sie dem Gemisch ein Gesamtreaktionsvolumen von 100 μl hinzu. Zuletzt Cy3B NHS-Ester hinzufügen. Bei Raumtemperatur 1 h oder 4 °C über Nacht einwirken lassen.
- Bereiten Sie den Atto647N-Locking-Strang vor, indem Sie den Amin-Locking-Strang mit Atto647N NHS-Ester konjugieren. Bereiten Sie 10x PBS und 1 M NaHCO3 Lösung vor. Mischen Sie 10 μl der 1 mM Amin-Locking-Strang-Lösung (10 nmol) mit 10 μl 10x PBS, 10 μl 1 M NaHCO3 und 60 μlH2O. Lösen Sie unmittelbar vor Gebrauch 50 μg Atto647N NHS-Ester in 10 μl DMSO auf und fügen Sie dem Gemisch ein Gesamtreaktionsvolumen von 100 μl hinzu. Zuletzt Atto647N NHS-Ester hinzufügen. Bei Raumtemperatur 1 h oder 4 °C über Nacht einwirken lassen.
- Entfernen Sie nach den Reaktionen Nebenprodukte, überschüssigen Farbstoff und Salze durch P2-Entsalzungsgelfiltration. Das Reaktionsgemisch wird mitH2Oauf ein Gesamtvolumen von 300 μL verdünnt, das für den nachfolgenden HPLC-Aufreinigungsschritt geeignet ist. Geben Sie 650 μl hydratisiertes P2-Gel in eine Zentrifugalvorrichtung und schleudern Sie es 1 Minute lang bei 18.000 x g herunter. Entfernen Sie die Flüssigkeit an der Unterseite des Geräts, geben Sie die Reaktionsmischung in die Säule mit P2-Gel, drehen Sie sie 1 Minute lang bei 18.000 x g herunter und sammeln Sie die Reaktionsmischung an der Unterseite des Geräts.
Anmerkungen: P2-Gel sollte mindestens 4 Stunden vor der Verwendung mitH2O hydratisiert werden. - Reinigen Sie das entsalzte Reaktionsgemisch mit HPLC unter Verwendung einer C18-Säule, die für die Oligonukleotidaufreinigung vorgesehen ist, mit Lösungsmittel A: 0,1 M TEAA inH2Ound B: ACN als mobile Phase für eine lineare Gradientenelution 10-100% B über 50 min bei einer Flussrate von 0,5 ml/min. Injizieren Sie das entsalzte Reaktionsgemisch in die Umkehrphasen-HPLC mit einer 500-μl-Injektionsschleife zur Aufreinigung. Sammeln Sie das Produkt mit einem Absorptionspeak für die DNA (260 nm) und einem Absorptionspeak für den Fluorophor (560 nm für Cy3B und 647 nm für Atto647N) und trocknen Sie es über Nacht in einem Vakuum-Zentrifugalkonzentrator (siehe Abbildung 2A).
- Rekonstituieren Sie das getrocknete Oligofarbstoffprodukt in 100 μl Wasser. Bestimmen Sie die Konzentration des Cy3B-Ligandenstrangs und des Atto647N-Locking-Strangs mit dem Nanodrop-Spektralphotometer. Stellen Sie sicher, dass das Farbmarkierungsverhältnis nahe bei 1:1 liegt. Korrigieren Sie bei Bedarf die 260 nm Absorption des Farbstoffs bei der Bestimmung der Oligonukleotidkonzentration.
- Validieren Sie das gereinigte Produkt mit MALDI-TOF-MS unter Verwendung von 3-HPA als Substrat in 50 % ACN/H2O mit 0,1 % TFA und 5 mg/ml Ammoniumcitrat unter Verwendung von 0,5 μl des Produkts bei 1-5 μM für die MALDI-TOF-MS-Probenvorbereitung. Ein Beispiel für ein Massenspektrum ist in Abbildung 2B zu finden.
- Lösen Sie den Haarnadelstrang und den Quencher-Ankerstrang in Wasser auf und achten Sie darauf, dass die Konzentration der Stammlösungen zwischen 50 und 100 μM liegt.
HINWEIS: Der Haarnadelstrang ist unmodifiziert und kann direkt von einem Anbieter individuell synthetisiert werden. Der Ankerstrang verfügt über eine Thiol-Verankerungsgruppe und einen Quencher BHQ2 und kann direkt von einem Hersteller kundenspezifisch synthetisiert werden. - Aliquotieren Sie alle Oligonukleotide. Für den kurzfristigen Gebrauch und die Lagerung lagern Sie diese Oligonukleotide bei 4 °C. Für die Langzeitlagerung frieren Sie sie ein und bewahren Sie sie bei -20 °C auf. Zu diesem Zeitpunkt sind alle Oligonukleotide bereit für die DNA-Spannungssonde.
HINWEIS: Wiederholte Gefrier-Auftau-Zyklen sind für Oligonukleotide unproblematisch.
2. Oberflächenvorbereitung
HINWEIS: Die Herstellung der Substrate der DNA-Haarnadel-Zugsonde dauert zwei Tage. Die DNA-Hairpin-Zugsonde wird auf Glasdeckgläsern funktionalisiert.
- Tag 1
- Legen Sie die 25-mm-Deckgläser auf ein Polytetrafluorethylen-Gestell in einem 50-ml-Becherglas. Jedes Gestell bietet Platz für bis zu 8 Deckgläser. Spülen Sie die Deckgläser ab, indem Sie sie dreimal in Wasser tauchen.
- Geben Sie 40 ml einer mit Wasser vermischten Ethanollösung im Verhältnis 1:1 in das Becherglas mit dem Gestell und den Deckgläsern und verschließen Sie das Becherglas mit einer Paraffinfolie.
- Beschallen Sie das Becherglas 15 Minuten lang in einem Ultraschallreiniger (Betriebsfrequenz 35 KHz), um die Deckgläser zu reinigen. Entsorgen Sie nach der Beschallung die Flüssigkeit und spülen Sie das Becherglas mit dem Gestell und den Deckgläsern mindestens 6 Mal mit Wasser aus, um verbleibende organische Lösungsmittel zu entfernen.
- Bereiten Sie frische Piranha-Lösung zu, indem Sie Schwefelsäure und Wasserstoffperoxid im Verhältnis 3:1 mischen. Um 40 ml Piranha-Lösung herzustellen, geben Sie zuerst 30 ml Schwefelsäure in ein sauberes 50-ml-Becherglas und fügen Sie dann langsam 10 mlH2O2hinzu. Die Piranha-Lösung erhitzt sich schnell und sprudelt, wennH2O2zugegeben wird. Mischen Sie den Piranha vorsichtig mit dem Ende einer Glaspipette.
- Übertragen Sie als Nächstes das Gestell, in dem sich die Deckgläser befinden, in das Becherglas mit der vorsichtig gemischten Piranha-Lösung zum Ätzen (Abbildung 3A). Lassen Sie die Piranha-Lösung hydroxylieren und reinigen Sie die Deckgläser 30 Minuten lang bei Raumtemperatur. Übertragen Sie das Gestell nach dem Piranha-Ätzen mit einer Pinzette aus Stahl oder Polytetrafluorethylen in ein sauberes 50-ml-Becherglas mit Wasser und spülen Sie es mindestens 6 Mal erneut mit Wasser ab.
ACHTUNG: Große Mengen organischer Substanzen können mit Piranha-Lösung heftig reagieren und eine Explosion verursachen. Seien Sie vorsichtig und arbeiten Sie immer mit Piranha-Lösung in einem Abzug. Achten Sie darauf, einen Laborkittel, Handschuhe und eine Schutzbrille zu tragen. Bewahren Sie niemals frische Piranha-Lösung in einem verschlossenen Behälter auf.
Anmerkungen: Das Verhältnis von Wasserstoffperoxid zu Schwefelsäure sollte unter 1:2 (v:v) gehalten werden und niemals 1:1 überschreiten. Wenn Sie das Gestell mit Deckgläsern in Piranha-Lösung tauchen, legen Sie diese langsam und vorsichtig in die Lösung. Entsorgen Sie die Lösung nicht sofort nach dem Ätzen, da sie noch aktiv und heiß ist. Lassen Sie es über Nacht im Becherglas, bevor Sie es in den Säureabfallbehälter gießen. - Tauchen Sie das Gestell mit den Deckgläsern in ein 50-ml-Becherglas mit 40-ml-Ethanol, um Wasser zu entfernen. Entsorgen Sie das Ethanol und wiederholen Sie den Vorgang 3 Mal, um sicherzustellen, dass das Wasser entfernt wurde.
- Tauchen Sie dann das Rack in 3%iges Aminopropyltriethoxysilan (APTES) (v/v) in 40 ml Ethanol, um 1 h bei Raumtemperatur mit dem -OH auf den Deckgläsern zu reagieren (Abbildung 3B).
Anmerkungen: Ethanol kann durch Aceton ersetzt werden. - Spülen Sie die Oberflächen 6 Mal ab, indem Sie sie in 40 ml Ethanol tauchen, und trocknen Sie sie dann 20 Minuten lang im Ofen bei 80 °C. Lagern Sie die getrockneten aminmodifizierten Deckgläser nach dem Abkühlen bei -20 °C für die zukünftige Verwendung (bis zu 6 Monate).
- Decken Sie die untere Innenseite von Kunststoff-Petrischalen mit einem Durchmesser von 10 cm mit Paraffinfolie ab. Die Paraffinfolie verhindert das Verrutschen der Deckgläser in der Petrischale und trägt dazu bei, dass die Lösung für die nächsten Schritte der Funktionalisierung auf den Deckgläsern verbleibt. Legen Sie die abgekühlten aminmodifizierten Deckgläser in die Petrischale. Die zu funktionalisierende Seite sollte nach oben zeigen.
- Um die Amingruppen auf den Deckgläsern zu modifizieren, werden 300 μl 0,5 % w/v Liponsäure PEG NHS (LA-PEG-SC) und 2,5 % w/v mPEG NHS (mPEG-SC) in 0,1 M NaHCO3 auf jedes Deckglas gegeben und 1 h bei Raumtemperatur inkubiert (Abbildung 3C). Für jedes 25-mm-Deckglas wiegen Sie 1,5 mg LA-PEG-SC und 7,5 mg mPEG-SC ein. Lösen Sie die NHS-Reagenzien unmittelbar vor der Zugabe zu den Oberflächen auf, da sie in wässriger Lösung bei Raumtemperatur eine kurze Halbwertszeit (~10 min) haben. Spülen Sie die Oberflächen nach der Reaktion 3 Mal mit Wasser ab.
HINWEIS: Die NHS-Reaktion kann bei 4 °C über Nacht durchgeführt werden. NHS-Reagenzien haben eine längere Halbwertszeit vor der Hydrolyse bei 4 °C, was etwa 4-6 h beträgt. Dies führt zu einer dreitägigen Oberflächenvorbereitung. - 100 μl 0,1 M NaHCO3 mit 1 mg/ml Sulfo-NHS-Acetat in einen Satz "Sandwich"-Deckgläser geben (zwei Deckgläser zueinander mit Reaktionspuffer dazwischen). Lassen Sie die Passivierung mindestens 30 Minuten einwirken. Um Reagenz zu sparen, könnte dieser Schritt mit 50 μl 1 mg/ml Sulfo-NHS-Acetat durchgeführt werden. Nach der Passivierung dreimal mit Wasser abspülen.
- Fügen Sie 0,5 ml Goldnanopartikel (AuNP, 8,8 nm, Gerbsäure, 0,05 mg/ml) zu jedem Deckglas hinzu und inkubieren Sie es 30 Minuten lang bei Raumtemperatur (Abbildung 3D). Um das Reagenz zu sparen, kann dieser Schritt auch durch Einlegen von zwei Deckgläsern erfolgen. Stellen Sie sicher, dass keine Salze aus den vorherigen Schritten im System vorhanden sind, um eine Aggregation von Goldnanopartikeln zu vermeiden. Lassen Sie die Deckgläser nach diesem Schritt nicht trocknen.
- In der Zwischenzeit werden 4,7 pN-Haarnadeln, Cy3B-Ligandenstränge und BHQ2-Ankerstränge, die die DNA-Spannungssonden bilden, in einem Verhältnis von 1,1:1:1 in 1 M NaCl bei 300 nM in einem PCR-Röhrchen vorhybridisiert. Glühen Sie die Stränge, indem Sie die Lösung 5 Minuten lang auf 95 °C erhitzen, und kühlen Sie sie dann allmählich ab, indem Sie die Temperatur über 30 Minuten in einem Thermocycler auf 20 °C senken.
- Spülen Sie die Deckgläser nach 30 Minuten Inkubation mit Goldnanopartikeln dreimal mit Wasser ab. Fügen Sie der getemperten DNA-Lösung zusätzlichen BHQ2-Ankerstrang (ab 100 μM Bestand) hinzu, um das Verhältnis zwischen BHQ2-Ankerstrang und Cy3B-Ligandenstrang 10:1 zu erhalten. Zu diesem Zeitpunkt sollte die DNA-Lösung 300 nM Zugsondenkonstrukt und 2,7 μM BHQ2-Strang enthalten. Fügen Sie 100 μl pro zwei Deckgläser hinzu, um das "Sandwich" herzustellen (Abbildung 3E).
- Legen Sie vorsichtig eine nasse Labortaschentuchkugel in die Petrischale (weg von Deckgläsern) und verschließen Sie die Schale mit Paraffinfolie, um ein Austrocknen der Lösung zu verhindern. Die Form mit Folie abdecken und über Nacht bei 4 °C inkubieren.
- Tag 2
- Waschen Sie die überschüssigen Sonden von den Deckgläsern mit 1x PBS ab. Überprüfen Sie die Oberflächenqualität der DNA-Zugsonde unter einem Epifluoreszenzmikroskop.
- Bereiten Sie 40 μg/ml Streptavidin in 1x PBS vor und inkubieren Sie es 30 Minuten lang bei Raumtemperatur auf Deckgläsern (Abbildung 3F). In der Regel reichen 100 μL für ein 25 mm Deckglas aus. Nach der Inkubation 3 Mal mit PBS spülen, um die überschüssige Menge an Streptavidin wegzuspülen.
- Präparation von 40 μg/ml biotinyliertem Antikörper/Liganden in 1x PBS. Fügen Sie 50-100 μl pro Sandwich hinzu und inkubieren Sie es 30 Minuten lang bei Raumtemperatur (Abbildung 3G). Nach der Inkubation dreimal mit PBS spülen, um die überschüssige Menge an biotinylierten Antikörpern/Liganden abzuwaschen.
- Montieren Sie die sauberen Bildgebungskammern mit Oberflächen sorgfältig. Oberflächen können beim Anziehen der Kammern leicht rissig werden. Geben Sie 0,5 bis 1 ml der Hank-Salzlösung (HBSS) in die Bildgebungskammern und halten Sie sie für die Bildgebung mit Zellen bereit (Abbildung 3H).
3. Zellrezeptorkräfte abbilden
- Bereiten Sie Immunzellen von Interesse in HBSS mit 1-2 x 106 Zellen/ml vor.
HINWEIS: In diesem Artikel werden OT-1 CD8+ naive Zellen als Beispiel verwendet. Reinigen Sie OT-1 CD8+ naive T-Zellen a aus der Milz von geopferten Mäusen mit dem MACS Maus CD8+ T-Zell-Isolationskit mit einem MACS-Separator gemäß den Anweisungen des Herstellers. Isolieren und reichern Sie die CD8+ T-Zellen an, indem Sie alle Nicht-CD8+ T-Zellen entfernen, an die der magnetisch abbauende Antikörpercocktail gebunden ist. Resuspendieren Sie gereinigte OT-1 CD8+ naive T-Zellen in HBSS bei 2 x 106 Zellen/ml und halten Sie sie vor der Verwendung auf Eis. - Überprüfen Sie die Qualität der Oberfläche der DNA-Haarnadel-Spannungssonde unter einem Fluoreszenzmikroskop (100-faches Objektiv) zur Qualitätskontrolle, bevor Sie Liganden hinzufügen oder Zellen plattieren. Abbildung und Quantifizierung der durchschnittlichen Hintergrundintensität im Cy3B-Kanal einer DNA-Hairpin-Spannungssondenoberfläche aus mindestens 5 verschiedenen Positionen und 3 Replikationen. Halten Sie die Aufnahmebedingungen für die Bildgebung konstant, damit dieser Wert als zuverlässiger Marker für die Oberflächenqualität und die Sondendichte verwendet werden kann (Abbildung 4C).
HINWEIS: Quantifizieren Sie die Anzahl der DNA-Stränge pro Gold-Nanopartikel und die Anzahl der Gold-Nanopartikel pro μm2 bei den ersten Malen der Oberflächenpräparation gemäß Literatur12, die als weiterer zuverlässiger Marker für die Oberflächenqualität verwendet werden kann. - Legen Sie ~4 x 104 - 10 x 104 Zellen auf jedes funktionalisierte Deckglas der DNA-Spannungssonde und lassen Sie sie bei Raumtemperatur ~15 Minuten lang anheften und verteilen.
- Wenn die Zellen auf die DNA-Haarnadel-Spannungssonden aufgetragen werden und sich auszubreiten beginnen, bilden Sie die Fluoreszenzsignale ab, die im Cy3B-Kanal mit dem 100x-Objektiv erzeugt werden (Abbildung 3I).
- Nachdem die Zellen begonnen haben, ein Echtzeit-Spannungssignal auf der Oberfläche der DNA-Haarnadel-Spannungssonde im Cy3B-Kanal zu erzeugen, nehmen Sie Bilder sowohl im Cy3B- als auch im Atto647N-Kanal auf (die TIRF-Mikroskopie liefert ein besseres Signal-Rausch-Verhältnis als die Epifluoreszenz). Anschließend wird der Atto647N-Strang in einer Endkonzentration von 200 nM zur mechanisch selektiven Hybridisierung in die Bildgebungskammern gegeben.
- Entfernen Sie nach 10 Minuten Inkubationszeit schnell und vorsichtig den Puffer mit dem fluoreszierenden Atto647N-Verriegelungsstrang und ersetzen Sie ihn durch frische Hank's balancierte Salze. Bild in den Kanälen Cy3B und Atto647N und bestimmen Sie den Korrelationskoeffizienten von Pearson mit der Fidschi-Software15.
- Führen Sie zu dem Zeitpunkt, der für die Untersuchung von Interesse ist, einen nicht fluoreszierenden Verriegelungsstrang in die Zellen in der Bildgebungskammer ein, um das Spannungssignal zu speichern. Bereiten Sie das Locking-Strangmaterial (100 μM) vor und geben Sie es in einer Endkonzentration von 1 μM zu den Zellen. Die Dauer der Sperre kann variieren, aber 10 Minuten ist die empfohlene Zeit.
- Erfassen Sie Zeitrafferfilme oder Endpunktbilder in Epifluoreszenz sowohl für die qualitative Spannungskartierung als auch für die quantitative Analyse nach Bedarf (Abbildung 3J und Abbildung 5).
Anmerkungen: Wenn eine Spannungsmessung zu mehreren Zeitpunkten gewünscht wird, leiten Sie das Löschen gespeicherter Spannungssignale durch Hinzufügen eines Entriegelungsstrangs ein. Um eine übermäßige Spülung zu vermeiden, wird eine höhere Endkonzentration des Entriegelungsstrangs bei 2 μM verwendet, um eine Zehenhalte-vermittelte Strangverlagerungsreaktion mit dem Verriegelungsstrang für 3 min zu initiieren, wodurch die gespeicherten Signale gelöscht werden (Abbildung 3J). Spülen Sie die überschüssigen Oligonukleotide vorsichtig mit HBSS ab. Die Oberfläche der DNA-Haarnadel-Spannungssonde und die Zellen sind bereit für eine weitere Runde der Spannungsspeicherung und -kartierung. Das Entsperren von Spannungssignalen ist nicht notwendig, wenn nur ein Zeitpunkt für die Studie von Interesse ist.
4. Datenanalyse
HINWEIS: Die Bildanalyse wird mit der Fidschi-Software durchgeführt, und die quantitative Analyse wird mit einer Analysesoftware durchgeführt.
- Korrigieren Sie jede Abweichung während der Bildaufnahme mit dem Befehl 3D-Drift korrigieren in der Registrierung im Menü Plugins .
- Entfernen Sie den Kamerahintergrund des Bildes mit dem Befehl Subtrahieren im Menü Verarbeiten .
- Bestimmen Sie den Pearson-Korrelationskoeffizienten mit der Funktion Kolokalisierung im Menü Analysieren .
- Mitteln und subtrahieren Sie den Fluoreszenzhintergrund, der von den ungeöffneten Sonden erzeugt wird, von drei verschiedenen lokalen Hintergrundregionen. Zeichnen Sie ROIs von Zellen entweder auf Bildern mit Hintergrundsubtraktion oder auf RCM-Bildern (Reflection Interference Contrast Microscopy) mit dem Freihandauswahl-Werkzeug Bild J. Messen Sie jede relevante Metrik der ROIs, z. B. integrierte Fluoreszenzintensität und Spannungsbelegung, mit dem Werkzeug " Messen " im Menü " Analysieren" (Abbildung 6).
- Exportieren Sie die Messwerte für die quantitative Analyse mit einer Analysesoftware.
- Zeichnen Sie die Daten mit einer beliebigen Analysesoftware auf.
Ergebnisse
Hier zeigen wir repräsentative Bilder zur Kontrolle der Oberflächenqualität (Abbildung 4). Eine hochwertige Oberfläche sollte einen sauberen Hintergrund im RICM-Kanal (Abbildung 4B) und eine gleichmäßige Fluoreszenzintensität im Cy3B-Kanal (Abbildung 4C) aufweisen. Bei gleichen bildgebenden Geräten und identischen Aufnahmebedingungen für die Fluoreszenzbildgebung sollte die Hintergrundfluoreszenzintensität bei der Durchfü...
Diskussion
Mit den hier vorgestellten detaillierten Verfahren kann man DNA-Haarnadel-Spannungssondensubstrate herstellen, um die von Immunzellen produzierte Rezeptorspannung zu kartieren und zu quantifizieren. Wenn Zellen auf das Substrat der DNA-Haarnadel-Spannungssonde plattiert werden, landen, heften sie sich an und breiten sich aus, während die Rezeptoren die Liganden sowohl chemisch als auch mechanisch wahrnehmen, wobei letzteres von unseren Sonden erkannt wird. In einigen Fällen kann es jedoch vorkommen, dass sich die Zelle...
Offenlegungen
Die Autoren erklären, dass kein Interessenkonflikt besteht.
Danksagungen
Diese Arbeit wurde durch die NIH Grants R01GM131099, NIH R01GM124472 und NSF CAREER 1350829 unterstützt. Wir danken der NIH Tetramer Facility für pMHC-Liganden. Diese Studie wurde zum Teil durch den Emory Comprehensive Glycomics Core unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| 3-hydroxypicolinic acid (3-HPA) | Sigma | 56197 | maldi-TOF-MS matrix |
| mPEG-SC | Biochempeg | MF001023-2K | surface prep |
| (3-Aminopropyl)triethoxysilane | Acros | AC430941000 | surface prep |
| 10x Red blood cell lysis buffer | Biolegend | 00-4333-57 | buffer |
| 8.8 nm gold nanoparticles, tannic acid | Nanocomposix | customized order | surface prep |
| Atto647N NHS ester | Sigma | 18373-1MG-F | fluorophore, oligo prep |
| Attofluor Cell Chamber, for microscopy | Thermo Fisher Scientific | A7816 | imaging |
| BD Syringes only with Luer-Lok | BD bioscience | 309657 | cells |
| biotinylated anti-mouse CD3e | ebioscience | 13-0031-82 | antibody/ligand |
| Biotinylated pMHC ovalbumin (SIINFEKL) | NIH Tetramer Core Facility at Emory University | NA | antibody/ligand |
| bovine serum albumin | Sigma | 735078001 | block non-specific interactions |
| Cell strainers | Biologix | 15-1100 | cells |
| Coverslip Mini-Rack, teflon | Thermo Fisher Scientific | C14784 | surface prep |
| Cy3B NHS ester | GE Healthcare | PA63101 | fluorophore, oligo prep |
| Dulbecco's phosphate-buffered saline (DPBS) | Corning | 21-031-CM | buffer |
| ethanol | Sigma | 459836 | surface prep |
| Hank’s balanced salts (HBSS) | Sigma | H8264 | buffer |
| hydrogen peroxide | Sigma | H1009 | surface prep |
| LA-PEG-SC | Biochempeg | HE039023-3.4K | surface prep |
| Midi MACS (LS) startup kit | Miltenyi Biotec | 130-042-301 | cells |
| mouse CD8+ T cell isolation kit | Miltenyi Biotec | 130-104-075 | cells |
| Nanosep MF centrifugal devices | Pall laboratory | ODM02C35 | oligo prep |
| No. 2 round glass coverslips | VWR | 48382-085 | surface prep |
| NTA-SAM | Dojindo Molecular Technologies | N475-10 | surface prep |
| P2 gel | Bio-rad | 1504118 | oligo prep |
| sufuric acid | EMD Millipore Corporation | SX1244-6 | surface prep |
| Sulfo-NHS acetate | Thermo Fisher Scientific | 26777 | surface prep |
| Equipment | |||
| Agilent AdvanceBio Oligonucleotide C18 column, 4.6 x 150 mm, 2.7 μm | 653950-702 | oligonucleotide preparation | |
| Barnstead Nanopure water purifying system | Thermo Fisher | water | |
| CFI Apo 100× NA 1.49 objective | Nikon | Microscopy | |
| Cy5 cube | CHROMA | Microscopy | |
| evolve electron multiplying charge coupled device (EMCCD) | Photometrics | Microscopy | |
| High-performance liquid chromatography | Agilent 1100 | oligonucleotide preparation | |
| Intensilight epifluorescence source | Nikon | Microscopy | |
| Matrix-assisted laser desorption/ionization time-of-flight mass spectrometer (MALDI-TOF-MS) | Voyager STR | oligonucleotide preparation | |
| Nanodrop 2000 UV-Vis Spectrophotometer | Thermo Fisher | oligonucleotide preparation | |
| Nikon Eclipse Ti inverted microscope | Nikon | Microscopy | |
| Nikon Perfect Focus System | Nikon | Microscopy | |
| NIS Elements software | Nikon | Microscopy | |
| quad band TIRF 405/488/561/647 cube | CHROMA | Microscopy | |
| RICM cube | CHROMA | Microscopy | |
| TIRF launcher with 488 nm (50 mW), 561 nm (50 mW), and 640 nm | Coherent | Microscopy | |
| TRITC cube | CHROMA | Microscopy | |
| oligo name | 5' modification / 3' modification | sequence (5' to 3') | Use |
| 15mer amine locking strand | 5' modification: no modification 3' modification: /3AmMO/ | AAA AAA CAT TTA TAC CCT ACC TA | locking real-time tension signal |
| 15mer Atto647N locking strand | 5' modification: Atto647N 3' modification: /3AmMO/ | AAA AAA CAT TTA TAC CCT ACC TA | locking real-time tension signal |
| 15mer non-fluoresccent locking strand | 5' modification: no modification 3' modification: no modification | A AAA AAC ATT TAT AC | locking real-time tension signal for quantitative analysis |
| 4.7 pN hairpin strand | 5' modification: no modification 3' modification: no modification | GTGAAATACCGCACAGATGCGT TTGTATAAATGTTTTTTTCATTTAT ACTTTAAGAGCGCCACGTAGCC CAGC | hairpin probe |
| amine ligand strand | 5' modification: /5AmMC6/ 3' modification: /3Bio/ | CGCATCTGTGCG GTA TTT CAC TTT | hairpin probe |
| BHQ2 anchor strand | 5' modification: /5ThiolMC6-D/ 3' modification: /3BHQ_2/ | TTTGCTGGGCTACGTGGCGCTCTT | hairpin probe |
| Cy3B ligand strand | 5' modification: Cy3B 3' modification: /3Bio/ | CGCATCTGTGCG GTA TTT CAC TTT | hairpin probe |
| unlocking strand | 5' modification: no modification 3' modification: no modification | TAG GTA GGG TAT AAA TGT TTT TTT C | unlocking accumulated tension signal |
Referenzen
- Dustin, M. L. T-cell activation through immunological synapses and kinapses. Immunological Reviews. 221 (1), 77-89 (2008).
- Spillane, K. M., Tolar, P. B cell antigen extraction is regulated by physical properties of antigen-presenting cells. Journal of Cell Biology. 216 (1), 217-230 (2017).
- Feng, Y., et al. Mechanosensing drives acuity of αβ T-cell recognition. Proceedings of the National Academy of Sciences. 114 (39), 8204-8213 (2017).
- Hong, J., et al. A TCR mechanotransduction signaling loop induces negative selection in the thymus. Nature Immunology. 19 (12), 1379-1390 (2018).
- Basu, R., et al. Cytotoxic T cells use mechanical force to potentiate target cell killing. Cell. 165 (1), 100-110 (2016).
- Bashour, K. T., et al. CD28 and CD3 have complementary roles in T-cell traction forces. Proceedings of the National Academy of Sciences. 111 (6), 2241-2246 (2014).
- Ma, V. P. Y., Salaita, K. DNA nanotechnology as an emerging tool to study mechanotransduction in living systems. Small. 15 (26), 1900961 (2019).
- Liu, Y., Yehl, K., Narui, Y., Salaita, K. Tension sensing nanoparticles for mechano-imaging at the living/nonliving interface. Journal of the American Chemical Society. 135 (14), 5320-5323 (2013).
- Glazier, R., et al. DNA mechanotechnology reveals that integrin receptors apply pN forces in podosomes on fluid substrates. Nature Communications. 10 (1), 1-13 (2019).
- Galior, K., Liu, Y., Yehl, K., Vivek, S., Salaita, K. Titin-based nanoparticle tension sensors map high-magnitude integrin forces within focal adhesions. Nano Letters. 16 (1), 341-348 (2016).
- Zhang, Y., Ge, C., Zhu, C., Salaita, K. DNA-based digital tension probes reveal integrin forces during early cell adhesion. Nature Communications. 5, 5167 (2014).
- Liu, Y., et al. DNA-based nanoparticle tension sensors reveal that T-cell receptors transmit defined pN forces to their antigens for enhanced fidelity. Proceedings of the National Academy of Sciences. 113 (20), 5610-5615 (2016).
- Zhang, Y., et al. Platelet integrins exhibit anisotropic mechanosensing and harness piconewton forces to mediate platelet aggregation. Proceedings of the National Academy of Sciences. 115 (2), 325-330 (2018).
- Huang, J., et al. The kinetics of two-dimensional TCR and pMHC interactions determine T-cell responsiveness. Nature. 464 (7290), 932-936 (2010).
- Ma, R., et al. DNA probes that store mechanical information reveal transient piconewton forces applied by T cells. Proceedings of the National Academy of Sciences. 116 (34), 16949-16954 (2019).
- Hui, E., et al. T cell costimulatory receptor CD28 is a primary target for PD-1-mediated inhibition. Science. 355 (6332), 1428-1433 (2017).
- Whitley, K. D., Comstock, M. J., Chemla, Y. R. Elasticity of the transition state for oligonucleotide hybridization. Nucleic Acids Research. 45 (2), 547-555 (2016).
- Brockman, J. M., et al. Live-cell super-resolved PAINT imaging of piconewton cellular traction forces. Nature Methods. 17 (10), 1018-1024 (2020).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten