Bu içeriği görüntülemek için JoVE aboneliği gereklidir. Oturum açın veya ücretsiz deneme sürümünü başlatın.
Method Article
Bağışıklık Hücreleri Tarafından Geçici Piconewton Reseptör Kuvvetlerini Haritalamak için DNA Gerilim Probları
Bu Makalede
Özet
Bu makalede, bağışıklık hücreleri tarafından uygulanan reseptör kuvvetlerini görüntülemek için DNA tabanlı gerilim problarını kullanmak için ayrıntılı bir protokol açıklanmaktadır. Bu yaklaşım, reseptör kuvvetlerini gerçek zamanlı olarak >4.7pN'yi haritalayabilir ve zaman içinde kuvvetleri entegre edebilir.
Özet
İki komşu hücre arasındaki kavşakta ve hücreler ile hücre dışı matris arasındaki kavşakta iletilen mekanik kuvvetler, gelişimden immünolojiye kadar birçok süreci düzenlemek için kritik öneme sahiptir. Bu nedenle, bu kuvvetleri moleküler ölçekte incelemek için araçlar geliştirmek kritik öneme sahiptir. Grubumuz, hücreler tarafından üretilen ve belirli ligandlara iletilen kuvvetleri ölçmek ve görselleştirmek için bir dizi moleküler gerilim sensörü geliştirdi. Moleküler gerilim sensörlerinin en hassas sınıfı, nükleik asit sap-döngü saç tokalarından oluşur. Bu sensörler, DNA saç tokalarının kuvvet altında mekanik olarak uzamasını ve açılmasını bildirmek için florofor-söndürücü çiftleri kullanır. DNA saç tokası gerginlik sensörleri ile ilgili bir zorluk, gerginliğin sona ermesi üzerine hızlı saç tokası ile geri dönüşümlü olmaları ve bu nedenle geçici kuvvetlerin kaydedilmesinin zor olmasıdır. Bu makalede, mekanik bilginin "depolanmasını" sağlamak için "kilitlenebilen" ve yeniden katlanması önlenebilen DNA gerilim sensörlerini hazırlama protokollerini açıklayacağız. Bu, kilidi kaldıran tamamlayıcı nükleik asitlerin eklenmesiyle daha sonra "silinebilen" oldukça geçici pikonewton kuvvetlerinin kaydedilmesine izin verir. Gerçek zamanlı gerilim haritalaması ve mekanik bilgi depolama arasında geçiş yapma yeteneği, T hücreleri tarafından bağışıklık fonksiyonlarının bir parçası olarak yaygın olarak kullanılan zayıf, kısa ömürlü ve daha az bol miktarda kuvvet ortaya çıkarır.
Giriş
Bağışıklık hücreleri, hedef hücrelerin yüzeylerini antijenler için sürekli tarayarak ve tarayarak, yüzeylerini 1,2 saplayarak patojenlere ve kanser hücrelerine karşı savunurlar. Antijen tanıma, hedef hücrelerin yüzeyinde eksprese edilen T hücre reseptörü (TCR) ile peptid-majör histokompatibilite kompleksi MHC (pMHC) kompleksi arasındaki bağlanma ile başlatılır. TCR-pMHC tanıma iki mobil hücre arasındaki kavşakta meydana geldiğinden, uzun zamandır mekanik kuvvetler yaşadığından şüphelenilmektedir. Dahası, bu, TCR kuvvetlerinin 3,4 işlevine katkıda bulunduğunu öne süren TCR aktivasyonunun mekanosensör modeline yol açtı. Mekanik kuvvetlerin T hücresi fonksiyonuna ne zaman, nerede ve nasıl katkıda bulunduğunu anlamak için, T hücreleri tarafından iletilen moleküler kuvvetleri görselleştirmek için araçlar geliştirmek zorunludur. Geleneksel olarak, çekiş kuvveti mikroskobu (TFM) ve mikrosütun dizileri gibi yöntemler, hücresel kuvvetleri araştırmak için kullanılır 5,6. Bununla birlikte, TFM ve mikrosütun dizilerinin kuvvet duyarlılığı nanonewton (nN) ölçeğindedir ve bu nedenle hücre reseptörleri tarafından iletilen moleküler pikonewton (pN) kuvvetlerini incelemek için genellikle yetersizdir7. Tespit için kuvveti ve uzamsal çözünürlüğü iyileştirmek için laboratuvarımız başlangıçta polietilen glikol (PEG) polimerleri7 kullanılarak sentezlenen moleküler gerilim problarının geliştirilmesine öncülük etti. Moleküler gerilim probları, bir florofor ve söndürücü ile çevrili uzatılabilir bir moleküler "yay" dan (PEG, protein, DNA) oluşur ve bir yüzeye sabitlenir. Probun ucuna uygulanan kuvvetler, florofor ve söndürücüyü ayırarak genişlemesine yol açar ve böylece güçlü bir floresan sinyali üretir (Şekil 1A)8,9,10.
Son on yılda, nükleik asitler11, proteinler10 vepolimerler 8'den yapılmış yay elementleri ile farklı moleküler gerilim probları sınıflarından oluşan bir kütüphane geliştirdik. Bunlar arasında, DNA tabanlı gerilim probları, birkaç pN'den ~ 20 pN11'e kadar kolayca ayarlanabilen en yüksek sinyal-gürültü oranını ve en yüksek kuvvet hassasiyetini sağlar. Bu gerçek zamanlı DNA gerilim problarını, fibroblastlar, kanser hücreleri, trombositler ve bağışıklık hücreleri11,12,13 dahil olmak üzere birçok farklı hücre tipi tarafından üretilen moleküler kuvvetleri incelemek için kullandık. Bu makalede, geleneksel floresan mikroskobu kullanılarak moleküler reseptör kuvvetlerini pN kuvvet çözünürlüğü ile haritalamak için DNA gerilim problarını bir yüzey üzerinde sentezlemek ve birleştirmek için kullanılan protokoller anlatılacaktır. Mevcut prosedür, floresan muhabiri tanıtmak için nükleik asitte kimyasal modifikasyonlar içerirken (Şekil 1B), modifikasyon ve saflaştırma adımlarının çoğunun özel DNA sentez şirketlerine dış kaynaklı olabileceğini belirtmek önemlidir. Bu nedenle, DNA gerilim probları teknolojisi kolaydır ve daha geniş hücre biyolojisi ve mekanobiyoloji toplulukları için erişilebilirdir.
Kısaca, DNA gerilim sensörlerini monte etmek için, bir DNA saç tokası bir koldaki floresan ligand ipliğine ve diğer koldaki bir söndürücü çapa ipliğine hibridize edilir ve daha sonra bir cam substrat üzerinde hareketsiz hale getirilir (Şekil 1C, gerçek zamanlı gerginlik). Mekanik kuvvetin yokluğunda, saç tokası kapatılır ve böylece floresan söndürülür. Bununla birlikte, uygulanan mekanik kuvvet F1/2'den (% 50'lik bir açılma olasılığına yol açan dengedeki kuvvet) daha büyük olduğunda, saç tokası mekanik olarak erir ve bir floresan sinyali üretilir.
Gerçek zamanlı DNA gerilim sensörüne dayanarak, birikmiş kuvvetleri haritalamak için protokolleri de açıklıyoruz; bu, bağışıklık hücreleri üzerindeki reseptörler ile doğal ligandları arasındaki etkileşimleri incelemek için özellikle yararlıdır. Bunun nedeni, bağışıklık reseptörlerinin sıklıkla kısa ömürlü bağlar göstermesidir 3,14. Biriken kuvvetler, tercihen açık DNA saç tokalarına bağlanan ve mekanik çekme olaylarıyla ilişkili floresan sinyallerinin depolanmasına izin veren bir "kilitleme" ipliği kullanılarak görüntülenir (Şekil 1C, kilitli gerilim). Kilitleme ipliği, saç tokasının mekanik olarak indüklenen erimesi üzerine açığa çıkan şifreli bir bağlama bölgesini bağlamak ve saç tokasının yeniden katlanmasını engelleyerek saç tokasını açık durumda kilitlemek, böylece gerginlik sinyalini depolamak ve birikmiş bir gerilim haritası oluşturmak için tasarlanmıştır. Dahası, kilitleme ipliği, tam tamamlayıcısı olan "kilit açma" ipliği ile ayak parmağı aracılı bir iplik yer değiştirme reaksiyonu sağlayan sekiz nükleotid ayak parmağı ile tasarlanmıştır. Kilit açma telinin eklenmesiyle, bağlı kilitleme teli saç tokası yapısından sıyrılır, depolanan gerginlik sinyali silinir ve saç tokasını gerçek zamanlı duruma geri döndürür.
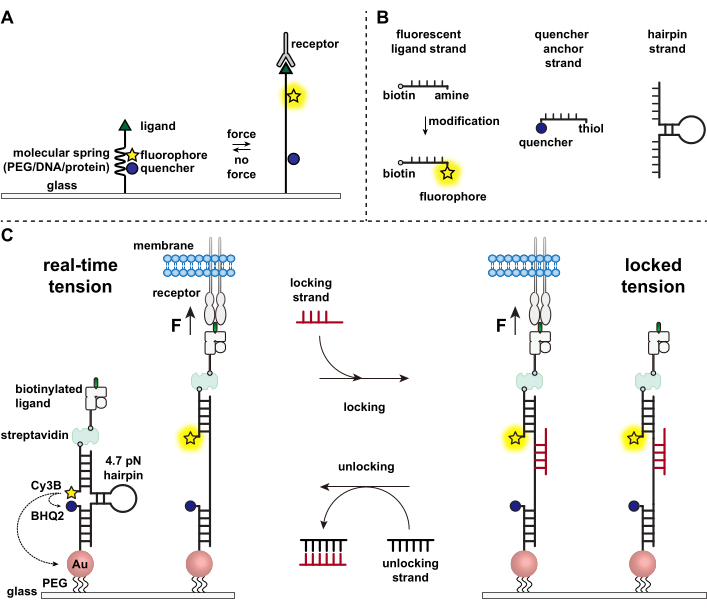
Resim 1: Son teknoloji moleküler gerilim problarının şeması . (A) Gerçek zamanlı moleküler gerilim probunun genel tasarımı, (B) DNA tabanlı gerilim probu yapısı için iplikçikler ve (C) DNA tabanlı gerilim probları ve bunların gerçek zamanlı durum ile kilitli durum arasında geçiş yapması. Bu şeklin daha büyük bir versiyonunu görmek için lütfen buraya tıklayın.
Ana protokol dört ana bölümden oluşur - oligonükleotid hazırlama, yüzey hazırlama, görüntüleme ve veri analizi. Bu protokol, laboratuvarımız ve diğerleri tarafından naif ve aktive edilmiş OT-1 CD8 + T hücreleri, OT-II CD4 + hücreleri ve hibridomlarda başarıyla gösterilmiştir ve T hücre reseptörü, programlanmış hücre ölüm reseptörü (PD1) ve lenfosit fonksiyonu ile ilişkili antijen 1 (LFA-1) kuvvetleri dahil olmak üzere farklı bağışıklık hücresi reseptörlerini sorgulamak için uygulanabilir. OT-1 CD8+ naif T hücreleri bu yazıda örnek hücre çizgisi olarak kullanılmıştır.
Protokol
OT-1 transgenik fareler, Emory Üniversitesi'ndeki Hayvan Kaynakları Bölümü Tesisi'nde yer almaktadır. Tüm deneyler Kurumsal Hayvan Bakımı ve Kullanımı Komitesi (IACUC) protokolü kapsamında onaylanmış ve gerçekleştirilmiştir.
1. Oligonükleotid preparatı
- Ligand iplikçik DNA'sını suda çözün (tüm protokol boyunca kullanılan 18.2 MΩ direnci). Vorteks yapın ve çözeltiyi bir masa üstü santrifüj ile döndürün. Suyun hacmini, son konsantrasyon 1 mM olacak şekilde ayarlayın. 260 nm'deki absorbansı ölçmek için bir nanodamla spektrofotometresi kullanarak konsantrasyonu doğrulayın ve oligonükleotidin yok olma katsayısına dayanarak nihai konsantrasyonu belirleyin.
NOT: Ligand ipliği, florofor ile konjuge olmak ve biyotinile ligandı sunmak için her terminusta, 5' amin ve 3' biotin'de bir modifikasyona sahiptir. Ligand ipliğindeki amin grubunun bir florofor ile konjuge edilmesi gerekir. Cy3B boyası, yüksek parlaklığı ve fotostabilitesi nedeniyle bu konjugasyon için kullanılır, ancak genellikle ticari olarak sunulmaz ve şirket içi konjugasyon gerektirir. Buna göre, aşağıdaki bölümde aminler ve NHS ester boyaları arasındaki konjugasyon açıklanmaktadır. Nükleik asit modifikasyonu için tesislere veya kaynaklara erişimi olmayan son kullanıcılar için, modifiye nükleik asitler bunun yerine Alexa ve Atto boya ailesi gibi parlak ve fotolanabilir boyalar sunan özel DNA sentezi satıcılarından satın alınabilir. - 10x PBS ve 1 M NaHCO3 çözeltileri hazırlayın. 1 mM amin ligand iplikçik çözeltisinin 10 μL'sini (10 nmol) 10 μL 10x PBS, 10 μL 1 M NaHCO3 ve 60 μLH2O ile karıştırın. Kullanımdan hemen önce 50 μg Cy3B NHS esterini 10 μL DMSO içinde çözün ve toplam 100 μL reaksiyon hacmi için karışıma ekleyin. Oda sıcaklığında gece boyunca 1 saat veya 4 °C reaksiyona girmesine izin verin.
- Amin kilitleme ipliğini Atto647N NHS esteri ile konjuge ederek Atto647N kilitleme ipliğini hazırlayın. 10x PBS ve 1 M NaHCO3 çözeltisi hazırlayın. 1 mM amin kilitleme ipliği çözeltisinin 10 μL'sini (10 nmol) 10 μL 10x PBS, 10 μL 1 M NaHCO3 ve 60 μLH2O ile karıştırın. Kullanımdan hemen önce 50 μg Atto647N NHS esterini 10 μL DMSO içinde çözün ve toplam 100 μL'lik bir reaksiyon hacmi için karışıma ekleyin. Oda sıcaklığında gece boyunca 1 saat veya 4 °C reaksiyona girmesine izin verin.
- Reaksiyonlardan sonra, P2 tuzdan arındırma jeli filtrasyonu ile yan ürünleri, fazla boyayı ve tuzları çıkarın. Reaksiyon karışımınıH2O ile toplam 300 μL hacme kadar seyreltin, bu da sonraki HPLC saflaştırma adımı için uygundur. Santrifüjlü bir cihaza 650 μL hidratlanmış P2 jeli ekleyin ve 1 dakika boyunca 18.000 x g'de döndürün. Cihazın altındaki sıvıyı çıkarın, reaksiyon karışımını P2 jeli içeren kolona ekleyin, 1 dakika boyunca 18.000 x g'de döndürün ve reaksiyon karışımını cihazın altında toplayın.
NOT: P2 jel,H2O ile kullanılmadan önce en az 4 saat nemlendirilmelidir. - Tuzdan arındırılmış reaksiyon karışımını, oligonükleotid saflaştırması için belirlenmiş bir C18 sütunu kullanarak,H2O'da A: 0,1 M TEAA ve B: ACN çözücüsü ile 50 dakika boyunca 0,5 mL / dak akış hızında 50 dakika boyunca% 10-100 B doğrusal gradyan elüsyonu için mobil faz olarak HPLC ile saflaştırın. Tuzdan arındırılmış reaksiyon karışımını saflaştırma için ters fazlı HPLC'ye 500 μL'lik bir enjeksiyon döngüsü ile enjekte edin. DNA için bir absorbans zirvesi (260 nm) ve florofor için bir absorbans zirvesi (Cy3B için 560 nm ve Atto647N için 647 nm) olan ürünü toplayın ve bunları bir gece boyunca vakumlu bir santrifüj yoğunlaştırıcıda kurutun (bkz. Şekil 2A).
- Kurutulmuş oligo-boya ürününü 100 μL suda yeniden oluşturun. Nanodrop spektrofotometresi ile Cy3B ligand ipliğinin ve Atto647N kilitleme ipliğinin konsantrasyonunu belirleyin. Boya etiketleme oranının 1:1'e yakın olduğundan emin olun. Oligonükleotid konsantrasyonunu belirlerken gerekirse boyanın 260 nm emilimi için doğrudur.
- MALDI-TOF-MS numune hazırlama için %0,1 TFA ile %50 ACN/S2O ve 1-5 μM'de ürünün 0,5 μL'sini kullanarak 5 mg/mL amonyum sitrat kullanarak substrat olarak 3-HPA kullanarak saflaştırılmış ürünü MALDI-TOF-MS ile doğrulayın. Örnek bir kütle spektrumu Şekil 2B'de bulunabilir.
- Saç tokası telini ve söndürücü ankraj telini suda çözün ve stok çözeltilerinin konsantrasyonunun 50 ila 100 μM arasında olduğundan emin olun.
NOT: Saç tokası teli değiştirilmemiştir ve doğrudan bir satıcıdan özel olarak sentezlenebilir. Ankraj ipliği bir tiol ankraj grubuna ve bir söndürücü BHQ2'ye sahiptir ve doğrudan bir satıcıdan özel olarak sentezlenebilir. - Aliquot tüm oligonükleotidler. Kısa süreli kullanım ve depolama için, bu oligonükleotidleri 4 ° C'de saklayın. Uzun süreli depolama için, dondurun ve -20 ° C'de tutun. Bu noktada, tüm oligonükleotidler DNA gerilim probu montajı için hazırdır.
NOT: Tekrarlanan donma-çözülme döngüleri oligonükleotidler için sorunlu değildir.
2. Yüzey hazırlığı
NOT: DNA saç tokası gerginlik probu substratlarının hazırlanması iki gün sürer. DNA saç tokası gerginlik probu cam kapaklar üzerinde işlevselleştirilecektir.
- 1. Gün
- 25 mm'lik kapak fişlerini 50 mL'lik bir beherde bir politetrafloroetilen rafa yerleştirin. Her raf 8 adede kadar kapak kapağı tutabilir. Kapakları üç kez suya batırarak durulayın.
- Raf ve kapakları içeren beherin içine suyla karıştırılmış 40 mL 1:1 oranında (v:v) etanol çözeltisi ekleyin ve beheri bir parafin filmi kullanarak kapatın.
- Kapakları temizlemek için beheri bir ultrasonik temizleyicide (çalışma frekansı 35 KHz) 15 dakika boyunca sonikleştirin. Sonikasyondan sonra, sıvıyı atın ve beheri rafla durulayın ve kalan organik çözücüyü çıkarmak için en az 6 kez suyla örtün.
- Sülfürik asit ve hidrojen peroksiti 3: 1 oranında karıştırarak taze Piranha çözeltisi hazırlayın. 40 mL Piranha çözeltisi yapmak için, önce temiz bir 50 mL beher içine 30 mL sülfürik asit ekleyin ve ardından yavaşça 10 mL H2O2ekleyin. Piranha çözeltisi,H2O2'nineklenmesiyle hızla ısınacak ve kabarcıklanacaktır. Piranha'yı bir cam pipetin ucunu kullanarak yavaşça karıştırın.
- Daha sonra, kapakları tutan rafı, aşındırma için hafifçe karıştırılmış Piranha çözeltisi içeren behere aktarın (Şekil 3A). Piranha çözeltisinin hidroksilasyon yapmasına izin verin ve kapakları oda sıcaklığında 30 dakika boyunca temizleyin. Piranha aşındırmasından sonra, çelik veya politetrafloroetilen cımbız kullanarak rafı su ile temiz bir 50 mL behere aktarın ve en az 6 kez suyla tekrar durulayın.
DİKKAT: Büyük miktarda organik madde Piranha çözeltisi ile kuvvetli bir şekilde reaksiyona girebilir ve patlamaya neden olabilir. Dikkatli olun ve her zaman bir duman davlumbazında Piranha çözeltisi ile çalışın. Bir laboratuvar önlüğü, eldiven ve güvenlik gözlüğü taktığınızdan emin olun. Taze Piranha çözeltisini asla kapalı bir kapta saklamayın.
NOT: Hidrojen peroksit-sülfürik asit oranı 1:2'nin (v:v) altında tutulmalı ve asla 1:1'i geçmemelidir. Rafı Piranha çözeltisine kapaklarla batırırken, yavaşça ve dikkatlice çözeltiye yerleştirin. Çözeltiyi aşındırmadan hemen sonra atmayın, çünkü hala aktif ve sıcaktır. Asit atık kabına dökmeden önce gece boyunca beherde bırakın. - Kapağı tutan rafı, suyu çıkarmak için 40 mL etanol içeren 50 mL'lik bir beherin içine daldırın. Etanol atılır ve suyun çıkarıldığından emin olmak için 3 kez tekrarlayın.
- Daha sonra rafı, oda sıcaklığında 1 saat boyunca kapaklardaki -OH ile reaksiyona girmek için 40 mL etanol içinde% 3 aminopropil trietoksi silan (APTES) (v / v) içine daldırın (Şekil 3B).
NOT: Etanol aseton ile değiştirilebilir. - Yüzeyleri 40 mL etanol içine batırarak 6 kez durulayın, ardından 80 ° C'de fırında 20 dakika kurutun. Soğuduktan sonra, kurutulmuş amin modifiye edilmiş kapak fişlerini ileride kullanmak üzere -20 ° C'de saklayın (6 aya kadar).
- 10 cm çapında plastik Petri kaplarının alt iç tarafını parafin film ile örtün. Parafin film, kapak kaymalarının Petri kabının içinde kaymasını önler ve bir sonraki işlevsellik adımları için çözümün kapak kaymalarında kalmasına yardımcı olur. Soğutulmuş amin modifiye edilmiş kapak fişlerini Petri kaplarına yerleştirin. İşlevselleştirilecek taraf yukarı bakmalıdır.
- Kapak fişlerindeki amin gruplarını değiştirmek için, her bir kapak kapağına 0,1 M NaHCO3'te 300 μL %0,5 lipoik asit PEG NHS (LA-PEG-SC) ve %2,5 w/v mPEG NHS (mPEG-SC) ekleyin ve oda sıcaklığında 1 saat inkübe edin (Şekil 3C). Her 25 mm'lik kapak kayması için 1,5 mg LA-PEG-SC ve 7,5 mg mPEG-SC ağırlığındadır. NHS reaktiflerini yüzeylere eklemeden hemen önce çözün, çünkü oda sıcaklığında sulu çözelti içinde kısa bir yarı ömre (~ 10 dakika) sahiptirler. Reaksiyondan sonra, yüzeyleri 3 kez su ile durulayın.
NOT: NHS reaksiyonu gece boyunca 4 ° C'de gerçekleştirilebilir. NHS reaktifleri, 4-6 saat civarında olan 4 ° C'de hidrolizden önce daha uzun yarı ömre sahiptir. Bu, üç günlük bir yüzey hazırlama prosedürü ile sonuçlanacaktır. - Bir dizi "sandviç" kapak fişine 1 mg/mL sülfo-NHS asetat içeren 100 μL 0,1 M NaHCO3 ekleyin (aralarında reaksiyon tamponu bulunan birbirine bakan iki kapak kayması). Pasivasyonun en az 30 dakika boyunca gerçekleşmesine izin verin. Reaktiften tasarruf etmek için, bu adım 50 μL 1 mg / mL sülfo-NHS asetat ile yapılabilir. Pasivasyondan sonra üç kez su ile durulayın.
- Her bir kapak kaymasına 0.5 mL altın nanopartikül (AuNP, 8.8 nm, tanik asit, 0.05 mg / mL) ekleyin ve oda sıcaklığında 30 dakika inkübe edin (Şekil 3D). Reaktifi kurtarmak için, bu adım iki kapak fişini sandviçleyerek de yapılabilir. Altın nanopartiküllerin toplanmasını önlemek için sistemde önceki adımlardan tuz bulunmadığından emin olun. Bu adımdan sonra kapakları kurumaya bırakmayın.
- Bu arada, DNA gerilim problarını oluşturan 4.7 pN saç tokası, Cy3B ligand ipliği ve BHQ2 ankraj ipliği, bir PCR tüpünde 300 nM'de 1 M NaCl'de 1.1: 1: 1 oranında bir oranda inşa edilir. Çözeltiyi 5 dakika boyunca 95 ° C'ye kadar ısıtarak telleri tavlayın, daha sonra bir termal döngüleyicide sıcaklığı 30 dakika boyunca 20 ° C'ye düşürerek kademeli olarak soğutun.
- Altın nanopartiküllerle 30 dakikalık inkübasyondan sonra kapakları üç kez suyla durulayın. BHQ2 ankraj ipliği ile Cy3B ligand ipliği arasındaki oranı 10:1 yapmak için tavlanmış DNA çözeltisine ek BHQ2 ankraj ipliği (100 μM stoktan) ekleyin. Bu noktada, DNA çözeltisi 300 nM gerilim probu yapısı ve 2.7 μM BHQ2 ipliği içermelidir. "Sandviç" yapmak için iki kapak fişi başına 100 μL ekleyin (Şekil 3E).
- Petri kabına (kapaklardan uzak) ıslak bir laboratuvar mendil topunu dikkatlice yerleştirin ve çözeltinin kurumasını önlemek için kabı parafin filmle kapatın. Çanağı folyo ile örtün ve gece boyunca 4 ° C'de inkübe edin.
- 2. Gün
- Kapaklardaki fazla probları 1x PBS ile yıkayın. Epifloresan mikroskop altında DNA gerilim probu yüzey kalitesini kontrol edin.
- 1x PBS'de 40 μg / mL streptavidin hazırlayın ve oda sıcaklığında 30 dakika boyunca kapak fişleri üzerinde inkübe edin (Şekil 3F). Genellikle, 25 mm'lik bir kapak kayması için 100 μL yeterlidir. Fazla miktarda streptavidini yıkamak için inkübasyondan sonra PBS ile 3 kez durulayın.
- 1x PBS'de 40 μg/mL biyotinile antikor/ligand hazırlayın. Sandviç başına 50-100 μL ekleyin ve oda sıcaklığında 30 dakika inkübe edin (Şekil 3G). Fazla miktarda biyotinile antikor/ligandı temizlemek için inkübasyondan sonra PBS ile üç kez durulayın.
- Temiz görüntüleme odalarını yüzeyleri olan bir şekilde dikkatlice monte edin. Odaları sıkarken yüzeyler kolayca çatlayabilir. Görüntüleme odalarına 0,5-1 mL Hank dengeli tuz çözeltisi (HBSS) ekleyin ve hücrelerle görüntülemeye hazır tutun (Şekil 3H).
3. Görüntüleme hücresi reseptör kuvvetleri
- HBSS'de ilgilenilen bağışıklık hücrelerini 1-2 x 106 hücre / mL'de hazırlayın.
NOT: OT-1 CD8+ naif hücreler bu makalede örnek olarak kullanılmıştır. OT-1 CD8+ naif T hücrelerini, üreticinin talimatlarını izleyerek MACS fare CD8+ T hücre izolasyon kitini bir MACS ayırıcı ile kullanarak kurban edilen farelerin dalaklarından arındırın. Manyetik tükenen antikor kokteylinin bağlı olduğu CD8 + T olmayan hücreleri çıkararak CD8 + T hücrelerini izole edin ve zenginleştirin. HBSS'deki saflaştırılmış OT-1 CD8 + naif T hücrelerini 2 x 106 hücre / mL'de tekrar askıya alın ve kullanmadan önce buzda tutun. - Ligandlar veya kaplama hücreleri eklemeden önce kalite kontrolü için DNA saç tokası gerilim probu yüzeyinin kalitesini bir floresan mikroskobu (100x hedef) altında kontrol edin. Bir DNA saç tokası gerilim probu yüzeyinin Cy3B kanalındaki ortalama arka plan yoğunluğunu en az 5 farklı konumdan ve 3 kopyadan görüntüleyin ve ölçün. Görüntüleme elde etme koşullarını tutarlı tutun, böylece bu değer yüzey kalitesi ve prob yoğunluğunun güvenilir bir belirteci olarak kullanılabilir (Şekil 4C).
NOT: Altın nanopartikül başına DNA iplikçik sayısını ve μm2 başına altın nanopartikül sayısını, yüzey kalitesinin bir başka güvenilir belirteci olarak kullanılabilecek literatür12'ye göre yüzey hazırlığının ilk birkaç kez sayın. - Her DNA gerilim probu üzerine ~ 4 x 10 4 - 10 x 104 hücreli plaka işlevsel kapak kayması ve oda sıcaklığında ~ 15 dakika boyunca yapışmalarını ve yayılmalarını sağlar.
- Hücreler DNA saç tokası gerilim problarına kaplanıp yayılmaya başladıkça, Cy3B kanalında üretilen floresan sinyallerini 100x hedefiyle görüntüleyin (Şekil 3I).
- Hücreler, Cy3B kanalındaki DNA saç tokası gerilim probu yüzeyinde gerçek zamanlı gerilim sinyali üretmeye başladıktan sonra, hem Cy3B hem de Atto647N kanallarında görüntüler elde edin (TIRF mikroskopisi, epifloresandan daha iyi sinyal-gürültü oranı sağlar). Daha sonra, mekanik olarak seçici hibridizasyon için görüntüleme odalarına 200 nM'lik son konsantrasyonda Atto647N ipliği ekleyin.
- 10 dakikalık inkübasyondan sonra, floresan Atto647N kilitleme ipliğini içeren tamponu hızlı ve nazik bir şekilde çıkarın ve taze Hank'in dengeli tuzlarıyla değiştirin. Hem Cy3B hem de Atto647N kanallarındaki görüntü tekrar ve Pearson'un Fiji yazılımı ile korelasyon katsayısını belirlemek15.
- Araştırmanın ilgilendiği zaman noktasında, gerilim sinyalini depolamak için görüntüleme odasındaki hücrelere floresan olmayan kilitleme ipliği ekleyin. Kilitleme ipliği stoğu (100 μM) hazırlayın ve hücrelere 1 μM'lik son bir konsantrasyonda ekleyin. Kilitleme süresi değişebilir, ancak önerilen süre 10 dakikadır.
- Gerektiğinde hem kalitatif gerilim haritalaması hem de kantitatif analiz için epifloresanda hızlandırılmış filmler veya son nokta görüntüleri elde edin (Şekil 3J ve Şekil 5).
NOT: Birden fazla zaman noktasında gerilim ölçümü isteniyorsa, bir kilit açma ipliği ekleyerek depolanan gerilim sinyallerinin silinmesini başlatın. Aşırı durulamayı önlemek için, 2 μM'de daha yüksek bir nihai kilit açma ipliği konsantrasyonu, saklanan sinyalleri silen kilitleme ipliği ile 3 dakika boyunca ayak parmağı aracılı bir iplik yer değiştirme reaksiyonu başlatmak için kullanılır (Şekil 3J). Fazla oligonükleotidleri HBSS ile nazikçe durulayın. DNA saç tokası gerilim probu yüzeyi ve hücreler başka bir gerilim depolama ve haritalama turu için hazırdır. Gerilim sinyallerinin kilidinin açılması, çalışmada yalnızca bir zaman noktası ilgi çekiciyse gerekli değildir.
4. Veri analizi
NOT: Görüntü analizi Fiji yazılımı kullanılarak, nicel analiz ise analiz yazılımı kullanılarak gerçekleştirilir.
- Eklentiler menüsünün altındaki Kayıt'taki Doğru 3B sürüklenme komutuyla görüntü alımı sırasında herhangi bir sapmayı düzeltin .
- İşlem menüsünün altındaki Çıkar komutuyla görüntünün kamera arka planını kaldırın.
- Pearson'ın korelasyon katsayısını, Analiz menüsü altındaki Eşyerelleştirme işleviyle belirleyin.
- Açılmamış problar tarafından üretilen floresan arka planın ortalamasını alın ve üç farklı yerel arka plan bölgesinden çıkarın. Image J Freehand Selections aracıyla arka plandan çıkarılmış görüntülere veya RICM (yansıma paraziti kontrast mikroskobu) görüntülerine hücrelerin YG'lerini çizin. YG'lerin ilgilendiği herhangi bir metriği ölçün, örneğin Analiz menüsü altındaki Ölçüm aracını kullanarak entegre floresan yoğunluğunu ve gerilim doluluğunu ölçün (Şekil 6).
- Analiz yazılımı ile nicel analiz için ölçümleri dışa aktarın.
- Verileri herhangi bir analiz yazılımıyla çizin.
Sonuçlar
Burada temsili yüzey kalite kontrol görüntülerini gösteriyoruz (Şekil 4). Yüksek kaliteli bir yüzey, RICM kanalında temiz bir arka plana (Şekil 4B) ve Cy3B kanalında (Şekil 4C) eşit floresan yoğunluğuna sahip olmalıdır. Aynı görüntüleme ekipmanı ve aynı floresan görüntüleme edinme koşullarıyla, DNA probları ile deneyler yapılırken arka plan floresan yoğunluğu her seferinde tutarlı ve tekrarlanabilir...
Tartışmalar
Burada verilen ayrıntılı prosedürlerle, bağışıklık hücreleri tarafından üretilen reseptör gerginliğini haritalamak ve ölçmek için DNA saç tokası gerginlik probu substratları hazırlanabilir. Hücreler DNA saç tokası gerilim probu substratına kaplandığında, reseptörler ligandları hem kimyasal hem de mekanik olarak algıladıkça iner, bağlanır ve yayılırlar, ikincisi problarımız tarafından tespit edilir. Bununla birlikte, bazı durumlarda hücreler yayılmayabilir (?...
Açıklamalar
Yazarlar çıkar çatışması olmadığını beyan ederler.
Teşekkürler
Bu çalışma NIH Grants R01GM131099, NIH R01GM124472 ve NSF CAREER 1350829 tarafından desteklenmiştir. pMHC ligandları için NIH Tetramer Tesisi'ne teşekkür ederiz. Bu çalışma, kısmen, Emory Comprehensive Glycomics Core tarafından desteklenmiştir.
Malzemeler
| Name | Company | Catalog Number | Comments |
| 3-hydroxypicolinic acid (3-HPA) | Sigma | 56197 | maldi-TOF-MS matrix |
| mPEG-SC | Biochempeg | MF001023-2K | surface prep |
| (3-Aminopropyl)triethoxysilane | Acros | AC430941000 | surface prep |
| 10x Red blood cell lysis buffer | Biolegend | 00-4333-57 | buffer |
| 8.8 nm gold nanoparticles, tannic acid | Nanocomposix | customized order | surface prep |
| Atto647N NHS ester | Sigma | 18373-1MG-F | fluorophore, oligo prep |
| Attofluor Cell Chamber, for microscopy | Thermo Fisher Scientific | A7816 | imaging |
| BD Syringes only with Luer-Lok | BD bioscience | 309657 | cells |
| biotinylated anti-mouse CD3e | ebioscience | 13-0031-82 | antibody/ligand |
| Biotinylated pMHC ovalbumin (SIINFEKL) | NIH Tetramer Core Facility at Emory University | NA | antibody/ligand |
| bovine serum albumin | Sigma | 735078001 | block non-specific interactions |
| Cell strainers | Biologix | 15-1100 | cells |
| Coverslip Mini-Rack, teflon | Thermo Fisher Scientific | C14784 | surface prep |
| Cy3B NHS ester | GE Healthcare | PA63101 | fluorophore, oligo prep |
| Dulbecco's phosphate-buffered saline (DPBS) | Corning | 21-031-CM | buffer |
| ethanol | Sigma | 459836 | surface prep |
| Hank’s balanced salts (HBSS) | Sigma | H8264 | buffer |
| hydrogen peroxide | Sigma | H1009 | surface prep |
| LA-PEG-SC | Biochempeg | HE039023-3.4K | surface prep |
| Midi MACS (LS) startup kit | Miltenyi Biotec | 130-042-301 | cells |
| mouse CD8+ T cell isolation kit | Miltenyi Biotec | 130-104-075 | cells |
| Nanosep MF centrifugal devices | Pall laboratory | ODM02C35 | oligo prep |
| No. 2 round glass coverslips | VWR | 48382-085 | surface prep |
| NTA-SAM | Dojindo Molecular Technologies | N475-10 | surface prep |
| P2 gel | Bio-rad | 1504118 | oligo prep |
| sufuric acid | EMD Millipore Corporation | SX1244-6 | surface prep |
| Sulfo-NHS acetate | Thermo Fisher Scientific | 26777 | surface prep |
| Equipment | |||
| Agilent AdvanceBio Oligonucleotide C18 column, 4.6 x 150 mm, 2.7 μm | 653950-702 | oligonucleotide preparation | |
| Barnstead Nanopure water purifying system | Thermo Fisher | water | |
| CFI Apo 100× NA 1.49 objective | Nikon | Microscopy | |
| Cy5 cube | CHROMA | Microscopy | |
| evolve electron multiplying charge coupled device (EMCCD) | Photometrics | Microscopy | |
| High-performance liquid chromatography | Agilent 1100 | oligonucleotide preparation | |
| Intensilight epifluorescence source | Nikon | Microscopy | |
| Matrix-assisted laser desorption/ionization time-of-flight mass spectrometer (MALDI-TOF-MS) | Voyager STR | oligonucleotide preparation | |
| Nanodrop 2000 UV-Vis Spectrophotometer | Thermo Fisher | oligonucleotide preparation | |
| Nikon Eclipse Ti inverted microscope | Nikon | Microscopy | |
| Nikon Perfect Focus System | Nikon | Microscopy | |
| NIS Elements software | Nikon | Microscopy | |
| quad band TIRF 405/488/561/647 cube | CHROMA | Microscopy | |
| RICM cube | CHROMA | Microscopy | |
| TIRF launcher with 488 nm (50 mW), 561 nm (50 mW), and 640 nm | Coherent | Microscopy | |
| TRITC cube | CHROMA | Microscopy | |
| oligo name | 5' modification / 3' modification | sequence (5' to 3') | Use |
| 15mer amine locking strand | 5' modification: no modification 3' modification: /3AmMO/ | AAA AAA CAT TTA TAC CCT ACC TA | locking real-time tension signal |
| 15mer Atto647N locking strand | 5' modification: Atto647N 3' modification: /3AmMO/ | AAA AAA CAT TTA TAC CCT ACC TA | locking real-time tension signal |
| 15mer non-fluoresccent locking strand | 5' modification: no modification 3' modification: no modification | A AAA AAC ATT TAT AC | locking real-time tension signal for quantitative analysis |
| 4.7 pN hairpin strand | 5' modification: no modification 3' modification: no modification | GTGAAATACCGCACAGATGCGT TTGTATAAATGTTTTTTTCATTTAT ACTTTAAGAGCGCCACGTAGCC CAGC | hairpin probe |
| amine ligand strand | 5' modification: /5AmMC6/ 3' modification: /3Bio/ | CGCATCTGTGCG GTA TTT CAC TTT | hairpin probe |
| BHQ2 anchor strand | 5' modification: /5ThiolMC6-D/ 3' modification: /3BHQ_2/ | TTTGCTGGGCTACGTGGCGCTCTT | hairpin probe |
| Cy3B ligand strand | 5' modification: Cy3B 3' modification: /3Bio/ | CGCATCTGTGCG GTA TTT CAC TTT | hairpin probe |
| unlocking strand | 5' modification: no modification 3' modification: no modification | TAG GTA GGG TAT AAA TGT TTT TTT C | unlocking accumulated tension signal |
Referanslar
- Dustin, M. L. T-cell activation through immunological synapses and kinapses. Immunological Reviews. 221 (1), 77-89 (2008).
- Spillane, K. M., Tolar, P. B cell antigen extraction is regulated by physical properties of antigen-presenting cells. Journal of Cell Biology. 216 (1), 217-230 (2017).
- Feng, Y., et al. Mechanosensing drives acuity of αβ T-cell recognition. Proceedings of the National Academy of Sciences. 114 (39), 8204-8213 (2017).
- Hong, J., et al. A TCR mechanotransduction signaling loop induces negative selection in the thymus. Nature Immunology. 19 (12), 1379-1390 (2018).
- Basu, R., et al. Cytotoxic T cells use mechanical force to potentiate target cell killing. Cell. 165 (1), 100-110 (2016).
- Bashour, K. T., et al. CD28 and CD3 have complementary roles in T-cell traction forces. Proceedings of the National Academy of Sciences. 111 (6), 2241-2246 (2014).
- Ma, V. P. Y., Salaita, K. DNA nanotechnology as an emerging tool to study mechanotransduction in living systems. Small. 15 (26), 1900961 (2019).
- Liu, Y., Yehl, K., Narui, Y., Salaita, K. Tension sensing nanoparticles for mechano-imaging at the living/nonliving interface. Journal of the American Chemical Society. 135 (14), 5320-5323 (2013).
- Glazier, R., et al. DNA mechanotechnology reveals that integrin receptors apply pN forces in podosomes on fluid substrates. Nature Communications. 10 (1), 1-13 (2019).
- Galior, K., Liu, Y., Yehl, K., Vivek, S., Salaita, K. Titin-based nanoparticle tension sensors map high-magnitude integrin forces within focal adhesions. Nano Letters. 16 (1), 341-348 (2016).
- Zhang, Y., Ge, C., Zhu, C., Salaita, K. DNA-based digital tension probes reveal integrin forces during early cell adhesion. Nature Communications. 5, 5167 (2014).
- Liu, Y., et al. DNA-based nanoparticle tension sensors reveal that T-cell receptors transmit defined pN forces to their antigens for enhanced fidelity. Proceedings of the National Academy of Sciences. 113 (20), 5610-5615 (2016).
- Zhang, Y., et al. Platelet integrins exhibit anisotropic mechanosensing and harness piconewton forces to mediate platelet aggregation. Proceedings of the National Academy of Sciences. 115 (2), 325-330 (2018).
- Huang, J., et al. The kinetics of two-dimensional TCR and pMHC interactions determine T-cell responsiveness. Nature. 464 (7290), 932-936 (2010).
- Ma, R., et al. DNA probes that store mechanical information reveal transient piconewton forces applied by T cells. Proceedings of the National Academy of Sciences. 116 (34), 16949-16954 (2019).
- Hui, E., et al. T cell costimulatory receptor CD28 is a primary target for PD-1-mediated inhibition. Science. 355 (6332), 1428-1433 (2017).
- Whitley, K. D., Comstock, M. J., Chemla, Y. R. Elasticity of the transition state for oligonucleotide hybridization. Nucleic Acids Research. 45 (2), 547-555 (2016).
- Brockman, J. M., et al. Live-cell super-resolved PAINT imaging of piconewton cellular traction forces. Nature Methods. 17 (10), 1018-1024 (2020).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır