É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Sondas de Tensão de DNA para Mapear as Forças Transitórias do Receptor de Piconewton por Células Imunes
Neste Artigo
Resumo
Este artigo descreve um protocolo detalhado para o uso de sondas de tensão baseadas em DNA para obter imagens das forças receptoras aplicadas por células imunes. Essa abordagem pode mapear forças receptoras >4.7pN em tempo real e pode integrar forças ao longo do tempo.
Resumo
As forças mecânicas transmitidas na junção entre duas células vizinhas e na junção entre as células e a matriz extracelular são críticas para a regulação de muitos processos que vão desde o desenvolvimento até a imunologia. Portanto, desenvolver ferramentas para estudar essas forças em escala molecular é fundamental. Nosso grupo desenvolveu um conjunto de sensores de tensão molecular para quantificar e visualizar as forças geradas pelas células e transmitidas a ligantes específicos. A classe mais sensível de sensores de tensão molecular é composta por grampos de alça de haste de ácido nucleico. Esses sensores usam pares fluoróforo-quencher para relatar a extensão mecânica e o desdobramento de pinos de cabelo de DNA sob força. Um desafio com os sensores de tensão hairpin de DNA é que eles são reversíveis com redobramento rápido do hairpin após o término da tensão e, portanto, as forças transitórias são difíceis de registrar. Neste artigo, descrevemos os protocolos de preparação de sensores de tensão de DNA que podem ser "travados" e impedidos de redobrar para permitir o "armazenamento" de informações mecânicas. Isso permite o registro de forças de piconewton altamente transitórias, que podem ser posteriormente "apagadas" pela adição de ácidos nucleicos complementares que removem a fechadura. Essa capacidade de alternar entre o mapeamento de tensão em tempo real e o armazenamento de informações mecânicas revela forças fracas, de curta duração e menos abundantes, que são comumente empregadas pelas células T como parte de suas funções imunológicas.
Introdução
As células imunes defendem-se contra patógenos e células cancerígenas rastejando e varrendo continuamente as superfícies das células-alvo em busca de antígenos, fixando sua superfície 1,2. O reconhecimento do antígeno é iniciado após a ligação entre o receptor de células T (TCR) e o complexo MHC (pMHC) do complexo peptídeo-principal de histocompatibilidade expresso na superfície das células-alvo. Como o reconhecimento do TCR-pMHC ocorre na junção entre duas células móveis, há muito se suspeita que ele experimente forças mecânicas. Além disso, isso levou ao modelo mecanosensor de ativação do TCR, que sugere que as forças do TCR contribuem para sua função 3,4. Para entender quando, onde e como as forças mecânicas contribuem para o funcionamento das células T, é imperativo desenvolver ferramentas para visualizar as forças moleculares transmitidas pelas células T. Tradicionalmente, métodos como a microscopia de força de tração (MFT) e os arranjos de micropilares são utilizados para investigar as forças celulares 5,6. No entanto, a sensibilidade à força dos arranjos de MTF e micropilares está na escala de nanonewton (nN) e, portanto, muitas vezes é insuficiente para estudar as forças moleculares de piconewton (pN) transmitidas por receptores celulares7. Para melhorar a força e a resolução espacial para detecção, nosso laboratório foi pioneiro no desenvolvimento de sondas de tensão molecular, que foram inicialmente sintetizadas usando polímeros de polietilenoglicol (PEG)7. As sondas de tensão molecular são compostas por uma "mola" molecular extensível (PEG, proteína, DNA) flanqueada por um fluoróforo e quencher e estão ancoradas em uma superfície. Forças aplicadas ao término da sonda levam à sua extensão, separando o fluoróforo e o quencher, gerando um forte sinal de fluorescência (Figura 1A)8,9,10.
Na última década, desenvolvemos uma biblioteca de diferentes classes de sondas de tensão molecular com elementos de mola feitos de ácidos nucléicos11, proteínas10 e polímeros8. Dentre estas, as sondas de tensão baseadas em DNA fornecem a maior relação sinal/ruído e a maior sensibilidade à força, que é facilmente sintonizada de alguns pN até ~20 pN11. Usamos essas sondas de tensão de DNA em tempo real para estudar as forças moleculares geradas por diversos tipos celulares, incluindo fibroblastos, células cancerosas, plaquetas e células imunes11,12,13. Este manuscrito descreverá protocolos para sintetizar e montar sondas de tensão de DNA em uma superfície para mapear forças de receptores moleculares com resolução de força de pN usando um microscópio de fluorescência convencional. Embora o procedimento atual inclua modificações químicas no ácido nucleico para introduzir o repórter fluorescente (Figura 1B), é importante notar que muitas das etapas de modificação e purificação podem ser terceirizadas para empresas de síntese de DNA personalizadas. Portanto, a tecnologia de sondas de tensão de DNA é fácil e acessível para as comunidades mais amplas de biologia celular e mecanobiologia.
Resumidamente, para montar sensores de tensão de DNA, um hairpin de DNA é hibridizado a uma fita fluorescente ligante em um braço e uma fita de âncora quencher no outro braço e, em seguida, imobilizado em um substrato de vidro (Figura 1C, tensão em tempo real). Na ausência de força mecânica, o hairpin é fechado e, assim, a fluorescência é apagada. No entanto, quando a força mecânica aplicada é maior que o F1/2 (a força em equilíbrio que leva a uma probabilidade de 50% de desdobramento), o hairpin derrete mecanicamente e um sinal fluorescente é gerado.
Com base no sensor de tensão do DNA em tempo real, também descrevemos protocolos para mapear forças acumuladas, o que é particularmente útil para estudar interações entre receptores em células imunes e seu ligante natural. Isso ocorre porque os receptores imunes frequentemente apresentam ligações de curta duração 3,14. As forças acumuladas são imageadas usando uma fita de "travamento" que se liga preferencialmente aos pinos de cabelo de DNA abertos e permite o armazenamento de sinais de fluorescência associados a eventos de tração mecânica (Figura 1C, tensão bloqueada). O fio de bloqueio é projetado para ligar um local de ligação críptico que é exposto após o derretimento mecanicamente induzido do hairpin e travar o hairpin no estado aberto, bloqueando a redobração do hairpin, armazenando assim o sinal de tensão e gerando um mapa de tensão acumulado. Além disso, a fita de travamento é projetada com um suporte de oito nucleotídeos, que permite uma reação de deslocamento de fita mediada por dedos dos pés com seu complemento completo, a fita de "destravamento". Com a adição do fio de desbloqueio, o fio de travamento preso é retirado da construção do hairpin, apagando o sinal de tensão armazenado e redefinindo o hairpin de volta ao estado em tempo real.
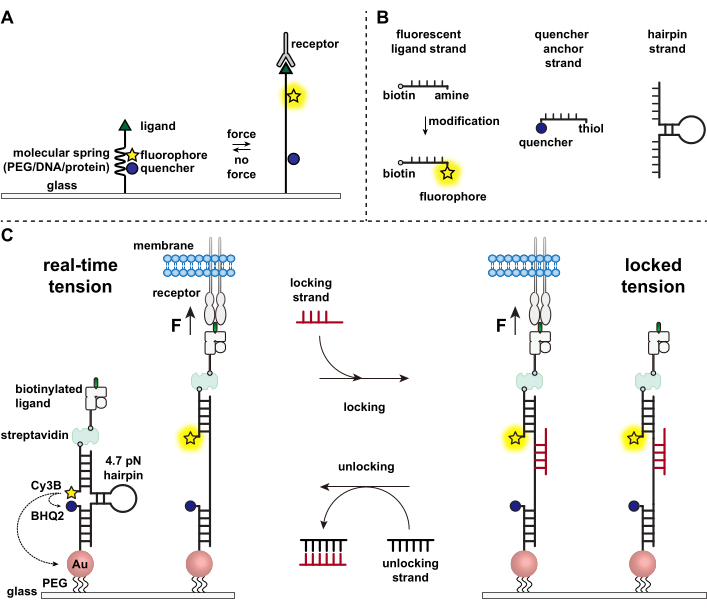
Figura 1: Esquema das sondas de tensão molecular de última geração. (A) Projeto geral de sonda de tensão molecular em tempo real, (B) Fitas para a construção de sonda de tensão baseada em DNA e (C) Sondas de tensão baseadas em DNA projetadas e sua alternância entre o estado em tempo real e o estado bloqueado. Clique aqui para ver uma versão maior desta figura.
O protocolo principal consiste em quatro seções principais - preparação de oligonucleotídeos, preparação de superfície, imagem e análise de dados. Este protocolo foi demonstrado com sucesso por nosso laboratório e outros em células T CD8+ OT-1 virgens e ativadas, células CD4+ OT-II, bem como hibridomas, e pode ser aplicado para interrogar diferentes receptores de células imunes, incluindo receptor de células T, receptor de morte celular programada (PD1) e forças do antígeno 1 associado à função linfocitária (LFA-1). Células T virgens de OT-1 CD8+ são usadas como um exemplo de linhagem celular neste artigo.
Protocolo
Os camundongos transgênicos OT-1 estão alojados na Divisão de Recursos Animais da Universidade de Emory. Todos os experimentos foram aprovados e realizados sob o protocolo da Comissão Institucional de Cuidados e Uso de Animais (IACUC).
1. Preparação de oligonucleotídeos
- Dissolver o DNA da fita ligante em água (resistividade de 18,2 MΩ, utilizada durante todo o protocolo). Vórtice e gire a solução com uma centrífuga de mesa. Ajuste o volume de água de tal forma que a concentração final seja de 1 mM. Validar a concentração usando um espectrofotômetro de nanogotas para medir a absorbância a 260 nm e determinar a concentração final com base no coeficiente de extinção do oligonucleotídeo.
NOTA: A fita ligante tem uma modificação em cada terminal, 5' amina e 3' biotina, para conjugar com o fluoróforo e apresentar o ligante biotinilado. O grupo amina na fita ligante precisa ser conjugado com um fluoróforo. O corante Cy3B é usado para esta conjugação devido ao seu alto brilho e fotoestabilidade, mas geralmente não é oferecido comercialmente e requer conjugação interna. Assim, a seção a seguir descreve a conjugação entre aminas e corantes ésteres do NHS. Para usuários finais que não têm acesso a instalações ou recursos para modificação de ácido nucleico, os ácidos nucleicos modificados podem ser comprados de fornecedores personalizados de síntese de DNA que oferecem corantes brilhantes e fotoestáveis, como a família de corantes Alexa e Atto. - Prepare soluções 10x PBS e 1 M NaHCO3 . Misturar 10 μL da solução de fita ligante de amina 1 mM (10 nmol) com 10 μL de 10x PBS, 10 μL de 1 M NaHCO3 e 60 μL de H2O. Dissolver 50 μg de Cy3B NHS ester em 10 μL de DMSO imediatamente antes do uso e adicionar à mistura para um volume total de reação de 100 μL. Adicione Cy3B NHS ester por último. Deixar reagir à temperatura ambiente durante 1 h ou 4 °C durante a noite.
- Prepare o fio de bloqueio Atto647N conjugando o fio de bloqueio de amina com o éster Atto647N NHS. Preparar 10x PBS e 1 M NaHCO3 solução. Misturar 10 μL da solução de fita de bloqueio de amina 1 mM (10 nmol) com 10 μL de 10x PBS, 10 μL de 1 M NaHCO3 e 60 μL de H2O. Dissolver 50 μg de éster Atto647N NHS em 10 μL de DMSO imediatamente antes do uso e adicionar à mistura para um volume total de reação de 100 μL. Adicione Atto647N NHS ester por último. Deixar reagir à temperatura ambiente durante 1 h ou 4 °C durante a noite.
- Após as reações, remover os subprodutos, o excesso de corante e os sais por filtração em gel dessalinizador P2. Diluir a mistura de reacção com H2O para um volume total de 300 μL, adequado para a etapa subsequente de purificação por HPLC. Adicionar 650 μL de gel P2 hidratado a um dispositivo centrífugo e girar para baixo a 18.000 x g por 1 min. Retire o líquido na parte inferior do dispositivo, adicione a mistura de reação à coluna que contém o gel P2, gire para baixo a 18.000 x g por 1 min e colete a mistura de reação na parte inferior do dispositivo.
NOTA: O gel P2 deve ser hidratado pelo menos 4 h antes do uso com H2O. - Purificar a mistura de reação dessalinizada com HPLC usando uma coluna C18 designada para purificação de oligonucleotídeos, com solvente A: 0,1 M TEAA em H2O e B: ACN como fase móvel para uma eluição de gradiente linear 10-100% B durante 50 min a uma taxa de fluxo de 0,5 mL/min. Injetar a mistura de reação dessalgada em HPLC de fase reversa com um circuito de injeção de 500 μL para purificação. Recolher o produto que tem um pico de absorbância para o ADN (260 nm) e um pico de absorbância para o fluoróforo (560 nm para Cy3B e 647 nm para Atto647N) e secá-los num concentrador centrífugo a vácuo durante a noite (ver Figura 2A).
- Reconstituir o produto oligocorante seco em 100 μL de água. Determinar a concentração da fita ligante Cy3B e da fita de bloqueio Atto647N com o espectrofotômetro de nanogotas. Certifique-se de que a proporção de rotulagem do corante esteja próxima de 1:1. Corrigir a absorbância de 260 nm do corante, se necessário, ao determinar a concentração de oligonucleotídeos.
- Validar o produto purificado com MALDI-TOF-MS usando 3-HPA como substrato em 50% ACN/H2O com TFA 0,1% e 5 mg/mL de citrato de amônio usando 0,5 μL do produto a 1-5 μM para preparação de amostra de MALDI-TOF-MS. Um exemplo de espectro de massa pode ser encontrado na Figura 2B.
- Dissolva o fio hairpin e o fio de âncora quencher em água e certifique-se de que a concentração das soluções estoque esteja entre 50 e 100 μM.
NOTA: O fio hairpin não é modificado e pode ser sintetizado diretamente personalizado a partir de um fornecedor. A fita âncora tem um grupo de ancoragem tiol e um quencher BHQ2 e pode ser sintetizada diretamente de um fornecedor. - Alíquota todos os oligonucleotídeos. Para utilização e armazenamento a curto prazo, conservar estes oligonucleótidos a 4 °C. Para armazenamento a longo prazo, congelar e mantê-los a -20 °C. Neste ponto, todos os oligonucleotídeos estão prontos para a montagem da sonda de tensão do DNA.
NOTA: Ciclos repetidos de congelamento-descongelamento não são problemáticos para oligonucleotídeos.
2. Preparação da superfície
NOTA: A preparação de substratos de sonda de tensão de DNA hairpin leva dois dias. A sonda de tensão do pino de DNA será funcionalizada sobre lamínulas de vidro.
- Dia 1
- Coloque as lamínulas de 25 mm em um rack de politetrafluoretileno em um copo de 50 mL. Cada rack pode conter até 8 lamínulas. Enxágue as lamínulas submergindo em água três vezes.
- Adicionar 40 ml de uma solução de etanol na proporção 1:1 (v:v) misturada com água ao copo que contém a cremalheira e as lamínulas e selar o copo utilizando uma película de parafina.
- Sonicate o copo por 15 min em um limpador ultrassônico (frequência de operação 35 KHz) para limpar as lamínulas. Após a sonicação, descarte o líquido e enxágue o copo com a cremalheira e cubra com água pelo menos 6 vezes para remover qualquer solvente orgânico restante.
- Preparar solução fresca de piranha misturando ácido sulfúrico e peróxido de hidrogênio na proporção de 3:1. Para fazer 40 mL de solução de piranha, adicione 30 mL de ácido sulfúrico a um copo limpo de 50 mL primeiro e, em seguida, adicione lentamente 10 mL de H 2 O2. A solução de piranha aquece rapidamente e borbulha após a adição do H 2 O2. Misture suavemente a piranha usando a extremidade de uma pipeta de vidro.
- Em seguida, transfira o rack que contém as lamínulas para o copo contendo solução de Piranha suavemente misturada para condicionamento ácido (Figura 3A). Deixe a solução de Piranha hidroxilato e limpe as lamínulas por 30 min à temperatura ambiente. Após a gravação da piranha, transfira o rack com uma pinça de aço ou politetrafluoretileno para um copo limpo de 50 mL com água e enxágue novamente com água pelo menos 6 vezes.
CUIDADO: Grandes quantidades de substâncias orgânicas podem reagir vigorosamente com a solução de piranha e podem causar explosão. Tenha cuidado e trabalhe sempre com solução de piranha em uma coifa de fumaça. Certifique-se de usar jaleco, luvas e óculos de segurança. Nunca guarde a solução fresca de piranha em um recipiente lacrado.
NOTA: A relação peróxido de hidrogênio/ácido sulfúrico deve ser mantida abaixo de 1:2 (v:v) e nunca deve exceder 1:1. Ao submergir o rack com lamínulas em solução Piranha, coloque-as na solução de forma lenta e cuidadosa. Não descarte a solução imediatamente após o condicionamento térmico, pois ela ainda está ativa e quente. Deixe-o no copo durante a noite antes de despejá-lo no recipiente de resíduos ácidos. - Mergulhe o rack que prende as lamínulas em um copo de 50 mL com 40 mL de etanol para remover a água. Descarte o etanol e repita 3 vezes para garantir que a água foi removida.
- Em seguida, imergir o rack em aminopropiltrietoxi-silano (APTES) a 3% (v/v) em 40 mL de etanol para reagir com o -OH nas lamínulas por 1 h à temperatura ambiente (Figura 3B).
NOTA: O etanol pode ser substituído por acetona. - Enxaguar as superfícies 6 vezes submergindo-as em 40 mL de etanol e, em seguida, secar em estufa a 80 °C por 20 min. Após o arrefecimento, guarde as lamínulas secas modificadas com amina a -20 °C para utilização futura (até 6 meses).
- Cubra a parte interna inferior de placas de Petri plásticas de 10 cm de diâmetro com filme de parafina. A película de parafina evita que as lamínulas deslizem para dentro da placa de Petri e ajuda a manter a solução para os próximos passos da funcionalização nas lamínulas. Coloque as tampas resfriadas modificadas com amina nas placas de Petri. O lado a ser funcionalizado deve estar voltado para cima.
- Para modificar os grupos amina nas lamínulas, adicionar 300 μL de ácido lipóico a 0,5% p/v PEG NHS (LA-PEG-SC) e 2,5% p/v mPEG NHS (mPEG-SC) em NaHCO3 0,1 M em cada lamínula e incubar por 1 h à temperatura ambiente (Figura 3C). Para cada lamínula de 25 mm, pesar 1,5 mg de LA-PEG-SC e 7,5 mg de mPEG-SC. Dissolva os reagentes do NHS imediatamente antes de adicionar às superfícies, pois eles têm uma meia-vida curta (~10 min) em solução aquosa à temperatura ambiente. Após a reação, enxaguar as superfícies 3 vezes com água.
NOTA: A reação do NHS pode ser realizada a 4 °C durante a noite. Os reagentes do NHS têm meia-vida mais longa antes da hidrólise a 4 °C, que é em torno de 4-6 h. Isso resultará em um procedimento de preparação de superfície de três dias. - Adicionar 100 μL de NaHCO3 0,1 M contendo 1 mg/mL de acetato de sulfo-NHS a um conjunto de lamínulas "sanduíche" (duas lamínulas voltadas uma para a outra com tampão de reação no meio). Permitir que a passivação ocorra por pelo menos 30 min. Para economizar reagente, essa etapa poderia ser feita com 50 μL de 1 mg/mL de acetato de sulfo-NHS. Enxaguar com água três vezes após a passivação.
- Adicionar 0,5 mL de nanopartículas de ouro (AuNP, 8,8 nm, ácido tânico, 0,05 mg/mL) a cada lamínula e incubar por 30 min à temperatura ambiente (Figura 3D). Para salvar o reagente, essa etapa pode ser feita com duas tampas também. Certifique-se de que nenhum sal esteja presente no sistema das etapas anteriores para evitar a agregação de nanopartículas de ouro. Não deixe as tampas secar após esta etapa.
- Enquanto isso, pré-hibridizar 4,7 pN hairpin, fita de ligante Cy3B e fita de âncora BHQ2 que formam as sondas de tensão de DNA constroem em uma proporção de 1,1:1:1 em 1 M NaCl a 300 nM em um tubo de PCR. Recozer os fios aquecendo a solução até 95 °C durante 5 minutos e, em seguida, arrefecer gradualmente diminuindo a temperatura para 20 °C durante 30 minutos num termociclador.
- Enxaguar as lamínulas com água três vezes após 30 min de incubação com nanopartículas de ouro. Adicione uma fita de ancoragem BHQ2 adicional (a partir de 100 μM de estoque) à solução de DNA recozido para fazer a relação entre a fita de ancoragem BHQ2 e a fita ligante Cy3B 10:1. Neste ponto, a solução de DNA deve conter 300 nM de construção de sonda de tensão e 2,7 μM de fita BHQ2. Adicionar 100 μL por duas lamínulas para fazer o "sanduíche" (Figura 3E).
- Coloque cuidadosamente uma bola de tecido de laboratório molhada na placa de Petri (longe de lamínulas) e sele a placa com filme de parafina para evitar que a solução seque. Cubra o prato com papel alumínio e incube a 4 °C durante a noite.
- Dia 2
- Lave as sondas em excesso das lamínulas com 1x PBS. Verifique a qualidade da superfície da sonda de tensão do DNA em um microscópio epifluorescente.
- Preparar 40 μg/mL de estreptavidina em 1x PBS e incubar em lamínulas por 30 min à temperatura ambiente (Figura 3F). Normalmente, 100 μL são suficientes para uma lamínula de 25 mm. Enxaguar com PBS 3 vezes após a incubação para lavar o excesso de estreptavidina.
- Preparar 40 μg/mL de anticorpo/ligante biotinilado em 1x PBS. Adicionar 50-100 μL por sanduíche e incubar por 30 min à temperatura ambiente (Figura 3G). Enxaguar com PBS três vezes após a incubação para lavar a quantidade excessiva de anticorpo/ligante biotinilado.
- Monte as câmaras de imagem limpas com superfícies cuidadosamente. As superfícies podem ser facilmente rachadas ao apertar as câmaras. Adicionar 0,5-1 mL de solução salina balanceada de Hank (HBSS) às câmaras de imagem e mantê-las prontas para aquisição de imagens com células (Figura 3H).
3. Forças de receptores celulares por imagem
- Preparar células imunes de interesse em HBSS em 1-2 x 106 células/mL.
NOTA: OT-1 CD8+ células virgens são usadas como exemplo neste artigo. Purificar células T virgens OT-1 CD8+ a partir do baço de camundongos sacrificados usando o kit de isolamento de células T CD8+ de camundongo MACS com um separador MACS seguindo as instruções do fabricante. Isolar e enriquecer as células T CD8+ removendo quaisquer células T não CD8+ às quais o coquetel de anticorpos de esgotamento magnético se ligou. Ressuspender as células T não VHS OT-1 CD8+ purificadas em HBSS a 2 x 106 células/mL e manter gelo antes do uso. - Verificar a qualidade da superfície da sonda de tensão do pino de DNA sob um microscópio de fluorescência (objetiva 100x) para controle de qualidade antes de adicionar ligantes ou plaquear células. Imagem e quantificação da intensidade média de fundo no canal Cy3B de uma superfície de sonda de tensão hairpin DNA a partir de pelo menos 5 posições diferentes e 3 réplicas. Manter as condições de aquisição das imagens consistentes para que esse valor possa ser usado como um marcador confiável da qualidade da superfície e da densidade da sonda (Figura 4C).
NOTA: Quantificar o número de fitas de DNA por nanopartícula de ouro e o número de nanopartículas de ouro por μm2 nas primeiras vezes de preparação da superfície de acordo com a literatura12, que pode ser usado como outro marcador confiável da qualidade da superfície. - Placa ~4 x 10 4 - 10 x 104 células em cada sonda de tensão de DNA funcionalizada coverslip e permitir que eles se fixem e espalhem por ~15 min à temperatura ambiente.
- À medida que as células são plaqueadas nas sondas de tensão do pino de DNA e começam a se espalhar, os sinais de fluorescência que são gerados no canal Cy3B com a objetiva de 100x (Figura 3I).
- Depois que as células começam a produzir sinal de tensão em tempo real na superfície da sonda de tensão hairpin do DNA no canal Cy3B, adquirem imagens nos canais Cy3B e Atto647N (a microscopia TIRF fornece melhor relação sinal-ruído do que a epifluorescência). Em seguida, adicionar fita Atto647N às câmaras de imagem em uma concentração final de 200 nM para hibridização mecanicamente seletiva.
- Após 10 min de incubação, remova rápida e suavemente o tampão que contém o fio de bloqueio fluorescente Atto647N e substitua por sais balanceados frescos de Hank. Imagem nos canais Cy3B e Atto647N novamente e determinação do coeficiente de correlação de Pearson com o software Fiji15.
- No momento de interesse para a investigação, introduzir fita de bloqueio não fluorescente nas células na câmara de imagem para armazenar o sinal de tensão. Preparar o caldo de fita de bloqueio (100 μM) e adicionar às células a uma concentração final de 1 μM. Pipetar suavemente para misturar. A duração do bloqueio pode variar, mas 10 min é o tempo recomendado.
- Adquira filmes de lapso de tempo ou imagens de ponto final em epifluorescência para mapeamento qualitativo de tensão e análise quantitativa, conforme necessário (Figura 3J e Figura 5).
NOTA: Se a medição de tensão em vários pontos de tempo for desejada, inicie o apagamento dos sinais de tensão armazenados pela adição de uma fita de desbloqueio. Para evitar o excesso de enxágue, uma concentração final mais alta da fita de destravamento a 2 μM é usada para iniciar uma reação de deslocamento da fita mediada pelo dedo do pé com a fita de travamento por 3 min, que apaga os sinais armazenados (Figura 3J). Enxágue suavemente o excesso de oligonucleotídeos com HBSS. A superfície da sonda de tensão do pino de cabelo do DNA e as células estão prontas para mais uma rodada de armazenamento e mapeamento de tensão. O desbloqueio de sinais de tensão não é necessário se apenas um ponto de tempo for de interesse no estudo.
4. Análise dos dados
NOTA: A análise de imagens é realizada usando o software Fiji, e a análise quantitativa é realizada usando o software de análise.
- Corrija qualquer desvio durante a aquisição de imagens com o comando Corrigir desvio 3D em Registro no menu Plug-ins .
- Remova o fundo da câmera da imagem com o comando Subtrair no menu Processo .
- Determine o coeficiente de correlação de Pearson com a função Colocalização no menu Analisar .
- Média e subtração do fundo de fluorescência produzido pelas sondas não abertas de três diferentes regiões de fundo local. Desenhe ROIs de células em imagens subtraídas em plano de fundo ou imagens RICM (microscopia de contraste de interferência de reflexão) com a ferramenta Image J Freehand Sselections . Meça qualquer métrica de interesse das ROIs, por exemplo, intensidade de fluorescência integrada e ocupação de tensão usando a ferramenta Medir no menu Analisar (Figura 6).
- Exporte as medições para análise quantitativa com software de análise.
- Plote os dados com qualquer software de análise.
Resultados
Aqui mostramos imagens representativas de controle de qualidade de superfície (Figura 4). Uma superfície de alta qualidade deve ter um fundo limpo no canal RICM (Figura 4B) e intensidade de fluorescência uniforme no canal Cy3B (Figura 4C). Com o mesmo equipamento de imagem e condições idênticas de aquisição de imagens de fluorescência, a intensidade de fluorescência de fundo deve ser consistente e reprodutível sempre ao r...
Discussão
Com os procedimentos detalhados fornecidos aqui, pode-se preparar substratos de sonda de tensão de DNA hairpin para mapear e quantificar a tensão do receptor produzida pelas células imunes. Quando as células são plaqueadas no substrato da sonda de tensão do pino de DNA, elas pousam, se ligam e se espalham à medida que os receptores detectam os ligantes química e mecanicamente, o último dos quais é detectado por nossas sondas. No entanto, em alguns casos, as células podem não se espalhar (...
Divulgações
Os autores declaram a inexistência de conflitos de interesse.
Agradecimentos
Este trabalho foi apoiado pelos NIH Grants R01GM131099, NIH R01GM124472 e NSF CAREER 1350829. Agradecemos ao NIH Tetramer Facility pelos ligantes pMHC. Este estudo foi apoiado, em parte, pelo Emory Comprehensive Glycomics Core.
Materiais
| Name | Company | Catalog Number | Comments |
| 3-hydroxypicolinic acid (3-HPA) | Sigma | 56197 | maldi-TOF-MS matrix |
| mPEG-SC | Biochempeg | MF001023-2K | surface prep |
| (3-Aminopropyl)triethoxysilane | Acros | AC430941000 | surface prep |
| 10x Red blood cell lysis buffer | Biolegend | 00-4333-57 | buffer |
| 8.8 nm gold nanoparticles, tannic acid | Nanocomposix | customized order | surface prep |
| Atto647N NHS ester | Sigma | 18373-1MG-F | fluorophore, oligo prep |
| Attofluor Cell Chamber, for microscopy | Thermo Fisher Scientific | A7816 | imaging |
| BD Syringes only with Luer-Lok | BD bioscience | 309657 | cells |
| biotinylated anti-mouse CD3e | ebioscience | 13-0031-82 | antibody/ligand |
| Biotinylated pMHC ovalbumin (SIINFEKL) | NIH Tetramer Core Facility at Emory University | NA | antibody/ligand |
| bovine serum albumin | Sigma | 735078001 | block non-specific interactions |
| Cell strainers | Biologix | 15-1100 | cells |
| Coverslip Mini-Rack, teflon | Thermo Fisher Scientific | C14784 | surface prep |
| Cy3B NHS ester | GE Healthcare | PA63101 | fluorophore, oligo prep |
| Dulbecco's phosphate-buffered saline (DPBS) | Corning | 21-031-CM | buffer |
| ethanol | Sigma | 459836 | surface prep |
| Hank’s balanced salts (HBSS) | Sigma | H8264 | buffer |
| hydrogen peroxide | Sigma | H1009 | surface prep |
| LA-PEG-SC | Biochempeg | HE039023-3.4K | surface prep |
| Midi MACS (LS) startup kit | Miltenyi Biotec | 130-042-301 | cells |
| mouse CD8+ T cell isolation kit | Miltenyi Biotec | 130-104-075 | cells |
| Nanosep MF centrifugal devices | Pall laboratory | ODM02C35 | oligo prep |
| No. 2 round glass coverslips | VWR | 48382-085 | surface prep |
| NTA-SAM | Dojindo Molecular Technologies | N475-10 | surface prep |
| P2 gel | Bio-rad | 1504118 | oligo prep |
| sufuric acid | EMD Millipore Corporation | SX1244-6 | surface prep |
| Sulfo-NHS acetate | Thermo Fisher Scientific | 26777 | surface prep |
| Equipment | |||
| Agilent AdvanceBio Oligonucleotide C18 column, 4.6 x 150 mm, 2.7 μm | 653950-702 | oligonucleotide preparation | |
| Barnstead Nanopure water purifying system | Thermo Fisher | water | |
| CFI Apo 100× NA 1.49 objective | Nikon | Microscopy | |
| Cy5 cube | CHROMA | Microscopy | |
| evolve electron multiplying charge coupled device (EMCCD) | Photometrics | Microscopy | |
| High-performance liquid chromatography | Agilent 1100 | oligonucleotide preparation | |
| Intensilight epifluorescence source | Nikon | Microscopy | |
| Matrix-assisted laser desorption/ionization time-of-flight mass spectrometer (MALDI-TOF-MS) | Voyager STR | oligonucleotide preparation | |
| Nanodrop 2000 UV-Vis Spectrophotometer | Thermo Fisher | oligonucleotide preparation | |
| Nikon Eclipse Ti inverted microscope | Nikon | Microscopy | |
| Nikon Perfect Focus System | Nikon | Microscopy | |
| NIS Elements software | Nikon | Microscopy | |
| quad band TIRF 405/488/561/647 cube | CHROMA | Microscopy | |
| RICM cube | CHROMA | Microscopy | |
| TIRF launcher with 488 nm (50 mW), 561 nm (50 mW), and 640 nm | Coherent | Microscopy | |
| TRITC cube | CHROMA | Microscopy | |
| oligo name | 5' modification / 3' modification | sequence (5' to 3') | Use |
| 15mer amine locking strand | 5' modification: no modification 3' modification: /3AmMO/ | AAA AAA CAT TTA TAC CCT ACC TA | locking real-time tension signal |
| 15mer Atto647N locking strand | 5' modification: Atto647N 3' modification: /3AmMO/ | AAA AAA CAT TTA TAC CCT ACC TA | locking real-time tension signal |
| 15mer non-fluoresccent locking strand | 5' modification: no modification 3' modification: no modification | A AAA AAC ATT TAT AC | locking real-time tension signal for quantitative analysis |
| 4.7 pN hairpin strand | 5' modification: no modification 3' modification: no modification | GTGAAATACCGCACAGATGCGT TTGTATAAATGTTTTTTTCATTTAT ACTTTAAGAGCGCCACGTAGCC CAGC | hairpin probe |
| amine ligand strand | 5' modification: /5AmMC6/ 3' modification: /3Bio/ | CGCATCTGTGCG GTA TTT CAC TTT | hairpin probe |
| BHQ2 anchor strand | 5' modification: /5ThiolMC6-D/ 3' modification: /3BHQ_2/ | TTTGCTGGGCTACGTGGCGCTCTT | hairpin probe |
| Cy3B ligand strand | 5' modification: Cy3B 3' modification: /3Bio/ | CGCATCTGTGCG GTA TTT CAC TTT | hairpin probe |
| unlocking strand | 5' modification: no modification 3' modification: no modification | TAG GTA GGG TAT AAA TGT TTT TTT C | unlocking accumulated tension signal |
Referências
- Dustin, M. L. T-cell activation through immunological synapses and kinapses. Immunological Reviews. 221 (1), 77-89 (2008).
- Spillane, K. M., Tolar, P. B cell antigen extraction is regulated by physical properties of antigen-presenting cells. Journal of Cell Biology. 216 (1), 217-230 (2017).
- Feng, Y., et al. Mechanosensing drives acuity of αβ T-cell recognition. Proceedings of the National Academy of Sciences. 114 (39), 8204-8213 (2017).
- Hong, J., et al. A TCR mechanotransduction signaling loop induces negative selection in the thymus. Nature Immunology. 19 (12), 1379-1390 (2018).
- Basu, R., et al. Cytotoxic T cells use mechanical force to potentiate target cell killing. Cell. 165 (1), 100-110 (2016).
- Bashour, K. T., et al. CD28 and CD3 have complementary roles in T-cell traction forces. Proceedings of the National Academy of Sciences. 111 (6), 2241-2246 (2014).
- Ma, V. P. Y., Salaita, K. DNA nanotechnology as an emerging tool to study mechanotransduction in living systems. Small. 15 (26), 1900961 (2019).
- Liu, Y., Yehl, K., Narui, Y., Salaita, K. Tension sensing nanoparticles for mechano-imaging at the living/nonliving interface. Journal of the American Chemical Society. 135 (14), 5320-5323 (2013).
- Glazier, R., et al. DNA mechanotechnology reveals that integrin receptors apply pN forces in podosomes on fluid substrates. Nature Communications. 10 (1), 1-13 (2019).
- Galior, K., Liu, Y., Yehl, K., Vivek, S., Salaita, K. Titin-based nanoparticle tension sensors map high-magnitude integrin forces within focal adhesions. Nano Letters. 16 (1), 341-348 (2016).
- Zhang, Y., Ge, C., Zhu, C., Salaita, K. DNA-based digital tension probes reveal integrin forces during early cell adhesion. Nature Communications. 5, 5167 (2014).
- Liu, Y., et al. DNA-based nanoparticle tension sensors reveal that T-cell receptors transmit defined pN forces to their antigens for enhanced fidelity. Proceedings of the National Academy of Sciences. 113 (20), 5610-5615 (2016).
- Zhang, Y., et al. Platelet integrins exhibit anisotropic mechanosensing and harness piconewton forces to mediate platelet aggregation. Proceedings of the National Academy of Sciences. 115 (2), 325-330 (2018).
- Huang, J., et al. The kinetics of two-dimensional TCR and pMHC interactions determine T-cell responsiveness. Nature. 464 (7290), 932-936 (2010).
- Ma, R., et al. DNA probes that store mechanical information reveal transient piconewton forces applied by T cells. Proceedings of the National Academy of Sciences. 116 (34), 16949-16954 (2019).
- Hui, E., et al. T cell costimulatory receptor CD28 is a primary target for PD-1-mediated inhibition. Science. 355 (6332), 1428-1433 (2017).
- Whitley, K. D., Comstock, M. J., Chemla, Y. R. Elasticity of the transition state for oligonucleotide hybridization. Nucleic Acids Research. 45 (2), 547-555 (2016).
- Brockman, J. M., et al. Live-cell super-resolved PAINT imaging of piconewton cellular traction forces. Nature Methods. 17 (10), 1018-1024 (2020).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados