このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
免疫細胞による一過性ピコニュートン受容体力をマッピングするためのDNA張力プローブ
要約
この論文では、DNAベースのテンションプローブを使用して免疫細胞によって加えられる受容体力を画像化するための詳細なプロトコルについて説明します。このアプローチは、受容体の力>4.7pNをリアルタイムでマッピングし、時間の経過とともに力を積分することができます。
要約
隣接する2つの細胞間の接合部および細胞と細胞外マトリックス間の接合部で伝達される機械的な力は、発生から免疫学に至るまでの多くのプロセスを調節するために重要です。したがって、分子スケールでこれらの力を研究するためのツールを開発することが重要です。私たちのグループは、細胞によって生成され、特定のリガンドに伝達される力を定量化および視覚化するための一連の分子張力センサーを開発しました。最も感度の高いクラスの分子張力センサーは、核酸ステムループヘアピンで構成されています。これらのセンサーは、蛍光色素と消光物質のペアを使用して、力によるDNAヘアピンの機械的伸長と展開について報告します。DNAヘアピン張力センサーの課題の1つは、張力の終了時にヘアピンが急速に折りたたまれて可逆的であるため、過渡力を記録するのが難しいことです。本稿では、機械的情報の「保存」を可能にするために「ロック」して折り畳みを防止できるDNA張力センサーを準備するためのプロトコルについて説明します。これにより、非常に過渡的なピコネウトン力の記録が可能になり、ロックを除去する相補的核酸を添加することで、その後「消去」することができます。リアルタイムの張力マッピングと機械的情報保存を切り替えるこの能力は、T細胞が免疫機能の一部として一般的に使用する、弱く、短命で、存在量の少ない力を明らかにします。
概要
免疫細胞は、標的細胞の表面を連続的に這い回って抗原を探し、その表面をスタッディングすることにより、病原体や癌細胞から防御します1,2。抗原認識は、T細胞受容体(TCR)と標的細胞の表面に発現するペプチド主要組織適合遺伝子複合体MHC(pMHC)複合体との間の結合時に開始される。TCR-pMHC認識は2つのモバイルセル間の接合部で発生するため、機械的な力を受けることが長い間疑われてきました。さらに、これはTCR活性化のメカノセンサーモデルにつながり、TCR力がその機能に寄与することを示唆しています3,4。機械的な力がいつ、どこで、どのようにT細胞の機能に寄与するかを理解するには、T細胞によって伝達される分子力を視覚化するツールを開発することが不可欠です。伝統的に、牽引力顕微鏡(TFM)やマイクロピラーアレイなどの方法が細胞力を調べるために使用されます5,6。しかし、TFMおよびマイクロピラーアレイの力感度はナノニュートン(nN)スケールであるため、細胞受容体によって伝達される分子ピコニュートン(pN)力を研究するには不十分なことがよくあります7。検出力と空間分解能を向上させるために、私たちの研究室は、ポリエチレングリコール(PEG)ポリマーを使用して最初に合成された分子張力プローブの開発を開拓しました7。分子張力プローブは、蛍光色素と消光物質に隣接する拡張可能な分子「バネ」(PEG、タンパク質、DNA)で構成され、表面に固定されています。プローブの末端に力を加えるとプローブが伸長し、蛍光色素と消光物質が分離され、強い蛍光シグナルが生成されます(図1A)8,9,10。
過去10年間にわたり、核酸11、タンパク質10、およびポリマー8から作られたスプリングエレメントを備えたさまざまなクラスの分子張力プローブのライブラリを開発してきました。これらの中で、DNAベースのテンションプローブは、最高のS/N比と最大の力感度を提供し、数pNから~20pNまで簡単に調整できます11。これらのリアルタイムDNAテンションプローブを使用して、線維芽細胞、癌細胞、血小板、免疫細胞など、さまざまな種類の細胞によって生成される分子力を研究しています11,12,13。この原稿では、従来の蛍光顕微鏡を使用してpN力分解能で分子受容体力をマッピングするために、表面上でDNA張力プローブを合成および組み立てるためのプロトコルについて説明します。現在の手順には、蛍光レポーターを導入するための核酸の化学修飾が含まれていますが(図1B)、修飾および精製手順の多くはカスタムDNA合成会社に委託できることに注意することが重要です。したがって、DNA張力プローブ技術は容易であり、より広範な細胞生物学およびメカノバイオロジーコミュニティが利用できます。
簡単に言うと、DNA張力センサーを組み立てるために、DNAヘアピンを一方のアームの蛍光リガンド鎖ともう一方のアームのクエンチャーアンカー鎖にハイブリダイズし、ガラス基板に固定化します(図1C、リアルタイム張力)。機械的な力がない場合、ヘアピンは閉じられ、蛍光は消光します。しかし、加えられた機械的な力がF1/2 (50%の確率で展開する平衡時の力)よりも大きい場合、ヘアピンは機械的に溶け、蛍光シグナルが発生します。
リアルタイムDNA張力センサーに基づいて、免疫細胞上の受容体とその天然リガンドとの間の相互作用を研究するのに特に役立つ蓄積された力をマッピングするためのプロトコルについても説明します。これは、免疫受容体がしばしば短命結合を示すためである3,14。蓄積された力は、開いたDNAヘアピンに優先的に結合し、機械的引っ張りイベントに関連する蛍光シグナルの保存を可能にする「ロック」鎖を使用して画像化されます(図1C、ロック張力)。ロッキングストランドは、ヘアピンの機械的に誘発された融解時に露出する不可解な結合部位を結合し、ヘアピンの再折り畳みをブロックして張力信号を格納し、蓄積された張力マップを生成することによってヘアピンを開いた状態にロックするように設計されています。さらに、ロッキング鎖は8ヌクレオチドのトーホールドで設計されており、その完全な補体である「ロック解除」鎖とのトーホールドを介した鎖置換反応を可能にします。ロック解除ストランドを追加すると、バインドされたロックストランドがヘアピン構造から剥がされ、保存されているテンション信号が消去され、ヘアピンがリアルタイム状態にリセットされます。
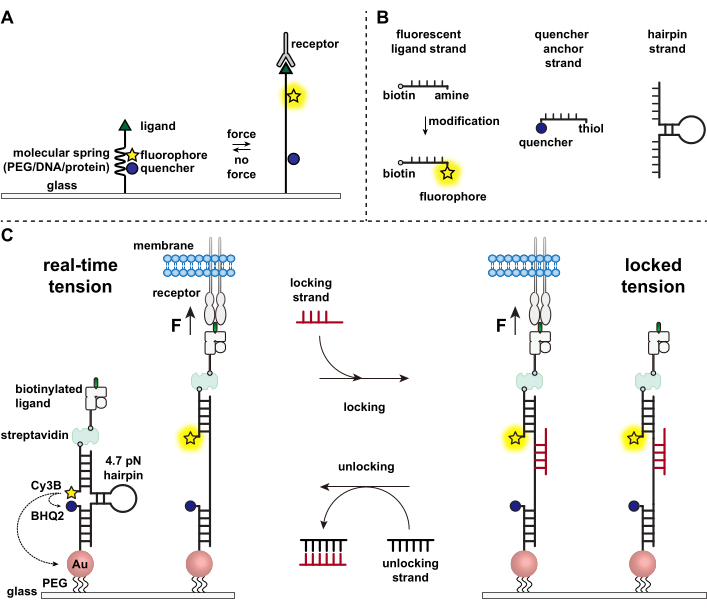
図1:最先端の分子張力プローブのスキーム 。 (A)リアルタイム分子張力プローブの一般的な設計、(B)DNAベースの張力プローブ構築のための鎖、および(C)設計されたDNAベースの張力プローブと、リアルタイム状態とロック状態の間のそれらの切り替え。 この図の拡大版を表示するには、ここをクリックしてください。
メインプロトコルは、オリゴヌクレオチド調製、表面処理、イメージング、およびデータ分析の4つの主要なセクションで構成されています。このプロトコルは、ナイーブで活性化されたOT-1 CD8+ T細胞、OT-II CD4 + 細胞、およびハイブリドーマにおいて私たちの研究室や他の人々によって首尾よく実証されており、T細胞受容体、プログラム細胞死受容体(PD1)、およびリンパ球機能関連抗原1(LFA-1)力を含むさまざまな免疫細胞受容体を調べるために適用できます。OT-1 CD8+ ナイーブT細胞は、この論文の細胞株の例として使用されています。
プロトコル
OT-1トランスジェニックマウスは、エモリー大学の動物資源施設に収容されています。すべての実験は、施設動物管理および使用委員会(IACUC)プロトコルの下で承認および実施されました。
1. オリゴヌクレオチド調製
- リガンド鎖DNAを水に溶解します(18.2 MΩの抵抗率、プロトコル全体で使用)。卓上遠心分離機で溶液を渦巻き、スピンダウンします。最終濃度が1 mMになるように水の量を調整します。ナノドロップ分光光度計を使用して濃度を検証し、260 nmでの吸光度を測定し、オリゴヌクレオチドの吸光係数に基づいて最終濃度を決定します。
注:リガンド鎖は、フルオロフォアと結合し、ビオチン化リガンドを提示するために、各末端、5'アミンおよび3'ビオチンに修飾されています。配位子鎖のアミン基は、蛍光色素と結合する必要があります。Cy3B色素は、その高い輝度と光安定性のためにこの結合に使用されますが、一般的には市販されておらず、社内の抱合が必要です。したがって、次のセクションでは、アミンとNHSエステル染料との間の結合について説明する。核酸修飾のための施設やリソースにアクセスできないエンドユーザーのために、修飾核酸は、AlexaやAttoファミリーの色素など、明るく光安定性のある色素を提供するカスタムDNA合成ベンダーから購入できます。 - 10x PBSおよび1 M NaHCO3 溶液を調製します。10 μLの1 mMアミンリガンド鎖溶液(10 nmol)を10 μLの10x PBS、10 μLの1 M NaHCO3、および60 μLのH2Oと混合します。 使用直前に50 μgのCy3B NHSエステルを10 μLのDMSOに溶解し、混合物に加えて総反応容量を100 μLにします。 最後にCy3B NHSエステルを追加します。室温で1時間または4°Cで一晩反応させる。
- アミンロックストランドをAtto647N NHSエステルと結合させて、Atto647Nロッキングストランドを調製します。10x PBSおよび1 M NaHCO3 溶液を調製します。10 μLの1 mMアミンロック鎖溶液(10 nmol)を10 μLの10x PBS、10 μLの1 M NaHCO3、および60 μLのH2Oと混合します。 使用直前に50 μgのAtto647N NHSエステルを10 μLのDMSOに溶解し、混合物に加えて総反応容量100 μLにします。 最後にAtto647N NHSエステルを追加します。室温で1時間または4°Cで一晩反応させる。
- 反応後、P2脱塩ゲルろ過により副生成物、余分な色素、塩を除去します。反応混合物をH2Oで希釈して総容量300μLにし、これは後続のHPLC精製ステップに適しています。650 μLの水和P2ゲルを遠心装置に加え、18,000 x g で1分間スピンダウンします。装置の下部にある液体を取り除き、反応混合物をP2ゲルを含むカラムに加え、18,000 x g で1分間スピンダウンし、装置の下部にある反応混合物を収集します。
注:P2ゲルは、H2Oで使用する少なくとも4時間前に水和する必要があります。 - オリゴヌクレオチド精製用に指定されたC18カラムを使用して、溶媒A:H2O中の0.1 M TEAAおよびB:ACNを移動相として、0.5 mL/minの流速で50分間にわたって10-100%Bの直線勾配溶出を行い、脱塩反応混合物をHPLCで精製します。脱塩した反応混合物を、精製用の500 μL注入ループを備えた逆相HPLCに注入します。DNAの吸光度ピーク(260 nm)と蛍光色素の吸光度ピーク(Cy3Bの場合は560 nm、Atto647Nの場合は647 nm)を持つ生成物を収集し、真空遠心濃縮器で一晩乾燥させます( 図2Aを参照)。
- 乾燥したオリゴ色素生成物を100 μLの水で再構成します。ナノドロップ分光光度計でCy3Bリガンド鎖およびAtto647Nロッキング鎖の濃度を測定します。色素の標識率が1:1に近いことを確認してください。オリゴヌクレオチド濃度を決定する際に必要に応じて、色素の260 nmの吸光度を修正します。
- MALDI-TOF-MSサンプル調製用に、0.1%TFAを含む50%ACN/H2Oおよび5 mg/mLクエン酸アンモニウムの基質として3-HPAを使用して、精製された製品を1〜5 μMで0.5 μLの製品を使用して検証します。マススペクトルの例を 図2Bに示します。
- ヘアピンストランドとクエンチャーアンカーストランドを水に溶かし、ストック溶液の濃度が50〜100μMであることを確認します。
注:ヘアピンストランドは変更されておらず、ベンダーから直接カスタム合成できます。アンカーストランドはチオールアンカー基とクエンチャーBHQ2を有し、ベンダーから直接カスタム合成することができます。 - すべてのオリゴヌクレオチドを分注します。短期間の使用および保存のために、これらのオリゴヌクレオチドを4°Cで保存してください。 長期保存の場合は、凍結して-20°Cに保管してください。 この時点で、すべてのオリゴヌクレオチドはDNA張力プローブアセンブリの準備ができています。
注:凍結融解サイクルの繰り返しは、オリゴヌクレオチドにとって問題ではありません。
2.表面処理
注:DNAヘアピンテンションプローブ基板の調製には2日かかります。DNAヘアピンテンションプローブは、ガラスカバーガラスに機能化されます。
- 1日目
- 25 mmのカバーガラスを50 mLビーカーのポリテトラフルオロエチレンラックに置きます。各ラックには、最大 8 つのカバーガラスを収納できます。カバーガラスを水に3回沈めてすすぎます。
- ラックとカバーガラスを含むビーカーに水と混合したエタノールの1:1の比率(v:v)溶液40 mLを加え、パラフィンフィルムを使用してビーカーを密封します。
- カバーガラスをきれいにするために超音波クリーナー(動作周波数35 KHz)で15分間ビーカーを超音波処理します。超音波処理後、液体を廃棄し、ラックとカバーガラスでビーカーを少なくとも6回水で洗い流して、残っている有機溶剤を除去します。
- 硫酸と過酸化水素を3:1の比率で混合して、新鮮なピラニア溶液を調製します。40 mLのピラニア溶液を作るには、最初にきれいな50 mLビーカーに30 mLの硫酸を加え、次に10 mLのH 2 O2をゆっくりと加えます。ピラニア溶液は、H 2 O2を添加すると急速に加熱および気泡化する。ガラスピペットの端を使用してピラニアを静かに混ぜます。
- 次に、カバーガラスを保持するラックを、エッチング用の穏やかに混合されたピラニア溶液を含むビーカーに移します(図3A)。ピラニア溶液をヒドロキシル化し、カバーガラスを室温で30分間洗浄します。ピラニアエッチング後、スチールまたはポリテトラフルオロエチレンピンセットを使用してラックを水できれいな50 mLビーカーに移し、少なくとも6回水で再度すすぎます。
注意: 大量の有機物質がピラニア溶液と激しく反応し、爆発を引き起こす可能性があります。注意して、常にドラフト内のピラニアソリューションを使用してください。白衣、手袋、安全ゴーグルを必ず着用してください。新鮮なピラニア溶液を密閉容器に保管しないでください。
注意: 過酸化水素と硫酸の比率は1:2(v:v)未満に保ち、1:1を超えてはなりません。Piranha溶液にカバーガラスでラックを沈めるときは、ゆっくりと慎重に溶液に入れます。それはまだアクティブで熱いので、エッチングの直後に溶液を捨てないでください。酸廃棄物容器に注ぐ前に、ビーカーに一晩置いておいてください。 - カバーガラスを保持しているラックを、40 mLエタノールを入れた50 mLビーカーに浸して水を取り除きます。エタノールを廃棄し、3回繰り返して、水が除去されたことを確認します。
- 次に、ラックを40 mLのエタノール中の3%アミノプロピルトリエトキシシラン(APTES)(v / v)に浸し、カバーガラスの-OHと室温で1時間反応させます(図3B)。
注:エタノールはアセトンに置き換えることができます。 - 表面を40 mLエタノールに浸して6回すすぎ、80°Cのオーブンで20分間乾燥させます。冷却後、乾燥したアミン変性カバーガラスを-20°Cで保管し、将来使用してください(最大6か月)。
- 直径10 cmのプラスチック製ペトリ皿の下内側をパラフィンフィルムで覆います。パラフィンフィルムは、カバーガラスがペトリ皿内で滑るのを防ぎ、機能化の次のステップのための溶液をカバーガラスにとどまらせるのに役立ちます。冷却したアミン変性カバーガラスをペトリ皿に入れます。機能化される側は上を向いている必要があります。
- カバーガラス上のアミン基を変更するには、0.1 M NaHCO3 中の0.5%w/vリポ酸PEG NHS(LA-PEG-SC)および2.5%w/v mPEG NHS(mPEG-SC)300 μLを各カバーガラスに加え、室温で1時間インキュベートします(図3C)。25 mmのカバーガラスごとに、1.5 mgのLA-PEG-SCと7.5 mgのmPEG-SCの重量を量ります。NHS試薬は室温の水溶液中で半減期が短い(~10分)ため、表面に加える直前に溶解してください。反応後、表面を水で3回すすぐ。
注:NHS反応は4°Cで一晩行うことができます。NHS試薬は、4°Cでの加水分解前の半減期が長く、約4〜6時間です。これにより、3日間の表面準備手順が行われます。 - 1 mg/mLのスルホ-NHSアセテートを含む0.1 M NaHCO3 を100 μLの「サンドイッチ」カバーガラスのセットに加えます(反応バッファーを間に挟んで、2つのカバーガラスを互いに向かせます)。パッシベーションを少なくとも30分間発生させます。試薬を節約するために、このステップは50 μLの1 mg/mLスルホ-NHSアセテートで行うことができます。不動態化後3回水ですすいでください。
- 0.5 mLの金ナノ粒子(AuNP、8.8 nm、タンニン酸、0.05 mg/mL)を各カバーガラスに加え、室温で30分間インキュベートします(図3D)。試薬を保存するために、このステップは2つのカバーガラスを挟むことによっても行うことができます。金ナノ粒子の凝集を避けるために、前の手順のシステムに塩が存在しないことを確認してください。この手順の後、カバーガラスを放置して乾かさないでください。
- 一方、DNAテンションプローブを形成する4.7 pNヘアピン、Cy3Bリガンド鎖、およびBHQ2アンカー鎖を、PCRチューブ中で300 nMで1 M NaCl中で1.1:1:1の比率でプレハイブリダイズします。溶液を95°Cまで5分間加熱してストランドをアニールし、サーマルサイクラーで30分間かけて温度を20°Cまで下げて徐々に冷却します。
- 金ナノ粒子とのインキュベーションの30分後にカバーガラスを水で3回すすいでください。BHQ2アンカー鎖(100 μMストックから)をアニーリングしたDNA溶液に追加して、BHQ2アンカー鎖とCy3Bリガンド鎖の比率を10:1にします。この時点で、DNA溶液には300 nMのテンションプローブコンストラクトと2.7 μM BHQ2鎖が含まれているはずです。カバーガラス2枚あたり100 μLを加えて「サンドイッチ」を作ります(図3E)。
- 湿ったラボティッシュボールをペトリ皿(カバーガラスから離して)に慎重に置き、溶液が乾かないようにパラフィンフィルムで皿を密封します。皿をホイルで覆い、4°Cで一晩インキュベートする。
- 2日目
- カバーガラスから余分なプローブを1x PBSで洗い流します。エピ蛍光顕微鏡でDNA張力プローブの表面品質を確認します。
- 1x PBSで40 μg/mLのストレプトアビジンを調製し、カバーガラス上で室温で30分間インキュベートします(図3F)。通常、25 mmのカバーガラスには100 μLで十分です。インキュベーション後にPBSで3回すすぎ、過剰量のストレプトアビジンを洗い流します。
- 40 μg/mL ビオチン化抗体/リガンドを 1x PBS で調製します。サンドイッチあたり50〜100 μLを加え、室温で30分間インキュベートします(図3G)。インキュベーション後にPBSで3回すすぎ、過剰量のビオチン化抗体/リガンドを洗い流します。
- 表面のある清潔なイメージングチャンバーを慎重に組み立てます。チャンバーを締め付けると、表面に簡単にひびが入る可能性があります。0.5〜1 mLのハンク平衡塩溶液(HBSS)をイメージングチャンバーに加え、細胞でイメージングできるように準備します(図3H)。
3. 細胞受容体力のイメージング
- HBSSで目的の免疫細胞を1-2 x 106 細胞/mLで調製します。
注:この論文では、OT-1 CD8+ナイーブ細胞を例として使用しています。製造元の指示に従って、MACSセパレーターを備えたMACSマウスCD8+ T細胞分離キットを使用して、屠殺されたマウスの脾臓からOT-1 CD8 +ナイーブT細胞aを精製します。磁気枯渇抗体カクテルが結合している非CD8+ T細胞を除去することにより、CD8+ T細胞を単離し、濃縮します。精製したOT-1 CD8+ナイーブT細胞をHBSS中の2 x 106細胞/mLで再懸濁し、使用前に氷上に保管してください。 - リガンドやプレーティング細胞を追加する前に、蛍光顕微鏡(対物レンズ100倍)でDNAヘアピンテンションプローブ表面の品質をチェックして品質管理を行います。少なくとも5つの異なる位置および3回の反復からのDNAヘアピン張力プローブ表面のCy3Bチャネルにおける平均バックグラウンド強度を画像化および定量化する。この値を表面品質とプローブ密度の信頼できるマーカーとして使用できるように、画像取得条件を一定に保ちます(図4C)。
注:文献12に従って、表面処理の最初の数回で、金ナノ粒子あたりのDNA鎖の数とμm2あたりの金ナノ粒子の数を定量化し、表面品質の別の信頼できるマーカーとして使用できます。 - ~4 x 10 4 - 10 x 10 4細胞を各DNA張力プローブ機能カバーガラスにプレートし、室温で~15分間付着して広げます。
- 細胞がDNAヘアピンテンションプローブにプレーティングされて広がり始めると、Cy3Bチャネルで生成された蛍光シグナルを100倍の対物レンズでイメージングします(図3I)。
- 細胞がCy3BチャネルのDNAヘアピン張力プローブ表面にリアルタイムの張力シグナルを生成し始めたら、Cy3BチャネルとAtto647Nチャネルの両方で画像を取得します(TIRF顕微鏡は落射蛍光よりも優れた信号対雑音比を提供します)。続いて、機械的に選択的なハイブリダイゼーションのために、Atto647N鎖を最終濃度200nMでイメージングチャンバーに加える。
- 10分間のインキュベーション後、蛍光Atto647Nロックストランドを含むバッファーを迅速かつ穏やかに除去し、新鮮なハンクバランス塩と交換します。Cy3BおよびAtto647Nチャネルの両方で再度画像を作成し、フィジーソフトウェア15とのピアソンの相関係数を決定します。
- 調査の対象時点で、非蛍光ロッキングストランドをイメージングチャンバー内の細胞に導入して、張力信号を保存します。ロッキングストランドストック(100 μM)を調製し、最終濃度1 μMで細胞に加えます。 穏やかにピペットで混ぜます。ロックの時間はさまざまですが、10分が推奨される時間です。
- 必要に応じて、定性的張力マッピングと定量分析の両方のために、落射蛍光のタイムラプス動画またはエンドポイント画像を取得します(図3J および 図5)。
注意: 複数の時点での張力測定が必要な場合は、ロック解除ストランドを追加して、保存されている張力信号の消去を開始します。過剰なすすぎを避けるために、2 μMのより高い最終濃度のロック解除ストランドを使用して、ロックストランドとのトーホールドを介したストランド置換反応を3分間開始し、保存された信号を消去します(図3J)。余分なオリゴヌクレオチドをHBSSでやさしく洗い流します。DNAヘアピンテンションプローブの表面と細胞は、テンションの保存とマッピングの別のラウンドの準備ができています。張力信号のロック解除は、研究に関心のある時点が1つだけである場合は必要ありません。
4.データ分析
注:画像分析はフィジーソフトウェアを使用して実行され、定量分析は分析ソフトウェアを使用して実行されます。
- プラグインメニューの登録にある3Dドリフトの修正コマンドを使用して、画像取得中のドリフトを修正します。
- [プロセス]メニューの[減算]コマンドを使用して、画像のカメラの背景を削除します。
- ピアソンの相関係数は、分析メニューの共局在化関数で求めます。
- 未開封プローブによって生成された蛍光バックグラウンドを3つの異なる局所バックグラウンド領域から平均して差し引きます。Image Jフリーハンド選択ツールを使用して、背景減算画像またはRICM(反射干渉コントラスト顕微鏡)画像のいずれかに細胞のROIを描画します。分析メニューの測定ツールを使用して、統合された蛍光強度や張力占有率など、ROIの関心のあるメトリックを測定します(図6)。
- 分析ソフトウェアで定量分析するために測定値をエクスポートします。
- 任意の分析ソフトウェアでデータをプロットします。
結果
代表的な表面品質管理画像を示します(図4)。高品質の表面は、RICMチャネルでクリーンなバックグラウンドを持ち(図4B)、Cy3Bチャネルで均一な蛍光強度を持つ必要があります(図4C)。同じイメージング機器と同一の蛍光イメージング取得条件では、DNAプローブで実験を行う場合、バックグラウンド蛍光強度は毎回一貫して再現性...
ディスカッション
ここで提供される詳細な手順を使用して、免疫細胞によって産生される受容体張力をマッピングおよび定量するためのDNAヘアピン張力プローブ基板を調製することができます。細胞がDNAヘアピンテンションプローブ基板にプレーティングされると、受容体が化学的および機械的にリガンドを感知し、後者はプローブによって検出されるため、細胞は着地、付着、拡散します。ただし、場合に?...
開示事項
著者は利益相反を宣言しません。
謝辞
この作業は、NIH Grants R01GM131099、NIH R01GM124472、およびNSF CAREER 1350829の支援を受けました。pMHCリガンドに関するNIHテトラマーファシリティに感謝します。この研究は、Emory Comprehensive Glycomics Coreによって部分的にサポートされました。
資料
| Name | Company | Catalog Number | Comments |
| 3-hydroxypicolinic acid (3-HPA) | Sigma | 56197 | maldi-TOF-MS matrix |
| mPEG-SC | Biochempeg | MF001023-2K | surface prep |
| (3-Aminopropyl)triethoxysilane | Acros | AC430941000 | surface prep |
| 10x Red blood cell lysis buffer | Biolegend | 00-4333-57 | buffer |
| 8.8 nm gold nanoparticles, tannic acid | Nanocomposix | customized order | surface prep |
| Atto647N NHS ester | Sigma | 18373-1MG-F | fluorophore, oligo prep |
| Attofluor Cell Chamber, for microscopy | Thermo Fisher Scientific | A7816 | imaging |
| BD Syringes only with Luer-Lok | BD bioscience | 309657 | cells |
| biotinylated anti-mouse CD3e | ebioscience | 13-0031-82 | antibody/ligand |
| Biotinylated pMHC ovalbumin (SIINFEKL) | NIH Tetramer Core Facility at Emory University | NA | antibody/ligand |
| bovine serum albumin | Sigma | 735078001 | block non-specific interactions |
| Cell strainers | Biologix | 15-1100 | cells |
| Coverslip Mini-Rack, teflon | Thermo Fisher Scientific | C14784 | surface prep |
| Cy3B NHS ester | GE Healthcare | PA63101 | fluorophore, oligo prep |
| Dulbecco's phosphate-buffered saline (DPBS) | Corning | 21-031-CM | buffer |
| ethanol | Sigma | 459836 | surface prep |
| Hank’s balanced salts (HBSS) | Sigma | H8264 | buffer |
| hydrogen peroxide | Sigma | H1009 | surface prep |
| LA-PEG-SC | Biochempeg | HE039023-3.4K | surface prep |
| Midi MACS (LS) startup kit | Miltenyi Biotec | 130-042-301 | cells |
| mouse CD8+ T cell isolation kit | Miltenyi Biotec | 130-104-075 | cells |
| Nanosep MF centrifugal devices | Pall laboratory | ODM02C35 | oligo prep |
| No. 2 round glass coverslips | VWR | 48382-085 | surface prep |
| NTA-SAM | Dojindo Molecular Technologies | N475-10 | surface prep |
| P2 gel | Bio-rad | 1504118 | oligo prep |
| sufuric acid | EMD Millipore Corporation | SX1244-6 | surface prep |
| Sulfo-NHS acetate | Thermo Fisher Scientific | 26777 | surface prep |
| Equipment | |||
| Agilent AdvanceBio Oligonucleotide C18 column, 4.6 x 150 mm, 2.7 μm | 653950-702 | oligonucleotide preparation | |
| Barnstead Nanopure water purifying system | Thermo Fisher | water | |
| CFI Apo 100× NA 1.49 objective | Nikon | Microscopy | |
| Cy5 cube | CHROMA | Microscopy | |
| evolve electron multiplying charge coupled device (EMCCD) | Photometrics | Microscopy | |
| High-performance liquid chromatography | Agilent 1100 | oligonucleotide preparation | |
| Intensilight epifluorescence source | Nikon | Microscopy | |
| Matrix-assisted laser desorption/ionization time-of-flight mass spectrometer (MALDI-TOF-MS) | Voyager STR | oligonucleotide preparation | |
| Nanodrop 2000 UV-Vis Spectrophotometer | Thermo Fisher | oligonucleotide preparation | |
| Nikon Eclipse Ti inverted microscope | Nikon | Microscopy | |
| Nikon Perfect Focus System | Nikon | Microscopy | |
| NIS Elements software | Nikon | Microscopy | |
| quad band TIRF 405/488/561/647 cube | CHROMA | Microscopy | |
| RICM cube | CHROMA | Microscopy | |
| TIRF launcher with 488 nm (50 mW), 561 nm (50 mW), and 640 nm | Coherent | Microscopy | |
| TRITC cube | CHROMA | Microscopy | |
| oligo name | 5' modification / 3' modification | sequence (5' to 3') | Use |
| 15mer amine locking strand | 5' modification: no modification 3' modification: /3AmMO/ | AAA AAA CAT TTA TAC CCT ACC TA | locking real-time tension signal |
| 15mer Atto647N locking strand | 5' modification: Atto647N 3' modification: /3AmMO/ | AAA AAA CAT TTA TAC CCT ACC TA | locking real-time tension signal |
| 15mer non-fluoresccent locking strand | 5' modification: no modification 3' modification: no modification | A AAA AAC ATT TAT AC | locking real-time tension signal for quantitative analysis |
| 4.7 pN hairpin strand | 5' modification: no modification 3' modification: no modification | GTGAAATACCGCACAGATGCGT TTGTATAAATGTTTTTTTCATTTAT ACTTTAAGAGCGCCACGTAGCC CAGC | hairpin probe |
| amine ligand strand | 5' modification: /5AmMC6/ 3' modification: /3Bio/ | CGCATCTGTGCG GTA TTT CAC TTT | hairpin probe |
| BHQ2 anchor strand | 5' modification: /5ThiolMC6-D/ 3' modification: /3BHQ_2/ | TTTGCTGGGCTACGTGGCGCTCTT | hairpin probe |
| Cy3B ligand strand | 5' modification: Cy3B 3' modification: /3Bio/ | CGCATCTGTGCG GTA TTT CAC TTT | hairpin probe |
| unlocking strand | 5' modification: no modification 3' modification: no modification | TAG GTA GGG TAT AAA TGT TTT TTT C | unlocking accumulated tension signal |
参考文献
- Dustin, M. L. T-cell activation through immunological synapses and kinapses. Immunological Reviews. 221 (1), 77-89 (2008).
- Spillane, K. M., Tolar, P. B cell antigen extraction is regulated by physical properties of antigen-presenting cells. Journal of Cell Biology. 216 (1), 217-230 (2017).
- Feng, Y., et al. Mechanosensing drives acuity of αβ T-cell recognition. Proceedings of the National Academy of Sciences. 114 (39), 8204-8213 (2017).
- Hong, J., et al. A TCR mechanotransduction signaling loop induces negative selection in the thymus. Nature Immunology. 19 (12), 1379-1390 (2018).
- Basu, R., et al. Cytotoxic T cells use mechanical force to potentiate target cell killing. Cell. 165 (1), 100-110 (2016).
- Bashour, K. T., et al. CD28 and CD3 have complementary roles in T-cell traction forces. Proceedings of the National Academy of Sciences. 111 (6), 2241-2246 (2014).
- Ma, V. P. Y., Salaita, K. DNA nanotechnology as an emerging tool to study mechanotransduction in living systems. Small. 15 (26), 1900961 (2019).
- Liu, Y., Yehl, K., Narui, Y., Salaita, K. Tension sensing nanoparticles for mechano-imaging at the living/nonliving interface. Journal of the American Chemical Society. 135 (14), 5320-5323 (2013).
- Glazier, R., et al. DNA mechanotechnology reveals that integrin receptors apply pN forces in podosomes on fluid substrates. Nature Communications. 10 (1), 1-13 (2019).
- Galior, K., Liu, Y., Yehl, K., Vivek, S., Salaita, K. Titin-based nanoparticle tension sensors map high-magnitude integrin forces within focal adhesions. Nano Letters. 16 (1), 341-348 (2016).
- Zhang, Y., Ge, C., Zhu, C., Salaita, K. DNA-based digital tension probes reveal integrin forces during early cell adhesion. Nature Communications. 5, 5167 (2014).
- Liu, Y., et al. DNA-based nanoparticle tension sensors reveal that T-cell receptors transmit defined pN forces to their antigens for enhanced fidelity. Proceedings of the National Academy of Sciences. 113 (20), 5610-5615 (2016).
- Zhang, Y., et al. Platelet integrins exhibit anisotropic mechanosensing and harness piconewton forces to mediate platelet aggregation. Proceedings of the National Academy of Sciences. 115 (2), 325-330 (2018).
- Huang, J., et al. The kinetics of two-dimensional TCR and pMHC interactions determine T-cell responsiveness. Nature. 464 (7290), 932-936 (2010).
- Ma, R., et al. DNA probes that store mechanical information reveal transient piconewton forces applied by T cells. Proceedings of the National Academy of Sciences. 116 (34), 16949-16954 (2019).
- Hui, E., et al. T cell costimulatory receptor CD28 is a primary target for PD-1-mediated inhibition. Science. 355 (6332), 1428-1433 (2017).
- Whitley, K. D., Comstock, M. J., Chemla, Y. R. Elasticity of the transition state for oligonucleotide hybridization. Nucleic Acids Research. 45 (2), 547-555 (2016).
- Brockman, J. M., et al. Live-cell super-resolved PAINT imaging of piconewton cellular traction forces. Nature Methods. 17 (10), 1018-1024 (2020).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved