Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Sondes de tension de l’ADN pour cartographier les forces transitoires du récepteur Piconewton par les cellules immunitaires
Dans cet article
Résumé
Cet article décrit un protocole détaillé pour l’utilisation de sondes de tension basées sur l’ADN pour imager les forces réceptrices appliquées par les cellules immunitaires. Cette approche permet de cartographier les forces du récepteur >4,7pN en temps réel et d’intégrer les forces au fil du temps.
Résumé
Les forces mécaniques transmises à la jonction entre deux cellules voisines et à la jonction entre les cellules et la matrice extracellulaire sont essentielles pour réguler de nombreux processus allant du développement à l’immunologie. Par conséquent, le développement des outils pour étudier ces forces à l’échelle moléculaire est essentiel. Notre groupe a développé une suite de capteurs de tension moléculaire pour quantifier et visualiser les forces générées par les cellules et transmises à des ligands spécifiques. La classe la plus sensible des capteurs de tension moléculaire est composée d’épingles à cheveux à boucle tige d’acide nucléique. Ces capteurs utilisent des paires fluorophore-quencher pour rendre compte de l’extension mécanique et du dépliage des épingles à cheveux ADN sous force. L’un des défis des capteurs de tension en épingle à cheveux à ADN est qu’ils sont réversibles avec un repliement rapide en épingle à cheveux à la fin de la tension et que les forces transitoires sont donc difficiles à enregistrer. Dans cet article, nous décrivons les protocoles de préparation des capteurs de tension de l’ADN qui peuvent être « verrouillés » et empêchés de se replier pour permettre le « stockage » des informations mécaniques. Cela permet d’enregistrer des forces de piconewton hautement transitoires, qui peuvent ensuite être « effacées » par l’ajout d’acides nucléiques complémentaires qui suppriment le verrou. Cette capacité à basculer entre la cartographie de tension en temps réel et le stockage d’informations mécaniques révèle des forces faibles, de courte durée et moins abondantes, qui sont couramment utilisées par les cellules T dans le cadre de leurs fonctions immunitaires.
Introduction
Les cellules immunitaires se défendent contre les agents pathogènes et les cellules cancéreuses en rampant et en balayant continuellement les surfaces des cellules cibles à la recherche d’antigènes, en cloutant leur surface 1,2. La reconnaissance de l’antigène est initiée lors de la liaison entre le récepteur des lymphocytes T (TCR) et le complexe peptide-majeur d’histocompatibilité CMH (pMHC) exprimé à la surface des cellules cibles. Parce que la reconnaissance TCR-pMHC se produit à la jonction entre deux cellules mobiles, il a longtemps été soupçonné de subir des forces mécaniques. De plus, cela a conduit au modèle mécanocapteur d’activation TCR, qui suggère que les forces TCR contribuent à sa fonction 3,4. Pour comprendre quand, où et comment les forces mécaniques contribuent au fonctionnement des lymphocytes T, il est impératif de développer des outils pour visualiser les forces moléculaires transmises par les lymphocytes T. Traditionnellement, des méthodes telles que la microscopie à force de traction (TFM) et les réseaux de micropiliers sont utilisés pour étudier les forces cellulaires 5,6. Cependant, la sensibilité aux forces de la TFM et des réseaux de micropiliers est à l’échelle du nanonewton (nN) et est donc souvent insuffisante pour étudier les forces moléculaires du piconewton (pN) transmises par les récepteurs cellulaires7. Pour améliorer la force et la résolution spatiale de la détection, notre laboratoire a été le pionnier du développement de sondes de tension moléculaire, qui ont été initialement synthétisées à l’aide de polymères de polyéthylèneglycol (PEG)7. Les sondes de tension moléculaire sont composées d’un « ressort » moléculaire extensible (PEG, protéine, ADN) flanqué d’un fluorophore et d’un quencher et sont ancrées sur une surface. Les forces appliquées à l’extrémité de la sonde conduisent à son extension, séparant le fluorophore et l’anti-quenceur, et générant ainsi un fort signal de fluorescence (Figure 1A)8,9,10.
Au cours de la dernière décennie, nous avons développé une bibliothèque de différentes classes de sondes de tension moléculaire avec des éléments à ressort fabriqués à partir d’acides nucléiques11, de protéines10 et de polymères8. Parmi celles-ci, les sondes de tension basées sur l’ADN fournissent le rapport signal sur bruit le plus élevé et la plus grande sensibilité à la force, qui est facilement réglée de quelques pN à ~20 pN11. Nous avons utilisé ces sondes de tension d’ADN en temps réel pour étudier les forces moléculaires générées par de nombreux types de cellules, y compris les fibroblastes, les cellules cancéreuses, les plaquettes et les cellules immunitaires11,12,13. Ce manuscrit décrira des protocoles pour synthétiser et assembler des sondes de tension d’ADN sur une surface afin de cartographier les forces des récepteurs moléculaires avec une résolution de force pN à l’aide d’un microscope à fluorescence conventionnel. Bien que la procédure actuelle comprenne des modifications chimiques de l’acide nucléique pour introduire le rapporteur fluorescent (figure 1B), il est important de noter que de nombreuses étapes de modification et de purification peuvent être sous-traitées à des entreprises de synthèse d’ADN personnalisées. Par conséquent, la technologie des sondes de tension de l’ADN est facile et accessible aux communautés plus larges de la biologie cellulaire et de la mécanobiologie.
Brièvement, pour assembler des capteurs de tension d’ADN, une épingle à cheveux à ADN est hybridée à un brin de ligand fluorescent sur un bras et à un brin d’ancrage de quencher sur l’autre bras, puis immobilisée sur un substrat de verre (Figure 1C, tension en temps réel). En l’absence de force mécanique, l’épingle à cheveux est fermée et la fluorescence est donc éteinte. Cependant, lorsque la force mécanique appliquée est supérieure à la F1/2 (la force à l’équilibre qui conduit à une probabilité de 50% de se déplier), l’épingle à cheveux fond mécaniquement et un signal fluorescent est généré.
En nous appuyant sur le capteur de tension de l’ADN en temps réel, nous décrivons également des protocoles pour cartographier les forces accumulées, ce qui est particulièrement utile pour étudier les interactions entre les récepteurs des cellules immunitaires et leur ligand naturel. En effet, les récepteurs immunitaires présentent souvent des liaisons de courte durée 3,14. Les forces accumulées sont imagées à l’aide d’un brin de « verrouillage » qui se lie préférentiellement aux épingles à cheveux d’ADN ouvertes et permet le stockage des signaux de fluorescence associés aux événements de traction mécanique (Figure 1C, tension verrouillée). Le brin de verrouillage est conçu pour lier un site de liaison cryptique qui est exposé lors de la fusion induite mécaniquement de l’épingle à cheveux et verrouiller l’épingle à cheveux à l’état ouvert en bloquant le repliement de l’épingle à cheveux, stockant ainsi le signal de tension et générant une carte de tension accumulée. De plus, le brin de verrouillage est conçu avec un toehold à huit nucléotides, ce qui permet une réaction de déplacement du brin médiée par le teil avec son complément complet, le brin de « déverrouillage ». Avec l’ajout du brin de déverrouillage, le brin de verrouillage lié est retiré de la construction en épingle à cheveux, effaçant le signal de tension stocké et réinitialisant l’épingle à cheveux à l’état temps réel.
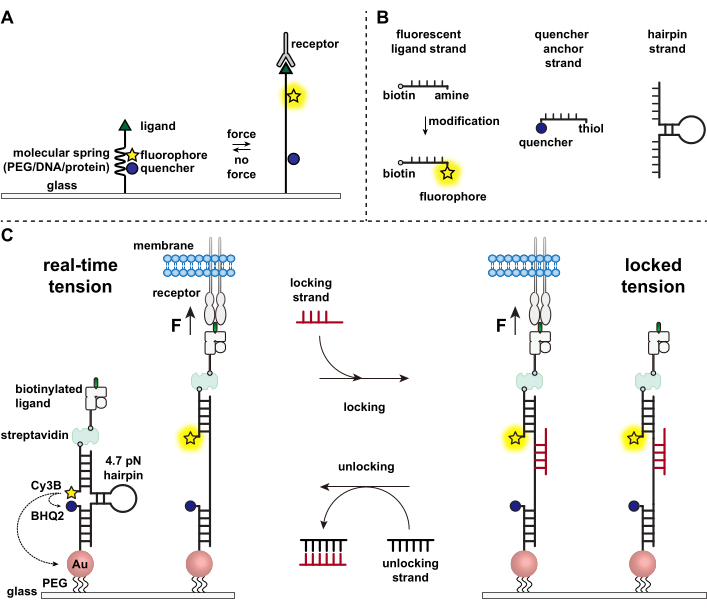
Figure 1 : Schéma des sondes de tension moléculaire de pointe. (A) Conception générale de sonde de tension moléculaire en temps réel, (B) Brins pour la construction de sonde de tension basée sur l’ADN, et (C) sondes de tension basées sur l’ADN et leur basculement entre l’état en temps réel et l’état verrouillé. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Le protocole principal comprend quatre sections principales: préparation des oligonucléotides, préparation de surface, imagerie et analyse des données. Ce protocole a été démontré avec succès par notre laboratoire et d’autres dans des lymphocytes T CD8+ OT-1 naïfs et activés, des cellules CD4+ OT-II, ainsi que des hybridomes, et peut être appliqué pour interroger différents récepteurs de cellules immunitaires, y compris le récepteur des cellules T, le récepteur de mort cellulaire programmée (PD1) et les forces de l’antigène 1 associé à la fonction lymphocytaire (LFA-1). Les lymphocytes T naïfs de CD8+ OT-1 sont utilisés comme exemple de lignée cellulaire dans cet article.
Protocole
Les souris transgéniques OT-1 sont hébergées à la Division of Animal Resources Facility de l’Université Emory. Toutes les expériences ont été approuvées et réalisées selon le protocole du Comité institutionnel de soin et d’utilisation des animaux (IACUC).
1. Préparation d’oligonucléotides
- Dissoudre l’ADN du brin de ligand dans l’eau (résistivité de 18,2 MΩ, utilisée tout au long du protocole). Vortex et faire tourner la solution avec une centrifugeuse de table. Réglez le volume d’eau de telle sorte que la concentration finale soit de 1 mM. Valider la concentration à l’aide d’un spectrophotomètre nanogoutte pour mesurer l’absorbance à 260 nm et déterminer la concentration finale en fonction du coefficient d’extinction de l’oligonucléotide.
NOTE: Le brin de ligand a une modification à chaque extrémité, 5' amine et 3' biotine, pour se conjuguer avec le fluorophore et présenter le ligand biotinylé. Le groupe amine dans le brin de ligand doit être conjugué avec un fluorophore. Le colorant Cy3B est utilisé pour cette conjugaison en raison de sa luminosité élevée et de sa photostabilité, mais il n’est généralement pas offert dans le commerce et nécessite une conjugaison interne. Par conséquent, la section suivante décrit la conjugaison entre les amines et les colorants esters du NHS. Pour les utilisateurs finaux qui n’ont pas accès à des installations ou à des ressources pour la modification des acides nucléiques, les acides nucléiques modifiés peuvent être achetés auprès de fournisseurs de synthèse d’ADN personnalisés qui offrent des colorants brillants et photostables, tels que la famille de colorants Alexa et Atto. - Préparez 10x PBS et 1 M NaHCO3 solutions. Mélanger 10 μL de la solution de brin de ligand amine 1 mM (10 nmol) avec 10 μL de PBS 10x, 10 μL de NaHCO3 1 M et 60 μL deH2O. Dissoudre 50 μg d’ester NHS Cy3B dans 10 μL de DMSO immédiatement avant utilisation et ajouter au mélange pour un volume de réaction total de 100 μL. Ajouter l’ester NHS Cy3B en dernier. Laisser réagir à température ambiante pendant 1 h ou 4 °C pendant la nuit.
- Préparer le brin de verrouillage Atto647N en conjuguant le brin de verrouillage d’amine avec l’ester NHS Atto647N. Préparer 10x PBS et 1 M NaHCO3 solution. Mélanger 10 μL de la solution de brin de verrouillage d’amine de 1 mM (10 nmol) avec 10 μL de PBS 10x, 10 μL de NaHCO3 1 M et 60 μL deH2O. Dissoudre 50 μg d’ester NHS Atto647N dans 10 μL de DMSO immédiatement avant utilisation et ajouter au mélange pour un volume de réaction total de 100 μL. Ajouter l’ester NHS Atto647N en dernier. Laisser réagir à température ambiante pendant 1 h ou 4 °C pendant la nuit.
- Après les réactions, éliminer les sous-produits, l’excès de colorant et les sels par filtration de gel dessalant P2. Diluer le mélange réactionnel avecH2Ojusqu’à obtenir un volume total de 300 μL, ce qui est approprié pour l’étape de purification CLHP suivante. Ajouter 650 μL de gel P2 hydraté à un dispositif centrifuge et faire tourner vers le bas à 18 000 x g pendant 1 min. Retirer le liquide au fond de l’appareil, ajouter le mélange réactionnel à la colonne contenant le gel P2, faire tourner vers le bas à 18 000 x g pendant 1 min et recueillir le mélange réactionnel au fond de l’appareil.
REMARQUE: Le gel P2 doit être hydraté au moins 4 h avant utilisation avecH2O. - Purifier le mélange réactionnel dessalé par CLHP à l’aide d’une colonne C18 désignée pour la purification des oligonucléotides, avec le solvant A: 0,1 M TEAA enH2Oet B: ACN comme phase mobile pour une élution linéaire gradient 10-100% B sur 50 min à un débit de 0,5 mL/min. Injecter le mélange réactionnel dessalé en CLHP en phase inverse avec une boucle d’injection de 500 μL pour la purification. Prélever le produit qui a un pic d’absorbance pour l’ADN (260 nm) et un pic d’absorbance pour le fluorophore (560 nm pour Cy3B et 647 nm pour Atto647N) et séchez-le dans un concentrateur centrifuge sous vide pendant une nuit (voir Figure 2A).
- Reconstituer le produit oligo-colorant séché dans 100 μL d’eau. Déterminez la concentration du brin de ligand Cy3B et du brin de verrouillage Atto647N avec le spectrophotomètre nanodrop. Assurez-vous que le rapport d’étiquetage du colorant est proche de 1:1. Corriger l’absorbance de 260 nm du colorant si nécessaire lors de la détermination de la concentration d’oligonucléotides.
- Valider le produit purifié avec MALDI-TOF-MS en utilisant la 3-HPA comme substrat dans 50 % d’ACN/H2Oavec 0,1 % de TFA et 5 mg/mL de citrate d’ammonium en utilisant 0,5 μL du produit à 1-5 μM pour la préparation de l’échantillon MALDI-TOF-MS. Un exemple de spectre de masse peut être trouvé à la figure 2B.
- Dissoudre le brin en épingle à cheveux et le brin d’ancrage de l’antisseur dans de l’eau et s’assurer que la concentration des solutions mères est comprise entre 50 et 100 μM.
REMARQUE: Le brin en épingle à cheveux n’est pas modifié et peut être synthétisé directement sur mesure par un fournisseur. Le brin d’ancrage a un groupe d’ancrage thiol et un quencher BHQ2 et peut être synthétisé directement sur mesure à partir d’un fournisseur. - Aliquote tous les oligonucléotides. Pour une utilisation et un stockage à court terme, conserver ces oligonucléotides à 4 °C. Pour un stockage à long terme, congelez-les et conservez-les à -20 °C. À ce stade, tous les oligonucléotides sont prêts pour l’assemblage de la sonde de tension de l’ADN.
REMARQUE : Les cycles répétés de gel-dégel ne sont pas problématiques pour les oligonucléotides.
2. Préparation de surface
REMARQUE: La préparation des substrats de sonde de tension en épingle à cheveux ADN prend deux jours. La sonde de tension ADN en épingle à cheveux sera fonctionnalisée sur des lamelles de couvercle en verre.
- Jour 1
- Placer les lamelles de couverture de 25 mm sur une grille en polytétrafluoroéthylène dans un bécher de 50 mL. Chaque rack peut contenir jusqu’à 8 lamelles de couverture. Rincer les lamelles en les plongeant trois fois dans l’eau.
- Ajouter 40 mL d’une solution d’éthanol à rapport 1:1 (v:v) mélangée à de l’eau dans le bécher contenant la grille et les lamelles de couverture, et sceller le bécher à l’aide d’un film de paraffine.
- Sonicer le bécher pendant 15 min dans un nettoyeur à ultrasons (fréquence de fonctionnement 35 KHz) pour nettoyer les lames de couverture. Après la sonication, jetez le liquide et rincez le bécher avec la grille et les lamelles de couverture avec de l’eau au moins 6 fois pour éliminer tout solvant organique restant.
- Préparer une solution fraîche de piranha en mélangeant de l’acide sulfurique et du peroxyde d’hydrogène dans un rapport de 3:1. Pour obtenir 40 ml de solution de piranha, ajouter d’abord 30 ml d’acide sulfurique dans un bécher propre de 50 ml, puis ajouter lentement 10 ml deH2O2. La solution de Piranha chauffera rapidement et fera des bulles lors de l’ajout du H2O2. Mélanger délicatement le piranha à l’aide de l’extrémité d’une pipette en verre.
- Ensuite, transférez la grille qui contient les lamelles de couverture dans le bécher contenant une solution de piranha légèrement mélangée pour la gravure (Figure 3A). Laisser la solution de Piranha s’hydroxyler et nettoyer les lamelles de couverture pendant 30 min à température ambiante. Après la gravure Piranha, transférer la grille à l’aide d’une pince à épiler en acier ou en polytétrafluoroéthylène dans un bécher propre de 50 ml avec de l’eau et rincer à nouveau à l’eau au moins 6 fois.
ATTENTION: De grandes quantités de substances organiques peuvent réagir vigoureusement avec la solution de Piranha et provoquer une explosion. Soyez prudent et travaillez toujours avec la solution Piranha dans une hotte. Assurez-vous de porter un sarrau de laboratoire, des gants et des lunettes de sécurité. Ne conservez jamais la solution fraîche de Piranha dans un récipient scellé.
NOTE: Le rapport peroxyde d’hydrogène/acide sulfurique doit être maintenu en dessous de 1:2 (v:v) et ne doit jamais dépasser 1:1. Lorsque vous immergez le rack avec des lamelles de couverture dans une solution Piranha, placez-les lentement et soigneusement dans la solution. Ne jetez pas la solution immédiatement après la gravure, car elle est encore active et chaude. Laissez-le dans le bécher pendant la nuit avant de le verser dans le récipient à déchets acides. - Plongez la grille contenant les lamelles de couverture dans un bécher de 50 ml avec de l’éthanol de 40 ml pour éliminer l’eau. Jetez l’éthanol et répétez 3 fois pour vous assurer que l’eau a été éliminée.
- Immergez ensuite la grille dans du triéthoxysilane aminopropylique à 3 % (APTES) (v/v) dans 40 mL d’éthanol pour réagir avec le -OH sur les lamelles de recouvrement pendant 1 h à température ambiante (Figure 3B).
NOTE: L’éthanol peut être remplacé par de l’acétone. - Rincer les surfaces 6 fois en les immergeant dans 40 ml d’éthanol, puis sécher au four à 80 °C pendant 20 min. Après refroidissement, conserver les lamelles de couverture séchées modifiées aux amines à -20 °C pour une utilisation ultérieure (jusqu’à 6 mois).
- Couvrir la face intérieure inférieure des boîtes de Petri en plastique de 10 cm de diamètre avec un film de paraffine. Le film de paraffine empêche les lamelles de couvercle de glisser à l’intérieur de la boîte de Petri et aide à garder la solution pour les prochaines étapes de fonctionnalisation sur les lamelles de couverture. Placez les lames de couverture refroidies modifiées aux amines dans les boîtes de Pétri. Le côté à fonctionnaliser doit être orienté vers le haut.
- Pour modifier les groupes amines sur les lames de couverture, ajouter 300 μL d’acide lipoïque PEG NHS (LA-PEG-SC) à 0,5 % p/v d’acide lipoïque PEG NHS (LA-PEG-SC) et 2,5 % p/v mPEG NHS (mPEG-SC) dans 0,1 M NaHCO3 sur chaque lamelle de couverture et incuber pendant 1 h à température ambiante (Figure 3C). Pour chaque lamelle de couverture de 25 mm, peser 1,5 mg de LA-PEG-SC et 7,5 mg de mPEG-SC. Dissoudre les réactifs NHS immédiatement avant de les ajouter aux surfaces, car ils ont une courte demi-vie (~10 min) en solution aqueuse à température ambiante. Après la réaction, rincer les surfaces 3 fois avec de l’eau.
REMARQUE: La réaction NHS peut être effectuée à 4 ° C pendant la nuit. Les réactifs NHS ont une demi-vie plus longue avant hydrolyse à 4 °C, soit environ 4-6 h. Cela se traduira par une procédure de préparation de surface de trois jours. - Ajouter 100 μL de NaHCO3 0,1 M contenant 1 mg/mL d’acétate de sulfo-NHS à un ensemble de lamelles de couverture « sandwich » (deux lamelles de couverture tournées l’une vers l’autre avec tampon de réaction entre les deux). Laisser la passivation se produire pendant au moins 30 minutes. Pour économiser le réactif, cette étape pourrait être effectuée avec 50 μL de 1 mg/mL d’acétate de sulfo-NHS. Rincer à l’eau trois fois après la passivation.
- Ajouter 0,5 mL de nanoparticules d’or (AuNP, 8,8 nm, acide tannique, 0,05 mg/mL) à chaque lamelle de couverture et incuber pendant 30 min à température ambiante (Figure 3D). Pour économiser le réactif, cette étape peut être effectuée en prenant également en sandwich deux lamelles de couverture. Assurez-vous qu’aucun sel n’est présent dans le système à partir des étapes précédentes pour éviter l’agrégation de nanoparticules d’or. Ne laissez pas sécher les lamelles de couverture après cette étape.
- Pendant ce temps, pré-hybrider l’épingle à cheveux 4,7 pN, le brin de ligand Cy3B et le brin d’ancrage BHQ2 qui forment les sondes de tension de l’ADN construisent à un rapport de 1,1: 1: 1 dans 1 M NaCl à 300 nM dans un tube PCR. Recuisez les brins en chauffant la solution jusqu’à 95 °C pendant 5 min, puis refroidissez progressivement en diminuant la température à 20 °C pendant 30 min dans un cycleur thermique.
- Rincer les lamelles de couverture avec de l’eau trois fois après 30 min d’incubation avec des nanoparticules d’or. Ajouter un brin d’ancrage BHQ2 supplémentaire (à partir de 100 μM de stock) à la solution d’ADN recuite pour obtenir le rapport entre le brin d’ancrage BHQ2 et le brin de ligand Cy3B 10:1. À ce stade, la solution d’ADN doit contenir 300 nM de construction de sonde de tension et 2,7 μM de brin BHQ2. Ajouter 100 μL par deux lamelles de couverture pour faire le « sandwich » (figure 3E).
- Placez soigneusement une boule de mouchoir humide de laboratoire dans la boîte de Petri (loin des lamelles de couverture) et scellez la boîte avec un film de paraffine pour éviter que la solution ne sèche. Couvrir le plat de papier d’aluminium et incuber à 4 °C pendant la nuit.
- Jour 2
- Lavez les sondes excédentaires des lamelles de couverture avec 1x PBS. Vérifiez la qualité de la surface de la sonde de tension de l’ADN au microscope épifluorescent.
- Préparer 40 μg/mL de streptavidine dans 1x PBS et incuber sur des lames de couverture pendant 30 minutes à température ambiante (figure 3F). Habituellement, 100 μL suffisent pour une lamelle de couverture de 25 mm. Rincer avec du PBS 3 fois après l’incubation pour éliminer l’excès de streptavidine.
- Préparer 40 μg/mL d’anticorps/ligand biotinylé dans 1x PBS. Ajouter 50-100 μL par sandwich et incuber pendant 30 min à température ambiante (Figure 3G). Rincer avec du PBS trois fois après l’incubation pour éliminer la quantité excessive d’anticorps / ligand biotinylés.
- Assemblez soigneusement les chambres d’imagerie propres avec les surfaces. Les surfaces peuvent être facilement fissurées lors du serrage des chambres. Ajoutez 0,5 à 1 mL de solution saline équilibrée (HBSS) de Hank aux chambres d’imagerie et gardez-les prêtes pour l’imagerie avec des cellules (Figure 3H).
3. Imagerie des forces des récepteurs cellulaires
- Préparer les cellules immunitaires d’intérêt dans HBSS à 1-2 x 106 cellules / mL.
REMARQUE : Les cellules naïves de CD8+ OT-1 sont utilisées à titre d’exemple dans le présent document. Purifier les lymphocytes T naïfs OT-1 CD8+ des rates de souris sacrifiées à l’aide du kit d’isolation des lymphocytes T CD8+ de souris MACS avec un séparateur MACS suivant les instructions du fabricant. Isoler et enrichir les lymphocytes T CD8+ en éliminant tous les lymphocytes T non CD8+ auxquels le cocktail d’anticorps appauvrissant magnétiquement est lié. Resuspendre les lymphocytes T naïfs OT-1 CD8+ purifiés dans le HBSS à raison de 2 x 106 cellules/ml et conserver sur la glace avant utilisation. - Vérifiez la qualité de la surface de la sonde de tension en épingle à cheveux de l’ADN sous un microscope à fluorescence (objectif 100x) pour le contrôle de la qualité avant d’ajouter des ligands ou des cellules de placage. Imagez et quantifiez l’intensité de fond moyenne dans le canal Cy3B d’une surface de sonde de tension en épingle à cheveux ADN à partir d’au moins 5 positions différentes et 3 réplications. Maintenir les conditions d’acquisition de l’imagerie cohérentes afin que cette valeur puisse être utilisée comme marqueur fiable de la qualité de surface et de la densité de la sonde (Figure 4C).
NOTE: Quantifier le nombre de brins d’ADN par nanoparticule d’or et le nombre de nanoparticules d’or par μm2 les premières fois de préparation de surface selon la littérature12, qui peut être utilisé comme un autre marqueur fiable de la qualité de surface. - Plaquer ~4 x 10 4 - 10 x 104 cellules sur chaque lamelle de couverture fonctionnalisée de la sonde de tension ADN et leur permettre de se fixer et de se répandre pendant ~15 min à température ambiante.
- Au fur et à mesure que les cellules sont plaquées sur les sondes de tension en épingle à cheveux de l’ADN et commencent à se propager, imagez les signaux de fluorescence générés dans le canal Cy3B avec l’objectif 100x (Figure 3I).
- Une fois que les cellules commencent à produire un signal de tension en temps réel sur la surface de la sonde de tension en épingle à cheveux de l’ADN dans le canal Cy3B, acquérir des images dans les canaux Cy3B et Atto647N (la microscopie TIRF donne un meilleur rapport signal sur bruit que l’épifluorescence). Par la suite, ajouter le brin Atto647N aux chambres d’imagerie à une concentration finale de 200 nM pour une hybridation mécaniquement sélective.
- Après 10 min d’incubation, retirez rapidement et délicatement le tampon contenant le brin de verrouillage fluorescent Atto647N et remplacez-le par des sels équilibrés frais de Hank. Image à nouveau dans les canaux Cy3B et Atto647N et déterminer le coefficient de corrélation de Pearson avec le logiciel Fidji15.
- Au moment d’intérêt pour l’étude, introduire un brin de verrouillage non fluorescent dans les cellules de la chambre d’imagerie pour stocker le signal de tension. Préparer le fil de verrouillage (100 μM) et ajouter aux cellules à une concentration finale de 1 μM. Pipeter doucement pour mélanger. La durée de verrouillage peut varier mais 10 min est le temps recommandé.
- Acquérir des films accélérés ou des images de point final en épifluorescence pour la cartographie qualitative de la tension et l’analyse quantitative au besoin (Figure 3J et Figure 5).
NOTE: Si une mesure de tension à plusieurs points temporels est souhaitée, initier l’effacement des signaux de tension stockés par l’ajout d’un brin de déverrouillage. Pour éviter un rinçage excessif, une concentration finale plus élevée de brin de déverrouillage à 2 μM est utilisée pour déclencher une réaction de déplacement du brin médiée par la prise du brin avec le brin de verrouillage pendant 3 minutes, ce qui efface les signaux stockés (Figure 3J). Rincer doucement les oligonucléotides en excès avec HBSS. La surface de la sonde de tension en épingle à cheveux de l’ADN et les cellules sont prêtes pour un autre cycle de stockage et de cartographie de la tension. Le déverrouillage des signaux de tension n’est pas nécessaire si un seul point temporel présente un intérêt dans l’étude.
4. Analyse des données
REMARQUE: L’analyse d’image est effectuée à l’aide d’un logiciel Fidji et l’analyse quantitative est effectuée à l’aide d’un logiciel d’analyse.
- Corrigez toute dérive pendant l’acquisition d’image avec la commande Corriger la dérive 3D dans Enregistrement sous le menu Plugins .
- Supprimez l’arrière-plan de la caméra de l’image à l’aide de la commande Soustraire dans le menu Processus .
- Déterminez le coefficient de corrélation de Pearson avec la fonction Colocalisation sous le menu Analyser .
- Moyenne et soustraction du fond de fluorescence produit par les sondes non ouvertes de trois régions de fond locales différentes. Dessinez des ROI de cellules sur des images soustraites en arrière-plan ou des images RICM (microscopie à contraste par interférence par réflexion) avec l’outil Image J Freehand Selections . Mesurez toute mesure d’intérêt des ROI, par exemple, l’intensité de fluorescence intégrée et l’occupation de la tension à l’aide de l’outil Mesure du menu Analyser (Figure 6).
- Exportez les mesures pour l’analyse quantitative avec un logiciel d’analyse.
- Tracez les données avec n’importe quel logiciel d’analyse.
Résultats
Nous montrons ici des images représentatives du contrôle de la qualité de surface (Figure 4). Une surface de haute qualité doit avoir un fond propre dans le canal RICM (Figure 4B) et une intensité de fluorescence uniforme dans le canal Cy3B (Figure 4C). Avec le même équipement d’imagerie et des conditions d’acquisition d’imagerie par fluorescence identiques, l’intensité de fluorescence de fond doit être constante et...
Discussion
Avec les procédures détaillées fournies ici, on peut préparer des substrats de sonde de tension en épingle à cheveux ADN pour cartographier et quantifier la tension du récepteur produite par les cellules immunitaires. Lorsque les cellules sont plaquées sur le substrat de la sonde de tension en épingle à cheveux de l’ADN, elles atterrissent, se fixent et se propagent lorsque les récepteurs détectent les ligands à la fois chimiquement et mécaniquement, ce dernier étant détecté par nos sondes. Cependant, ...
Déclarations de divulgation
Les auteurs ne déclarent aucun conflit d’intérêts.
Remerciements
Ce travail a été soutenu par les subventions NIH R01GM131099, NIH R01GM124472 et NSF CAREER 1350829. Nous remercions le NIH Tetramer Facility pour les ligands pMHC. Cette étude a été appuyée, en partie, par le Emory Comprehensive Glycomics Core.
matériels
| Name | Company | Catalog Number | Comments |
| 3-hydroxypicolinic acid (3-HPA) | Sigma | 56197 | maldi-TOF-MS matrix |
| mPEG-SC | Biochempeg | MF001023-2K | surface prep |
| (3-Aminopropyl)triethoxysilane | Acros | AC430941000 | surface prep |
| 10x Red blood cell lysis buffer | Biolegend | 00-4333-57 | buffer |
| 8.8 nm gold nanoparticles, tannic acid | Nanocomposix | customized order | surface prep |
| Atto647N NHS ester | Sigma | 18373-1MG-F | fluorophore, oligo prep |
| Attofluor Cell Chamber, for microscopy | Thermo Fisher Scientific | A7816 | imaging |
| BD Syringes only with Luer-Lok | BD bioscience | 309657 | cells |
| biotinylated anti-mouse CD3e | ebioscience | 13-0031-82 | antibody/ligand |
| Biotinylated pMHC ovalbumin (SIINFEKL) | NIH Tetramer Core Facility at Emory University | NA | antibody/ligand |
| bovine serum albumin | Sigma | 735078001 | block non-specific interactions |
| Cell strainers | Biologix | 15-1100 | cells |
| Coverslip Mini-Rack, teflon | Thermo Fisher Scientific | C14784 | surface prep |
| Cy3B NHS ester | GE Healthcare | PA63101 | fluorophore, oligo prep |
| Dulbecco's phosphate-buffered saline (DPBS) | Corning | 21-031-CM | buffer |
| ethanol | Sigma | 459836 | surface prep |
| Hank’s balanced salts (HBSS) | Sigma | H8264 | buffer |
| hydrogen peroxide | Sigma | H1009 | surface prep |
| LA-PEG-SC | Biochempeg | HE039023-3.4K | surface prep |
| Midi MACS (LS) startup kit | Miltenyi Biotec | 130-042-301 | cells |
| mouse CD8+ T cell isolation kit | Miltenyi Biotec | 130-104-075 | cells |
| Nanosep MF centrifugal devices | Pall laboratory | ODM02C35 | oligo prep |
| No. 2 round glass coverslips | VWR | 48382-085 | surface prep |
| NTA-SAM | Dojindo Molecular Technologies | N475-10 | surface prep |
| P2 gel | Bio-rad | 1504118 | oligo prep |
| sufuric acid | EMD Millipore Corporation | SX1244-6 | surface prep |
| Sulfo-NHS acetate | Thermo Fisher Scientific | 26777 | surface prep |
| Equipment | |||
| Agilent AdvanceBio Oligonucleotide C18 column, 4.6 x 150 mm, 2.7 μm | 653950-702 | oligonucleotide preparation | |
| Barnstead Nanopure water purifying system | Thermo Fisher | water | |
| CFI Apo 100× NA 1.49 objective | Nikon | Microscopy | |
| Cy5 cube | CHROMA | Microscopy | |
| evolve electron multiplying charge coupled device (EMCCD) | Photometrics | Microscopy | |
| High-performance liquid chromatography | Agilent 1100 | oligonucleotide preparation | |
| Intensilight epifluorescence source | Nikon | Microscopy | |
| Matrix-assisted laser desorption/ionization time-of-flight mass spectrometer (MALDI-TOF-MS) | Voyager STR | oligonucleotide preparation | |
| Nanodrop 2000 UV-Vis Spectrophotometer | Thermo Fisher | oligonucleotide preparation | |
| Nikon Eclipse Ti inverted microscope | Nikon | Microscopy | |
| Nikon Perfect Focus System | Nikon | Microscopy | |
| NIS Elements software | Nikon | Microscopy | |
| quad band TIRF 405/488/561/647 cube | CHROMA | Microscopy | |
| RICM cube | CHROMA | Microscopy | |
| TIRF launcher with 488 nm (50 mW), 561 nm (50 mW), and 640 nm | Coherent | Microscopy | |
| TRITC cube | CHROMA | Microscopy | |
| oligo name | 5' modification / 3' modification | sequence (5' to 3') | Use |
| 15mer amine locking strand | 5' modification: no modification 3' modification: /3AmMO/ | AAA AAA CAT TTA TAC CCT ACC TA | locking real-time tension signal |
| 15mer Atto647N locking strand | 5' modification: Atto647N 3' modification: /3AmMO/ | AAA AAA CAT TTA TAC CCT ACC TA | locking real-time tension signal |
| 15mer non-fluoresccent locking strand | 5' modification: no modification 3' modification: no modification | A AAA AAC ATT TAT AC | locking real-time tension signal for quantitative analysis |
| 4.7 pN hairpin strand | 5' modification: no modification 3' modification: no modification | GTGAAATACCGCACAGATGCGT TTGTATAAATGTTTTTTTCATTTAT ACTTTAAGAGCGCCACGTAGCC CAGC | hairpin probe |
| amine ligand strand | 5' modification: /5AmMC6/ 3' modification: /3Bio/ | CGCATCTGTGCG GTA TTT CAC TTT | hairpin probe |
| BHQ2 anchor strand | 5' modification: /5ThiolMC6-D/ 3' modification: /3BHQ_2/ | TTTGCTGGGCTACGTGGCGCTCTT | hairpin probe |
| Cy3B ligand strand | 5' modification: Cy3B 3' modification: /3Bio/ | CGCATCTGTGCG GTA TTT CAC TTT | hairpin probe |
| unlocking strand | 5' modification: no modification 3' modification: no modification | TAG GTA GGG TAT AAA TGT TTT TTT C | unlocking accumulated tension signal |
Références
- Dustin, M. L. T-cell activation through immunological synapses and kinapses. Immunological Reviews. 221 (1), 77-89 (2008).
- Spillane, K. M., Tolar, P. B cell antigen extraction is regulated by physical properties of antigen-presenting cells. Journal of Cell Biology. 216 (1), 217-230 (2017).
- Feng, Y., et al. Mechanosensing drives acuity of αβ T-cell recognition. Proceedings of the National Academy of Sciences. 114 (39), 8204-8213 (2017).
- Hong, J., et al. A TCR mechanotransduction signaling loop induces negative selection in the thymus. Nature Immunology. 19 (12), 1379-1390 (2018).
- Basu, R., et al. Cytotoxic T cells use mechanical force to potentiate target cell killing. Cell. 165 (1), 100-110 (2016).
- Bashour, K. T., et al. CD28 and CD3 have complementary roles in T-cell traction forces. Proceedings of the National Academy of Sciences. 111 (6), 2241-2246 (2014).
- Ma, V. P. Y., Salaita, K. DNA nanotechnology as an emerging tool to study mechanotransduction in living systems. Small. 15 (26), 1900961 (2019).
- Liu, Y., Yehl, K., Narui, Y., Salaita, K. Tension sensing nanoparticles for mechano-imaging at the living/nonliving interface. Journal of the American Chemical Society. 135 (14), 5320-5323 (2013).
- Glazier, R., et al. DNA mechanotechnology reveals that integrin receptors apply pN forces in podosomes on fluid substrates. Nature Communications. 10 (1), 1-13 (2019).
- Galior, K., Liu, Y., Yehl, K., Vivek, S., Salaita, K. Titin-based nanoparticle tension sensors map high-magnitude integrin forces within focal adhesions. Nano Letters. 16 (1), 341-348 (2016).
- Zhang, Y., Ge, C., Zhu, C., Salaita, K. DNA-based digital tension probes reveal integrin forces during early cell adhesion. Nature Communications. 5, 5167 (2014).
- Liu, Y., et al. DNA-based nanoparticle tension sensors reveal that T-cell receptors transmit defined pN forces to their antigens for enhanced fidelity. Proceedings of the National Academy of Sciences. 113 (20), 5610-5615 (2016).
- Zhang, Y., et al. Platelet integrins exhibit anisotropic mechanosensing and harness piconewton forces to mediate platelet aggregation. Proceedings of the National Academy of Sciences. 115 (2), 325-330 (2018).
- Huang, J., et al. The kinetics of two-dimensional TCR and pMHC interactions determine T-cell responsiveness. Nature. 464 (7290), 932-936 (2010).
- Ma, R., et al. DNA probes that store mechanical information reveal transient piconewton forces applied by T cells. Proceedings of the National Academy of Sciences. 116 (34), 16949-16954 (2019).
- Hui, E., et al. T cell costimulatory receptor CD28 is a primary target for PD-1-mediated inhibition. Science. 355 (6332), 1428-1433 (2017).
- Whitley, K. D., Comstock, M. J., Chemla, Y. R. Elasticity of the transition state for oligonucleotide hybridization. Nucleic Acids Research. 45 (2), 547-555 (2016).
- Brockman, J. M., et al. Live-cell super-resolved PAINT imaging of piconewton cellular traction forces. Nature Methods. 17 (10), 1018-1024 (2020).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon