È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Visualizzazione della dinamica di diffusione dei nanorodi d'oro sulla membrana cellulare utilizzando la microscopia a campo scuro a nanoparticella singola
In questo articolo
Riepilogo
Qui, mostriamo l'uso della tradizionale microscopia a campo scuro per monitorare la dinamica delle nanorodi d'oro (AuNR) sulla membrana cellulare. La posizione e l'orientamento dei singoli AuR vengono rilevati utilizzando ImageJ e MATLAB, e gli stati diffusivi degli AuR sono caratterizzati da un'analisi di tracciamento di singole particelle.
Abstract
L'analisi della dinamica di diffusione delle nanoparticelle sulla membrana cellulare gioca un ruolo significativo nella migliore comprensione del processo di assorbimento cellulare e fornisce una base teorica per la progettazione razionale della somministrazione di nano-medicina. L'analisi del single particle tracking (SPT) potrebbe sondare la posizione e l'orientamento delle singole nanoparticelle sulla membrana cellulare, e rivelare i loro stati traslazionali e rotazionali. Qui, mostriamo come usare la tradizionale microscopia a campo scuro per monitorare la dinamica delle nanorodi d'oro (AuNR) sulla membrana cellulare viva. Mostriamo anche come estrarre la posizione e l'orientamento degli AuNAR utilizzando ImageJ e MATLAB e come caratterizzare gli stati diffusivi degli AuNR. L'analisi statistica di centinaia di particelle mostra che i singoli AuRS eseguono il moto browniano sulla superficie della membrana cellulare U87 MG. Tuttavia, l'analisi individuale a traiettoria lunga mostra che gli AuRS hanno due tipi distintamente diversi di stati di movimento sulla membrana, vale a dire il trasporto a lungo raggio e il confinamento ad area limitata. I nostri metodi SPT possono essere potenzialmente utilizzati per studiare la diffusione della superficie o delle particelle intracellulari in diverse cellule biologiche e possono diventare un potente strumento per le indagini su meccanismi cellulari complessi.
Introduzione
La dinamica delle nanoparticelle (NP) sulla membrana è strettamente associata al processo di assorbimento cellulare, che è essenziale per la comprensione delle funzioni cellulari, delle infezioni virali o batteriche e dello sviluppo di sistemi artificiali di somministrazione nanomedicale1,2. La tecnica di single particle tracking (SPT) è uno strumento robusto per caratterizzare i comportamenti eterogenei dei PNP3,4. In generale, la membrana cellulare è fluidica, il che significa che i componenti come proteine e lipidi possono muoversi lateralmente nel piano della membranaplasmatica 5,6,7. La complessità spaziotemporale dell'organizzazione e della struttura della membrana può portare all'eterogeneità spaziotemporale dell'interazione tra NP e membrana. Pertanto, la visualizzazione diretta del movimento dei PNP sulla membrana richiede sia un'alta risoluzione spaziale che temporale.
La microscopia a tracciamento di singole particelle che monitora la localizzazione delle singole particelle nelle cellule viventi con una risoluzione spaziale di decine di nanometri e una risoluzione temporale di millisecondi è stata ben sviluppata per studiare la dinamica dei PN o delle molecole di membrana8,9. Le tecniche di imaging microscopico a base di fluorescenza sono diventate strumenti preziosi per osservare np/molecolenell'ambiente delle cellule viventi 9,10,11,12. Ad esempio, la microscopia a fluorescenza a riflessione interna totale, che immagini strati sottili (~100 nm) del campione all'interfaccia substrato/soluzione con un'alta risoluzione spaziotemporale è stata ampiamente utilizzata negli studi sulla dinamica delle molecole di membrana13,14. Tuttavia, gli svantaggi intrinseci dei singoli fluorofori, come la bassa intensità e la rapida fotobleaching irreversibile, riducono l'accuratezza e la durata del tracciamento13. Pertanto, i PNP plasmonici non fluorescenti, che sostituiscono le sonde fluorescenti, hanno attirato sempre più attenzione negli studi di imaging a lungo termine a causa delle loro caratteristiche otticheuniche 15. Sulla base dei segnali di dispersione delle sonde NP plasmoniche, diversi tipi di tecnologie di imaging microscopico ottico sono stati utilizzati per studiare il meccanismo dei processi biologici, come la microscopia a campo scuro (DFM)16,la microscopia a dispersione interferometrica (iSCAT)17 e la microscopia a contrasto di interferenza differenziale (DICM)18. Inoltre, la dinamica di movimento e rotazione degli AuR può essere ottenuta utilizzando DFM e DICM18,19,20,21,22. Tipicamente, in un esperimento SPT, il movimento dell'oggetto viene registrato dal microscopio ottico, e quindi analizzato dai metodi di analisi SPT3. Le traiettorie risolte nel tempo e gli angoli orientativi generati dai singoli NP sono normalmente stocastico ed eterogeneo, quindi è necessario presentare abbondanti informazioni dinamiche con vari metodi di analisi.
Qui forniamo un protocollo integrato che monitora la dinamica degli AuNR sulla membrana cellulare utilizzando DFM, estrae la posizione e l'orientamento degli AuNR con ImageJ e MATLAB e caratterizza la diffusione degli AuNR con metodi di analisi SPT. Come dimostrazione, mostriamo qui come utilizzare il protocollo SPT per visualizzare la dinamica degli AuNR non modificati (CTAB-AuNRs, sintetizzati dalla molecola di bromuro di ammonio di cetilotrimetilammonio come agente protettivo) sulla membrana cellulare U87 MG. È stato dimostrato che i CTAB-AuNR possono adsorbire le proteine nell'ambiente biologico, muoversi sulla membrana cellulare equindi inserire le cellule 2,20,22. La cellula U87 MG è il tumore più comune e maligno del sistema nervoso centrale e i suoi recettori della membrana sono espressi in modo anomalo. I recettori della membrana possono interagire con le proteine sugli AuNR, che influenzano la dinamica degli AuNR. Il nostro protocollo è generalmente applicabile ad altri esperimenti SPT nel campo della biologia.
Protocollo
1. Coltura cellulare
- Preparare il mezzo completo per le cellule U87 MG aggiungendo siero bovino fetale (concentrazione finale 10%) e penicillina-streptomicina (concentrazione finale 1%) al mezzo essenziale minimo (MEM). Utilizzare il piatto di coltura cellulare in plastica per la sottocoltura delle cellule.
- Celle di passaggio da 2 a 3 volte a settimana.
- Rimuovere il mezzo di coltura e sciacquare lo strato cellulare con la salina tamponata dal fosfato di Dulbecco (D-PBS) 2~3 volte quando è confluente (80%~90%).
- Aggiungere da 1,0 a 2,0 mL di soluzione di tripside-EDTA al piatto di coltura cellulare e osservare le cellule al microscopio invertito fino a quando le cellule non diventano rotonde (3 ~ 5 min).
- Aggiungere 3,0 mL di mezzo completo preparato e disperdere le cellule con pipettando delicatamente.
- Aggiungere la sospensione cellulare (1 mL) al nuovo piatto di coltura con mezzo cellulare fresco (3 mL) e risospendire le cellule.
- Mantenere le cellule a 37 °C e al 5% di CO2 in atmosfera umidificata.
2. Preparazione del vetrino al microscopio
NOTA: Le cellule U87 MG di terza o decima generazione ad alta attività sono utilizzate negli esperimenti SPT.
- Sterilizzare 22 mm × 22 mm di coverslips già puliti con soluzione Piranha immergendosi in etanolo (99,9%).
- Utilizzare le flesse per esegnare la copertura dalla soluzione di etanolo (fase 2.1) e sterilizzare bruciando etanolo sulla fiamma. Una volta bruciato tutto l'etanolo, posizionare le copertine in un piatto di coltura cellulare di plastica (35 x 10 mm) riempito con 2 mL di mezzo cellulare (senza rosso fenolo).
- Aggiungere 50 μL della sospensione cellulare dal passaggio 1.2.3 sul coverslip e spingere delicatamente il piatto avanti e indietro e a sinistra e a destra per distribuire uniformemente le cellule. Posizionare in un'atmosfera umidificata.
- Quando le cellule U87 MG sul coverslip raggiungono il 20%-40% di confluenza (~12 h), aggiungere 20 μL di CTAB-AuNRs (138 pM) nel piatto e disperdersi. Incubare in atmosfera umidificata per 5 minuti.
- Aggiungere 100 μL del mezzo di coltura (senza rosso fenolo) dal piatto al passaggio 2.4 nella scanalatura dello scivolo di vetro scanalato (Figura 1) che viene prepulito con soluzione di Piranha.
- Togliere il coverslip dal piatto e invertirlo sulla parte superiore della scanalatura dello scivolo di vetro del microscopio (Figura 1). Sigillare con smalto per unghie, lasciarlo asciugare e posizionarlo sul palco per eseguire esperimenti SPT.
3. Esecuzione di esperimenti di tracciamento di singole particelle con microscopia a campo scuro (Figura 1).
- Posizionare una goccia di olio sul condensatore a campo scuro immerso nell'olio (NA 1.43-1.20) e ruotare la manopola per fare in modo che il condensatore contatti lo scivolo di vetro.
- Metti una goccia di olio sulla parte superiore del vetro di copertura e ruota la manopola di messa a fuoco per far toccare l'olio con l'obiettivo di immersione dell'olio 60x (NA 0,7-1,25).
- Accendere la sorgente luminosa e ruotare leggermente la manopola di messa a fuoco per mettere a fuoco il piano di imaging.
NOTA: nel campo visivo, lo sfondo è nero, le celle sono luminose e le CTAB-AuNR (proporzioni~2:1, Figura 2) sono piccole macchie di dispersione colorate (rosse, gialle o verdi). - Acquisire la luce di dispersione del campione da una fotocamera CMOS a colori. Fate clic su "Iconafotocamera " nel software per registrare ed esportare il formato TIFF per salvare le immagini.
4. Acquisizione dei dati
- Estrarre una singola traiettoria a lungo termine
- Operare come descritto nella figura 3 per convertire le immagini a campo scuro delle serie temporali dalla modalità "colore RGB" alla modalità "8 bit". Nell'immagine J fare clic su Immagine | Digitare | 8 bit. Per regolare il contrasto, fare clic su Immagine | Regolare | Luminosità | Contrasto.
- Selezionate una particella di destinazione e tagliate gli sfondi delle serie temporali eliminando lo sfondo con "CTRL+X".
- Aprire la finestra rilevamento particelle e collegamento particelle facendo clic su "Plugins | Sistema di monitoraggio delle particelle | Tracker di particelle".
- Impostate Raggio su 6, Taglio a 0 e Percentile allo 0,01%.
NOTA: per rilevare la particella, regolare sopra tre parametri con l'assistenza di Anteprima. Assicurarsi che il raggio sia leggermente più grande della particella mirata e più piccolo della più piccola separazione tra particelle. Percentile è il limite inferiore di distribuzione dell'intensità che deve essere particelle candidate. - Impostate l'intervallo di collegamento su 5 e lo spostamento su 10.
NOTA: per collegare la particella tra fotogrammi adiacenti consecutivi, regolate i due parametri precedenti. Lo spostamento è il pixel massimo che una particella può spostare tra due fotogrammi successivi e Intervallo di collegamenti è il numero di fotogrammi consecutivi da considerare quando si determina la migliore corrispondenza corrispondente. - Fate clic su "OK" per aprire la finestra Risultati particletracker (ParticleTracker Results) per visualizzare i risultati.
- Fate clic su "Visualizza tutte le traiettorie" per ispezionare le traiettorie generate.
- Fate clic sul menu "Ricollegareparticelle " nella parte superiore per ricollegare le particelle rilevate con diversi parametri di intervallo di collegamento e percentile, se la traiettoria generata dal software non corrisponde alla traiettoria in movimento dell'AuNR.
- Fate clic su "Salva reportcompleto " per salvare i risultati se la traiettoria generata dal software e la traiettoria mobile dell'AuNR sono abbinate.
NOTA: nella figura 4 viene illustrato un esempio di estrazione di una singola traiettoria a lungo termine con l'immagine J.
- Estrazione di multi-traiettorie
NOTA: un esempio di estrazione di più traiettorie con le Figi è illustrato nella figura 5. Ci sono diverse fasi e ogni fase costituisce un passo nel processo di tracciamento. Il risultato di ogni passaggio viene visualizzato immediatamente, il che consente all'utente di tornare a riadattare le impostazioni quando l'output non è soddisfacente.- Operare come descritto nella figura 3 per convertire le immagini a campo scuro delle serie temporali dalla modalità "colore RGB" alla modalità "8 bit". Nel percorso Immagine J fare clic su "Immagine | Digitare | 8 bit". Per regolarne il contrasto, fate clic su "Image | Regolare | Luminosità | Contrasto".
- Aprite il pannello iniziale facendo clic su "Plugins | Monitoraggio | TrackMate". Fare clic su "Avanti".
- Scegliete il "Rilevatore LoG" dal menu a discesa e fate clic su "Avanti".
- Regolare i parametri del pannello di configurazione del rilevatore. Impostate il diametro blob stimato su 10, impostate Soglia su 0 e selezionate "Fai localizzazione sottopixi pixel".
- Fate clic su "Avanti" per aprire il pannello di filtraggio spot iniziale.
NOTA: è possibile regolare diversi parametri per ottimizzare ulteriormente i punti di destinazione. In questo esempio, nessun altro parametri è stato regolato. - Fate clic su "Avanti" e selezionate "HyperStack displayer" dal menu a discesa.
- Fate clic su "Avanti" per aprire il pannello filtraggio Spot. Impostare la qualità sopra 1,88, X sopra 38,86, Y sopra 56,54.
- Fate clic su "Avanti" e selezionate "Simple LAP tracker" dal menu a discesa. Configurare il semplice tracker LAP regolando tre parametri, o almeno il linking max distance = 15, gap-closing max distance = 15 e Gap-closing max frame gap = 5.
NOTA: la distanza di collegamento-massimo è lo spostamento massimo di un punto tra due fotogrammi. La distanza massima di chiusura dello spazio è lo spostamento massimo di due segmenti. Lo spazio massimo di chiusura del telaio è il fotogramma più grande tra due punti da colmare. - Fare clic su "Avanti" per tenere traccia. Al termine, continuare a fare clic su "Avanti".
- Impostare filtri sulle tracce, ad esempio impostare il numero di punti nella traccia superiore a 300.
- Continuate a fareclic su" Avanti " fino all'apertura del pannello di salvataggio finale. Selezionate "Esporta tracce in file XML" dal menu a discesa, quindi fate clic su "Esegui" per salvarle in formato CSV.
- Valori R/G
NOTA: le intensità di scattering degli AuNR mirati nei canali R e G sono ottenute da immagini a colori darkfield utilizzando un codice scritto in MATLAB (https://github.com/fenggeqd/JOVE-2020/tree/master/RGandPolarangle) e il principio di estrazione è presentato nella figura 6.- Usate la funzione di xycoordination.m per trovare la coordinata pixel centrale dell'AuNR in ogni fotogramma in base alla coordinata x-y (estratta da ImageJ/Fiji).
- Usa la funzione di RGextraction.m per delimitare una matrice di 3 x 3 pixel, estrarre i 9 valori di intensità di dispersione dei canali R o G e calcolare un valore medio (μ, definito come R o G).
NOTA: la matrice di 3 x 3 pixel è centrata sulle coordinate dei pixel ottenute dal passaggio 4.3.1.
- Angolo polare
NOTA: L'angolo polare è l'angolo tra la longitudine dell'AuNR e l'asse ottico (come mostrato nella figura 1), che può riflettere la dinamica di rotazione spaziale (asse Z) dell'AuNR.- Utilizzare la funzione di polarangle.m calcolare gli angoli polari (θ) con il metodo differenziale a doppio canale22,
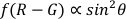 .
.
- Utilizzare la funzione di polarangle.m calcolare gli angoli polari (θ) con il metodo differenziale a doppio canale22,
5. Analisi dei dati
NOTA: Un quadro di analisi dei dati sistematico e solido è essenziale per le prestazioni e l'efficienza dei metodi di analisi SPT. Viene utilizzato il software personalizzato scritto in MATLAB (https://github.com/fenggeqd/JOVE-2020/tree/master/Analysis_parameters). Per disegnare i grafici viene utilizzato un software di grafica eanalisi (vedere Tabella dei materiali).
- Parametri di analisi
- Utilizzare script di csv_data_extract_dis_vel_ss.m e csv_data_MSD.m per calcolare i parametri dinamici in base alle formule mostrate nella tabella 1.
NOTA: Questi parametri vengono utilizzati per analizzare la dinamica degli AuNR e sono costituiti da tre parti. (1) Parametri relativi alla traiettoria: spostamento, dimensione del passo, velocità, raggio di rotazione (Rg)e angolo di rotazione (Ta); (2) Parametri MSD: coefficiente di diffusione (Dt) edesponente anormale della diffusione (α); e (3) Parametri relativi alla rotazione: angolo polare e labilità rotazionale (σ).
- Utilizzare script di csv_data_extract_dis_vel_ss.m e csv_data_MSD.m per calcolare i parametri dinamici in base alle formule mostrate nella tabella 1.
| Misure | Definizione | Significato fisico | |
| Spostamento | 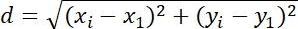 | Cambiamenti nella posizione degli oggetti | |
| Dimensioni del passo | 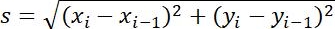 | Distanza tra due punti adiacenti | |
| Velocità |  | Velocità del movimento degli oggetti | |
| Rg | 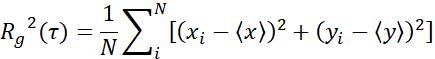 | Spostamento dell'intervallo di oggetti in un intervallo di tempo specifico | |
| Ta | 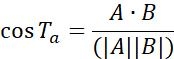 | Direzione del movimento degli oggetti tra due punti adiacenti | |
| Msd | 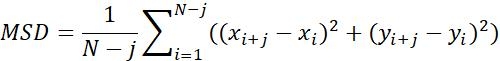 | Distanza media di spostamento degli oggetti in un intervallo di tempo specifico | |
| Dt | 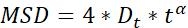 | Capacità di diffusione degli oggetti | |
| Α | 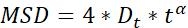 | Diffusione normale (α~1) | |
| Angolo polare |  | Informazioni sull'orientamento 3D degli oggetti | |
| Σ |  | Grado di dispersione del set di dati dell'angolo polare | |
Tabella 1: Tre tipi di parametri utilizzati per l'analisi. Questi includono parametri correlati alla traiettoria (spostamento, dimensione del passo, velocità, Rg e Ta), parametri MSD (MSD, Dt e α) e parametri relativi alla rotazione (angolo polare e labilità rotazionale).
- Analisi visiva della traiettoria
NOTA: la visualizzazione della traiettoria può presentare intuitivamente l'eterogeneità spaziotemporale del moto delle particelle e la distribuzione della traiettoria (coordinata) dei parametri dinamici, come la traiettoria di mappatura del tempo, la traiettoria di mappatura Rg- e Dt- e la traiettoria di mappatura dell'angolo polare. Le traiettorie di mappatura sono state disegnate utilizzando il software di grafica e analisi.- Impostate la coordinata x come X, y coordinate come Y e il tempo (Rg, Dt, angolo polare) come Z.
- Fare clic suPlot | Grafico a dispersione | Plot di mappatura del colore".
- Aggiungere la barra dei colori.
- Analisi MSD
NOTA: L'attività di movimento e la modalità di movimento delle particelle possono essere ottenute mediante analisi MSD23. Più grande è la Dt,più attivo è il moto di diffusione delle particelle. quando α~1, le particelle fanno un normale movimento di diffusione, altrimenti eseguono un movimento di diffusione anomalo.- Dati MSD-τ
- Impostare l'intervallo di tempo (τ) come X, i dati MSD come Y.
- Fare clic suPlot | Grafico a dispersione"
- Adatta i dati facendo clic su "Analizza | Raccordo | Raccordo curva non lineare (Funzione: Allometrico)".
- Figura doppia logaritmica MSD-τ
NOTA: La figura a doppio logaritmico MSD-τ, la cui pendenza è α intercetta è Dt, può presentare intuitivamente il moto delle particelle.- Impostare l'intervallo di tempo logaritmico come X e l'MSD logaritmico come Y.
- Fare clic suPlot | Spargi trama".
- Adatta i dati facendo clic su "Analizza | Raccordo | Raccordo a curva lineare".
- Figura D-τ (DMS/4t)
NOTA: D=MSD/4τ, è una funzione del tempo τ e del fattore di anomalia α, e la figura D-τ mostra direttamente il cambiamento del coefficiente di diffusione con il tempo. Quando D aumenta con il tempo, α è maggiore di 1 e le particelle fanno movimento di superdiffusione.- Impostare l'intervallo di tempo logaritmico (τ) come X e il logaritmico D come Y.
- Fare clic suPlot | Spargi trama".
- Adatta i dati facendo clic su "Analizza | Raccordo | Raccordo curva non lineare (Funzione: "Allometrico")".
NOTA: Più brevi sono le traiettorie, maggiore è l'imprecisione delle stime di diffusione. In generale, sonostati ottenuti D t α attraverso l'analisi MSD-τ del tempo a lungo intervallo (> 30 τ). Tuttavia, il raccordo semplice e ruvido levigerà i dettagli del movimento. Pertanto, l'analisi MSD-τ del tempo a breve intervallo (< 10 τ) deve essere eseguita per analizzare il comportamento di movimento delle particelle in breve tempo.
- Dati MSD-τ
- Analisi statistica
- Analisi statistica multi-particelle
NOTA: L'analisi statistica multi-particelle può riflettere lo stato di moto delle particelle in una regione spaziale, che indica indirettamente l'ambiente di eterogeneità spaziale. Ad esempio, se l'istogramma di Dt mostra una distribuzione su larga scala o multi-picchi, significa che le attività di movimento delle particelle sono eterogenee.- Impostare i parametri dinamici (ad esempio Dt, Rg, spostamento massimo) come Y.
- Fare clic suPlot | Istogramma".
- Fare doppio clic sull'istogramma e impostare le dimensioni della divisione o il numero di divisioni. Fare clic su "Applica".
- Analisi statistica a particella singola
NOTA: L'analisi statistica delle singole particelle può mostrare il comportamento di movimento delle singole particelle, che riflette anche indirettamente l'eterogeneità spaziotemporale dell'ambiente circostante.- Più fotogrammi
- Calcolare i parametri dinamici (ad esempio Ta, dimensione del passo, angolo polare) di tutti i fotogrammi di una singola traiettoria lunga e copiarli nella tabella Origine e impostare come Y.
- Fare clic suPlot | Istogramma".
- Fare doppio clic sull'istogramma e impostare le dimensioni della divisione o il numero di divisioni. Fare clic su "Applica".
NOTA: Se sia Ta che step size mostrano una piccola distribuzione di valore, le particelle espongono un movimento di super-diffusione in piccoli gradini.
- Spostamento delle finestre
- Calcolare i parametri dinamici (ad esempio Rg, Dt) di tutti i fotogrammi di singola traiettoria lunga tramite il metodo della finestra mobile (11 fotogrammi) e copiarli nella tabella Origin e impostare come Y.
- Fare clic suPlot | Istogramma".
- Fare doppio clic sull'istogramma e impostare le dimensioni della divisione o il numero di divisioni. Fare clic su "Applica".
- Più fotogrammi
- Analisi statistica multi-particelle
- Analisi delle serie temporali
NOTA: L'analisi statistica può rivelare lo stato di movimento dei PNP e l'analisi delle serie temporali può presentare il comportamento del movimento come integratore. Combinando diversi parametri delle serie temporali, può discriminare il comportamento di movimento dei PNP a livello temporale e spaziale.- Impostare il tempo come X, i parametri delle serie temporali come Y (ad esempio spostamento, angolo polare e Rg).
- Fare clic suPlot | Diagramma a più riquadri | Trama impilata | Linea + Simbolo".
Risultati
Nel protocollo sono stati utilizzati i CTAB-AuNR non modificati da 40 x 85 nm. Come mostrato nella figura 2B, il suo massimo plasmonico longitudinale a è di ~ 650 nm (regione rossa) e la risonanza trasversale è a 520 nm (regione verde). Letterature precedenti hanno rivelato che le proprietà ottiche (come l'intensità LSPR) degli AuNR plasmonici cambieranno significativamente con il lorodiametro 20,22. Nella fi...
Discussione
Il protocollo presentato viene utilizzato per studiare la dinamica degli AuNR sulla membrana cellulare. Il protocollo è composto da quattro parti, tra cui l'imaging microscopico, l'estrazione dei dati, il calcolo dei parametri dinamici e i metodi di analisi dei dati, e ogni parte è flessibile e universale. Pertanto, ci sono molte possibili applicazioni future, ad esempio, studiando il movimento delle molecole di membrana legate all'NP sulla membrana, la dinamica dell'endocitosi dei recettori etichettati NP, l'analisi d...
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Questo lavoro è stato supportato dalla National Natural Science Foundation of China con numeri di sovvenzione di 21425519, 91853105 e 21621003.
Materiali
| Name | Company | Catalog Number | Comments |
| CTAB coated gold nanorods(CTAB-AuNRs) | Nanoseedz | NR-40-650 | 85 nm * 40 nm |
| Color CMOS camera | Olympus | DP74 | Japan |
| Coverslips | Citoglas | z10212222C | 22*22 mm |
| Dark-field microscopy | Nikon | 80i | upright microscope |
| Fetal bovine serum (FBS) | Gibco | 10099141 | |
| Fiji | National Institutes of Health | 2.0.0-rc-69/1.52 p | a distribution of ImageJ |
| Grooved glass slide | Sail brand | 7103 | Single concave |
| Image J | National Institutes of Health | 1.52 j | |
| MATLAB | MathWorks | R2019b | |
| MATLAB Code | https://github.com/fenggeqd/JOVE-2020 | ||
| Minimum essential medium (MEM) | Gibco | 10-010-CVR | with phenol red |
| Minimum essential medium (MEM) | Gibco | 51200038 | no phenol red |
| Origin | OriginLab | Origin Pro 2018C | |
| Penicillin-streptomycin | Gibco | 15140122 | |
| Plastic cell culture dishes | Falcon | 353002 | |
| Plastic cell culture dishes | Falcon | 353001 | 35*10 mm |
| U87 MG cell | American Type Culture Collection | ATCC HTB-14 | a human primary glioblastoma cell line |
Riferimenti
- Rees, P., Wills, J. W., Brown, M. R., Barnes, C. M., Summers, H. D. The origin of heterogeneous nanoparticle uptake by cells. Nature Communication. 10 (1), 2341 (2019).
- Behzadi, S., et al. Cellular uptake of nanoparticles: journey inside the cell. Chemical Society Reviews. 46 (14), 4218-4244 (2017).
- Shen, H., et al. Single Particle Tracking: From Theory to Biophysical Applications. Chemical Reviews. 117 (11), 7331-7376 (2017).
- Saxton, M. J. Single-particle tracking: connecting the dots. Nature Methods. 5 (8), 671-672 (2008).
- Kusumi, A., et al. Dynamic organizing principles of the plasma membrane that regulate signal transduction: commemorating the fortieth anniversary of Singer and Nicolson's fluid-mosaic model. Annual Review of Cell and Developmental Biology. 28, 215-250 (2012).
- Jacobson, K., Liu, P., Lagerholm, B. C. The Lateral Organization and Mobility of Plasma Membrane Components. Cell. 177 (4), 806-819 (2019).
- Sezgin, E., Levental, I., Mayor, S., Eggeling, C. The mystery of membrane organization: composition, regulation and roles of lipid rafts. Nature Reviews Molecular Cell Biology. 18 (6), 361-374 (2017).
- von Diezmann, A., Shechtman, Y., Moerner, W. E. Three-Dimensional Localization of Single Molecules for Super-Resolution Imaging and Single-Particle Tracking. Chemical Reviews. 117 (11), 7244-7275 (2017).
- Rosenberg, J., Huang, J. Visualizing Surface T-Cell Receptor Dynamics Four-Dimensionally Using Lattice Light-Sheet Microscopy. Journal of Visualized Experiments. (155), e59914 (2020).
- Kusumi, A., Tsunoyama, T. A., Hirosawa, K. M., Kasai, R. S., Fujiwara, T. K. Tracking single molecules at work in living cells. Nature Chemical Biology. 10 (7), 524-532 (2014).
- Rocha, J. M., Gahlmann, A. Single-Molecule Tracking Microscopy - A Tool for Determining the Diffusive States of Cytosolic Molecules. Journal of Visualized Experiments. (151), e59387 (2019).
- Uphoff, S., Sherratt, D. J., Kapanidis, A. N. Visualizing protein-DNA interactions in live bacterial cells using photoactivated single-molecule tracking. Journal of Visualized Experiments. (85), e511177 (2014).
- Pinaud, F., Clarke, S., Sittner, A., Dahan, M. Probing cellular events, one quantum dot at a time. Nature Methods. 7 (4), 275-285 (2010).
- Mehidi, A., et al. Transient Activations of Rac1 at the Lamellipodium Tip Trigger Membrane Protrusion. Current Biology. 29 (17), 2852-2866 (2019).
- Ye, Z., Wang, X., Xiao, L. Single-Particle Tracking with Scattering-Based Optical Microscopy. Analytical Chemistry. 91 (24), 15327-15334 (2019).
- Pan, Q., Zhao, H., Lin, X., He, Y. Spatiotemporal Heterogeneity of Reactions in Solution Observed with High-Speed Single-Nanorod Rotational Sensing. Angewandte Chemie. International Ed. in English. 58 (25), 8389-8393 (2019).
- Taylor, R. W., et al. Interferometric scattering microscopy reveals microsecond nanoscopic protein motion on a live cell membrane. Nature Photonics. 13 (7), 480-487 (2019).
- Chen, K., et al. Characteristic rotational behaviors of rod-shaped cargo revealed by automated five-dimensional single particle tracking. Nature Communication. 8 (1), 887 (2017).
- Xu, D., He, Y., Yeung, E. S. Y. Direct observation of the orientation dynamics of single protein-coated nanoparticles at liquid/solid interfaces. Angewandte Chemie. International Ed. in English. 53 (27), 6951-6955 (2014).
- Lehui, X., Yan, H., Edward, S. Y. Three Dimensional Orientational Imaging of Nanoparticles with Darkfield Microscopy. Analytical Chemistry. 82, 5268-5274 (2010).
- Ge, F., Xue, J., Wang, Z., Xiong, B., He, Y. Real-time observation of dynamic heterogeneity of gold nanorods on plasma membrane with darkfield microscopy. Science China Chemistry. 62, 1072-1081 (2019).
- Tinevez, J. Y., et al. TrackMate: An open and extensible platform for single-particle tracking. Methods. 115, 80-90 (2017).
- Janczura, J., Weron, A. Ergodicity testing for anomalous diffusion: small sample statistics. The Journal of Chemical Physics. 142 (14), 144103 (2015).
- Kim, D. H., et al. Single particle tracking-based reaction progress kinetic analysis reveals a series of molecular mechanisms of cetuximab-induced EGFR processes in a single living cell. Chemical Science. 8 (7), 4823-4832 (2017).
- Kurzthaler, C., et al. Probing the Spatiotemporal Dynamics of Catalytic Janus Particles with Single-Particle Tracking and Differential Dynamic Microscopy. Physical Review Letters. 121 (7), 078001 (2018).
- Lin, X., Pan, Q., He, Y. In situ detection of protein corona on single particle by rotational diffusivity. Nanoscale. 11 (39), 18367-18374 (2019).
- Wei, L., et al. Sub-diffraction-limit localization imaging of a plasmonic nanoparticle pair with wavelength-resolved dark-field microscopy. Nanoscale. 9 (25), 8747-8755 (2017).
- Cheng, X., Dai, D., Xu, D., He, Y., Yeung, E. S. Subdiffraction-limited plasmonic imaging with anisotropic metal nanoparticles. Analytical Chemistry. 86 (5), 2303-2307 (2014).
- Belyy, V., et al. PhotoGate microscopy to track single molecules in crowded environments. Nature Communication. 8, 13978 (2017).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon