Method Article
Generación eficiente y consistente de epitelio pigmentario retiniano/cordoides de montaje plano a partir de ojos humanos para análisis histológico
En este artículo
Resumen
Describimos un método para separar eficientemente el epitelio pigmentario de la retina (EPR) de la retina en los ojos humanos y generar montones planos completos de EPR / coroides para análisis histológicos y morfométricos del EPR.
Resumen
El epitelio pigmentario de la retina (EPR) y la retina son tejidos conectados funcional y estructuralmente que trabajan juntos para regular la percepción de la luz y la visión. Las proteínas en la superficie apical del EPR están estrechamente asociadas con las proteínas en la superficie del segmento externo del fotorreceptor, lo que dificulta la separación consistente del EPR de los fotorreceptores/retina. Desarrollamos un método para separar eficientemente la retina del EPR de los ojos humanos para generar montajes planos completos de EPR / coroides y retina para el análisis celular separado de los fotorreceptores y las células del EPR. Una inyección intravítrea de una solución de alta osmolaridad de D-manitol, un azúcar no transportado por el EPR, indujo la separación del EPR y la retina a través de toda la cámara posterior sin causar daño a las uniones celulares del EPR. No se observaron parches de EPR adheridos a la retina. El marcado de faloidina de la actina mostró la preservación de la forma del EPR y permitió el análisis morfométrico de todo el epitelio. Se desarrolló un software basado en inteligencia artificial (IA) para reconocer y segmentar con precisión los bordes celulares del EPR y cuantificar 30 métricas de forma diferentes. Este método de disección es altamente reproducible y puede extenderse fácilmente a otros modelos animales.
Introducción
El epitelio pigmentario de la retina (EPR) y la retina neural están fuertemente interconectados entre sí debido a la fuerte dependencia fisiológica de los fotorreceptores en el EPR. Durante la disección, la separación mecánica de la retina neural del EPR causa el desgarro de las células del EPR, con las porciones apicales del EPR unidas a los segmentos externos de los fotorreceptores de la retina. El grado de adhesión EPR-retina es tan grande que la cantidad de pigmento que queda en la retina después de la separación se utiliza para cuantificar la fuerza de la adhesión retiniana1. Específicamente, las uniones estrechas del EPR y la estructura de actina que las conecta, que se encuentran en el lado apical, se rompen durante la separación mecánica. Por lo tanto, la tinción de los montajes planos del EPR para los bordes de las celdas da como resultado una monocapa irregular en la que muchas células tienen bordes faltantes. Este efecto se exacerba cuando el tejido se fija con paraformaldehído (PFA) antes de la disección, a medida que las proteínas se reticulan.
Estudios sobre la administración intravítrea de fármacos han demostrado que las inyecciones de soluciones hiperosmóticas en la cámara posterior inducen desprendimiento de retina 2,3. En estos experimentos, 50 μL de diferentes soluciones, que van desde 1.000 mOsm a 2.400 mOsm, inyectados en el vítreo medio causaron desprendimiento de retina en cuestión de minutos. En particular, incluso después de largas exposiciones a soluciones de alta osmolaridad, las uniones estrechas del EPR aparecieron intactas en las imágenes microscópicas electrónicas de transmisión de ojos de conejo y mono3. Siguiendo una estrategia similar, inyectamos en el vítreo medio una solución hiperosmótica de D-manitol para inducir un desprendimiento de retina eficiente antes de realizar la disección del EPR. Como el D-manitol no es transportado por el EPR4, se mantiene una alta concentración intravítrea, generando un gradiente osmótico. La separación eficiente del EPR y la retina en toda la cámara posterior garantiza la preservación de las uniones celulares del EPR y permite el estudio de la morfometría del EPR en todo el montaje plano. Además, desarrollamos un software basado en inteligencia artificial (IA) que reconoce y segmenta los bordes celulares del EPR etiquetados con fluorescencia, cuantifica 30 métricas de forma diferentes y produce mapas de calor de cada métrica para la visualización 5,6.
Protocolo
Los globos humanos de cadáveres se obtuvieron de la Advanced Sight Network (Birmingham, AL). El trabajo realizado en tejido de cadáver está exento por la Junta de Revisión Institucional de los NIH del comité de ética de investigación.
1. Envío de globo ocular
- Después de la enucleación, envíe globos oculares frescos en un recipiente lleno de DPBS 1x helado con Ca 2+ y Mg2+.
NOTA: Es mejor diseccionar el ojo dentro de las 24 h después de la enucleación. La morfología del EPR no se altera durante este período de tiempo.
2. Preparación del molde de silicona
- Corte los 20 mm inferiores de un tubo de fondo redondo de 25 mm de diámetro. Colóquelo en la base de un barco de pesaje cuadrado (81 mm x 81 mm x 25 mm).
- Mezcle los dos componentes del kit de elastómero de silicio en una proporción de 10: 1, prestando atención a no atrapar el aire. Vierta la mezcla en el bote de pesaje que contiene la pieza esférica del tubo de fondo redondo.
- Curar la silicona a temperatura ambiente durante la noche. Retire el bote de pesaje y el tubo de fondo redondo del molde de silicona curado.
3. Disección del EPR
- Limpie la esclerótica del globo ocular fresco de los músculos y el tejido conectivo. Asegure el ojo al molde de silicona con agujas de 27 G enhebradas a través de la esclerótica. Llenar la cavidad del molde con DPBS 1x con Ca2+ y Mg 2+.
NOTA: Preste atención a no romper la cámara ocular. Las agujas solo deben pasar a través de la esclerótica. - Use una jeringa de 1 ml y una aguja de 21 g para inyectar ~ 400 μL de solución de D-manitol de 1,700 mOsm en el vítreo. Inserte la aguja a través de la pars plana para evitar perforar la cámara anterior del ojo. Deje el ojo a temperatura ambiente durante ~ 45 min.
- Corte la cámara anterior al nivel de la pars plana con un par de tijeras finas y fórceps. Llene la cámara posterior del ojo con DPBS 1x con Ca 2+ y Mg2+. Bajo el microscopio estereoscópico, localice la mácula (la mancha amarilla en la retina).
- Si hay un cortador vítreo quirúrgico disponible, retire el vítreo y reemplácelo con DPBS 1x con Ca 2+ y Mg2+. Alternativamente, intente levantar el vítreo con fórceps y córtelo con tijeras finas.
- Prestando atención a preservar la región macular, corte el ojo en cuadrantes: nasal, temporal, superior e inferior. Retire las agujas si están en el camino.
- Transfiera la cámara posterior del ojo a una placa de Petri de 100 mm que contenga DPBS 1x con Ca 2+ y Mg2+. Antes de extraer la retina, marque el pétalo que contiene la mácula haciendo un pequeño corte (en forma de V) en el margen ciliar. Levante y corte todo el vítreo que se encuentra en la retina.
- Levante suavemente la retina desde múltiples lados con una espátula curva o un par de fórceps para verificar si la retina se ha desprendido del EPR y para permitir que circule algo de líquido entre las dos capas.
NOTA: La retina todavía estará unida en la misma periferia (márgenes ciliares) y en el nervio óptico. - Cortar la retina de los márgenes ciliares en todos los pétalos, asegurándose de no rayar el EPR. Coloque el tejido en PFA al 4% e incube durante ~ 1 h. Lavar 3x con DPBS 1x con Ca 2+ y Mg2+. Transfiera el tejido a un recipiente lleno de DPBS 1x con Ca 2+ y Mg2+ y guárdelo a 4 °C.
NOTA: En este punto, la retina solo está unida al nervio óptico. Este es un punto de pausa en este experimento. - Transfiera la muestra a una placa de Petri de 100 mm que contenga DPBS 1x con Ca 2+ y Mg2+. Perfore la cabeza del nervio óptico con un punzón de biopsia de 1,5 mm y recoja la retina. Almacenar la retina neural en DPBS 1x con Ca 2+ y Mg2+ a 4 °C.
NOTA: Antes de perforar la cabeza del nervio óptico, asegúrese de cortar el nervio óptico tanto como sea posible en el lado escleral. Esto aumentará la precisión del golpe. Perforar el nervio óptico después de que el montaje plano se fija en PFA al 4% reduce el daño a las células RPE ubicadas alrededor del nervio óptico. - Retire la esclerótica del EPR / coroides levantando suavemente la capa de EPR / coroides de la periferia y cortando los vasos coroideos y el tejido conectivo que se encuentran entre la esclerótica y el EPR con un par de tijeras de resorte Vannas. Una vez que el EPR/coroides esté completamente separado de la esclerótica, recoja la capa del EPR/coroides. Transfiera el tejido a un recipiente lleno de DPBS 1x con Ca 2+ y Mg2+ y guárdelo a 4°C.
NOTA: En este momento, el experimento se puede pausar.
4. Tinción
- Transfiera el EPR/coroides a un pocillo de una placa de 6 pocillos. Bloquear y permeabilizar la muestra en DPBS 1x con Ca 2+ y Mg 2+ con albúmina sérica bovina (BSA) al 1%, Tween20 al 0,5% y Triton X-100 al 0,5% durante 1 h a temperatura ambiente.
- Incubar la muestra con faloidina conjugada con un fluoróforo 647 en una dilución 1:250 en DPBS 1x con Ca 2+ y Mg 2+ con BSA al 1%, Tween20 al 0,5% y Triton X-100 al 0,5% durante 1 h a temperatura ambiente. Lavar 3x en DPBS 1x con Ca 2+ y Mg2+.
- Transfiera la muestra de EPR/coroides a un portaobjetos de vidrio de 50 mm x 75 mm y aplanarlo. Corte cada "pétalo" en dos para que la muestra sea más plana. Presta atención a la mácula. Dibuja un contorno del flat mount con un bolígrafo hidrófobo.
- Para apagar la autofluorescencia de lipofuscina, agregue 500 μL de la solución de autofluorescencia diluida a 1:20 en etanol al 70% e incube a temperatura ambiente durante 2 min.
- Lavar bien (al menos 3x) en DPBS 1x con Ca 2+ y Mg2+. Retire el DPBS y agregue el medio de montaje. Coloque una cubierta de vidrio en el montaje plano y selle con esmalte de uñas.
- Imagen del montaje plano con un microscopio de fluorescencia (preferiblemente usando un objetivo de 10x o 20x).
5. Análisis REShAPE
NOTA: Como el algoritmo basado en IA REShAPE fue entrenado en imágenes 10x y 20x, se recomienda encarecidamente usar un objetivo 10x o 20x al crear imágenes. De lo contrario, las imágenes deberán reescalarse en consecuencia.
- Si las imágenes se adquieren con más de un canal fluorescente, aísle el canal utilizado para adquirir los bordes celulares. Exporte las imágenes como archivos TIF en escala de grises de 16 bits.
NOTA: Las imágenes adquiridas con la extensión de archivo .czi no necesitan ser exportadas como archivos TIF, pero el canal que contiene los bordes de la celda todavía necesita ser aislado. - Instale el software en plataformas Windows x64 o Linux (Centos 7).
NOTA: El software y las instrucciones de instalación se pueden encontrar en https://github.com/nih-nei/REShAPE. - Abra el software (Figura 1).
- En la pestaña Directorios , seleccione las carpetas de entrada y salida. En el directorio de entrada, seleccione la carpeta que contiene las imágenes. En el directorio de salida, deje que el software cambie automáticamente la ruta a " directorio de entrada / Procesado". Como alternativa, cambie el directorio de salida manualmente.
NOTA: El software iterará a través de todas las imágenes contenidas en la carpeta de entrada. - En la pestaña Opciones de segmentación NN, elija la siguiente configuración para la segmentación de imágenes:
- Tamaño del lote: escriba un valor de 20 en el cuadro de texto como un buen punto de partida, pero bájelo si el sistema se queda sin unidades de procesamiento de gráficos (GPU).
NOTA: La imagen original se divide en mosaicos más pequeños para su procesamiento. El tamaño del lote especifica cuántos mosaicos se pueden procesar a la vez. - ¿Usar GPU?: Si el sistema no tiene suficientes recursos de GPU, marque la casilla No.
- Escala interna: Usando el menú desplegable, cambie la escala interna de Ninguno en los casos en que las imágenes no se adquirieron con un objetivo de 10x o 20x. Por ejemplo, reduzca la escala de las imágenes de 40x a la mitad del tamaño con el botón 1/2 . Las opciones de reescalado disponibles en el menú desplegable son las siguientes: 5x, 2x, Ninguno, 1/2, 1/5.
NOTA: El aprendizaje automático se entrenó con imágenes de 10x y 20x. Es posible que no reconozca los bordes de las celdas de las imágenes tomadas con otros aumentos. En este caso, las imágenes binarias aparecerán completamente negras. - Filtro Arti: Utilice la pestaña del filtro de artefactos para eliminar partículas muy brillantes (residuos o pequeñas partes de la retina o tejido conectivo alrededor del nervio óptico) que pueden estar presentes en la imagen y pueden interferir con la segmentación del borde celular. No se utiliza ningún filtro de forma predeterminada. Las opciones de filtro artificial en el menú desplegable son las siguientes: Regular, Fuerte y Débil.
- Tamaño del lote: escriba un valor de 20 en el cuadro de texto como un buen punto de partida, pero bájelo si el sistema se queda sin unidades de procesamiento de gráficos (GPU).
- En la pestaña Opción de imagen en mosaico, inserte un valor en el cuadro de texto para especificar la cantidad de superposición entre los mosaicos de imagen ajustando el parámetro Mosaicos de relleno por . Una superposición de 100 píxeles generalmente funciona bien.
- En la pestaña Opciones de gráfico de salida , ajuste la configuración para la generación de mapas de calor:
- ¿Reconstruir imágenes en mosaico?: si el sistema no tiene suficientes recursos para el análisis de imágenes grandes, marque la casilla No para permitir que el software complete el análisis guardando mosaicos individuales sin reconstruir toda la imagen.
- Crear imágenes en color: Para generar mapas de calor, selecciona Todo en el menú desplegable.
- Para Colorear, elija cualquiera de las diferentes paletas de colores disponibles en el menú desplegable: Térmica, Verde, Mpl-magma, Fase, Fuego, Chorro, Cian Caliente.
- Para Formato de imagen, guarde las imágenes como TIF o PNG seleccionando una de las opciones de la pestaña.
- Con respecto a la función ¿Usar límites manuales? , marque la casilla No para permitir que el software use los valores mínimos y máximos detectados en cada imagen. Marque la casilla Sí para ajustar manualmente el rango de valores para cada mapa de calor métrico de forma, y haga clic en el botón Establecer límites para elegir rangos para los parámetros individuales insertando valores en los cuadros de texto. Después de cambiar los valores de interés, haga clic en Guardar. Haga clic en Cargar valores predeterminados para restablecer todos los límites.
NOTA: Utilice límites manuales si es necesario comparar mapas de calor de varias imágenes, como cuando se comparan los efectos de diferentes compuestos en la forma de la célula. De esta manera, se utiliza el mismo rango de valores. El conjunto de valores que deben introducirse manualmente varía en función del tipo de muestra. Se recomienda ejecutar algunas iteraciones para elegir el rango óptimo.
- En la ficha Restricciones de tamaño de celda para análisis, seleccione un umbral de tamaño de celda para el análisis:
- En Tamaño de celda inferior, inserte en el cuadro de texto el tamaño de la celda más pequeña que se incluirá en el análisis.
- En Tamaño de celda superior, inserte en el cuadro de texto el tamaño de la celda más grande que se incluirá en el análisis.
NOTA: La unidad de tamaño de celda cambia de píxeles a micrómetros al cuadrado dependiendo de la opción elegida en la ficha Conversión automática de unidades .
- En la ficha Conversión automática de unidades , elija la unidad preferida para el análisis:
- En ¿Convertir píxeles en unidades reales?, marque la casilla No para ejecutar el análisis en unidades de píxeles. Marque la casilla Sí para ejecutar el análisis en micrómetros.
- En Longitud de la barra de escala (píxeles), introduzca el valor de píxel en el cuadro de texto.
- En Longitud de la barra de escala (micras), introduzca en el cuadro de texto la distancia correspondiente en micrómetros.
- Para iniciar el análisis, pulse Ir por ello.
NOTA: El software también puede medir la viabilidad celular cuando las células se tiñen con 4',6-diamidino-2-fenilindol (DAPI) y yoduro de propidio (pestaña de opciones CZI ), pero esto no se aplica a los montajes planos de RPE.
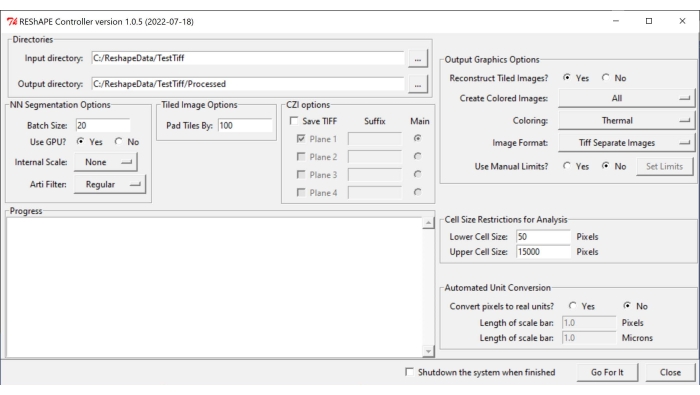
Figura 1: Interfaz gráfica de usuario de REShALE. La GUI tiene diferentes pestañas para seleccionar los directorios de trabajo (pestaña Directorios), modificar las opciones de segmentación (pestañas Opciones de segmentación NN y Opciones de imagen en mosaico), especificar los parámetros para el análisis (pestañas Restricciones de tamaño de celda para análisis y Conversión automática de unidades) y para la generación de mapas de calor (pestaña Opciones de gráficos de salida). Abreviatura: GUI = interfaz gráfica de usuario. Haga clic aquí para ver una versión más grande de esta figura.
Resultados
Este protocolo da como resultado una imagen de un solo plano de un montaje plano, donde la ubicación de la celda y las 30 métricas de forma se miden para cada celda RPE identificada correctamente (Figura 2). Una carpeta llamada "Procesada" se genera automáticamente dentro del directorio de entrada. Esta carpeta contiene cuatro subdirectorios, denominados "Análisis", "Codificado por colores", "Archivos combinados" e "Imágenes segmentadas", y algunos archivos temporales generados durante el análisis. La carpeta "Archivos combinados" contiene una hoja de cálculo con todas las medidas de forma y una hoja de cálculo con las frecuencias de los recuentos de vecinos de celda de todos los archivos combinados. La carpeta "Análisis" contiene una hoja de cálculo con todas las medidas de forma y una hoja de cálculo con las frecuencias de los recuentos de vecinos de celda para cada imagen por separado. El directorio "Imágenes segmentadas" contiene las máscaras binarias finales de los bordes de celda RPE; Se puede utilizar para evaluar la calidad de la segmentación. El directorio "Codificado por colores" contiene mapas de calor para cada medida de forma para visualizar los patrones de forma en cada imagen. Las definiciones y abreviaturas de las métricas de forma se pueden encontrar en la Tabla 1.
A veces, los soportes planos de EPR pueden contener piezas residuales de retina que no se eliminaron limpiamente, especialmente alrededor del nervio óptico. La tinción de faloidina de la muestra da como resultado una señal fuerte proveniente de la retina, y esto puede causar problemas para la segmentación del borde celular del EPR. Algunos mosaicos aparecerán completamente negros, mientras que los mosaicos circundantes mostrarán una segmentación normal. Otros objetos brillantes que pueden estar presentes en la imagen también causarán la generación de mosaicos negros (Figura 3). En estos casos, elegir una de las opciones de filtrado (Débil, Regular, Fuerte) disponibles en el menú desplegable Filtro arti evitará la formación de mosaicos negros.
REShAPE toma imágenes en escala de grises de 8 bits o 16 bits como entrada, pero no imágenes RGB. El uso de imágenes RGB para el análisis REShAPE producirá imágenes binarias completamente negras. Si esto ocurre, la conversión de las imágenes RGB a escala de grises producirá imágenes binarias segmentadas correctamente (Figura 4). En algunas ocasiones en las que los bordes del EPR no se reconocen correctamente, por ejemplo, si la tinción no es óptima o si la muestra está dañada por un rasguño (Figura 5A), se pueden identificar grandes grupos de células como una sola célula muy grande (Figura 5B). En este caso, los objetos grandes se pueden excluir del análisis reduciendo el umbral de tamaño de celda (Figura 5C). Esto se puede lograr insertando un valor inferior en el cuadro de texto Tamaño de celda superior . Sin embargo, esto resultará en un cambio en el rango del mapa de calor. Si un investigador decide hacerlo, también es posible mantener el rango original del mapa de calor (Figura 5D) marcando la casilla Sí en la función ¿Usar límites manuales? Posteriormente, el investigador debe hacer clic izquierdo en el botón Establecer límites e insertar los valores deseados en los cuadros de texto para especificar los límites manuales.
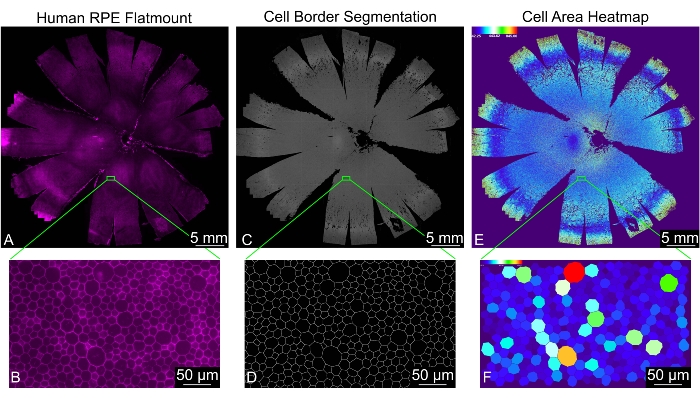
Figura 2: Análisis morfométrico completo de una monocapa completa de EPR humano. (A) Una vista de bajo aumento de un RPE/coroides humano completo de montaje plano (magenta: faloidina). (B) Una vista ampliada de las células RPE teñidas con faloidina. (C) segmentación generada por REShAPE de los bordes de la celda RPE para un RPE/coroides humano completo de montaje plano y (D) la vista ampliada correspondiente. (E) Un mapa de calor generado por software que ilustra el área celular de las células RPE individuales en todo el montaje plano humano. La escala térmica en la esquina superior izquierda muestra el rango de valores utilizados. (F) La vista ampliada correspondiente que muestra celdas individuales de RPE coloreadas por área. Barras de escala = (B,D,F) 50 μm, (A,C,E) 5 mm. Abreviatura: RPE = epitelio pigmentario de la retina. Haga clic aquí para ver una versión más grande de esta figura.
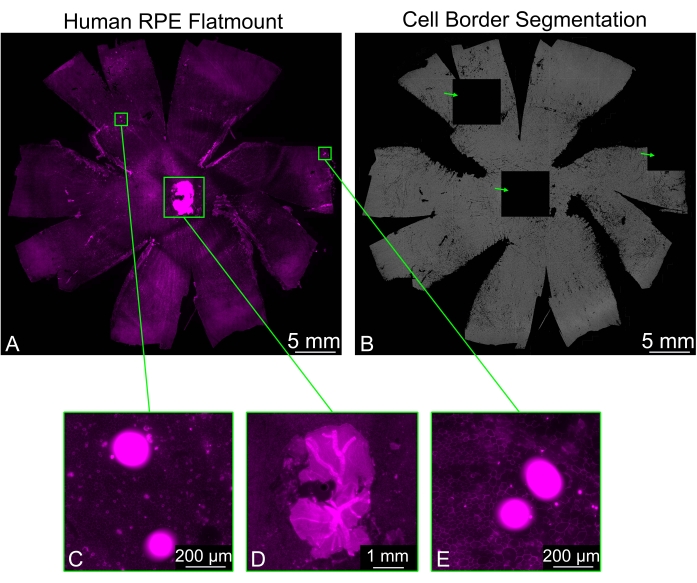
Figura 3: Filtrado de artefactos brillantes. (A) Un RPE humano de montaje plano teñido para bordes celulares (magenta: faloidina) puede presentar áreas brillantes (rectángulos verdes) que interfieren con la segmentación. (B) La segmentación del borde de la celda RPE de todo el montaje plano contiene tres mosaicos completamente negros (flechas verdes) correspondientes a regiones brillantes de fluorescencia. (C,E) Dos de los azulejos negros corresponden a áreas que contienen puntos brillantes, que posiblemente sean algunos escombros. (D) Una de las baldosas negras fue generada por un pedazo de retina neural alrededor del nervio óptico que no se eliminó correctamente. Las piezas de la retina neural son considerablemente más brillantes que la capa RPE y dificultan la segmentación celular. Haga clic aquí para ver una versión más grande de esta figura.

Figura 4: Requisito de imagen de entrada. Las celdas RPE teñidas para los bordes de celda se guardaron como (A) RGB o como (B) imágenes de 16 bits en escala de grises para el análisis REShAPE. (C) La salida del análisis de imagen RBG es una imagen binaria negra, (D) mientras que el análisis de la imagen en escala de grises produce un binario correctamente segmentado de los bordes de las celdas. REShAPE solo puede analizar imágenes en escala de grises de 8 bits o 16 bits. Barras de escala = 50 μm. Haga clic aquí para ver una versión más grande de esta figura.

Figura 5: Resultados subóptimos . (A) Una imagen de una porción de la monocapa de EPR donde las células teñidas con faloidina se rayaron accidentalmente. (B) Un mapa de calor de las celdas del EPR coloreadas por la dimensión del área celular. Un umbral de tamaño de celda superior grande incluye objetos grandes en el análisis. (C) Un mapa de calor del área de la celda en el que se eligió un umbral de tamaño de celda superior más pequeño para excluir objetos grandes del análisis. (D) Un mapa de calor del área de la celda en el que se eligió un umbral de tamaño de celda superior más pequeño y se establecieron límites manuales para mantener el rango de mapa de calor utilizado originalmente. Haga clic aquí para ver una versión más grande de esta figura.
Tabla 1: Parámetros REShALE. La tabla informa la definición de cada parámetro y las abreviaturas utilizadas en las hojas de cálculo sin procesar (archivos "_Data.csv") y para los mapas de calor. Haga clic aquí para descargar esta tabla.
Discusión
La separación consistente y eficiente del EPR humano y las retinas se puede lograr utilizando este protocolo. Este método permite el estudio de las diferencias regionales en la forma del EPR en retinas humanas completas5. Un paso crucial en el protocolo es la separación física del EPR y la retina. Si los dos tejidos no están completamente separados en algunas áreas, uno debe levantar suavemente la retina, asegurándose de no romper los tejidos. El análisis REShAPE de grandes montajes planos puede requerir el uso de sistemas con considerables recursos de RAM. En este caso, el reensamblaje de toda la imagen se puede deshabilitar para permitir que el software finalice con éxito el análisis a pesar de la falta de recursos de procesamiento.
La principal limitación del uso de REShAPE para segmentar los montajes planos de EPR humano es que el algoritmo de IA se entrenó principalmente en imágenes de RPE derivado de células madre pluripotentes inducidas. Como consecuencia, la segmentación de los montajes planos del EPR humano es menos precisa. Las células del EPR de donantes envejecidos contienen una gran cantidad de lipofuscina7, y el amplio espectro de su autofluorescencia interfiere con la segmentación del borde celular. En el futuro, se utilizarán más imágenes de montajes planos de RPE para mejorar la segmentación del borde celular en este tipo de muestra. A pesar de esta limitación, REShAPE fue entrenado específicamente para reconocer y segmentar los bordes de las células del EPR y funciona mejor que otros métodos existentes, como la segmentación de células del EPR Voronoi8 y CellProfiler9 .
Además, en comparación con la segmentación manual 10, REShAPE ofrece la ventaja de analizar imágenes grandes rápidamente (se probaron ~ 130,000 píxeles x130,000 píxeles). En conclusión, este método de disección es altamente reproducible y puede extenderse fácilmente a otros modelos animales. Además, el software se puede utilizar para estudiar la forma del EPR en montajes planos de ojos o en modelos de cultivo celular para examinar el efecto de ciertos tratamientos. Finalmente, la versatilidad de REShAPE lo hace ampliamente aplicable para el análisis de otros tipos de células epiteliales.
Divulgaciones
Los autores no tienen conflictos de intereses que revelar.
Agradecimientos
Agradecemos al núcleo histológico del Instituto Nacional del Ojo (NEI) por el uso del Zeiss Axio Scan.Z1. También agradecemos a los donantes, sus familias, a Advancing Sight Network y al Lions Eye Institute por su generosidad. Este trabajo fue apoyado por los fondos NEI IRP (número de subvención ZIA EY000533-04).
Materiales
| Name | Company | Catalog Number | Comments |
| Biopsy punch 1.5 mm | Acuderm Inc. | P1525 | |
| Bovine albumin | MP Biomedicals | 160069 | |
| Coverglass 50 x 75 mm, #1.5 thickness | Brain Research Laboratories | 5075-1.5D | |
| Curved spatula | Katena | K3-6600 | |
| D-Mannitol | Sigma | M9546 | |
| DPBS 1x with Ca2+ and Mg2+ | Gibco | 14040-133 | |
| Fine Scissors | Fine Science Tools | 14558-11 | |
| Fluormount-G | Southern Biotech | 0100-01 | |
| Forceps - Dumont #5 | Fine Science Tools | 11252-23 | |
| Microscope slides 50 x 75 x 1.2 mm | Brain Research Laboratories | 5075 | |
| Needles 21 G x 1-1/2" hypodermic | Becton Dickinson (BD) | 305167 | |
| Needles 27 G x 1-1/4" hypodermic | Becton Dickinson (BD) | 305136 | |
| Paraformaldehyde 16% | Electron Microscopy Sciences | 15710 | |
| Petri dish 100 mm | Corning | 430167 | |
| Phalloidin-iFluor 647 | Abcam | ab176759 | |
| Razor blades | PAL (Personna) | 62-0177 | |
| Round bottom tubes 50 mL | Newegg | 9SIA4SR9M88854 | |
| Silicon Elastomer Kit | Dow Corning Corporation | 4019862 | |
| Square weighing boat (81 mm x 81 mm x 25 mm) | Sigma | W2876 | |
| Surgical Vitrectomy System | BD Visitrec | 585100 | optional |
| Syringe 1 mL | Becton Dickinson (BD) | 309659 | |
| Triton X-100 | Sigma | T9284 | |
| TrueBlack | Biotium | 23007 | autofluorescence quencher |
| Tween 20 | Affymetrix | 20605 | |
| Vannas Spring Scissors - 3 mm cutting edge | Fine Science Tools | 15000-10 |
Referencias
- Endo, E. G., Yao, X. Y., Marmor, M. F. Pigment adherence as a measure of retinal adhesion: Dependence on temperature. Investigative Ophthalmology and Visual Science. 29 (9), 1390-1396 (1988).
- Marmor, M. F. Retinal detachment from hyperosmotic intravitreal injection. Investigative Ophthalmology and Visual Science. 18 (12), 1237-1244 (1979).
- Marmor, M. F., Martin, L. J., Tharpe, S. Osmotically induced retinal detachment in the rabbit and primate. Electron microscopy of the pigment epithelium. Investigative Ophthalmology and Visual Science. 19 (9), 1016-1029 (1980).
- Ban, Y., Rizzolo, L. A culture model of development reveals multiple properties of RPE tight junctions. Molecular Vision. 3, 18 (1997).
- Ortolan, D., et al. Single-cell-resolution map of human retinal pigment epithelium helps discover subpopulations with differential disease sensitivity. Proceedings of the National Academy of Sciences of the United States of America. 119 (19), 2117553119 (2022).
- Schaub, N. J., et al. Deep learning predicts function of live retinal pigment epithelium from quantitative microscopy. Journal of Clinical Investigation. 130 (2), 1010-1023 (2020).
- Beatty, S., Koh, H. -. H., Phil, M., Henson, D., Boulton, M. The role of oxidative stress in the pathogenesis of age-related macular degeneration. Survey of Ophthalmology. 45 (2), 115-134 (2000).
- Liu, Z., Kocaoglu, O. P., Miller, D. T. 3D imaging of retinal pigment epithelial cells in the living human retina. Investigative Ophthalmology and Visual Science. 57 (9), 533-543 (2016).
- Bhatia, S. K., et al. Analysis of RPE morphometry in human eyes. Molecular Vision. 22, 898-916 (2016).
- vonder Emde, L., et al. Histologic cell shape descriptors for the retinal pigment epithelium in age-related macular degeneration: A comparison to unaffected eyes. Translational Vision Science & Technology. 11 (8), 19 (2022).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados