A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
سير العمل التجريبي وتحليل البيانات للمسافة البادئة النانوية للمادة اللينة
In This Article
Summary
يقدم البروتوكول سير عمل كامل لتجارب المسافة البادئة النانوية للمواد اللينة ، بما في ذلك الهلاميات المائية والخلايا. أولا ، الخطوات التجريبية للحصول على بيانات التحليل الطيفي للقوة مفصلة ؛ بعد ذلك ، يتم تفصيل تحليل هذه البيانات من خلال برنامج Python مفتوح المصدر تم تطويره حديثا ، وهو مجاني للتنزيل من GitHub.
Abstract
تشير المسافة البادئة النانوية إلى فئة من التقنيات التجريبية حيث يتم استخدام مسبار القوة الميكرومترية لتحديد الخواص الميكانيكية المحلية للمواد الحيوية والخلايا اللينة. اكتسب هذا النهج دورا مركزيا في مجالات البيولوجيا الميكانيكية وتصميم المواد الحيوية وهندسة الأنسجة ، للحصول على توصيف ميكانيكي مناسب للمواد اللينة بدقة مماثلة لحجم الخلايا المفردة (ميكرومتر). الاستراتيجية الأكثر شيوعا للحصول على مثل هذه البيانات التجريبية هي استخدام مجهر القوة الذرية (AFM). في حين أن هذه الأداة توفر دقة غير مسبوقة في القوة (وصولا إلى pN) والفضاء (sub-nm) ، فإن قابليتها للاستخدام غالبا ما تكون محدودة بسبب تعقيدها الذي يمنع القياسات الروتينية للمؤشرات المتكاملة للخواص الميكانيكية ، مثل معامل يونغ (E). اكتسب جيل جديد من المسافات البادئة النانوية ، مثل تلك القائمة على تقنية استشعار الألياف الضوئية ، شعبية مؤخرا لسهولة تكاملها مع السماح بتطبيق قوى دون nN بدقة مكانية ميكرومتر ، وبالتالي فهي مناسبة لاستكشاف الخصائص الميكانيكية المحلية للهلاميات المائية والخلايا.
في هذا البروتوكول ، يتم تقديم دليل خطوة بخطوة يوضح بالتفصيل الإجراء التجريبي للحصول على بيانات المسافة البادئة النانوية على الهلاميات المائية والخلايا باستخدام nanoinenter الاستشعار عن الألياف الضوئية المتاح تجاريا. في حين أن بعض الخطوات خاصة بالأداة المستخدمة هنا ، يمكن اعتبار البروتوكول المقترح كدليل لأجهزة المسافة البادئة النانوية الأخرى ، بشرط أن يتم تكييف بعض الخطوات وفقا لإرشادات الشركة المصنعة. علاوة على ذلك ، يتم تقديم برنامج Python جديد مفتوح المصدر مزود بواجهة مستخدم رسومية سهلة الاستخدام لتحليل بيانات المسافة البادئة النانوية ، مما يسمح بفحص المنحنيات المكتسبة بشكل غير صحيح ، وتصفية البيانات ، وحساب نقطة الاتصال من خلال إجراءات رقمية مختلفة ، والحساب التقليدي ل E ، بالإضافة إلى تحليل أكثر تقدما مناسب بشكل خاص لبيانات المسافة البادئة النانوية أحادية الخلية.
Introduction
تم تأسيس الدور الأساسي للميكانيكا في علم الأحياء في الوقت الحاضر 1,2. من الأنسجة الكاملة إلى الخلايا المفردة ، يمكن للخواص الميكانيكية أن تبلغ عن الحالة الفيزيولوجية المرضية للمادة الحيوية قيد التحقيق 3,4. على سبيل المثال ، أنسجة الثدي المصابة بالسرطان أكثر صلابة من الأنسجة السليمة ، وهو مفهوم هو أساس اختبار الجس الشعبي5. والجدير بالذكر أنه تبين مؤخرا أن مرض الفيروس التاجي 2019 (COVID-19) الناجم عن فيروس كورونا 2 المسبب للمتلازمة التنفسية الحادة الوخيمة (SARS-CoV-2) يتم تأكيده من خلال التغيرات في الخواص الميكانيكية لخلايا الدم ، بما في ذلك انخفاض تشوه كرات الدم الحمراء وانخفاض تصلب الخلايا الليمفاوية والعدلات مقارنة بخلايا الدم من الأفراد الساذجين SARS-CoV-2 6.
بشكل عام ، تتشابك ميكانيكا الخلايا والأنسجة بطبيعتها: كل نسيج له خصائص ميكانيكية محددة تؤثر في نفس الوقت وتعتمد على تلك الموجودة في الخلايا المكونة والمصفوفة خارج الخلية (ECM)5. لهذا السبب ، غالبا ما تتضمن استراتيجيات دراسة الميكانيكا في علم الأحياء ركائز هندسية مع محفزات ميكانيكية ذات صلة من الناحية الفسيولوجية لتوضيح سلوك الخلية استجابة لتلك المحفزات. على سبيل المثال ، أظهر العمل الأساسي الذي قام به إنجلر وزملاؤه أن التزام الخلايا الجذعية الوسيطة يتم التحكم فيه بواسطة مرونة المصفوفة ، كما تمت دراسته على الهلاميات المائية ثنائية الأبعاد اللينة والقاسية (PAAm)7.
توجد العديد من الاستراتيجيات لتوصيف المادة الحيوية قيد التحقيق ميكانيكيا ، متفاوتة في النطاق المكاني (أي من المحلي إلى السائب) وفي طريقة التشوه (على سبيل المثال ، المحوري مقابل القص) ، وبالتالي تسفر عن معلومات مختلفة ، والتي تحتاج إلى تفسير دقيق3،8،9،10. عادة ما يتم التعبير عن ميكانيكا المواد الحيوية اللينة من حيث الصلابة. ومع ذلك ، تعتمد الصلابة على كل من خصائص المواد والهندسة ، في حين أن الوحدات المرنة هي خصائص أساسية للمادة ومستقلة عن هندسة المادة11. على هذا النحو ، ترتبط وحدات المرونة المختلفة بصلابة عينة معينة ، ويشمل كل معامل مرن مقاومة المادة لنمط معين من التشوه (على سبيل المثال ، المحوري مقابل القص) في ظل ظروف حدودية مختلفة (على سبيل المثال ، التمدد الحر مقابل الحبس)11,12. تسمح تجارب المسافة البادئة النانوية بتحديد الخواص الميكانيكية من خلال E والتي ترتبط بالتشوه أحادي المحور (المسافة البادئة) عندما لا تكون المادة الحيوية محصورة جانبيا10،11،12.
الطريقة الأكثر شيوعا لتحديد E للأنظمة البيولوجية على المستوى المجهري هي AFM13،14،15،16. AFM هي أداة قوية للغاية مع دقة القوة وصولا إلى مستوى pN والدقة المكانية وصولا إلى مقياس دون نانومتر. علاوة على ذلك ، توفر AFM مرونة قصوى من حيث الاقتران بالأدوات البصرية والميكانيكية التكميلية ، مما يوسع قدراتها على استخراج ثروة من المعلومات من المادة الحيوية قيد التحقيق13. ومع ذلك ، تأتي هذه الميزات الجذابة مع حاجز أمام الدخول يمثله تعقيد الإعداد التجريبي. يتطلب AFM تدريبا مكثفا قبل أن يتمكن المستخدمون من الحصول على بيانات قوية ، وغالبا ما يكون استخدامه للتوصيف الميكانيكي اليومي للمواد البيولوجية غير مبرر ، خاصة عندما لا تكون قوتها الفريدة ودقتها المكانية مطلوبة.
لهذا السبب ، اكتسبت فئة جديدة من nanoindenter شعبية مؤخرا بسبب سهولة استخدامها ، بينما لا تزال تقدم بيانات قابلة للمقارنة AFM مع دقة قوة دون nN ودقة مكانية ميكرومتر ، مما يعكس القوى التي تمارسها وتدركها الخلايا على مقاييس الطول ذات الصلة2. على وجه الخصوص ، اكتسبت أجهزة المسافة البادئة النانوية ذات الطويق الأعلى القائمة على تقنية استشعار الألياف الضوئية17,18 شعبية بين الباحثين النشطين في مجال البيولوجيا الميكانيكية وخارجها ؛ وتم نشر ثروة من الأعمال التي تبلغ عن الخصائص الميكانيكية للمواد الحيوية باستخدام هذه الأجهزة ، بما في ذلك الخلايا19،20 ، الهلاميات المائية8،21 ، والأنسجة22،23. على الرغم من قدرات هذه الأنظمة على سبر الخواص الميكانيكية الديناميكية المحلية (أي معامل التخزين والفقد) ، تظل التجارب شبه الثابتة التي تنتج E هي الخيار الأكثر شيوعا8،19،20،21. باختصار ، تتكون تجارب المسافة البادئة النانوية شبه الساكنة من وضع مسافة بادئة للعينة بسرعة ثابتة تصل إلى نقطة ضبط محددة إما بأقصى إزاحة أو قوة أو عمق مسافة بادئة ، وتسجيل كل من القوة والموضع الرأسي للناتئ في ما يسمى منحنيات مسافة القوة (F-z). ثم يتم تحويل منحنيات F-z إلى منحنيات المسافة البادئة للقوة (F-δ) من خلال تحديد نقطة الاتصال (CP) ، وتزويدها بنموذج ميكانيكا اتصال مناسب (عادة نموذج Hertz13) لحساب E.
في حين أن تشغيل nanoindenter أعلى الطويق يشبه قياسات AFM ، هناك خصوصيات تستحق الدراسة. في هذا العمل ، يتم توفير دليل خطوة بخطوة للحصول بقوة على منحنيات F-z من الخلايا والهلاميات المائية التي تحاكي الأنسجة باستخدام nanoinenter المتاح تجاريا ، من أجل تشجيع توحيد الإجراءات التجريبية بين مجموعات البحث باستخدام هذا الجهاز وغيره من الأجهزة المماثلة. بالإضافة إلى ذلك ، يتم تقديم المشورة حول أفضل طريقة لإعداد عينات وخلايا هيدروجيل لإجراء تجارب المسافة البادئة النانوية ، جنبا إلى جنب مع نصائح استكشاف الأخطاء وإصلاحها على طول المسار التجريبي.
علاوة على ذلك ، يعتمد الكثير من التباين في نتائج المسافة البادئة النانوية (أي E وتوزيعها) على الإجراء المحدد المستخدم لتحليل البيانات ، وهو أمر غير تافه. لمعالجة هذه المشكلة ، يتم توفير تعليمات لاستخدام برنامج مفتوح المصدر تم تطويره حديثا ومبرمج في Python ومجهز بواجهة مستخدم رسومية سهلة الاستخدام (GUI) لتحليل الدفعات لمنحنيات F-z . يسمح البرنامج بفحص البيانات بسرعة ، وتصفية البيانات ، وحساب CP من خلال إجراءات عددية مختلفة ، والحساب التقليدي ل E ، بالإضافة إلى تحليل أكثر تقدما يسمى أطياف المرونة24 ، مما يسمح بتقدير معامل يونغ الأكبر للخلية ، ومعامل يونغ لقشرة الأكتين ، وسمك قشرة الأكتين. يمكن تنزيل البرنامج بحرية من GitHub ويمكن تكييفه بسهولة لتحليل البيانات الناشئة من أنظمة أخرى عن طريق إضافة محلل بيانات مناسب. يتم التأكيد على أنه يمكن استخدام هذا البروتوكول لأجهزة المسافة البادئة النانوية الأخرى ذات الطويق الأعلى ، وأجهزة المسافة البادئة النانوية الأخرى بشكل عام ، بشرط أن يتم تكييف بعض الخطوات وفقا لإرشادات الأداة المحددة. تم تلخيص البروتوكول بشكل تخطيطي في الشكل 1.
Protocol
1. تحضير الركائز / الخلايا لقياسات المسافة البادئة النانوية
- اتبع الخطوات الواردة في البروتوكول التكميلي لإعداد الهلاميات المائية / الخلايا PAAm لتجارب المسافة البادئة النانوية. يتم تلخيص الإجراء في الشكل 2.
ملاحظة: تم اختيار الهلاميات المائية PAAm لأنها الهلاميات المائية الأكثر شيوعا المستخدمة في مجال علم الأحياء الميكانيكي. ومع ذلك ، فإن البروتوكول ينطبق بالتساوي على أي نوع من هيدروجيل25 (انظر المناقشة ، تعديلات الطريقة).
2. بدء تشغيل الجهاز واختيار المسبار ومعايرة المسبار
- اتبع الخطوات الواردة في البروتوكول التكميلي لبدء تشغيل الجهاز. للحصول على تفاصيل فنية حول تشغيل المسافات النانوية ذات المسافات البادئة النانوية ذات الألياف الضوئية ، تحقق من هذه المراجع17,18.
- حدد مسبار المسافة البادئة النانوية كما هو موضح أدناه.
ملاحظة: جميع المجسات المتاحة تجاريا مثل تلك المستخدمة في هذا البروتوكول مجهزة بطرف كروي (الشكل 3 أ). لذلك ، يضيق الاختيار إلى متغيرين: صلابة الكابولي ونصف قطر الطرف (الشكل 3 ب).- اختر صلابة الكابولي (k في N / m) التي تتطابق مع صلابة العينة المتوقعة للحصول على أفضل النتائج15 (انظر الشكل 3C والمناقشة ، الخطوات الحاسمة في البروتوكول). بالنسبة للخلايا ، حدد مسبارا به k في النطاق 0.01-0.09 نيوتن / م. بالنسبة للهلاميات المائية ، حدد مسبارا به k في النطاق 0.1-0.9 نيوتن / م ، مما ينتج عنه نتائج مثالية للمواد الهلامية مع E المتوقع بين بضعة كيلو باسكال و 100 كيلو باسكال (انظر النتائج التمثيلية).
- اختر نصف قطر الطرف (R في ميكرومتر) وفقا للدقة المكانية المطلوبة لعملية المسافة البادئة. بالنسبة للخلايا الصغيرة مثل خلايا الكلى الجنينية البشرية 293T (HEK293T) (متوسط قطر ~ 10-15 ميكرومتر26) ، حدد كرة ذات R = 3 ميكرومتر. بالنسبة للهلاميات المائية ، حدد كرة ب R = 10-250 ميكرومتر لاستكشاف الخواص الميكانيكية للمادة الحيوية على منطقة تلامس كبيرة وتجنب عدم التجانس المحلي.
- راجع الشكل 3C وأي إرشادات إضافية للشركة المصنعة لتحديد المسبار المناسب.
- بمجرد تحديد المسبار ، اتبع الخطوات الواردة في البروتوكول التكميلي لتثبيته على nanoindenter.
3. معايرة المسبار
ملاحظة: الخطوات التالية خاصة بأجهزة المسافة البادئة النانوية ذات الحلقات العلوية القائمة على تقنية استشعار الألياف الضوئية ، وهي مفصلة لإصدار البرنامج 3.4.1. بالنسبة لأجهزة المسافة البادئة النانوية الأخرى ، اتبع الخطوات الموصى بها من قبل الشركة المصنعة للجهاز.
- في النافذة الرئيسية للبرنامج ، انقر فوق تهيئة. ستظهر قائمة المعايرة. أدخل تفاصيل المسبار (يمكن العثور عليها على جانب صندوق المسبار ؛ k في N / m ، R في μm ، وعامل المعايرة في الهواء) في مربعات الإدخال.
- قم بإعداد طبق المعايرة: طبق بتري زجاجي سميك ذو قاع مسطح (انظر المناقشة ، الخطوات الحاسمة في البروتوكول). املأ الطبق بنفس وسيط طبق العينة (يمكن أن يكون هذا أيضا هواء). طابق درجة حرارة الوسط مع درجة حرارة العينة.
- ضع طبق المعايرة تحت المسبار. إذا لزم الأمر ، حرك المسبار من حامل ذراع nanoindenter وأمسكه بيد واحدة لتوفير مساحة لوضع طبق المعايرة. حرك المسبار إلى مكانه.
- نفذ الخطوتين التاليتين للمعايرة في السائل. في حالة القياس في الهواء ، استخدم ركيزة polytetrafluoroethylene المتوفرة للمعايرة وانتقل إلى الخطوة 5.
- قم بترطيب المسبار مسبقا بقطرة من الإيثانول بنسبة 70٪ باستخدام ماصة باستور مع نهاية الماصة في ملامسة خفيفة مع الطويق الزجاجي ، حتى تنزلق القطرة فوق الطرف الكابولي والكروي27 (انظر المناقشة ، الخطوات الحاسمة في البروتوكول).
- حرك ذراع nanoindenter يدويا لأسفل حتى يتم غمر المسبار بالكامل ، ولكن لا يزال بعيدا عن قاع طبق بتري. إذا لزم الأمر ، أضف المزيد من الوسائط إلى طبق المعايرة. انتظر 5 دقائق حتى يتم الوصول إلى ظروف التوازن في السائل.
- في قائمة تهيئة البرنامج ، انقر فوق مسح الطول الموجي. ستعرض شاشة مقياس التداخل شريط تقدم وستعرض نافذة Live Signal على برنامج الكمبيوتر النمط الموضح في الشكل S1A ، على اليسار. للتحقق مما إذا كان الفحص البصري ناجحا أم لا، انتقل إلى لوحة مسح الطول الموجي في مربع مقياس التداخل. إذا نجحت ، يجب أن تكون الموجة الجيبية مرئية (الشكل 1 أ ، يمين). راجع مناقشة (استكشاف أخطاء الأسلوب وإصلاحها) في حالة ظهور خطأ.
- في قائمة التهيئة ، انقر فوق Find Surface ، والذي سيخفض المسبار تدريجيا حتى يتم الوصول إلى عتبة محددة في انحناء الكابولي. يتوقف المسبار عن الحركة عند ملامسة طبق بتري الزجاجي.
- تحقق مما إذا كان المسبار ملامسا للسطح. حرك المسبار لأسفل بمقدار 1 ميكرومتر باستخدام زر السهم المتجه لأسفل y في النافذة الرئيسية للبرنامج. راقب الإشارة الخضراء (انحراف الكابولي) في النافذة الحية للبرنامج بحثا عن التغييرات في خط الأساس مع كل خطوة ، عندما يكون الكابولي على اتصال بالركيزة (الشكل S1B). إذا لم يكن هناك تغيير ، فإن الكابولي ليس على اتصال (انظر الخطوة التالية).
- قم بزيادة قيمة العتبة في قائمة الخيارات ضمن علامة التبويب البحث عن السطح من قيمتها الافتراضية 0.01 بخطوة 0.01 في المرة الواحدة وكرر خطوة البحث عن السطح حتى في الاتصال. بدلا من ذلك ، قم بإسقاط المسبار للاتصال به في خطوات صغيرة 1 ميكرومتر حتى يبدأ خط الأساس الأخضر في التحول عند كل خطوة لأسفل.
ملاحظة: بالنسبة لأخف الكابوليات (k = 0.025 Nm-1) ، قم بزيادة قيمة Threshold في قائمة الخيارات ضمن علامة التبويب Find Surface قبل تنفيذ الخطوة 6. ابدأ من قيمة عتبة 0.06 أو 0.07 وقم بزيادتها حتى 0.1 إذا لزم الأمر. وذلك لأن الضوضاء البيئية من المحتمل أن تتسبب في انحناء الكابولي فوق العتبة قبل الاتصال. بالنسبة للمجسات اللينة ، يؤدي تقليل سرعة الاقتراب (ميكرومتر / ثانية) في نفس القائمة أيضا إلى تحسين الإجراء. - في قائمة التهيئة ، انقر فوق معايرة.
- تحقق من نافذة الإشارة الحية للبرنامج وتأكد من أن كل من إزاحة بيزو وإشارات انحراف الكابولي تتحرك لأعلى في نفس الوقت (الشكل S1C).
- إذا كان هناك عدم تطابق في الوقت المناسب ، فإن المسبار ليس على اتصال كامل بالزجاج. تحرك لأسفل المسبار بخطوات 1 ميكرومتر حتى يتغير خط الأساس لإشارة الكابولي (انظر الخطوة 7) وكرر الخطوة 9.
- إذا لم تتغير إشارة الكابولي على الإطلاق أثناء خطوة المعايرة ، فإن المسبار بعيد عن السطح. قم بزيادة عتبة التلامس بشكل متكرر (انظر الخطوة 8) حتى يتم العثور على السطح بشكل صحيح وكرر المعايرة من البداية بدءا من مسح الطول الموجي.
- عند اكتمال المعايرة، تحقق من عوامل المعايرة القديمة والجديدة في النافذة المنبثقة المعروضة. إذا كان عامل المعايرة الجديد في النطاق الصحيح ، فانقر فوق استخدام عامل جديد. إذا فشلت المعايرة، وكان العامل الجديد إما NaN أو ليس في النطاق المتوقع، فراجع مناقشة (استكشاف أخطاء الأسلوب وإصلاحها) للحصول على الحل.
ملاحظة: يجب أن يكون العامل الجديد ~ n مرات أقل من العامل المتوفر في صندوق المسبار إذا تم إجراء المعايرة في وسط سائل بمعامل انكسار n (n = 1.33 للماء). إذا تم إجراء المعايرة في الهواء ، فيجب أن تكون عوامل المعايرة الجديدة والقديمة متساوية تقريبا. - تحقق مما إذا كانت دائرة إزالة التشكيل قد تمت معايرتها بشكل صحيح على النحو التالي. انتقل إلى علامة التبويب إزالة التشكيل على سطح مكتب مقياس التداخل. اضغط برفق على الطاولة البصرية أو على nanoindenter لإحداث ضوضاء كافية. يجب أن تغطي الدائرة البيضاء المكونة من نقاط بيانات منفصلة الدائرة الحمراء تقريبا (الشكل S1D).
- إذا لم تتداخل الدائرة البيضاء مع الدائرة الحمراء ، أو ظهر تحذير على شاشة مقياس التداخل ، فإن دائرة إزالة التشكيل تحتاج إلى إعادة معايرة. يمكن القيام بذلك بطريقتين كما هو موضح أدناه.
- اضغط باستمرار على جسم nanoindenter لإحداث دائرة كاملة من الضوضاء واضغط على زر المعايرة على مقياس التداخل.
- أدخل في اتصال مع الركيزة الزجاجية واضغط معايرة من قائمة تهيئة في النافذة الرئيسية للبرنامج. لا تقم بحفظ عامل المعايرة. في هذه المرحلة ، تحقق مرة أخرى وتأكد من تداخل الدائرة البيضاء مع الدائرة الحمراء.
ملاحظة: إذا لم يتم إزاحة الإشارة قليلا من دائرة إزالة التشكيل فحسب ، بل أصبحت صغيرة جدا أو غير مرئية على الإطلاق ، فهذا يعني أن الكابولي عالق بالألياف الضوئية. اتبع إرشادات استكشاف الأخطاء وإصلاحها لهذه المشكلة (راجع المناقشة، استكشاف الأخطاء وإصلاحها للأسلوب) وكرر الخطوتين 13.1 أو 13.2. بمجرد عودة الكابولي إلى وضعه الأفقي (الشكل 3 أ) ، ستتعافى الإشارة إلى دائرة إزالة التشكيل.
- تحقق من المعايرة عن طريق إجراء مسافة بادئة على الركيزة الزجاجية مباشرة بعد المعايرة كما هو موضح أدناه.
- قم بتحميل ملف تجربة أو إنشائه بالنقر فوق تكوين التجربة وإضافة خطوة البحث عن السطح وخطوة المسافة البادئة . بالنسبة لخطوة المسافة البادئة، استخدم إعدادات وضع الإزاحة الافتراضية وقم بتغيير الحد الأقصى للإزاحة إلى مسافة المعايرة (3000 نانومتر) لإزاحة المسبار ضد الركيزة الصلبة.
- انقر فوق تشغيل التجربة وتحقق من دائرة إزالة التشكيل في نافذة مقياس التداخل. تحقق من الإشارة البيضاء وتأكد من أنها أعلى الدائرة الحمراء أثناء المسافة البادئة.
- تحقق من النتائج على النافذة الرئيسية للبرنامج في الرسم البياني لبيانات الوقت ، وتأكد من أن إزاحة بيزو (الخط الأزرق) تساوي انحراف الكابولي (الخط الأخضر) حيث تبدأ المسافة البادئة في التلامس ولا يتوقع حدوث تشوه مادي. إذا لم تكن الإشارات متوازية، فراجع مناقشة (استكشاف أخطاء الطريقة وإصلاحها).
- قم بتغيير المسار المحلي في قائمة المعايرة عن طريق تعيين مسار حفظ المعايرة إلى دليل مناسب.
- عندما تتم معايرة المسبار بنجاح ، حرك بيزو لأعلى بمقدار 500 ميكرومتر.
4. قياس معامل يونغ للمواد اللينة
- المسافة البادئة النانوية للهلاميات المائية
- قم بتحميل طبق بتري الذي يحتوي على العينة (العينات) على مسرح المجهر وانقل مسبار nanoindenter يدويا إلى موضع x-y المطلوب فوق العينة.
- حرك المسبار يدويا في المحلول ، مع الحرص على ترك 1-2 مم بين المسبار وسطح العينة في هذه المرحلة. انتظر 5 دقائق حتى يتوازن المسبار في الوسط.
- ركز المستوى z للمجهر الضوئي بحيث يكون المسبار مرئيا بوضوح.
- قم بإجراء مسافة بادئة واحدة لضبط المعلمات التجريبية كما هو موضح أدناه.
- قم بتكوين تجربة جديدة في النافذة الرئيسية للبرنامج. انقر فوق تكوين التجربة ، والتي ستفتح نافذة جديدة. أضف خطوة البحث عن Surface . يمكن تغيير جميع معلمات خطوة Find Surface في قائمة الخيارات الخاصة بالبرنامج إذا لزم الأمر.
ملاحظة: سيقوم Find Surface بخفض المسبار حتى يتم العثور على السطح ، ثم سيسحب المسبار إلى مسافة محددة بواسطة Z فوق السطح (μm) ، فوق سطح العينة. إذا كان الكابولي المحدد شديد الصلابة بالنسبة للعينة أو كانت العينة لزجة ، فمن المحتمل أن يظل المسبار على اتصال بالعينة بعد الخطوة ، مما سيؤدي إلى منحنى بدون خط أساس (الشكل 4C). لحل هذه المشكلة ، قم بزيادة Z فوق السطح (ميكرومتر). - أضف خطوة المسافة البادئة . حدد علامة التبويب الملف الشخصي وانقر فوق التحكم في الإزاحة. اترك ملف تعريف المسافة البادئة الافتراضي.
- انقر فوق تشغيل التجربة في نافذة البرنامج الرئيسية. سيجد هذا السطح ويؤدي مسافة بادئة واحدة. إذا كانت المسافة البادئة الفردية لا تبدو كما هو متوقع ، فاضبط المعلمات التجريبية كما هو موضح في الشكل 4 والمناقشة (استكشاف أخطاء الطريقة وإصلاحها).
- قم بتكوين تجربة جديدة في النافذة الرئيسية للبرنامج. انقر فوق تكوين التجربة ، والتي ستفتح نافذة جديدة. أضف خطوة البحث عن Surface . يمكن تغيير جميع معلمات خطوة Find Surface في قائمة الخيارات الخاصة بالبرنامج إذا لزم الأمر.
- بمجرد أن تبدو المسافة البادئة كما هو مطلوب ، قم بتكوين فحص المصفوفة بحيث يتم وضع مسافة بادئة لمساحة كافية من العينة. انقر فوق تكوين التجربة ، وأضف خطوة Find Surface مع المعلمات التجريبية المحددة مسبقا وأضف خطوة Matrix Scan .
- بالنسبة للهلاميات المائية المسطحة ، قم بتكوين مسح مصفوفة يحتوي على 50-100 نقطة (أي 5 × 10 أو 10 × 10 بوصة x و y) متباعدة عند 10-100 ميكرومتر (أي dx = dy = 10-100 ميكرومتر). انقر فوق استخدام موضع المرحلة لبدء مسح المصفوفة من موضع المرحلة الحالي. تأكد من تحديد المربع البحث التلقائي عن السطح للعثور على السطح عند كل مسافة بادئة باستخدام المعلمات التجريبية المحددة.
- لتجنب الإفراط في أخذ العينات، اضبط حجم الخطوة على ضعف نصف قطر التلامس على الأقل (
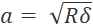 ، حيث δ هو عمق المسافة البادئة).
، حيث δ هو عمق المسافة البادئة). - قم بإعداد ملف تعريف مسح المصفوفة في التحكم في الإزاحة. تأكد من أن الملف الشخصي لا ينتهك افتراضات نموذج هيرتز (انظر المناقشة ، الخطوات الحاسمة في البروتوكول).
- اترك عدد المقاطع إلى 5، وهي القيمة الافتراضية، واستخدم ملف تعريف الإزاحة الافتراضي. إذا لزم الأمر ، قم بتغيير ملف تعريف الإزاحة من حيث الحد الأقصى للإزاحة والوقت لكل مقطع منحدر ، مما سيؤثر على أقصى عمق للمسافة البادئة ومعدل الإجهاد ، على التوالي. لا تتجاوز معدلات الإجهاد > 10 ميكرومتر / ثانية (انظر المناقشة ، حدود الطريقة).
- أدخل قيمة لسرعة الاقتراب ، والتي تحدد مدى سرعة إزاحة المسبار نحو العينة قبل الاتصال. طابق سرعة التراجع مع سرعة الاقتراب (انظر الملاحظة أدناه).
ملاحظة: بالنسبة للناتئ الناعم والبيئات الصاخبة ، يوصى بسرعة اقتراب تتراوح بين 1000 و 2000 نانومتر / ثانية. بالنسبة للناتئ الأكثر صلابة والبيئات الخاضعة للرقابة ، يمكن زيادة ذلك. - احفظ التجربة التي تم تكوينها في مسار التجربة المطلوب وحدد دليلا حيث سيتم حفظ البيانات في حفظ المسار، في علامة التبويب عام في نافذة تكوين التجربة . انقر فوق تشغيل التجربة.
- لتجنب الإفراط في أخذ العينات، اضبط حجم الخطوة على ضعف نصف قطر التلامس على الأقل (
- بمجرد اكتمال مسح المصفوفة ، ارفع المسبار بمقدار 200-500 ميكرومتر وانقل المسبار إلى منطقة مختلفة من العينة بعيدا بما فيه الكفاية عن المنطقة الأولى.
- كرر التجربة مرتين على الأقل بحيث يتم الحصول على بيانات كافية عن كل عينة (أي مسحين على الأقل لكل عينة ، تحتوي كل منهما على 50-100 منحني).
- المسافة البادئة النانوية للخلايا
- قم بتحميل العينة على المجهر كما هو موضح أعلاه.
- بالنسبة للمسافة البادئة أحادية الخلية ، ركز المستوى z بحيث تكون كل من الخلايا والمسبار مرئيين عند تكبير 20x أو 40x ، اعتمادا على حجم الخلية وانتشارها.
- حرك المسبار فوق الخلية المراد وضع مسافة بادئة لها.
- قم بتكوين تجربة جديدة في النافذة الرئيسية للبرنامج. انقر فوق تكوين التجربة ، والتي ستفتح نافذة جديدة. أضف خطوة البحث عن السطح والمسافة البادئة مع المعلمات الافتراضية في وضع الإزاحة.
- انقر فوق تشغيل التجربة ، والتي ستجد السطح وتؤدي مسافة بادئة واحدة. تحقق مما إذا كانت المسافة البادئة ناجحة. إذا كان المنحنى لا يبدو كما هو متوقع ، فاضبط المعلمات التجريبية (انظر الشكل 4 والمناقشة ، استكشاف الأخطاء وإصلاحها للطريقة).
- إذا نجحت المسافة البادئة، فأضف فحص مصفوفة إلى التجربة. اتبع الخطوات المعطاة لتجارب المسافة البادئة النانوية للهلاميات المائية ؛ قم بتكوين فحص المصفوفة بحيث يسمح حجم الخطوة بوضع مسافة بادئة لمساحة صغيرة من الخلية ؛ 25 نقطة متباعدة عند 0.5-5 ميكرومتر لخلايا HEK293T.
- اعتمادا على حجم الخلية ، قم بتكييف مسح المصفوفة للتأكد من أن الطرف لا يضع مسافة بادئة خارج حد الخلية ، أي إجراء هندسة خريطة مختلفة أو فحص نقاط أقل.
- انقر فوق تشغيل التجربة وانتظر حتى تكتمل.
- بمجرد اكتمال مسح المصفوفة ، ارفع المسبار بعيدا عن الاتصال (50 ميكرومتر في المستوى z ).
- حرك المسبار فوق خلية جديدة وكرر العملية (راجع المناقشة ، الخطوات الهامة في البروتوكول).
- تنظيف المسبار وإيقاف تشغيل الأداة
- اتبع الخطوات الواردة في البروتوكول التكميلي لتنظيف المسبار وإيقاف تشغيل جهاز المسافة البادئة النانوية.
5. تحليل البيانات
- تنزيل البرنامج وتثبيته
- اتبع الخطوات الواردة في البروتوكول التكميلي لتنزيل وتثبيت البرنامج لتحليل البيانات28,29.
- فحص منحنيات F-z وإنتاج مجموعة بيانات نظيفة بتنسيق JSON
- قم بتشغيل prepare.py من سطر الأوامر على كمبيوتر المختبر كما هو موضح في الخطوات من 2 إلى 3.
- إذا كنت تستخدم جهاز كمبيوتر يعمل بنظام Windows ، فاضغط مع الاستمرار على مفتاح shift أثناء النقر بزر الماوس الأيمن فوق مجلد NanoPrepare وانقر فوق فتح نافذة PowerShell هنا. اكتب الأمر python prepare.py واضغط على مفتاح Enter . ستظهر واجهة المستخدم الرسومية على شاشتك (الشكل S2).
- إذا كنت تستخدم جهاز كمبيوتر يعمل بنظام التشغيل MacOS ، فانقر بزر الماوس الأيمن فوق مجلد NanoPrepare وانقر فوق محطة طرفية جديدة في المجلد. اكتب الأمر python3 prepare.py واضغط على مفتاح Enter ، والذي سيشغل واجهة المستخدم الرسومية (الشكل S2).
- حدد تنسيق بيانات O11NEW من القائمة المنسدلة. إذا لم يتم تحميل البيانات بشكل صحيح ، فأعد تشغيل واجهة المستخدم الرسومية وحدد O11OLD.
ملاحظة: يعمل تنسيق O11NEW مع البيانات التي تم الحصول عليها باستخدام أجهزة المسافة البادئة النانوية لاستشعار الألياف الضوئية ذات الحلقة العلوية مع إصدار البرنامج 3.4.1. سيعمل هذا التنسيق أيضا مع إصدارات البرامج السابقة ، على الأقل تلك التي تنتمي إلى nanoindenter المثبتة في 2019-2020. - انقر فوق تحميل مجلد. حدد مجلدا يحتوي على البيانات المراد تحليلها - مسح مصفوفة مفردة أو عمليات مسح مصفوفة متعددة. سيتم ملء الرسم البياني العلوي (منحنيات Raw) بمجموعة البيانات التي تم تحميلها. لتصور منحنى معين ، انقر فوقه. سيؤدي ذلك إلى تمييزه باللون الأخضر وإظهاره على الرسم البياني السفلي (المنحنى الحالي).
- قم بتنظيف مجموعة البيانات باستخدام علامات التبويب الموجودة في الجزء الأيمن من واجهة المستخدم الرسومية كما هو موضح أدناه.
- استخدم زر المقطع لتحديد المقطع الصحيح المراد تحليله، وهو الجزء الأمامي من منحنيات F-z . يعتمد الرقم المحدد على عدد الأجزاء المحددة في برنامج المسافة البادئة النانوية عند إجراء التجارب.
- استخدم زر Crop 50 nm لاقتصاص المنحنيات بمقدار 50 نانومتر في أقصى اليسار (إذا تم تحديد L ) أو اليمين (إذا تم تحديد R ) أو كلا الجانبين (إذا تم تحديد كل من R و L). انقر فوق هذا الزر عدة مرات لاقتصاص بقدر ما هو مطلوب. استخدم هذا لإزالة القطع الأثرية الموجودة في بداية / نهاية منحنيات F-z .
- افحص علامة التبويب الكابولي بحثا عن ثابت الزنبرك وهندسة الطرف ونصف قطر الطرف. افحص علامة التبويب للتأكد من قراءة بيانات التعريف بشكل صحيح.
- استخدم علامة التبويب فحص لتعيين حد قوة يتجاهل كل المنحنيات التي لم تصل إلى القوة المحددة. سيتم تمييز المنحنيات المهملة باللون الأحمر.
- استخدم زر التبديل اليدوي لإزالة المنحنيات التي لم يتم الحصول عليها بشكل صحيح يدويا. قم بإزالة أي منحنيات بالنقر فوق المنحنى المحدد وتحديد OUT ، والذي سيبرز المنحنى باللون الأحمر.
- انقر فوق حفظ JSON. أدخل اسما مناسبا لمجموعة البيانات التي تم تنظيفها ، وهو ملف JSON واحد. أرسل ملف JSON إلى الكمبيوتر حيث تم تثبيت برنامج NanoAnalysis.
6. تحليل البيانات الرسمية
- قم بتشغيل ملف nano.py من سطر الأوامر بالانتقال إلى مجلد NanoAnalysis وتشغيل محطة طرفية كما هو موضح سابقا. اكتب الأمر python nano.py أو python3 nano.py (حسب نظام التشغيل) واضغط على مفتاح Enter . ستظهر واجهة المستخدم الرسومية على شاشتك (الشكل S3).
- في الجزء العلوي الأيسر من واجهة المستخدم الرسومية ، انقر فوق تحميل التجربة وحدد ملف JSON. سيؤدي هذا إلى ملء قائمة الملفات والرسم البياني لمنحنيات Raw الذي يعرض مجموعة البيانات من حيث منحنيات F-z. يتم عرض كل من المحورين F و z بالنسبة لإحداثيات CP ، والتي يتم حسابها في الخلفية عند تحميل مجموعة البيانات (انظر الملاحظة التالية). في مربع الإحصائيات ، تحقق من قيم المعلمات الثلاثة: تمتنشيط N ، وفشل N ، واستبعاد N.
ملاحظة: يمثل Nالمنشط عدد المنحنيات التي سيتم تحليلها في تحليل أطياف هيرتز / المرونة اللاحقة وتظهر باللون الأسود في الرسم البياني للمنحنيات الأولية . يمثل Nfailed عدد المنحنيات التي تعذر العثور على CP موثوق بها وتظهر باللون الأزرق في الرسم البياني. سيتم تجاهل هذه المنحنيات تلقائيا في التحليل اللاحق. عند فتح البرنامج ، قد يتم نقل بعض المنحنيات تلقائيا إلى المجموعة الفاشلة. وذلك لأن CP يتم حسابه عند فتح البرنامج باستخدام خوارزمية Threshold الافتراضية (انظر أدناه). يمثل Nالمستبعد المنحنيات التي تم تحديدها يدويا ليتم استبعادها من التحليل وستظهر باللون الأحمر في الرسم البياني (انظر أدناه). - تحقق مما إذا كان عدد المنحنيات الفاشلة والمستبعدة والمنشطة معقولا. تحقق من الرسم البياني للمنحنيات الأولية لتصور المنحنيات .
- لتصور منحنى معين بمزيد من التفصيل ، انقر فوقه. سيؤدي هذا إلى تمييزه باللون الأخضر وإظهاره على الرسم البياني للمنحنى الحالي . بمجرد تحديد منحنى واحد ، سيتم ملء معلمات R و k (التي يجب أن تكون هي نفسها لجميع المنحنيات) في مربع الإحصائيات في واجهة المستخدم الرسومية.
- قم بتغيير حالة منحنى معين باستخدام مربع التبديل . انقر فوق المنحنى المحدد الذي تريد تغيير حالته ، ثم انقر فوق إما مفعل أو فاشل أو مستبعد. يتم تحديث العدد في مربع الإحصائيات تلقائيا.
ملاحظة: لتغيير طريقة عرض مجموعة البيانات في الرسم البياني للمنحنيات الأولية ، استخدم المربع عرض . انقر فوق الكل لإظهار جميع المنحنيات (أي تم تنشيطها وفشلها واستبعادها في الألوان المعنية). انقر فوق فشل لإظهار المنحنيات المنشطة والفاشلة وانقر فوق تم التنشيط لإظهار المنحنيات المنشطة فقط. لإعادة تعيين حالة جميع المنحنيات بين التنشيط والمستبعد ، انقر فوق نشط أو مستبعد في مربع إعادة التعيين . - بمجرد تنظيف مجموعة البيانات بشكل أكبر ، اتبع خط أنابيب تحليل البيانات الموضح أدناه.
- قم بتصفية أي ضوضاء موجودة في المنحنيات باستخدام المرشحات المطبقة في واجهة المستخدم الرسومية (صندوق التصفية) ، أي مرشح مخصص يسمى مرشح البروز ، ومرشح Savitzky Golay30,31 (SAVGOL) ، ومرشح تجانس يعتمد على حساب متوسط البيانات في نافذة معينة (مرشح وسيط). راجع المناقشة (الخطوات الهامة في البروتوكول) للحصول على تفاصيل حول عوامل التصفية.
- افحص المنحنيات المصفاة في الرسم البياني للمنحنى الحالي . يظهر المنحنى المصفى باللون الأسود بينما يظهر الإصدار غير المصفى من المنحنى باللون الأخضر.
ملاحظة: ينصح بتصفية البيانات بأقل قدر ممكن للحفاظ على ميزات الإشارة الأصلية. قد تؤدي التصفية الزائدة إلى تخفيف أي اختلافات موجودة في البيانات. العمل مع مرشح البروز المنشط يكفي لتحليل هيرتز لبيانات المسافة البادئة النانوية. إذا كانت البيانات صاخبة بشكل خاص ، فقد يتم تطبيق مرشحات SAVGOL أو الوسيط بشكل إضافي. - حدد خوارزمية للعثور على CP. في مربع نقطة الاتصال ، اختر أحد سلسلة الإجراءات العددية التي تم تنفيذها في البرنامج ، وهي جودة الملاءمة (GoF) 32 أو نسبة الفروق (RoV) 32 أو المشتق الثاني 33 أو العتبة 33. راجع المناقشة ، الخطوات الهامة في البروتوكول للحصول على تفاصيل حول الخوارزميات.
ملاحظة: CP هو النقطة التي يتلامس فيها المسبار مع المادة ويحتاج إلى تحديد من أجل تحويل بيانات F-z إلى بيانات F-δ (بالنسبة للمواد اللينة ، δ محدود ويحتاج إلى حساب). سيتم تطبيق الخوارزمية المحددة على جميع المنحنيات النشطة لمجموعة البيانات ، وسيتم نقل المنحنيات التي تفشل فيها الخوارزمية في تحديد موقع CP بقوة إلى المجموعة الفاشلة. - اضبط معلمات الخوارزمية لتناسب مجموعة البيانات الخاصة بك بحيث يتم تحديد موقع CP بشكل صحيح كما هو مفصل في المناقشة ، الخطوات الحاسمة في البروتوكول. لعرض مكان العثور على CP على منحنى واحد ، حدد المنحنى بالنقر فوقه وانقر فوق فحص. تحقق من النافذة المنبثقة التي تظهر لتحديد مكان CP.
- تحقق مما إذا كان الخط الأحمر الذي يظهر ، وهو المعلمة التي حسبتها الخوارزمية في منطقة الاهتمام المحددة ، له قيمة قصوى أو دنيا تتوافق مع موقع CP (على سبيل المثال ، بالنسبة إلى GoF ، تكون المعلمة هي R2). كرر هذه العملية لجميع المنحنيات إذا لزم الأمر.
ملاحظة: محاور النافذة المنبثقة في إحداثيات مطلقة بحيث يمكن إظهار موقع CP. على العكس من ذلك ، يتم عرض محاور المنحنيات الخام والرسم البياني لمنحنى التيار بالنسبة إلى CP ، أي أن موقع CP هو (0,0). - انقر على تحليل هيرتز. سيؤدي هذا إلى إنشاء ثلاثة رسوم بيانية موصوفة أدناه.
- تحقق من منحنيات F-δ الفردية في مجموعة البيانات مع متوسط ملاءمة هرتز (خط أحمر متقطع). اضبط المسافة البادئة بالنانومتر التي تم تركيب طراز هرتز عليها في مربع النتائج ضمن الحد الأقصى للمسافة البادئة (نانومتر). اضبطه على ~ 10٪ من R كحد أقصى حتى يكون نموذج Hertz صالحا (انظر المناقشة ، الخطوات الهامة في البروتوكول).
- تحقق من متوسط منحنى F-δ مع نطاق خطأ يظهر انحرافا معياريا واحدا (SD) مع متوسط ملاءمة هرتز (خط أحمر متقطع). تصور متوسط ملاءمة هرتز على الرسم البياني للمنحنيات الخام كمرجع وتناسب هرتز لكل منحنى على منحنى التيار.
- تحقق من مخطط التشتت ل E الناشئ عن تركيب نموذج Hertz لكل منحنى على حدة.
- انقر فوق اسم الملف أو منحنى F-z أو منحنى F-δ أو نقطة على مخطط التشتت لتمييز المنحنى في كل مخطط. إذا ظهرت نقطة بيانات في مخطط التشتت خارج توزيع البيانات ، فانقر عليها وافحص المنحنى الذي تنتمي إليه. تحقق من تحديد موقع CP بشكل صحيح بالنقر فوق الزر فحص . استبعاد المنحنى من التحليل إذا لزم الأمر.
- افحص مربع النتائج بحثا عن المتوسط E المحسوب و SD الخاص به (Eγ ± σ) وتأكد من أنها معقولة للتجربة المحددة.
- في مربع حفظ ، انقر فوق هرتز. في النافذة المنبثقة، أدخل اسم الملف والدليل. بمجرد الانتهاء ، انقر فوق حفظ. سيتم إنشاء ملف .tsv. افتح ملف .tsv في أي برنامج تفضيل إضافي واستخدم القيم للتحليل الإحصائي والمزيد من التخطيط.
ملاحظة: يحتوي الملف على E التي تم الحصول عليها من كل منحنى ومتوسط E وSD لها. بالإضافة إلى ذلك، يحتوي الملف على البيانات الوصفية المرتبطة التحليل، بما في ذلك عدد المنحنيات التي تم تحليلها، R، k، والحد الأقصى المسافة البادئة المستخدمة لنموذج هيرتز. - هذه الخطوة اختيارية. انقر فوق متوسط F-Ind لتصدير متوسط القوة ومتوسط المسافة البادئة ، جنبا إلى جنب مع SD واحد في القوة.
- للحصول على بيانات المسافة البادئة النانوية للخلية ، انقر فوق تحليل أطياف المرونة (انظر النتائج التمثيلية والمناقشة). افحص المخططين المنتجين ، وهما E كدالة لعمق المسافة البادئة (E (δ)) لكل منحنى ، ومتوسط E (δ) مع نطاق خطأ يظهر SD واحدا (خط أحمر متصل ومنطقة مظللة) مثبتا بواسطة نموذج (خط أسود متقطع) ، والذي يسمح بتقدير معامل يونغ لقشرة الأكتين في الخلية ، معامل يونغ الأكبر للخلية ، وسمك قشرة الأكتين. بالإضافة إلى ذلك ، تحقق من متوسط E (δ) في الرسم البياني العلوي باللون الأحمر.
- تأكد من تحديد مربع الاستيفاء ، مما يضمن حساب المشتق اللازم لإجراء تحليل أطياف المرونة على الإشارة المقحمة (انظر النتائج التمثيلية).
- افحص مربع النتائج ، وأبلغ عن معامل يونغ للقشرة (E 0 ± σ) ، ومعامل يونغ الأكبر للخلية (Eb ± σ) ، وسمك القشرة (d 0 ± σ).
ملاحظة: قد تظهر أطياف المرونة المتوسطة صاخبة في البداية ، مع تذبذبات جيبية بارزة. نتيجة لذلك ، قد لا يتم تركيب المعادلة (3) بشكل صحيح. إذا كانت هذه هي الحالة ، فإن زيادة طول نافذة مرشح SAVGOL34 بشكل متكرر يحل هذه المشكلة. - بمجرد الانتهاء من التحليل ، انقر فوق ES في مربع حفظ . سيؤدي هذا إلى تصدير ملف .tsv في الدليل المحدد ، يحتوي على متوسط المرونة كدالة لمتوسط عمق المسافة البادئة ونصف قطر الاتصال ، والبيانات الوصفية المرتبطة بالتجربة (انظر أعلاه) ، ومعلمات النموذج المقدرة الموضحة أعلاه. أخيرا ، تم الإبلاغ أيضا عن متوسط المرونة التي تتجاهل الاعتماد على δ و SD الخاص به.
- أغلق البرنامج وأدخل النتائج المحفوظة في أي برنامج آخر مفضل لمزيد من رسم البيانات وإجراء التحليل الإحصائي.
- هذه الخطوة اختيارية: تصدير الرسوم البيانية من واجهة المستخدم الرسومية بالنقر بزر الماوس الأيمن على الرسم البياني وتحديد تصدير. قم بتصدير الرسم البياني في .svg ، بحيث تكون المعلمات مثل الخط وحجم الخط ونمط الخط وما إلى ذلك. يمكن تحريرها في برنامج آخر من اختيارك.
ملاحظة: يمكن برمجة خوارزميات ومرشحات CP المخصصة وإضافتها إلى الخوارزميات الموجودة بالفعل. انظر الملاحظة التكميلية 1 للاطلاع على التفاصيل.
النتائج
باتباع البروتوكول ، يتم الحصول على مجموعة من منحنيات F-z. من المرجح أن تحتوي مجموعة البيانات على منحنيات جيدة ، ومنحنيات يجب التخلص منها قبل متابعة التحليل. بشكل عام ، يجب التخلص من المنحنيات إذا كان شكلها مختلفا عن الشكل الموضح في الشكل 4 أ. يوضح الشكل 5AI...
Discussion
يوضح هذا البروتوكول كيفية الحصول بقوة على بيانات المسافة البادئة النانوية للتحليل الطيفي للقوة باستخدام محرك نانوي عالي الطويق متاح تجاريا على كل من الهلاميات المائية والخلايا المفردة. بالإضافة إلى ذلك ، يتم توفير تعليمات لاستخدام برنامج مفتوح المصدر مبرمج في Python يشتمل على سير عمل دقيق ...
Disclosures
ليس لدى المؤلفين ما يكشفون عنه.
Acknowledgements
تعترف GC و MAGO بجميع أعضاء CeMi. تقر MSS بالدعم من خلال منحة برنامج EPSRC (EP / P001114 / 1).
GC: البرمجيات (المساهمة في تطوير البرمجيات والخوارزميات) ، التحليل الرسمي (تحليل بيانات المسافة البادئة النانوية) ، التحقق من الصحة ، التحقيق (تجارب المسافة البادئة النانوية على المواد الهلامية بولي أكريلاميد) ، تنظيم البيانات ، الكتابة (المسودة الأصلية ، المراجعة والتحرير) ، التصور (الأشكال والرسوم البيانية). MAGO: التحقيق (إعداد عينات المواد الهلامية والخلايا ، تجارب المسافة البادئة النانوية على الخلايا) ، الكتابة (المسودة الأصلية ، المراجعة والتحرير) ، التصور (الأشكال والرسوم البيانية). NA: التحقق من الصحة والكتابة (المراجعة والتحرير). IL: البرمجيات (المساهمة في تطوير البرمجيات والخوارزميات) ، والتحقق من الصحة ، والكتابة (المراجعة والتحرير) ؛ MV: وضع المفاهيم ، البرمجيات (تصميم وتطوير البرامج والخوارزميات الأصلية) ، التحقق من الصحة ، الموارد ، الكتابة (المسودة الأصلية ، المراجعة والتحرير) ، الإشراف ، إدارة المشروع ، اقتناء التمويل MSS: الموارد ، الكتابة (المراجعة والتحرير) ، الإشراف ، إدارة المشروع ، اقتناء التمويل. قرأ جميع المؤلفين المخطوطة النهائية ووافقوا عليها.
Materials
| Name | Company | Catalog Number | Comments |
| 12 mm coverslips | VWR | 631-1577P | |
| 35 mm cell treated culture dishes | Greiner CELLSTAR | 627160 | |
| Acrylamide | Sigma-Aldrich | A4058 | |
| Acrylsilane | Alfa Aesar | L16400 | |
| Ammonium Persulfate | Merk | 7727-54-0 | |
| Bisacrylamide | Merk | 110-26-9 | |
| Chiaro nanoindenter | Optics 11 Life | no catologue number | |
| Ethanol | general | ||
| Fetal bovine serum | Gibco | 16140071 | |
| High glucose DMEM | Gibco | 11995065 | |
| Isopropanol | general | ||
| Kimwipe | Kimberly Clark | 21905-026 | |
| Microscope glass slides | VWR | 631-1550P | |
| MilliQ system | Merk Millipore | ZR0Q008WW | |
| OP1550 Interferometer | Optics11 Life | no catalogue number | |
| Optics 11 Life probe (k = 0.02-0.005 N/m, R = 3-3.5 um) | Optics 11 Life | no catologue number | |
| Optics 11 Life probe (k = 0.46-0.5 N/m, R = 50-55 um) | Optics 11 Life | no catologue number | |
| Penicillin/Streptomycin | Gibco | 15140122 | |
| RainX rain repellent | RainX | 26012 | |
| Standard petri dishes (90 mm) | Thermo Scientific | 101RTIRR | |
| Tetramethylethylenediamine | Sigma-Aldrich | 110-18-9 | |
| Vaccum dessicator | Thermo Scientific | 531-0250 | |
| Software | |||
| Data acquisition software (v 3.4.1) | Optics 11 Life | ||
| GitHub Desktop (Optional) | Microsoft | ||
| Python 3 | Python Software Foundation | ||
| Visual Studio Code (Optional) | Microsoft |
References
- Discher, D. E., Janmey, P. Y. W. Tissue cells feel and respond to the stiffness of their substrate. Science. 310 (5751), 1139-1143 (2005).
- Roca-Cusachs, P., Conte, V., Trepat, X. Quantifying forces in cell biology. Nature Cell Biology. 19 (7), 742-751 (2017).
- Guimarães, C. F., Gasperini, L., Marques, A. P., Reis, R. L. The stiffness of living tissues and its implications for tissue engineering. Nature. Reviews. Materials. 5, 351-370 (2020).
- Moeendarbary, E., Harris, A. R. Cell mechanics: Principles, practices, and prospects. Wiley Interdisciplinary. Reviews. Systems. Biology and Medicine. 6 (5), 371-388 (2014).
- Butcher, D. T., Alliston, T., Weaver, V. M. A tense situation: Forcing tumour progression. Nature Reviews Cancer. 9 (2), 108-122 (2009).
- Kubánková, M., et al. Physical phenotype of blood cells is altered in COVID-19. Biophysical Journal. 120 (14), 2838-2847 (2021).
- Engler, A. J., Sen, S., Sweeney, H. L., Discher, D. E. Matrix elasticity directs stem cell lineage specification. Cell. 126, 677-689 (2006).
- Ciccone, G., et al. What caging force cells feel in 3D hydrogels: A rheological perspective. Advanced Healthcare Materials. 9 (17), 2000517 (2020).
- Wu, P. H., et al. A comparison of methods to assess cell mechanical properties. Nature Methods. 15 (7), 491-498 (2018).
- McKee, C. T., Last, J. A., Russell, P., Murphy, C. J. Indentation versus tensile measurements of young's modulus for soft biological tissues. Tissue Engineering B Reviews. 17 (3), 155-164 (2011).
- Humphrey, J. D., Delange, S. L. . An Introduction to Biomechanics Solids and Fluids, Analysis and Design. , 271-371 (2004).
- Prevedel, R., Diz-Muñoz, A., Ruocco, G., Antonacci, G. Brillouin microscopy: an emerging tool for mechanobiology. Nature Methods. 16 (10), 969-977 (2019).
- Krieg, M., et al. Atomic force microscopy-based mechanobiology. Nature Reviews Physics. 1, 41-57 (2019).
- Norman, M. D. A., Ferreira, S. A., Jowett, G. M., Bozec, L., Gentleman, E. Measuring the elastic modulus of soft culture surfaces and three-dimensional hydrogels using atomic force microscopy. Nature Protocols. 16 (5), 2418-2449 (2021).
- Gavara, N. A beginner's guide to atomic force microscopy probing for cell mechanics. Microscopy Research and Technique. 80 (1), 75-84 (2017).
- Whitehead, A. J., Kirkland, N. J., Engler, A. J. Atomic force microscopy for live-cell and hydrogel measurement. Methods in Molecular Biology. 2299, 217-226 (2021).
- Chavan, D., et al. Ferrule-top nanoindenter: An optomechanical fiber sensor for nanoindentation. The Review of Scientific Instruments. 83 (11), 115110 (2012).
- Van Hoorn, H., Kurniawan, N. A., Koenderink Ac, G. H., Iannuzzi, D. Local dynamic mechanical analysis for heterogeneous soft matter using ferrule-top indentation. Soft Matter. 12 (12), 3066-3073 (2016).
- Baldini, F., et al. Biomechanics of cultured hepatic cells during different steatogenic hits. Journal of the Mechanical Behavior of Biomedical Materials. 97, 296-305 (2019).
- Emig, R., et al. Piezo1 channels contribute to the regulation of human atrial fibroblast mechanical properties and matrix stiffness sensing. Cells. 10 (3), 663 (2021).
- Dobre, O., et al. A hydrogel platform that incorporates laminin isoforms for efficient presentation of growth factors - Neural growth and osteogenesis. Advanced Functional Materials. 31 (21), 2010225 (2021).
- Antonovaite, N., Beekmans, S. V., Hol, E. M., Wadman, W. J., Iannuzzi, D. Regional variations in stiffness in live mouse brain tissue determined by depth-controlled indentation mapping. Scientific Reports. 8 (1), 12517 (2018).
- Wu, G., Gotthardt, M., Gollasch, M. Assessment of nanoindentation in stiffness measurement of soft biomaterials: kidney, liver, spleen and uterus. Scientific Reports. 10 (1), 18784 (2020).
- Lüchtefeld, I., et al. Elasticity spectra as a tool to investigate actin cortex mechanics. Journal of Nanobiotechnology. 18 (1), 147 (2020).
- Caliari, S. R., Burdick, J. A. A practical guide to hydrogels for cell culture. Nature Methods. 13 (5), 405-414 (2016).
- Blumlein, A., Williams, N., McManus, J. J. The mechanical properties of individual cell spheroids. Scientific Reports. 7 (1), 7346 (2017).
- Mirsandi, H., et al. Influence of wetting conditions on bubble formation from a submerged orifice. Experiments in Fluids. 61, 83 (2020).
- Vassalli, M., Ciccone, G. . CellMechLab/NanoPrepare: v0.1.1. Zendo. , (2021).
- Vassalli, M., Ciccone, G., Lüchtefeld, I. CellMechLab/nanoindentation: v1.0.0. Zendo. , (2021).
- Savitzky, A., Golay, M. J. E. Smoothing and differentiation of data by simplified least squares procedures. Analytical Chemistry. 36 (8), 1627-1639 (1964).
- Schafer, R. W. What is a savitzky-golay filter. IEEE Signal Processing Magazine. 28 (4), 111-117 (2011).
- Gavara, N. Combined strategies for optimal detection of the contact point in AFM force-indentation curves obtained on thin samples and adherent cells. Scientific Reports. 6, 21267 (2016).
- Lin, D. C., Dimitriadis, E. K., Horkay, F. Robust strategies for automated AFM force curve analysis-I. Non-adhesive indentation of soft, inhomogeneous materials. Journal of Biomechanical Engineering. 129 (3), 430-440 (2007).
- Virtanen, P., et al. SciPy 1.0: fundamental algorithms for scientific computing in Python. Nature Methods. 17 (3), 261-272 (2020).
- Tse, J. R., Engler, A. J. Preparation of hydrogel substrates with tunable mechanical properties. Current Protocols in Cell Biology. , (2010).
- Pharr, G. M., Oliver, W. C., Brotzen, F. R. On the generality of the relationship among contact stiffness, contact area, and elastic modulus during indentation. Journal of Materials Research. 7 (3), 613-617 (1992).
- Kumar, R., Saha, S., Sinha, B. Cell spread area and traction forces determine myosin-II-based cortex thickness regulation. Biochimica et Biophysica Acta - Molecular Cell Research. 1866 (12), 118516 (2019).
- Kunda, P., Pelling, A. E., Liu, T., Baum, B. Moesin controls cortical rigidity, cell rounding, and spindle morphogenesis during mitosis. Current Biology: CB. 18 (2), 91-101 (2008).
- Allen, M., Poggiali, D., Whitaker, K., Marshall, T. R., Kievit, R. A. Raincloud plots: a multi-platform tool for robust data visualization. Wellcome Open Research. 4, 63 (2019).
- Clark, A. G., Paluch, E. Mechanics and regulation of cell shape during the cell cycle. Results and Problems in Cell Differentiation. 53, 31-73 (2011).
- Langan, T. J., Rodgers, K. R., Chou, R. C. Synchronization of mammalian cell cultures by serum deprivation. Methods in Molecular Biology. 1524, 97-105 (2017).
- Hodgkinson, T., et al. The use of nanovibration to discover specific and potent bioactive metabolites that stimulate osteogenic differentiation in mesenchymal stem cells. Science Advances. 7 (9), 7921 (2021).
- Kloxin, A. M., Kloxin, C. J., Bowman, C. N., Anseth, K. S. Mechanical properties of cellularly responsive hydrogels and their experimental determination. Advanced Materials (Deerfield Beach, Fla). 22 (31), 3484-3494 (2010).
- Loessner, D., et al. Functionalization, preparation and use of cell-laden gelatin methacryloyl-based hydrogels as modular tissue culture platforms. Nature Protocols. 11 (4), 727-746 (2016).
- Yue, K., et al. Synthesis, properties, and biomedical applications of gelatin methacryloyl (GelMA) hydrogels. Biomaterials. 73, 254-271 (2015).
- Sarrigiannidis, S. O., et al. A tough act to follow: collagen hydrogel modifications to improve mechanical and growth factor loading capabilities. Materials Today. Bio. 10, 100098 (2021).
- Karoutas, A., et al. The NSL complex maintains nuclear architecture stability via lamin A/C acetylation. Nature Cell Biology. 21 (10), 1248-1260 (2019).
- Ryu, H., et al. Transparent, compliant 3D mesostructures for precise evaluation of mechanical characteristics of organoids. Advanced Materials (Deerfield Beach, Fla). 33 (25), 2100026 (2021).
- Carvalho, D. T. O., Feijão, T., Neves, M. I., Da Silva, R. M. P., Barrias, C. C. Directed self-assembly of spheroids into modular vascular beds for engineering large tissue constructs Biofabrication Directed self-assembly of spheroids into modular vascular beds for engineering large tissue constructs. Biofabrication. 13 (3), 035008 (2021).
- Chaudhuri, O., Cooper-White, J., Janmey, P. A., Mooney, D. J., Shenoy, V. B. Effects of extracellular matrix viscoelasticity on cellular behaviour. Nature. 584 (7822), 535-546 (2020).
- Cantini, M., Donnelly, H., Dalby, M. J., Salmeron-Sanchez, M. The plot thickens: The emerging role of matrix viscosity in cell mechanotransduction. Advanced Healthcare Materials. 9 (8), 1901259 (2020).
- Elosegui-Artola, A. The extracellular matrix viscoelasticity as a regulator of cell and tissue dynamics. Current Opinion in Cell Biology. 72, 10-18 (2021).
- Chaudhuri, O. Viscoelastic hydrogels for 3D cell culture. Biomaterials Science. 5 (8), 1480-1490 (2017).
- Nguyen, T. D., Gu, Y. Determination of strain-rate-dependent mechanical behavior of living and fixed osteocytes and chondrocytes using atomic force microscopy and inverse finite element analysis. Journal of Biomechanical Engineering. 136 (10), 101004 (2014).
- Mokbel, M., Hosseini, K., Aland, S., Fischer-Friedrich, E. The Poisson Ratio of the Cellular Actin Cortex Is Frequency Dependent. Biophysical Journal. 118 (8), 1968-1976 (2020).
- Javanmardi, Y., Colin-York, H., Szita, N., Fritzsche, M., Moeendarbary, E. Quantifying cell-generated forces: Poisson's ratio matters. Communications Physics. 4, 237 (2021).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved