需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
软物质纳米压痕的实验和数据分析工作流程
摘要
该协议为软材料纳米压痕实验(包括水凝胶和细胞)提供了完整的工作流程。首先,详细介绍了获取力谱数据的实验步骤;然后,通过新开发的开源Python软件详细分析这些数据,该软件可以从GitHub免费下载。
摘要
纳米压痕是指一类实验技术,其中使用微米力探针来量化软生物材料和细胞的局部机械性能。这种方法在机械生物学,生物材料设计和组织工程领域获得了核心作用,以获得与单细胞(μm)大小相当的软材料的适当机械表征。获取此类实验数据的最流行策略是使用原子力显微镜(AFM);虽然该仪器在力(低至pN)和空间(亚纳米)方面提供了前所未有的分辨率,但其可用性通常受到其复杂性的限制,这阻碍了对机械性能积分指标(例如杨氏模量(E))的常规测量。新一代纳米压痕,例如基于光纤传感技术的纳米压痕,最近因其易于集成而广受欢迎,同时允许以μm空间分辨率施加亚nN力,因此适用于探测水凝胶和细胞的局部机械性能。
在该协议中,提出了一个分步指南,详细介绍了使用市售的套圈顶部光纤传感纳米压痕仪获取水凝胶和细胞上的纳米压痕数据的实验程序。虽然某些步骤特定于本文中使用的仪器,但所提出的方案可以作为其它纳米压痕装置的指南,前提是某些步骤根据制造商的指南进行调整。此外,提出了一种新的开源Python软件,该软件配备了用于分析纳米压痕数据的用户友好图形用户界面,该软件允许筛选错误获取的曲线,数据过滤,通过不同的数值程序计算接触点, E的常规计算,以及特别适用于单细胞纳米压痕数据的更高级分析。
引言
力学在生物学中的基本作用现已确立1,2。从整个组织到单细胞,机械性能可以告知所研究的生物材料的病理生理状态3,4。例如,受癌症影响的乳腺组织比健康组织更硬,这一概念是流行的触诊测试5的基础。值得注意的是,最近已经表明,与SARS-CoV-2幼稚个体的血细胞相比,血细胞机械性能的变化强调了由严重急性呼吸综合征冠状病毒2(SARS-CoV-2)引起的2019冠状病毒病(COVID-19),包括红细胞变形性降低,淋巴细胞和中性粒细胞硬度降低6。
一般来说,细胞和组织的力学本质上是相互交织的:每个组织都具有特定的机械性能,同时影响并依赖于组成细胞和细胞外基质(ECM)的机械性能5。正因为如此,研究生物学力学的策略通常涉及工程基质与生理相关的机械刺激,以阐明细胞对这些刺激的反应行为。例如,Engler及其同事的开创性工作表明,间充质干细胞谱系的承诺受基质弹性控制,正如在软性和硬性二维聚丙烯酰胺(PAAm)水凝胶上研究的那样7。
存在许多机械表征所研究生物材料的策略,在空间尺度(即局部到整体)和变形模式(例如,轴向与剪切)上有所不同,因此产生不同的信息,需要仔细解释3,8,9,10。软生物材料的力学通常用刚度来表示。然而,刚度取决于材料属性和几何形状,而弹性模量是材料的基本属性,与材料的几何形状无关11。因此,不同的弹性模量与给定样品的刚度有关,每个弹性模量包括材料在不同边界条件下(例如,自由膨胀与约束)下对特定变形模式(例如,轴向与剪切)的抵抗力11,12。纳米压痕实验允许通过E量化机械性能,当生物材料未横向限制时,与单轴变形(压痕)相关10,11,12。
在微观尺度上量化生物系统E的最常用方法是AFM13,14,15,16。AFM是一种非常强大的工具,其力分辨率低至pN级,空间分辨率低至亚纳米级。此外,AFM在与互补的光学和机械工具耦合方面提供了极大的灵活性,扩展了其从正在研究的生物材料中提取大量信息的能力13。然而,这些吸引人的功能伴随着以实验设置的复杂性为代表的进入壁垒。在用户获得可靠数据之前,AFM需要经过广泛的培训,并且将其用于生物材料的日常机械表征通常是不合理的,特别是当不需要其独特的力和空间分辨率时。
正因为如此,一类新型纳米压痕器最近因其易用性而广受欢迎,同时仍然提供具有亚nN力分辨率和μm空间分辨率的AFM可比数据,反映细胞在相关长度尺度上施加和感知的力2。特别是,基于光纤传感技术17,18的套圈顶部纳米压痕器件在活跃在机械生物学及其他领域的研究人员中越来越受欢迎;并且已经发表了大量报告使用这些装置的生物材料的机械性能的工作,包括细胞19,20,水凝胶8,21和组织22,23。尽管这些系统能够探测局部动态机械性能(即存储和损耗模量),但产生E的准静态实验仍然是最受欢迎的选择8,19,20,21。简而言之,准静态纳米压痕实验包括以恒定速度压痕样品,直至由最大位移、力或压痕深度定义的设定点,并在所谓的力-距离(F-z)曲线中记录悬臂的力和垂直位置。然后通过识别接触点(CP)将F-z曲线转换为力压痕(F-δ)曲线,并拟合适当的接触力学模型(通常是赫兹模型13)来计算E。
虽然套圈顶部纳米压痕的操作类似于AFM测量,但有一些特殊性值得考虑。在这项工作中,提供了使用市售套圈顶部纳米压痕仪从细胞和组织模拟水凝胶中稳健地获取 F-z 曲线的分步指南,以鼓励使用该装置和其他类似设备的研究小组之间的实验程序标准化。此外,还提供了有关如何最好地制备水凝胶样品和细胞以进行纳米压痕实验的建议,以及实验途径中的故障排除提示。
此外,纳米压痕结果(即E及其分布)的大部分变异性取决于用于分析数据的特定程序,这是不平凡的。为了解决这个问题,提供了使用新开发的开源软件的说明,该软件用Python编程,并配备了用户友好的图形用户界面(GUI),用于F-z曲线的批量分析。该软件允许快速数据筛选,数据过滤,通过不同的数值程序计算CP,E的常规计算,以及称为弹性光谱24的更高级分析,允许估计细胞的体积杨氏模量,肌动蛋白皮层的杨氏模量和肌动蛋白皮层的厚度。该软件可以从GitHub免费下载,并且可以通过添加适当的数据解析器轻松调整以分析来自其他系统的数据。需要强调的是,该协议可用于其它套圈顶部纳米压痕装置,以及一般的其它纳米压痕装置,前提是某些步骤根据特定仪器的指南进行调整。该协议在图1中进行了示意性总结。
研究方案
1. 制备用于纳米压痕测量的底物/细胞
- 按照 补充方案中 给出的步骤制备用于纳米压痕实验的PAAm水凝胶/细胞。 图 2 总结了该过程。
注意:已选择PAAm水凝胶,因为它们是机械生物学领域最常用的水凝胶。然而,该方案同样适用于任何类型的水凝胶25 (参见 讨论,方法的修改)。
2. 启动设备、探头选择和探头校准
- 按照 补充协议 中给出的步骤启动设备。有关光纤套圈顶部纳米压痕操作的技术细节,请查看这些参考文献17,18。
- 如下所述选择纳米压痕探针。
注意:所有市售探头(例如本协议中使用的探头)都配备了球形尖端(图3A)。因此,选择范围缩小到两个变量:悬臂的刚度和尖端半径(图3B)。- 选择与预期样品刚度相匹配的悬臂刚度(k ,单位为N / m),以获得最佳结果15 (参见 图3C 和 讨论,协议中的关键步骤)。对于细胞,选择 k 在0.01-0.09 N / m范围内的探针。对于水凝胶,选择 k 在0.1-0.9 N/m范围内的探针,对于预期 E 在几kPa至100 kPa之间的凝胶,可获得最佳结果(参见 代表性结果)。
- 根据压痕过程所需的空间分辨率选择尖端半径(R 单位为 μm)。对于小细胞,如人胚胎肾293T(HEK293T)细胞(平均直径~10-15μm26),选择一个 R = 3μm的球体。对于水凝胶,选择一个 R = 10-250μm的球体,以探测生物材料在大接触面积上的机械性能,并避免局部异质性。
- 请参阅 图 3C 和任何其他制造商指南以选择合适的探头。
- 选择探头后,按照 补充协议 中的步骤将其安装在纳米压痕上。
3. 探头校准
注意:以下步骤特定于基于光纤传感技术的套圈顶部纳米压痕设备,并且针对软件版本3.4.1进行了详细说明。对于其他纳米压痕设备,请按照设备制造商建议的步骤操作。
- 在软件的主窗口中,单击 初始化。将出现校准菜单。输入探头详细信息(可在探头盒的侧面找到; k 以N/m为单位, R 以μm为单位,校准因子以空气为单位)在输入框中。
- 准备校准皿:一个平底的厚玻璃培养皿(参见 讨论,协议中的关键步骤)。用与样品培养皿相同的介质填充培养皿(也可以是空气)。使培养基的温度与样品的温度相匹配。
- 将校准盘放在探头下方。如果需要,将探头从纳米压痕仪的臂支架上滑出,并用一只手握住它,为放置校准皿腾出空间。将探头滑回到位。
- 执行以下两个步骤在液体中校准。如果在空气中测量,请使用提供的聚四氟乙烯基材进行校准,然后跳至步骤5。
- 使用巴斯德移液器用一滴70%乙醇预润湿探头,移液器的末端与玻璃套圈轻轻接触,使液滴滑过悬臂和球形尖端27 (参见 讨论,协议中的关键步骤)。
- 手动向下滑动纳米压痕仪的臂,直到探针完全浸没,但仍远离培养皿底部。如果需要,在校准皿中加入更多培养基。等待5分钟,使液体达到平衡条件。
- 在软件的 初始化 菜单中,单击 扫描波长。干涉仪的屏幕将显示进度条,计算机软件上的 实时信号 窗口将显示 图S1A(左)所示的模式。要检查光学扫描是否成功,请导航到干涉仪框上的 波长扫描 面板。如果成功,应该可以看到正弦波(图1A,右)。如果出现错误,请参阅 讨论 (方法的故障排除)。
- 在" 初始化 "菜单中,单击 "查找表面",这将逐渐降低探头,直到达到悬臂弯曲的设定阈值。当与玻璃培养皿接触时,探头停止移动。
- 检查探头是否与表面接触。使用软件主窗口上的 y 向下箭头按钮将探头向下移动1μm。当悬臂与基板接触时,观察软件实时 窗口中 的绿色信号(悬臂偏转),以了解每一步基线的变化(图 S1B)。如果没有变化,则悬臂不接触(见下一步)。
- 将"查找曲面"选项卡下的"选项"菜单中的"阈值"从默认值 0.01 一次增加 0.01,然后重复"查找曲面"步骤直至接触。或者,以 1 μm 的小步长将探针向下接触,直到绿色基线在每个向下的步骤开始移动。
注意: 对于最软的悬臂 (k = 0.025 Nm-1),请在执行步骤 6 时先验地增加"查找表面"选项卡下"选项"菜单中的"阈值"。从阈值 0.06 或 0.07 开始,如有必要,将其增加到 0.1。这是因为环境噪音可能会导致悬臂在接触前弯曲超过阈值。对于软探头,在同一菜单中降低接近速度(μm/s)也可以改进程序。 - 在" 初始化 "菜单中,单击" 校准"。
- 检查软件的实时信号窗口,确保压电陶瓷的位移和悬臂的偏转 信号 同时向上移动(图S1C)。
- 如果时间不匹配,则探头没有与玻璃完全接触。以1μm的步长向下移动探头,直到悬臂信号的基线发生变化(参见步骤7),然后重复步骤9。
- 如果在 校准 步骤中悬臂信号根本没有变化,则探头离表面很远。反复增加接触阈值(参见步骤8),直到正确找到表面,然后从头开始从波长扫描开始重复校准。
- 校准完成后,在弹出的弹出窗口中检查新旧校准因子。如果新的校准因子在正确的范围内,请单击使用新因子。如果校准失败,并且新因子为 NaN 或不在预期范围内,请参阅讨论(方法故障排除)以获取解决方案。
注意:如果在折射率为n(水为n = 1.33)的液体介质中进行校准,则新系数应比探头包装盒上提供的系数低~n倍。如果在空气中进行校准,则新旧校准因子应大致相等。 - 检查解调环是否已正确校准,如下所示。导航到干涉仪桌面上的 解调 选项卡。轻轻敲击光学工作台或纳米压痕仪以产生足够的噪音。由离散数据点组成的白色圆圈应近似覆盖红色圆圈(图S1D)。
- 如果白色圆圈不与红色圆圈重叠,或者干涉仪显示屏上出现警告,则需要重新校准解调圆圈。这可以通过两种方式完成,如下所述。
- 连续敲击纳米压痕仪的主体以产生一整圈噪声,然后按下干涉仪上的 校准 按钮。
- 进入与玻璃基板接触的物体,然后从软件主窗口的初始化菜单中按校准。不要保存校准因子。此时,再次检查并确保白色圆圈与红色圆圈重叠。
注意:如果信号不仅从解调圈中略微偏移,而且变得非常小或根本不可见,则意味着悬臂粘在光纤上。按照此问题的疑难解答建议进行操作(请参阅 讨论,方法疑难解答),然后重复步骤 13.1 或 13.2。一旦悬臂回到其水平位置(图3A),信号将恢复到解调圈。
- 如下所述,在校准后直接在玻璃基板上执行压痕来验证校准。
- 通过单击"配置实验"并添加"查找曲面"步骤和"缩进"步骤来加载或创建实验文件。对于压痕步骤,使用默认位移模式设置并将最大位移更改为校准距离(3,000 nm),以使探头靠在刚性基板上。
- 单击 运行实验 并在干涉仪窗口中检查解调圆圈。检查白色信号,并确保它在压痕过程中位于红色圆圈的顶部。
- 在 时间数据 图中的软件主窗口中检查结果,并确保压电陶瓷的位移(蓝线)等于悬臂的挠度(绿线),因为压痕从接触开始,并且预计不会发生材料变形。如果信号不并行,请参阅 讨论 (方法故障排除)。
- 通过将校准保存路径设置为适当的目录来更改校准菜单中的本地 路径 。
- 成功校准探头后,将压电陶瓷向上移动 500 μm。
4. 测量软材料的杨氏模量
- 水凝胶的纳米压痕
- 将含有样品的培养皿装载到显微镜载物台上,并手动将纳米压痕仪的探针移动到样品上方所需的 x-y 位置。
- 手动将探针滑入溶液中,注意在此阶段在探针和样品表面之间留出1-2毫米。等待5分钟,使探头在培养基中平衡。
- 聚焦光学显微镜的 z 平面,使探头清晰可见。
- 执行单个压痕以调整实验参数,如下所述。
- 在软件的主窗口中配置新实验。单击"配置实验",这将打开一个新窗口。添加"查找曲面"步骤。如果需要,可以在软件的"选项"菜单中更改"查找表面"步骤的所有参数。
注意:查找表面将降低探针,直到找到表面,然后将探针缩回由Z以上表面(μm)定义的距离,高于样品表面。如果所选悬臂对于样品来说太硬或样品粘稠,则在步骤之后,探针可能仍与样品接触,这将导致没有基线的曲线(图4C)。要解决此问题,请增加表面上的Z值(μm)。 - 添加缩 进 步骤。选择 配置文件 选项卡,然后单击位 移控制。保留默认缩进配置文件。
- 单击主软件窗口上的 运行实验 。这将找到表面并执行单个缩进。如果单个压痕看起来与预期不符,请按照 图4 和 讨论 (方法故障排除)中所述调整实验参数。
- 在软件的主窗口中配置新实验。单击"配置实验",这将打开一个新窗口。添加"查找曲面"步骤。如果需要,可以在软件的"选项"菜单中更改"查找表面"步骤的所有参数。
- 压痕看起来符合预期后,配置矩阵扫描,以便缩进样品的足够区域。单击" 配置实验",使用先前确定的实验参数添加" 查找表面 "步骤,并添加 "矩阵扫描 "步骤。
- 对于扁平水凝胶,配置包含50-100个点(即x和y中的5 x 10或10 x 10)的基质扫描,间隔为10-100μm(即dx = dy = 10-100μm)。单击使用载物台位置从当前载物台位置开始矩阵扫描。确保勾选"自动查找表面"框,以使用设置的实验参数在每个压痕处查找表面。
- 为避免过采样,请将步长设置为接触半径的至少两倍(
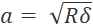 其中 δ 是压痕深度)。
其中 δ 是压痕深度)。 - 在位移控制中设置矩阵扫描配置文件。确保配置文件不违反赫兹模型的假设(请参阅 讨论,协议中的关键步骤)。
- 将线段数保留为 5(默认值),并使用默认位移轮廓。如有必要,根据每个倾斜段的最大位移和时间更改位移剖面,这将分别影响最大压痕深度和应变率。不要超过10μm/s>应变率(参见 讨论,方法的局限性)。
- 输入接近速度值,该值确定探头在接触前向样品移动的速度。使缩回速度与接近速度相匹配(请参阅下面的注释)。
注意:对于软悬臂和嘈杂的环境,建议接近速度为 1,000-2,000 nm/s。对于较硬的悬臂和受控环境,可以增加这一点。 - 将配置的试验保存在所需的实验路径中,并在"配置试验"窗口的"常规"选项卡中选择将数据保存在"保存路径"中的目录。单击"运行实验"。
- 为避免过采样,请将步长设置为接触半径的至少两倍(
- 基质扫描完成后,将探针升高200-500μm,并将探针移动到样品的不同区域,远离第一个区域。
- 重复实验至少两次,以便在每个样品上获得足够的数据(即,每个样品至少两次矩阵扫描,每个包含50-100条曲线)。
- 细胞的纳米压痕
- 如上所述将样品加载到显微镜上。
- 对于单细胞压痕,聚焦 z 平面,使细胞和探针在 20 倍或 40 倍放大倍率下都可见,具体取决于细胞大小和扩散。
- 将探针移到要缩进的单元格上方。
- 在软件的主窗口中配置新实验。单击" 配置实验",这将打开一个新窗口。在置换模式下使用默认参数添加" 查找曲面 和 缩进 "步骤。
- 单击" 运行实验",这将找到表面并执行单个压痕。检查缩进是否成功。如果曲线看起来与预期不符,请调整实验参数(参见 图4 和 讨论,方法的故障排除)。
- 如果压痕成功,则向实验添加矩阵扫描。遵循水凝胶纳米压痕实验给出的步骤;配置矩阵扫描,使步长允许缩进细胞的一小部分区域;HEK293T细胞的25点间隔为0.5-5μm。
- 根据像元大小,调整矩阵扫描以确保针尖不会缩进超出像元限制,即执行不同的地图几何形状或探测更少的点。
- 单击" 运行实验 "并等待其完成。
- 基质扫描完成后,将探头脱离接触( z 平面50μm)。
- 将探针移动到新细胞上方并重复该过程(请参阅 讨论,协议中的关键步骤)。
- 清洁探头并关闭仪器
- 按照 补充协议 中给出的步骤清洁探针并关闭纳米压痕装置。
5. 数据分析
- 下载和安装软件
- 按照 补充协议 中给出的步骤下载并安装数据分析软件28,29.
- 筛选 F-z 曲线并生成 JSON 格式的清理数据集
- 从实验室计算机上的命令行启动 prepare.py ,如步骤 2 到 3 中所述。
- 如果使用Windows计算机,请在右键单击 NanoPrepare 文件夹的同时按住shift键,然后单击 在此处打开PowerShell窗口。键入 python prepare.py 命令,然后按 Enter 键。屏幕上将弹出一个 GUI(图 S2)。
- 如果使用MacOS计算机,请右键单击 NanoPrepare 文件夹,然后单击 "文件夹中的新终端"。键入 python3 prepare.py 命令并按 Enter 键,这将启动 GUI(图 S2)。
- 从下拉列表中选择 O11新 数据格式。如果数据未正确加载,请重新启动 GUI 并选择 O11OLD。
注意: O11NEW 格式适用于使用软件版本 3.4.1 的套圈顶部光纤传感纳米压痕设备获得的数据。这种格式也适用于以前的软件版本,至少是那些属于2019-2020年安装的纳米压痕器的版本。 - 单击 加载文件夹。选择包含要分析的数据的文件夹 - 单矩阵扫描或多矩阵扫描。顶部图表(原始曲线)将使用上传的数据集进行填充。要可视化特定曲线,请单击它。这将以绿色突出显示它,并将其显示在底部图形(当前曲线)上。
- 使用 GUI 右侧的选项卡清理数据集,如下所述。
- 使用"线段"按钮选择要分析的正确线段,即 F-z 曲线的前向线段。具体数量取决于进行实验时在纳米压痕软件中选择的片段数。
- 使用裁剪 50 nm 按钮将最左侧(如果勾选 L)、右侧(如果勾选 R)或两侧(如果同时勾选 R 和 L)的曲线裁剪 50 nm。多次单击此按钮可根据需要裁剪尽可能多的内容。使用此选项可消除 F-z 曲线起点/终点处的伪影。
- 检查 悬臂 卡舌的弹簧常数、尖端几何形状和尖端半径。检查选项卡以确保已正确读取元数据。
- 使用 "筛选 "选项卡设置力阈值,该阈值将丢弃所有未达到给定力的曲线。丢弃的曲线将以红色突出显示。
- 使用"手动切换"按钮手动删除未正确采集的曲线。通过单击特定曲线并选择 OUT来删除任何曲线,这将以红色突出显示曲线。
- 单击 保存 JSON。为清理的数据集输入适当的名称,即单个 JSON 文件。将 JSON 文件发送到安装了 NanoAnalysis 软件的计算机。
6. 形式化的数据分析
- 通过导航到 NanoAnalysis 文件夹并启动终端,从命令行启动 nano.py 文件,如前所述。键入 python nano.py 或 python3 nano.py 命令(取决于操作系统),然后按 Enter 键。屏幕上将弹出一个 GUI(图 S3)。
- 在 GUI 的左上角,单击" 加载试验 "并选择 JSON 文件。这将填充文件列表和 原始曲线 图,以 F-z 曲线显示数据集。 F 轴和 z 轴都相对于 CP 坐标显示,CP 坐标在加载数据集时在后台计算(请参阅以下注释)。在 统计信息 框中,选中三个参数的值:N已激活、N 失败 和 N 已排除。
注意:激活 的N表示将在后续赫兹/弹性谱分析中分析的曲线数量,并在 原始曲线 图中显示为黑色。N失败 表示无法找到可靠 CP 并在图形中显示为蓝色的曲线数。这些曲线将在后续分析中自动丢弃。打开软件后,某些曲线可能会自动移动到失败的集合。这是因为 CP 是在使用默认阈值算法打开软件时计算的(见下文)。排除 的 N 表示手动选择要从分析中排除的曲线,并将在图形中显示为红色(见下文)。 - 检查失败曲线、排除曲线和激活曲线的数量是否合理。检查原始曲线图以可视化 曲线 。
- 要更详细地可视化特定曲线,请单击它。这将以绿色突出显示它,并将其显示在 当前曲线 图上。选择单个曲线后, R 和 k 参数(对于所有曲线必须相同)将填充到 GUI 的 "统计 "框中。
- 使用 切换 框更改给定曲线的状态。单击要更改其状态的特定曲线,然后单击"已 激活"、"失败"或 "已排除"。" 统计信息 "框中的计数会自动更新。
注: 要更改"原始曲线"图形中数据集的视图,请使用"视图"框。单击"全部"以显示所有曲线(即,激活、失败和以相应的颜色排除)。单击"失败"以显示已激活和失败的曲线,然后单击"已激活"以仅显示激活的曲线。要重置已激活和已排除之间所有曲线的状态,请单击"重置"框中的"已激活"或"已排除"。 - 进一步清理数据集后,请按照下面概述的数据分析管道进行操作。
- 使用GUI(过滤框)中实现的滤波器过滤曲线中存在的任何噪声,即称为突出滤波器的自定义滤波器,Savitzky Golay30,31(SAVGOL)滤波器,以及基于计算给定窗口中数据的中位数的平滑滤波器(中值滤波器)。有关过滤器的详细信息,请参阅讨论(协议中的关键步骤)。
- 检查 "当前曲线 "图形中的过滤曲线。过滤后的曲线显示为黑色,而未过滤的曲线版本以绿色显示。
注意:建议尽可能少地过滤数据,以保留原始信号的特征。过度过滤可能会消除数据中存在的任何差异。使用激活的突出滤光片足以对纳米压痕数据进行赫兹分析。如果数据特别嘈杂,则可以额外应用SAVGOL或中值滤波器。 - 选择算法以查找 CP。在"接触点"框中,选择已在软件中实现的一系列数值过程之一,即拟合优度 (GoF)32、方差比 (RoV)32、二阶导数 33 或阈值 33。有关算法的详细信息,请参阅讨论,协议中的关键步骤。
注意:CP是探头与材料接触的点,需要识别才能将F-z数据转换为F-δ数据(对于软材料,δ是有限的,需要计算)。所选算法将应用于数据集的所有活动曲线,算法无法稳健定位CP的曲线将被移动到失败集。 - 调整算法的参数以适合您的数据集,以便正确定位 CP,如 协议中的关键步骤讨论中所述。要查看在单个曲线上找到 CP 的位置,请单击曲线并单击" 检查"来选择该曲线。检查出现的弹出窗口以识别 CP 所在的位置。
- 检查出现的红线(即算法在所选感兴趣区域中计算的参数)是否具有对应于CP位置的最大值或最小值(例如,对于GoF,参数为 R2)。如果需要,对所有曲线重复此过程。
注: 弹出窗口的轴为绝对坐标,以便显示 CP 的位置。相反, 原始 曲线和 电流曲线 图的轴相对于 CP 显示,即 CP 的位置为 (0,0)。 - 单击 赫兹分析。这将生成下面描述的三个图形。
- 检查数据集中的各个 F-δ 曲线以及平均赫兹拟合(红色虚线)。调整赫兹模型在" 结果 "框中的最大 缩进 (nm) 下的缩进(以 nm 为单位)。将其设置为最大 R 的 ~10%,赫兹模型才有效(请参阅 讨论,协议中的关键步骤)。
- 检查平均 F-δ 曲线,误差带显示一个标准偏差 (SD) 以及平均赫兹拟合(红色虚线)。可视化原始曲线图上的平均赫兹拟合以供参考,并可视化当前曲线上每条曲线的赫兹拟合。
- 检查 E 的散点图,该散点图源自将赫兹模型拟合到每条单独的曲线。
- 单击文件名、 F-z 曲线、 F-δ 曲线或散点图上的点以突出显示每个图中的曲线。如果散点图中的数据点似乎位于数据分布之外,请单击该数据点并检查其所属的曲线。通过单击" 检查 "按钮验证是否正确定位了 CP。如有必要,从分析中排除曲线。
- 检查计算平均值 E 及其 SD(Eγ ± σ)的结果框,并确保它们对于给定实验是合理的。
- 在" 保存 "框中,单击" 赫兹"。在弹出窗口中,输入文件名和目录。完成后,单击 保存。将创建一个 .tsv 文件。在任何其他首选软件中打开 .tsv 文件,并使用这些值进行统计分析和进一步绘制。
注意:该文件包含从每条曲线获得的 E 和平均值 E 及其 SD。 此外,该文件还包含与分析关联的元数据,包括分析的曲线数、R、k 和用于赫兹模型的最大缩进。 - 此步骤是可选的。单击 平均 F-Ind 以导出平均力和平均压痕,以及力中的一个 SD。
- 有关细胞纳米压痕数据,请单击弹性光谱分析(参见代表性结果和讨论)。检查生成的两个图,即 E 作为每条曲线的压痕深度 (E(δ)) 的函数,以及误差带的平均 E(δ) 显示一个 SD(红色实线和阴影区域)由模型(黑色虚线)拟合,该模型允许估计细胞的肌动蛋白皮层的杨氏模量,细胞的体积杨氏模量, 和肌动蛋白皮层的厚度。此外,检查顶部图中的平均 E(δ) 红色。
- 确保选中插 值 框,以确保在插值信号上计算执行弹性谱分析所需的导数(参见 代表性结果)。
- 检查结果框,报告皮层的杨氏模量(E 0 ± σ),细胞的体积杨氏模量(Eb ± σ)和皮层厚度(d 0 ± σ)。
注意:平均弹性谱起初可能出现噪声,并伴有明显的正弦振荡。因此,方程(3)可能无法正确拟合。如果是这种情况,迭代增加平滑SAVGOL滤波器34 的窗口长度可以解决此问题。 - 分析完成后,单击"保存"框中的"ES"。这将导出指定目录中的 .tsv 文件,其中包含平均压痕深度和接触半径函数的平均弹性、与实验关联的元数据(见上文)以及上面解释的估计模型参数。最后,还报告了不考虑对δ及其SD的依赖性的平均弹性。
- 关闭软件并将保存的结果输入任何其他首选软件,以进一步绘制数据并执行统计分析。
- 此步骤是可选的:通过右键单击图形并选择 导出,从 GUI 导出图形。.svg导出图形,以便参数如字体、字体大小、线条样式等。可以在所选的其他软件中进行编辑。
注意:自定义 CP 算法和滤波器可以编程并添加到现有的算法和滤波器中。详见 补充说明1 。
结果
按照协议,获得一组F-z曲线。数据集很可能包含良好的曲线,以及在继续分析之前要丢弃的曲线。通常,如果曲线的形状与图4A所示的形状不同,则应丢弃曲线。图5AI显示了在NanoPrepare GUI中上传的预期E 0.8 KPa35的软PAAm水凝胶上获得的~100条曲线的数据集。大多数曲线呈现清晰、平坦的基线、过渡区域和与材料表观刚度成比?...
讨论
该协议展示了如何在水凝胶和单细胞上使用市售的套圈顶部纳米压痕仪稳健地获取力谱纳米压痕数据。此外,还提供了使用用Python编程的开源软件的说明,该软件包括用于分析纳米压痕数据的精确工作流程。
协议中的关键步骤
在遵循此协议时,以下步骤已被确定为特别重要。
样品制备
至关重要的是,在开始测量之前?...
披露声明
作者没有什么可透露的。
致谢
GC和MAGO承认CeMi的所有成员。MSS通过EPSRC计划赠款(EP / P001114 / 1) 获得 支持。
GC:软件(对软件开发和算法的贡献),形式分析(纳米压痕数据分析),验证,调查(聚丙烯酰胺凝胶的纳米压痕实验),数据管理,写作(原始草稿,审查和编辑),可视化(图形和图形)。 MAGO:调查(凝胶和细胞样品的制备,细胞的纳米压痕实验),写作(原始草稿,审查和编辑),可视化(图形和图形)。 NA:验证、写作(审查和编辑)。 IL:软件(对软件开发和算法的贡献)、验证、编写(审查和编辑); MV:概念化,软件(原始软件和算法的设计和开发),验证,资源,写作(原始草案,审查和编辑),监督,项目管理,资金获取 MSS:资源,写作(审查和编辑),监督,项目管理,资金获取。所有作者都阅读并批准了最终手稿。
材料
| Name | Company | Catalog Number | Comments |
| 12 mm coverslips | VWR | 631-1577P | |
| 35 mm cell treated culture dishes | Greiner CELLSTAR | 627160 | |
| Acrylamide | Sigma-Aldrich | A4058 | |
| Acrylsilane | Alfa Aesar | L16400 | |
| Ammonium Persulfate | Merk | 7727-54-0 | |
| Bisacrylamide | Merk | 110-26-9 | |
| Chiaro nanoindenter | Optics 11 Life | no catologue number | |
| Ethanol | general | ||
| Fetal bovine serum | Gibco | 16140071 | |
| High glucose DMEM | Gibco | 11995065 | |
| Isopropanol | general | ||
| Kimwipe | Kimberly Clark | 21905-026 | |
| Microscope glass slides | VWR | 631-1550P | |
| MilliQ system | Merk Millipore | ZR0Q008WW | |
| OP1550 Interferometer | Optics11 Life | no catalogue number | |
| Optics 11 Life probe (k = 0.02-0.005 N/m, R = 3-3.5 um) | Optics 11 Life | no catologue number | |
| Optics 11 Life probe (k = 0.46-0.5 N/m, R = 50-55 um) | Optics 11 Life | no catologue number | |
| Penicillin/Streptomycin | Gibco | 15140122 | |
| RainX rain repellent | RainX | 26012 | |
| Standard petri dishes (90 mm) | Thermo Scientific | 101RTIRR | |
| Tetramethylethylenediamine | Sigma-Aldrich | 110-18-9 | |
| Vaccum dessicator | Thermo Scientific | 531-0250 | |
| Software | |||
| Data acquisition software (v 3.4.1) | Optics 11 Life | ||
| GitHub Desktop (Optional) | Microsoft | ||
| Python 3 | Python Software Foundation | ||
| Visual Studio Code (Optional) | Microsoft |
参考文献
- Discher, D. E., Janmey, P. Y. W. Tissue cells feel and respond to the stiffness of their substrate. Science. 310 (5751), 1139-1143 (2005).
- Roca-Cusachs, P., Conte, V., Trepat, X. Quantifying forces in cell biology. Nature Cell Biology. 19 (7), 742-751 (2017).
- Guimarães, C. F., Gasperini, L., Marques, A. P., Reis, R. L. The stiffness of living tissues and its implications for tissue engineering. Nature. Reviews. Materials. 5, 351-370 (2020).
- Moeendarbary, E., Harris, A. R. Cell mechanics: Principles, practices, and prospects. Wiley Interdisciplinary. Reviews. Systems. Biology and Medicine. 6 (5), 371-388 (2014).
- Butcher, D. T., Alliston, T., Weaver, V. M. A tense situation: Forcing tumour progression. Nature Reviews Cancer. 9 (2), 108-122 (2009).
- Kubánková, M., et al. Physical phenotype of blood cells is altered in COVID-19. Biophysical Journal. 120 (14), 2838-2847 (2021).
- Engler, A. J., Sen, S., Sweeney, H. L., Discher, D. E. Matrix elasticity directs stem cell lineage specification. Cell. 126, 677-689 (2006).
- Ciccone, G., et al. What caging force cells feel in 3D hydrogels: A rheological perspective. Advanced Healthcare Materials. 9 (17), 2000517 (2020).
- Wu, P. H., et al. A comparison of methods to assess cell mechanical properties. Nature Methods. 15 (7), 491-498 (2018).
- McKee, C. T., Last, J. A., Russell, P., Murphy, C. J. Indentation versus tensile measurements of young's modulus for soft biological tissues. Tissue Engineering B Reviews. 17 (3), 155-164 (2011).
- Humphrey, J. D., Delange, S. L. . An Introduction to Biomechanics Solids and Fluids, Analysis and Design. , 271-371 (2004).
- Prevedel, R., Diz-Muñoz, A., Ruocco, G., Antonacci, G. Brillouin microscopy: an emerging tool for mechanobiology. Nature Methods. 16 (10), 969-977 (2019).
- Krieg, M., et al. Atomic force microscopy-based mechanobiology. Nature Reviews Physics. 1, 41-57 (2019).
- Norman, M. D. A., Ferreira, S. A., Jowett, G. M., Bozec, L., Gentleman, E. Measuring the elastic modulus of soft culture surfaces and three-dimensional hydrogels using atomic force microscopy. Nature Protocols. 16 (5), 2418-2449 (2021).
- Gavara, N. A beginner's guide to atomic force microscopy probing for cell mechanics. Microscopy Research and Technique. 80 (1), 75-84 (2017).
- Whitehead, A. J., Kirkland, N. J., Engler, A. J. Atomic force microscopy for live-cell and hydrogel measurement. Methods in Molecular Biology. 2299, 217-226 (2021).
- Chavan, D., et al. Ferrule-top nanoindenter: An optomechanical fiber sensor for nanoindentation. The Review of Scientific Instruments. 83 (11), 115110 (2012).
- Van Hoorn, H., Kurniawan, N. A., Koenderink Ac, G. H., Iannuzzi, D. Local dynamic mechanical analysis for heterogeneous soft matter using ferrule-top indentation. Soft Matter. 12 (12), 3066-3073 (2016).
- Baldini, F., et al. Biomechanics of cultured hepatic cells during different steatogenic hits. Journal of the Mechanical Behavior of Biomedical Materials. 97, 296-305 (2019).
- Emig, R., et al. Piezo1 channels contribute to the regulation of human atrial fibroblast mechanical properties and matrix stiffness sensing. Cells. 10 (3), 663 (2021).
- Dobre, O., et al. A hydrogel platform that incorporates laminin isoforms for efficient presentation of growth factors - Neural growth and osteogenesis. Advanced Functional Materials. 31 (21), 2010225 (2021).
- Antonovaite, N., Beekmans, S. V., Hol, E. M., Wadman, W. J., Iannuzzi, D. Regional variations in stiffness in live mouse brain tissue determined by depth-controlled indentation mapping. Scientific Reports. 8 (1), 12517 (2018).
- Wu, G., Gotthardt, M., Gollasch, M. Assessment of nanoindentation in stiffness measurement of soft biomaterials: kidney, liver, spleen and uterus. Scientific Reports. 10 (1), 18784 (2020).
- Lüchtefeld, I., et al. Elasticity spectra as a tool to investigate actin cortex mechanics. Journal of Nanobiotechnology. 18 (1), 147 (2020).
- Caliari, S. R., Burdick, J. A. A practical guide to hydrogels for cell culture. Nature Methods. 13 (5), 405-414 (2016).
- Blumlein, A., Williams, N., McManus, J. J. The mechanical properties of individual cell spheroids. Scientific Reports. 7 (1), 7346 (2017).
- Mirsandi, H., et al. Influence of wetting conditions on bubble formation from a submerged orifice. Experiments in Fluids. 61, 83 (2020).
- Vassalli, M., Ciccone, G. . CellMechLab/NanoPrepare: v0.1.1. Zendo. , (2021).
- Vassalli, M., Ciccone, G., Lüchtefeld, I. CellMechLab/nanoindentation: v1.0.0. Zendo. , (2021).
- Savitzky, A., Golay, M. J. E. Smoothing and differentiation of data by simplified least squares procedures. Analytical Chemistry. 36 (8), 1627-1639 (1964).
- Schafer, R. W. What is a savitzky-golay filter. IEEE Signal Processing Magazine. 28 (4), 111-117 (2011).
- Gavara, N. Combined strategies for optimal detection of the contact point in AFM force-indentation curves obtained on thin samples and adherent cells. Scientific Reports. 6, 21267 (2016).
- Lin, D. C., Dimitriadis, E. K., Horkay, F. Robust strategies for automated AFM force curve analysis-I. Non-adhesive indentation of soft, inhomogeneous materials. Journal of Biomechanical Engineering. 129 (3), 430-440 (2007).
- Virtanen, P., et al. SciPy 1.0: fundamental algorithms for scientific computing in Python. Nature Methods. 17 (3), 261-272 (2020).
- Tse, J. R., Engler, A. J. Preparation of hydrogel substrates with tunable mechanical properties. Current Protocols in Cell Biology. , (2010).
- Pharr, G. M., Oliver, W. C., Brotzen, F. R. On the generality of the relationship among contact stiffness, contact area, and elastic modulus during indentation. Journal of Materials Research. 7 (3), 613-617 (1992).
- Kumar, R., Saha, S., Sinha, B. Cell spread area and traction forces determine myosin-II-based cortex thickness regulation. Biochimica et Biophysica Acta - Molecular Cell Research. 1866 (12), 118516 (2019).
- Kunda, P., Pelling, A. E., Liu, T., Baum, B. Moesin controls cortical rigidity, cell rounding, and spindle morphogenesis during mitosis. Current Biology: CB. 18 (2), 91-101 (2008).
- Allen, M., Poggiali, D., Whitaker, K., Marshall, T. R., Kievit, R. A. Raincloud plots: a multi-platform tool for robust data visualization. Wellcome Open Research. 4, 63 (2019).
- Clark, A. G., Paluch, E. Mechanics and regulation of cell shape during the cell cycle. Results and Problems in Cell Differentiation. 53, 31-73 (2011).
- Langan, T. J., Rodgers, K. R., Chou, R. C. Synchronization of mammalian cell cultures by serum deprivation. Methods in Molecular Biology. 1524, 97-105 (2017).
- Hodgkinson, T., et al. The use of nanovibration to discover specific and potent bioactive metabolites that stimulate osteogenic differentiation in mesenchymal stem cells. Science Advances. 7 (9), 7921 (2021).
- Kloxin, A. M., Kloxin, C. J., Bowman, C. N., Anseth, K. S. Mechanical properties of cellularly responsive hydrogels and their experimental determination. Advanced Materials (Deerfield Beach, Fla). 22 (31), 3484-3494 (2010).
- Loessner, D., et al. Functionalization, preparation and use of cell-laden gelatin methacryloyl-based hydrogels as modular tissue culture platforms. Nature Protocols. 11 (4), 727-746 (2016).
- Yue, K., et al. Synthesis, properties, and biomedical applications of gelatin methacryloyl (GelMA) hydrogels. Biomaterials. 73, 254-271 (2015).
- Sarrigiannidis, S. O., et al. A tough act to follow: collagen hydrogel modifications to improve mechanical and growth factor loading capabilities. Materials Today. Bio. 10, 100098 (2021).
- Karoutas, A., et al. The NSL complex maintains nuclear architecture stability via lamin A/C acetylation. Nature Cell Biology. 21 (10), 1248-1260 (2019).
- Ryu, H., et al. Transparent, compliant 3D mesostructures for precise evaluation of mechanical characteristics of organoids. Advanced Materials (Deerfield Beach, Fla). 33 (25), 2100026 (2021).
- Carvalho, D. T. O., Feijão, T., Neves, M. I., Da Silva, R. M. P., Barrias, C. C. Directed self-assembly of spheroids into modular vascular beds for engineering large tissue constructs Biofabrication Directed self-assembly of spheroids into modular vascular beds for engineering large tissue constructs. Biofabrication. 13 (3), 035008 (2021).
- Chaudhuri, O., Cooper-White, J., Janmey, P. A., Mooney, D. J., Shenoy, V. B. Effects of extracellular matrix viscoelasticity on cellular behaviour. Nature. 584 (7822), 535-546 (2020).
- Cantini, M., Donnelly, H., Dalby, M. J., Salmeron-Sanchez, M. The plot thickens: The emerging role of matrix viscosity in cell mechanotransduction. Advanced Healthcare Materials. 9 (8), 1901259 (2020).
- Elosegui-Artola, A. The extracellular matrix viscoelasticity as a regulator of cell and tissue dynamics. Current Opinion in Cell Biology. 72, 10-18 (2021).
- Chaudhuri, O. Viscoelastic hydrogels for 3D cell culture. Biomaterials Science. 5 (8), 1480-1490 (2017).
- Nguyen, T. D., Gu, Y. Determination of strain-rate-dependent mechanical behavior of living and fixed osteocytes and chondrocytes using atomic force microscopy and inverse finite element analysis. Journal of Biomechanical Engineering. 136 (10), 101004 (2014).
- Mokbel, M., Hosseini, K., Aland, S., Fischer-Friedrich, E. The Poisson Ratio of the Cellular Actin Cortex Is Frequency Dependent. Biophysical Journal. 118 (8), 1968-1976 (2020).
- Javanmardi, Y., Colin-York, H., Szita, N., Fritzsche, M., Moeendarbary, E. Quantifying cell-generated forces: Poisson's ratio matters. Communications Physics. 4, 237 (2021).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。