Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Experimenteller und Datenanalyse-Workflow für die Nanoindentation weicher Materie
In diesem Artikel
Zusammenfassung
Das Protokoll stellt einen vollständigen Workflow für Experimente zur Nanoindentation weicher Materialien dar, einschließlich Hydrogele und Zellen. Zunächst werden die experimentellen Schritte zur Erfassung von Kraftspektroskopiedaten detailliert beschrieben. Dann wird die Analyse solcher Daten durch eine neu entwickelte Open-Source-Python-Software detailliert, die kostenlos von GitHub heruntergeladen werden kann.
Zusammenfassung
Nanoindentation bezieht sich auf eine Klasse von experimentellen Techniken, bei denen eine mikrometrische Kraftsonde verwendet wird, um die lokalen mechanischen Eigenschaften von weichen Biomaterialien und Zellen zu quantifizieren. Dieser Ansatz hat eine zentrale Rolle in den Bereichen Mechanobiologie, Biomaterialdesign und Tissue Engineering eingenommen, um eine korrekte mechanische Charakterisierung weicher Materialien mit einer Auflösung zu erhalten, die mit der Größe einzelner Zellen (μm) vergleichbar ist. Die beliebteste Strategie, um solche experimentellen Daten zu gewinnen, ist die Verwendung eines Rasterkraftmikroskops (AFM); Während dieses Instrument eine beispiellose Auflösung in Kraft (bis hin zu pN) und Raum (Sub-nm) bietet, ist seine Verwendbarkeit oft durch seine Komplexität eingeschränkt, die Routinemessungen von integralen Indikatoren mechanischer Eigenschaften wie dem Elastizitätsmodul (E) verhindert. Eine neue Generation von Nanoindentern, wie sie auf der optischen Fasersensortechnologie basieren, hat in letzter Zeit an Popularität gewonnen, da sie sich leicht integrieren lässt und gleichzeitig sub-nN-Kräfte mit μm-Ortsauflösung aufbringen kann, wodurch sie zur Untersuchung lokaler mechanischer Eigenschaften von Hydrogelen und Zellen geeignet sind.
In diesem Protokoll wird eine Schritt-für-Schritt-Anleitung vorgestellt, die das experimentelle Verfahren zur Erfassung von Nanoindentationsdaten auf Hydrogelen und Zellen unter Verwendung eines kommerziell erhältlichen Ferrule-Top-Nanofaser-Sensor-Nanoindenters beschreibt. Während einige Schritte spezifisch für das hier verwendete Instrument sind, kann das vorgeschlagene Protokoll als Leitfaden für andere Nanoindentationsvorrichtungen verwendet werden, vorausgesetzt, einige Schritte werden gemäß den Richtlinien des Herstellers angepasst. Darüber hinaus wird eine neue Open-Source-Python-Software vorgestellt, die mit einer benutzerfreundlichen grafischen Benutzeroberfläche für die Analyse von Nanoindentationsdaten ausgestattet ist, die das Screening von falsch erfassten Kurven, die Datenfilterung, die Berechnung des Kontaktpunkts durch verschiedene numerische Verfahren, die konventionelle Berechnung von E sowie eine erweiterte Analyse ermöglicht, die sich besonders für einzellige Nanoindentationsdaten eignet.
Einleitung
Die grundlegende Rolle der Mechanik in der Biologie ist heute etabliert 1,2. Von ganzen Geweben bis hin zu einzelnen Zellen können mechanische Eigenschaften über den pathophysiologischen Zustand des untersuchten Biomaterials 3,4 informieren. Zum Beispiel ist Brustgewebe, das von Krebs betroffen ist, steifer als gesundes Gewebe, ein Konzept, das die Grundlage des beliebten Palpationstests5 ist. Insbesondere wurde kürzlich gezeigt, dass die Coronavirus-Krankheit 2019 (COVID-19), die durch das schwere akute respiratorische Syndrom Coronavirus 2 (SARS-CoV-2) verursacht wird, durch Veränderungen der mechanischen Eigenschaften von Blutzellen unterstrichen wird, einschließlich einer verminderten Verformbarkeit der Erythrozyten und einer verminderten Lymphozyten- und Neutrophilensteifigkeit im Vergleich zu Blutzellen von SARS-CoV-2-naiven Personen6.
Im Allgemeinen ist die Mechanik von Zellen und Geweben inhärent miteinander verflochten: Jedes Gewebe hat spezifische mechanische Eigenschaften, die gleichzeitig die Eigenschaften der konstituierenden Zellen und der extrazellulären Matrix (ECM) beeinflussen und von ihnen abhängen5. Aus diesem Grund beinhalten Strategien zur Untersuchung der Mechanik in der Biologie oft technische Substrate mit physiologisch relevanten mechanischen Reizen, um das Zellverhalten als Reaktion auf diese Reize aufzuklären. Zum Beispiel zeigten die bahnbrechenden Arbeiten von Engler und Kollegen, dass die Verpflichtung der mesenchymalen Stammzelllinie durch die Matrixelastizität gesteuert wird, wie an weichen und steifen zweidimensionalen Polyacrylamid (PAAm) -Hydrogelen untersucht7.
Es gibt viele Strategien zur mechanischen Charakterisierung des untersuchten Biomaterials, die in der räumlichen Skala (d.h. lokal bis zur Masse) und in der Art der Verformung (z. B. axial vs. Scherung) variieren und folglich unterschiedliche Informationen liefern, die einer sorgfältigen Interpretation bedürfen 3,8,9,10. Die Mechanik weicher Biomaterialien wird üblicherweise in Form von Steifigkeit ausgedrückt. Die Steifigkeit hängt jedoch sowohl von den Materialeigenschaften als auch von der Geometrie ab, während elastische Module grundlegende Eigenschaften eines Materials sind und unabhängig von der Geometrie des Materialssind 11. Daher beziehen sich verschiedene Elastizitätsmodule auf die Steifigkeit einer bestimmten Probe, und jeder Elastizitätsmodul umfasst den Widerstand des Materials gegen eine bestimmte Verformungsart (z. B. axial vs. Scherung) unter verschiedenen Randbedingungen (z. B. freie Expansion vs. Einschluss)11,12. Nanoindentationsexperimente ermöglichen die Quantifizierung mechanischer Eigenschaften durch das E, das mit einer einachsigen Verformung (Indentation) verbunden ist, wenn das Biomaterial nicht seitlich begrenzt ist10,11,12.
Die beliebteste Methode zur Quantifizierung von E von biologischen Systemen auf der Mikroskala ist AFM13,14,15,16. AFM ist ein extrem leistungsfähiges Werkzeug mit Kraftauflösung bis auf pN-Ebene und räumlicher Auflösung bis in die Sub-nm-Skala. Darüber hinaus bietet AFM extreme Flexibilität in Bezug auf die Kopplung mit komplementären optischen und mechanischen Werkzeugen und erweitert seine Fähigkeiten, eine Fülle von Informationen aus dem untersuchten Biomaterial zu extrahieren13. Diese attraktiven Merkmale sind jedoch mit einer Eintrittsbarriere verbunden, die durch die Komplexität des Versuchsaufbaus dargestellt wird. AFM erfordert umfangreiche Schulungen, bevor Benutzer robuste Daten erfassen können, und seine Verwendung für die tägliche mechanische Charakterisierung biologischer Materialien ist oft ungerechtfertigt, insbesondere wenn seine einzigartige Kraft und räumliche Auflösung nicht erforderlich sind.
Aus diesem Grund hat eine neue Klasse von Nanoindentern in letzter Zeit aufgrund ihrer Benutzerfreundlichkeit an Popularität gewonnen, während sie immer noch AFM-vergleichbare Daten mit Sub-nN-Kraftauflösung und μm räumlicher Auflösung bieten, die Kräfte widerspiegeln, die von Zellen über relevante Längenskalen ausgeübt und wahrgenommenwerden 2. Insbesondere Ferrule-Top-Nanoindentationsgeräte, die auf der optischen Fasersensortechnologie17,18 basieren, haben bei Forschern, die auf dem Gebiet der Mechanobiologie und darüber hinaus tätig sind, an Popularität gewonnen; Und eine Fülle von Arbeiten, die über die mechanischen Eigenschaften von Biomaterialien unter Verwendung dieser Geräte berichten, einschließlich Zellen19,20, Hydrogele8,21 und Gewebe22,23 wurden veröffentlicht. Trotz der Fähigkeit dieser Systeme, lokale dynamisch-mechanische Eigenschaften (d.h. Speicher- und Verlustmodul) zu untersuchen, bleiben quasistatische Experimente mit E die beliebteste Wahl 8,19,20,21. Kurz gesagt, quasistatische Nanoindentationsexperimente bestehen darin, die Probe mit einer konstanten Geschwindigkeit bis zu einem Sollwert einzudrücken, der entweder durch eine maximale Verschiebung, Kraft oder Eindringtiefe definiert ist, und sowohl die Kraft als auch die vertikale Position des Cantilever in sogenannten Kraft-Weg-Kurven (F-z) aufzuzeichnen. F-z-Kurven werden dann durch die Identifizierung des Kontaktpunkts (CP) in Krafteindringkurven (F-δ) umgewandelt und mit einem geeigneten Kontaktmechanikmodell (normalerweise dem Hertz-Modell13) ausgestattet, um E zu berechnen.
Während der Betrieb von Ferrule-Top-Nanoindentern AFM-Messungen ähnelt, gibt es Besonderheiten, die es wert sind, berücksichtigt zu werden. In dieser Arbeit wird eine Schritt-für-Schritt-Anleitung zur robusten Erfassung von F-z-Kurven aus Zellen und gewebenachahmenden Hydrogelen unter Verwendung eines kommerziell erhältlichen Ferrule-Top-Nanoindenters bereitgestellt, um die Standardisierung experimenteller Verfahren zwischen Forschungsgruppen zu fördern, die dieses und andere ähnliche Geräte verwenden. Darüber hinaus werden Ratschläge gegeben, wie Hydrogelproben und -zellen am besten für die Durchführung von Nanoindentationsexperimenten vorbereitet werden können, zusammen mit Tipps zur Fehlerbehebung entlang des experimentellen Weges.
Darüber hinaus hängt ein Großteil der Variabilität der Nanoindentationsergebnisse (d. H. E und seine Verteilung) von dem spezifischen Verfahren ab, das zur Analyse von Daten verwendet wird, was nicht trivial ist. Um dieses Problem zu lösen, werden Anleitungen zur Verwendung einer neu entwickelten, in Python programmierten Open-Source-Software bereitgestellt, die mit einer benutzerfreundlichen grafischen Benutzeroberfläche (GUI) für die Batch-Analyse von F-z-Kurven ausgestattet ist. Die Software ermöglicht ein schnelles Datenscreening, das Filtern von Daten, die Berechnung des CP durch verschiedene numerische Verfahren, die konventionelle Berechnung von E sowie eine erweiterte Analyse namens Elastizitätsspektren24, die es ermöglicht, den Elastizitätsmodul der Zelle, den Elastizitätsmodul des Aktincortex und die Dicke des Aktinkortex zu schätzen. Die Software kann kostenlos von GitHub heruntergeladen werden und kann leicht angepasst werden, um Daten aus anderen Systemen zu analysieren, indem ein geeigneter Datenparser hinzugefügt wird. Es wird betont, dass dieses Protokoll für andere Ferrule-Top-Nanoindentationsvorrichtungen und andere Nanoindentationsgeräte im Allgemeinen verwendet werden kann, vorausgesetzt, einige Schritte werden gemäß den Richtlinien des spezifischen Instruments angepasst. Das Protokoll ist in Abbildung 1 schematisch zusammengefasst.
Protokoll
1. Vorbereitung von Substraten/Zellen für Nanoindentationsmessungen
- Befolgen Sie die im Zusatzprotokoll angegebenen Schritte zur Vorbereitung von PAAm-Hydrogelen/-Zellen für Nanoindentationsexperimente. Das Verfahren ist in Abbildung 2 zusammengefasst.
HINWEIS: PAAm-Hydrogele wurden ausgewählt, da sie die am häufigsten verwendeten Hydrogele im Bereich der Mechanobiologie sind. Das Protokoll ist jedoch gleichermaßen auf jede Art von Hydrogel25 anwendbar (siehe Diskussion, Änderungen des Verfahrens).
2. Inbetriebnahme des Geräts, Sondenauswahl und Sondenkalibrierung
- Befolgen Sie die im Zusatzprotokoll angegebenen Schritte zum Starten des Geräts. Technische Details zum Betrieb von optischen Faser-Ferrule-Top-Nanoindentern finden Sie in diesen Referenzen17,18.
- Wählen Sie die Nanoeindringsonde wie unten beschrieben aus.
HINWEIS: Alle handelsüblichen Sonden, wie sie in diesem Protokoll verwendet werden, sind mit einer kugelförmigen Spitze ausgestattet (Abbildung 3A). Daher beschränkt sich die Auswahl auf zwei Variablen: die Steifigkeit des Auslegers und den Spitzenradius (Abbildung 3B).- Wählen Sie die Steifigkeit eines Auslegers (k in N/m), die der erwarteten Probensteifigkeit entspricht, um beste Ergebnissezu erzielen 15 (siehe Abbildung 3C und Diskussion, kritische Schritte im Protokoll). Wählen Sie für Zellen eine Sonde mit k im Bereich 0,01-0,09 N/m. Wählen Sie für Hydrogele eine Sonde mit k im Bereich von 0,1-0,9 N/m, die optimale Ergebnisse für Gele mit erwartetem E zwischen einigen kPa und 100 kPa liefert (siehe Repräsentative Ergebnisse).
- Wählen Sie den Spitzenradius (R in μm) entsprechend der gewünschten räumlichen Auflösung des Eindringvorgangs. Für kleine Zellen wie Human Embryonic Kidney 293T (HEK293T) Zellen (durchschnittlicher Durchmesser von ~10-15 μm26) wählen Sie eine Kugel mit R = 3 μm. Wählen Sie für Hydrogele eine Kugel mit R = 10-250 μm, um die mechanischen Eigenschaften des Biomaterials über eine große Kontaktfläche zu untersuchen und lokale Heterogenitäten zu vermeiden.
- Konsultieren Sie Abbildung 3C und alle zusätzlichen Herstellerrichtlinien, um die geeignete Sonde auszuwählen.
- Sobald die Sonde ausgewählt wurde, befolgen Sie die Schritte im Zusatzprotokoll , um sie auf dem Nanoindenter zu montieren.
3. Sondenkalibrierung
HINWEIS: Die folgenden Schritte gelten speziell für Ferrule-Top-Nanoindentationsgeräte, die auf der optischen Fasersensortechnologie basieren, und sind für die Softwareversion 3.4.1 detailliert. Befolgen Sie bei anderen Nanoindentationsgeräten die vom Gerätehersteller empfohlenen Schritte.
- Klicken Sie im Hauptfenster der Software auf Initialisieren. Ein Kalibrierungsmenü wird angezeigt. Geben Sie die Sondendetails ein (zu finden auf der Seite der Sondenbox; k in N/m, R in μm und der Kalibrierfaktor in Luft) in den Eingabefeldern.
- Bereiten Sie die Kalibrierschale vor: eine dicke Glas-Petrischale mit flachem Boden (siehe Diskussion, kritische Schritte im Protokoll). Füllen Sie die Schale mit dem gleichen Medium wie die Probenschale (dies kann auch Luft sein). Passen Sie die Temperatur des Mediums an die der Probe an.
- Legen Sie die Kalibrierschale unter die Sonde. Ziehen Sie die Sonde bei Bedarf aus dem Armhalter des Nanoindringers heraus und halten Sie sie in einer Hand, um Platz für die Platzierung der Kalibrierschale zu schaffen. Schieben Sie die Sonde wieder in Position.
- Führen Sie die nächsten beiden Schritte zur Kalibrierung in Flüssigkeit durch. Wenn Sie in Luft messen, verwenden Sie das mitgelieferte Polytetrafluorethylensubstrat für die Kalibrierung und fahren Sie mit Schritt 5 fort.
- Befeuchten Sie die Sonde mit einem Tropfen 70% Ethanol unter Verwendung einer Pasteur-Pipette mit dem Ende der Pipette in leichtem Kontakt mit der Glasferrule, damit der Tropfen über den Ausleger und die kugelförmige Spitze27 gleitet (siehe Diskussion, kritische Schritte im Protokoll).
- Schieben Sie den Arm des Nanoindringers manuell nach unten, bis die Sonde vollständig eingetaucht ist, aber immer noch weit weg vom Boden der Petrischale. Geben Sie bei Bedarf mehr Medium in die Kalibrierschale. Warten Sie 5 Minuten, bis Gleichgewichtsbedingungen in der Flüssigkeit erreicht sind.
- Klicken Sie im Menü Initialisieren der Software auf Scan-Wellenlänge. Der Bildschirm des Interferometers zeigt einen Fortschrittsbalken und das Live-Signal-Fenster der Computersoftware zeigt das in Abbildung S1A links gezeigte Muster an. Um zu überprüfen, ob der optische Scan erfolgreich war, navigieren Sie zum Bereich Wellenlängen-Scan in der Interferometerbox. Bei Erfolg sollte eine Sinuswelle sichtbar sein (Abbildung 1A, rechts). Siehe Diskussion (Fehlerbehebung der Methode), wenn ein Fehler angezeigt wird.
- Klicken Sie im Menü Initialisieren auf Oberfläche suchen, wodurch die Sonde schrittweise abgesenkt wird, bis ein eingestellter Schwellenwert in der Biegung des Auslegers erreicht ist. Die Sonde hört auf, sich zu bewegen, wenn Kontakt mit der Glas-Petrischale hergestellt wird.
- Prüfen Sie, ob die Sonde mit der Oberfläche in Berührung kommt. Bewegen Sie die Sonde um 1 μm nach unten, indem Sie die Pfeiltaste y nach unten im Hauptfenster der Software verwenden. Beobachten Sie das grüne Signal (Auslenkung des Auslegers) im Live-Fenster der Software für Änderungen der Basislinie bei jedem Schritt, wenn der Ausleger mit dem Substrat in Kontakt kommt (Abbildung S1B). Wenn es keine Änderung gibt, ist der Ausleger nicht in Kontakt (siehe nächster Schritt).
- Erhöhen Sie den Schwellenwert im Menü Optionen auf der Registerkarte Oberfläche suchen von seinem Standardwert 0,01 um jeweils 0,01, und wiederholen Sie den Schritt Oberfläche suchen , bis er in Kontakt kommt. Alternativ können Sie die Sonde in kleinen Schritten von 1 μm zum Kontakt bringen, bis sich die grüne Grundlinie bei jedem Schritt nach unten zu verschieben beginnt.
Hinweis: Erhöhen Sie für die weichsten Ausleger (k = 0,025 Nm-1) den Schwellenwert im Menü Optionen auf der Registerkarte Oberfläche suchen a priori nach dem Ausführen von Schritt 6. Beginnen Sie mit einem Schwellenwert von 0,06 oder 0,07 und erhöhen Sie ihn bei Bedarf auf 0,1. Dies liegt daran, dass Umgebungslärm wahrscheinlich dazu führt, dass sich der Ausleger vor dem Kontakt über den Schwellenwert beugt. Bei weichen Sonden verbessert die Verringerung der Annäherungsgeschwindigkeit (μm/s) im selben Menü ebenfalls das Verfahren. - Klicken Sie im Menü Initialisieren auf Kalibrieren.
- Überprüfen Sie das Live-Signal-Fenster der Software und stellen Sie sicher, dass sich sowohl die Verschiebung des Piezos als auch die Auslenkungssignale des Auslegers gleichzeitig nach oben bewegen (Abbildung S1C).
- Wenn die Zeit nicht übereinstimmt, ist die Sonde nicht vollständig mit dem Glas in Kontakt. Bewegen Sie die Sonde in Schritten von 1 μm nach unten, bis sich die Grundlinie des Cantilever-Signals ändert (siehe Schritt 7), und wiederholen Sie Schritt 9.
- Wenn sich das Cantilever-Signal während des Kalibrierungsschritts überhaupt nicht ändert, ist die Sonde weit von der Oberfläche entfernt. Erhöhen Sie die Kontaktschwelle iterativ (siehe Schritt 8), bis die Oberfläche korrekt gefunden wird, und wiederholen Sie die Kalibrierung von Anfang an, beginnend mit dem Wellenlängenscan.
- Wenn die Kalibrierung abgeschlossen ist, überprüfen Sie die alten und neuen Kalibrierungsfaktoren im angezeigten Popup-Fenster. Wenn der neue Kalibrierfaktor im richtigen Bereich liegt, klicken Sie auf Neuen Faktor verwenden. Wenn die Kalibrierung fehlschlägt und der neue Faktor entweder NaN ist oder nicht im erwarteten Bereich liegt, finden Sie eine Lösung unter Diskussion (Fehlerbehebung der Methode).
HINWEIS: Der neue Faktor sollte ~n mal niedriger sein als der auf der Sondenverpackung angegebene Faktor, wenn die Kalibrierung in einem flüssigen Medium mit Brechungsindex n ( n = 1,33 für Wasser) durchgeführt wurde. Wenn die Kalibrierung in Luft durchgeführt wurde, sollten die neuen und alten Kalibrierfaktoren ungefähr gleich sein. - Überprüfen Sie, ob der Demodulationskreis wie folgt korrekt kalibriert wurde. Navigieren Sie zur Registerkarte Demodulation auf dem Interferometer-Desktop. Klopfen Sie vorsichtig auf den optischen Tisch oder auf den Nanoindenter, um genügend Rauschen zu erzeugen. Ein weißer Kreis, der aus diskreten Datenpunkten besteht, sollte den roten Kreis ungefähr abdecken (Abbildung S1D).
- Wenn sich der weiße Kreis nicht mit dem roten Kreis überschneidet oder eine Warnung auf dem Display des Interferometers angezeigt wird, muss der Demodulationskreis neu kalibriert werden. Dies kann auf zwei Arten erfolgen, wie unten beschrieben.
- Tippen Sie kontinuierlich auf den Körper des Nanoindenters, um einen vollständigen Rauschkreis zu induzieren, und drücken Sie die Taste Calibrate (Kalibrieren ) am Interferometer.
- Geben Sie Kontakt mit dem Glassubstrat ein und drücken Sie Kalibrieren aus dem Menü Initialisieren des Hauptfensters der Software. Speichern Sie den Kalibrierungsfaktor nicht. Überprüfen Sie an dieser Stelle erneut, und stellen Sie sicher, dass sich der weiße Kreis mit dem roten Kreis überlappt.
HINWEIS: Wenn das Signal nicht nur leicht vom Demodulationskreis verschoben wird, sondern sehr klein geworden ist oder überhaupt nicht sichtbar ist, bedeutet dies, dass der Cantilever an der optischen Faser haftet. Befolgen Sie die Hinweise zur Fehlerbehebung für dieses Problem (siehe Diskussion, Fehlerbehebung der Methode), und wiederholen Sie entweder die Schritte 13.1 oder 13.2. Sobald der Ausleger wieder in seine horizontale Position zurückkehrt (Abbildung 3A), kehrt das Signal zum Demodulationskreis zurück.
- Überprüfen Sie die Kalibrierung, indem Sie direkt nach der Kalibrierung wie unten beschrieben einen Einzug auf dem Glassubstrat durchführen.
- Laden oder erstellen Sie eine Testdatei, indem Sie auf Experiment konfigurieren klicken und einen Schritt "Oberfläche suchen " und einen Schritt " Einzug " hinzufügen. Verwenden Sie für den Eindringschritt die Standardeinstellungen für den Verdrängungsmodus und ändern Sie die maximale Verschiebung auf den Kalibrierabstand (3.000 nm), um die Sonde gegen das steife Substrat zu verschieben.
- Klicken Sie auf Experiment ausführen und überprüfen Sie den Demodulationskreis im Interferometerfenster. Überprüfen Sie das weiße Signal und stellen Sie sicher, dass es sich während des Einrückens über dem roten Kreis befindet.
- Überprüfen Sie die Ergebnisse im Hauptfenster der Software im Zeitdatendiagramm und stellen Sie sicher, dass die Verschiebung des Piezos (blaue Linie) gleich der Auslenkung des Auslegers (grüne Linie) ist, da die Vertiefung in Kontakt beginnt und keine Materialverformung erwartet wird. Wenn die Signale nicht parallel sind, finden Sie weitere Informationen unter Diskussion (Fehlerbehebung bei der Methode).
- Ändern Sie den lokalen Pfad im Kalibrierungsmenü, indem Sie den Kalibrierungsspeicherpfad auf ein entsprechendes Verzeichnis setzen.
- Wenn die Sonde erfolgreich kalibriert wurde, bewegen Sie den Piezo um 500 μm nach oben.
4. Messung des Elastizitätsmoduls weicher Materialien
- Nanoindentation von Hydrogelen
- Laden Sie die Petrischale mit der Probe(n) auf den Mikroskoptisch und bewegen Sie die Sonde des Nanoindenters manuell in eine gewünschte x-y-Position über der Probe.
- Schieben Sie die Sonde manuell in Lösung, wobei Sie darauf achten, dass in diesem Stadium 1-2 mm zwischen der Sonde und der Probenoberfläche verbleiben. Warten Sie 5 Minuten, bis die Sonde im Medium ausgeglichen ist.
- Fokussieren Sie die z-Ebene des Lichtmikroskops so, dass die Sonde deutlich sichtbar ist.
- Führen Sie einen einzelnen Einzug durch, um die experimentellen Parameter wie unten beschrieben abzustimmen.
- Konfigurieren Sie ein neues Experiment im Hauptfenster der Software. Klicken Sie auf Experiment konfigurieren, um ein neues Fenster zu öffnen. Fügen Sie einen Schritt "Oberfläche suchen " hinzu. Alle Parameter des Schritts Oberfläche suchen können bei Bedarf im Menü Optionen der Software geändert werden.
HINWEIS: Die Suchfläche senkt die Sonde ab, bis die Oberfläche gefunden wird, und zieht die Sonde dann auf einen Abstand zurück, der durch Z über der Oberfläche (μm) über der Probenoberfläche definiert ist. Wenn der ausgewählte Cantilever zu steif für die Probe ist oder die Probe klebrig ist, ist es wahrscheinlich, dass die Sonde nach dem Schritt immer noch in Kontakt mit der Probe ist, was zu einer Kurve ohne Basislinie führt (Abbildung 4C). Um dieses Problem zu lösen, erhöhen Sie das Z über der Oberfläche (μm). - Fügen Sie einen Einzugsschritt hinzu. Wählen Sie die Registerkarte Profil und klicken Sie auf Verschiebungssteuerung. Behalten Sie das Standardeinzugsprofil bei.
- Klicken Sie im Hauptfenster der Software auf Experiment ausführen . Dadurch wird die Oberfläche gefunden und ein einzelner Einzug ausgeführt. Wenn der einzelne Einzug nicht wie erwartet aussieht, passen Sie die experimentellen Parameter wie in Abbildung 4 und Diskussion (Fehlerbehebung der Methode) beschrieben an.
- Konfigurieren Sie ein neues Experiment im Hauptfenster der Software. Klicken Sie auf Experiment konfigurieren, um ein neues Fenster zu öffnen. Fügen Sie einen Schritt "Oberfläche suchen " hinzu. Alle Parameter des Schritts Oberfläche suchen können bei Bedarf im Menü Optionen der Software geändert werden.
- Sobald der Einzug wie gewünscht aussieht, konfigurieren Sie den Matrixscan so, dass ein ausreichender Bereich der Probe eingerückt wird. Klicken Sie auf Experiment konfigurieren, fügen Sie einen Schritt Oberfläche suchen mit den zuvor ermittelten experimentellen Parametern hinzu und fügen Sie einen Matrix Scan-Schritt hinzu.
- Konfigurieren Sie für flache Hydrogele einen Matrixscan mit 50-100 Punkten (d. h. 5 x 10 oder 10 x 10 in x und y) im Abstand von 10-100 μm (d. h . dx = dy = 10-100 μm). Klicken Sie auf Bühnenposition verwenden , um den Matrixscan von der aktuellen Bühnenposition aus zu starten. Stellen Sie sicher, dass das Kontrollkästchen Oberfläche automatisch suchen aktiviert ist, um die Oberfläche bei jedem Einzug mithilfe der festgelegten experimentellen Parameter zu finden.
- Um eine Überabtastung zu vermeiden, stellen Sie die Schrittweite auf mindestens den doppelten Kontaktradius ein (
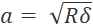 , wobei δ die Einzugstiefe ist).
, wobei δ die Einzugstiefe ist). - Richten Sie das Matrix-Scan-Profil in der Verschiebungssteuerung ein. Stellen Sie sicher, dass das Profil nicht gegen die Annahmen des Hertz-Modells verstößt (siehe Diskussion, kritische Schritte im Protokoll).
- Behalten Sie die Anzahl der Segmente auf 5 (Standardwert) bei, und verwenden Sie das Standardverschiebungsprofil. Ändern Sie ggf. das Verdrängungsprofil in Bezug auf maximale Verschiebung und Zeit für jedes geneigte Segment, was sich auf die maximale Eindringtiefe bzw. die Dehnungsrate auswirkt. Dehnungsraten > 10 μm/s nicht überschreiten (siehe Diskussion, Einschränkungen der Methode).
- Geben Sie einen Wert für die Annäherungsgeschwindigkeit ein, der bestimmt, wie schnell die Sonde vor dem Kontakt in Richtung der Probe verschoben wird. Passen Sie die Rückzugsgeschwindigkeit an die Anfahrgeschwindigkeit an (siehe Hinweis unten).
HINWEIS: Für weiche Ausleger und laute Umgebungen wird eine Annäherungsgeschwindigkeit von 1.000-2.000 nm/s empfohlen. Für steifere Ausleger und kontrollierte Umgebungen kann dieser erhöht werden. - Speichern Sie das konfigurierte Experiment im gewünschten Experimentpfad, und wählen Sie im Fenster Test konfigurieren auf der Registerkarte Speicherpfad auf der Registerkarte Allgemein ein Verzeichnis aus, in dem die Daten gespeichert werden sollen. Klicken Sie auf Test ausführen.
- Um eine Überabtastung zu vermeiden, stellen Sie die Schrittweite auf mindestens den doppelten Kontaktradius ein (
- Sobald der Matrixscan abgeschlossen ist, heben Sie die Sonde um 200-500 μm an und bewegen Sie die Sonde in einen anderen Bereich der Probe, der ausreichend weit vom ersten Bereich entfernt ist.
- Wiederholen Sie das Experiment mindestens zweimal, damit genügend Daten zu jeder Probe erfasst werden (d. h. mindestens zwei Matrixscans pro Probe mit jeweils 50-100 Kurven).
- Nanoindentation von Zellen
- Laden Sie die Probe wie oben beschrieben auf das Mikroskop.
- Für den Einzelzelleinzug fokussieren Sie die z-Ebene so, dass sowohl die Zellen als auch die Sonde je nach Zellgröße und -verteilung bei 20- oder 40-facher Vergrößerung sichtbar sind.
- Bewegen Sie die Sonde über die einzurückende Zelle.
- Konfigurieren Sie ein neues Experiment im Hauptfenster der Software. Klicken Sie auf Experiment konfigurieren, um ein neues Fenster zu öffnen. Fügen Sie einen Schritt "Fläche suchen " und " Einzug " mit Standardparametern im Verschiebungsmodus hinzu.
- Klicken Sie auf Run Experiment, um die Oberfläche zu finden und einen einzelnen Einzug durchzuführen. Überprüfen Sie, ob der Einzug erfolgreich war. Wenn die Kurve nicht wie erwartet aussieht, passen Sie die experimentellen Parameter an (siehe Abbildung 4 und Diskussion, Fehlerbehebung der Methode).
- Wenn der Einzug erfolgreich war, fügen Sie dem Experiment einen Matrixscan hinzu. Befolgen Sie die Schritte für die Nanoindentationsexperimente von Hydrogelen. Konfigurieren Sie den Matrix-Scan so, dass die Schrittweite es ermöglicht, einen kleinen Bereich der Zelle einzurücken. 25 Punkte im Abstand von 0,5-5 μm für HEK293T-Zellen.
- Passen Sie je nach Zellengröße den Matrixscan an, um sicherzustellen, dass die Spitze nicht außerhalb der Zellengrenze einrückt, d. H. Führen Sie eine andere Kartengeometrie durch oder tasten Sie weniger Punkte ab.
- Klicken Sie auf Experiment ausführen und warten Sie, bis es abgeschlossen ist.
- Sobald der Matrix-Scan abgeschlossen ist, heben Sie die Sonde außer Kontakt (50 μm in der z-Ebene ).
- Bewegen Sie die Sonde über eine neue Zelle, und wiederholen Sie den Vorgang (siehe Diskussion, kritische Schritte im Protokoll).
- Sonde reinigen und Gerät ausschalten
- Befolgen Sie die im Zusatzprotokoll angegebenen Schritte, um die Sonde zu reinigen und die Nanoindentationsvorrichtung auszuschalten.
5. Datenanalyse
- Herunterladen und Installieren der Software
- Folgen Sie den Schritten im Zusatzprotokoll, um die Software für die Datenanalyse28,29 herunterzuladen und zu installieren.
- Screening von F-z-Kurven und Erstellung von bereinigten Datensätzen im JSON-Format
- Starten Sie prepare.py über die Befehlszeile auf dem Laborcomputer, wie in den Schritten 2 bis 3 beschrieben.
- Wenn Sie einen Windows-Computer verwenden, halten Sie die Umschalttaste gedrückt, während Sie mit der rechten Maustaste auf den Ordner NanoPrepare klicken, und klicken Sie auf PowerShell-Fenster hier öffnen. Geben Sie den Befehl python prepare.py ein und drücken Sie die Eingabetaste . Eine grafische Benutzeroberfläche wird auf Ihrem Bildschirm angezeigt (Abbildung S2).
- Wenn Sie einen MacOS-Computer verwenden, klicken Sie mit der rechten Maustaste auf den Ordner NanoPrepare und klicken Sie auf Neues Terminal unter Ordner. Geben Sie den Befehl python3 prepare.py ein und drücken Sie die Eingabetaste , um die GUI zu starten (Abbildung S2).
- Wählen Sie das Datenformat O11NEW aus der Dropdown-Liste aus. Wenn die Daten nicht korrekt geladen werden, starten Sie die GUI neu und wählen Sie O11OLD.
HINWEIS: Das O11NEW-Format funktioniert für Daten, die mit Ferrule-Top-Nanoindentationsgeräten mit Glasfasererkennung und Softwareversion 3.4.1 erhalten wurden. Dieses Format funktioniert auch für frühere Softwareversionen, zumindest für Nanoindenter, die in den Jahren 2019-2020 installiert wurden. - Klicken Sie auf Ordner laden. Wählen Sie einen Ordner aus, der die zu analysierenden Daten enthält - einzelner Matrix-Scan oder mehrere Matrix-Scans. Das obere Diagramm (Rohkurven) wird mit dem hochgeladenen Datensatz gefüllt. Um eine bestimmte Kurve zu visualisieren, klicken Sie darauf. Dadurch wird es grün hervorgehoben und in der unteren Grafik (Current Curve) angezeigt.
- Bereinigen Sie den Datensatz mithilfe der Registerkarten im rechten Teil der GUI, wie unten beschrieben.
- Verwenden Sie die Schaltfläche Segment, um das richtige zu analysierende Segment auszuwählen, bei dem es sich um das Vorwärtssegment der F-z-Kurven handelt. Die genaue Anzahl hängt von der Anzahl der Segmente ab, die in der Nanoindentationssoftware bei der Durchführung von Experimenten ausgewählt werden.
- Verwenden Sie die Schaltfläche 50 nm zuschneiden, um die Kurven ganz links (wenn L angekreuzt ist), rechts (wenn R angekreuzt ist) oder beidseitig (wenn sowohl R als auch L angekreuzt sind) um 50 nm zuzuschneiden. Klicken Sie mehrmals auf diese Schaltfläche, um so viel wie nötig zuzuschneiden. Verwenden Sie diese Option, um Artefakte am Anfang/Ende von F-z-Kurven zu entfernen.
- Überprüfen Sie die Auslegerlasche auf Federkonstante, Spitzengeometrie und Spitzenradius. Überprüfen Sie die Registerkarte, um sicherzustellen, dass die Metadaten korrekt gelesen wurden.
- Verwenden Sie die Registerkarte Raster, um einen Kraftschwellenwert festzulegen, der alle Kurven verwirft, die die angegebene Kraft nicht erreicht haben. Verworfene Kurven werden rot hervorgehoben.
- Verwenden Sie die Schaltfläche Manueller Umschalter , um Kurven, die nicht korrekt erfasst wurden, manuell zu entfernen. Entfernen Sie alle Kurven, indem Sie auf die spezifische Kurve klicken und OUT auswählen, wodurch die Kurve rot hervorgehoben wird.
- Klicken Sie auf JSON speichern. Geben Sie einen geeigneten Namen für das bereinigte Dataset ein, bei dem es sich um eine einzelne JSON-Datei handelt. Senden Sie die JSON-Datei an den Computer, auf dem die NanoAnalysis-Software installiert wurde.
6. Formale Datenanalyse
- Starten Sie die nano.py Datei über die Befehlszeile, indem Sie zum Ordner NanoAnalysis navigieren und wie zuvor beschrieben ein Terminal starten. Geben Sie den Befehl python nano.py oder python3 nano.py ein (je nach Betriebssystem) und drücken Sie die Eingabetaste . Eine grafische Benutzeroberfläche wird auf Ihrem Bildschirm angezeigt (Abbildung S3).
- Klicken Sie oben links in der GUI auf Load Experiment und wählen Sie die JSON-Datei aus. Dadurch werden die Dateiliste und das Diagramm Rohkurven gefüllt, das den Datensatz in Form von F-z-Kurven anzeigt. Sowohl die F- als auch die z-Achse werden relativ zu den CP-Koordinaten angezeigt, die beim Laden des Datensatzes im Hintergrund berechnet werden (siehe folgende Anmerkung). Aktivieren Sie im Feld Statistiken die Werte der drei Parameter: Naktiviert, N fehlgeschlagen und Nausgeschlossen.
HINWEIS:N aktiviert stellt die Anzahl der Kurven dar, die in der nachfolgenden Hertz-/Elastizitätsspektrenanalyse analysiert werden und im Diagramm Rohkurven schwarz angezeigt werden. Nfehlgeschlagen stellt die Anzahl der Kurven dar, auf denen kein zuverlässiger CP gefunden werden konnte, und wird im Diagramm blau dargestellt. Diese Kurven werden in der nachfolgenden Analyse automatisch verworfen. Beim Öffnen der Software werden einige Kurven möglicherweise automatisch in den fehlerhaften Satz verschoben. Dies liegt daran, dass der CP beim Öffnen der Software mit dem Standard-Schwellenwertalgorithmus berechnet wird (siehe unten). N ausgeschlossen stellt die Kurven dar, die manuell ausgewählt wurden, um aus der Analyseausgeschlossen zu werden, und wird im Diagramm rot angezeigt (siehe unten). - Überprüfen Sie, ob die Anzahl der fehlgeschlagenen, ausgeschlossenen und aktivierten Kurven angemessen ist. Überprüfen Sie das Diagramm "Rohkurven ", um die Kurven zu visualisieren.
- Um eine bestimmte Kurve genauer zu visualisieren, klicken Sie darauf. Dadurch wird es grün hervorgehoben und im Diagramm der aktuellen Kurve angezeigt. Sobald eine einzelne Kurve ausgewählt wurde, werden die R - und k-Parameter (die für alle Kurven gleich sein müssen) im Feld Statistik der GUI ausgefüllt.
- Ändern Sie den Status einer bestimmten Kurve mithilfe des Felds Umschalten . Klicken Sie auf die spezifische Kurve, deren Status Sie ändern möchten, und klicken Sie dann entweder auf Aktiviert, Fehlgeschlagen oder Ausgeschlossen. Die Anzahl im Feld Statistiken wird automatisch aktualisiert.
HINWEIS: Um die Ansicht des Datensatzes im Diagramm Rohkurven zu ändern, verwenden Sie das Feld Ansicht. Klicken Sie auf Alle, um alle Kurven (d.h. aktiviert, fehlgeschlagen und ausgeschlossen) in den jeweiligen Farben anzuzeigen. Klicken Sie auf Fehlgeschlagen, um die aktivierten und die fehlgeschlagenen Kurven anzuzeigen, und klicken Sie auf Aktiviert, um nur die aktivierten Kurven anzuzeigen. Um den Status aller Kurven zwischen aktiviert und ausgeschlossen zurückzusetzen, klicken Sie im Feld Zurücksetzen auf Aktiviert oder Ausgeschlossen. - Nachdem das Dataset weiter bereinigt wurde, folgen Sie der unten beschriebenen Datenanalyse-Pipeline.
- Filtern Sie alle in den Kurven vorhandenen Geräusche mit den in der GUI (Filterbox) implementierten Filtern, nämlich einem benutzerdefinierten Filter namens Prominenzfilter, dem Savitzky Golay30,31 (SAVGOL) -Filter und einem Glättungsfilter, der auf der Berechnung des Medians der Daten in einem bestimmten Fenster basiert (Medianfilter). Weitere Informationen zu Filtern finden Sie unter Diskussion (kritische Schritte im Protokoll).
- Überprüfen Sie die gefilterten Kurven im Diagramm Aktuelle Kurve . Die gefilterte Kurve wird schwarz dargestellt, während die nicht gefilterte Version der Kurve grün dargestellt wird.
HINWEIS: Es wird empfohlen, die Daten so wenig wie möglich zu filtern, um die Merkmale des Originalsignals zu erhalten. Übermäßiges Filtern kann Unterschiede in den Daten ausgleichen. Für die Hertz-Analyse von Nanoindentationsdaten reicht das Arbeiten mit aktiviertem Prominenzfilter aus. Wenn die Daten besonders verrauscht sind, kann zusätzlich ein SAVGOL- oder Medianfilter angewendet werden. - Wählen Sie einen Algorithmus aus, um den CP zu finden. Wählen Sie im Feld Kontaktpunkt eines aus einer Reihe von numerischen Verfahren aus, die in der Software implementiert wurden, nämlich die Anpassungsgüte (GoF)32, das Varianzverhältnis (RoV)32, die zweite Ableitung 33 oder den Schwellenwert 33. Weitere Informationen zu den Algorithmen finden Sie unter Diskussion, kritische Schritte im Protokoll.
HINWEIS: Der CP ist der Punkt, an dem die Sonde mit dem Material in Kontakt kommt und identifiziert werden muss, um F-z-Daten in F-δ-Daten umzuwandeln (bei weichen Materialien ist δ endlich und muss berechnet werden). Der ausgewählte Algorithmus wird auf alle aktiven Kurven des Datensatzes angewendet, und Kurven, bei denen der Algorithmus den CP nicht robust lokalisieren kann, werden in den fehlerhaften Satz verschoben. - Passen Sie die Parameter des Algorithmus an Ihren Datensatz an, so dass der CP korrekt lokalisiert wird, wie in der Diskussion, kritische Schritte im Protokoll beschrieben. Um zu sehen, wo der CP auf einer einzelnen Kurve gefunden wurde, wählen Sie die Kurve aus, indem Sie darauf klicken und auf Prüfen klicken. Überprüfen Sie das angezeigte Popup-Fenster, um festzustellen, wo sich der CP befindet.
- Überprüfen Sie, ob die angezeigte rote Linie, d. h. der Parameter, den der Algorithmus in der ausgewählten Region von Interesse berechnet hat, einen Maximal- oder Minimalwert hat, der der Position des CP entspricht (z. B. für die GoF ist der Parameter R2). Wiederholen Sie diesen Vorgang bei Bedarf für alle Kurven.
HINWEIS: Die Achsen des Pop-ups sind in absoluten Koordinaten, so dass die Position des CP angezeigt werden kann. Umgekehrt werden die Achsen des Diagramms Rohkurven und Stromkurve relativ zum CP angezeigt, d.h. die Position des CP ist (0,0). - Klicken Sie auf Hertz-Analyse. Dadurch werden drei Diagramme generiert, die unten beschrieben werden.
- Überprüfen Sie einzelne F-δ-Kurven im Datensatz zusammen mit der durchschnittlichen Hertz-Anpassung (rote gestrichelte Linie). Passen Sie den Einzug in nm, bis zu dem das Hertz-Modell angepasst ist, im Feld Ergebnisse unter Max. Einzug (nm) an. Setzen Sie es auf maximal ~10% von R , damit das Hertz-Modell gültig ist (siehe Diskussion, kritische Schritte im Protokoll).
- Überprüfen Sie die durchschnittliche F-δ-Kurve mit Fehlerband, das eine Standardabweichung (SD) zusammen mit der durchschnittlichen Hertz-Anpassung (rote gestrichelte Linie) anzeigt. Visualisieren Sie die durchschnittliche Hertz-Anpassung im Rohkurvendiagramm als Referenz und die Hertz-Anpassung für jede Kurve auf der aktuellen Kurve.
- Überprüfen Sie das Punktdiagramm von E , das sich aus der Anpassung des Hertz-Modells an jede einzelne Kurve ergibt.
- Klicken Sie entweder auf den Dateinamen, die F-z-Kurve , die F-δ-Kurve oder einen Punkt im Punktdiagramm, um die Kurve in jedem Diagramm hervorzuheben. Wenn ein Datenpunkt im Punktdiagramm außerhalb der Verteilung der Daten zu liegen scheint, klicken Sie darauf, und überprüfen Sie die Kurve, zu der er gehört. Überprüfen Sie, ob der CP korrekt gefunden wurde, indem Sie auf die Schaltfläche Prüfen klicken. Schließen Sie die Kurve ggf. aus der Analyse aus.
- Überprüfen Sie das Feld Ergebnisse auf den berechneten Mittelwert E und seinen SD (Eγ ± σ) und stellen Sie sicher, dass sie für das gegebene Experiment angemessen sind.
- Klicken Sie im Feld Speichern auf Hertz. Geben Sie im Popup-Fenster den Dateinamen und das Verzeichnis ein. Wenn Sie fertig sind, klicken Sie auf Speichern. Eine TSV-Datei wird erstellt. Öffnen Sie die .tsv-Datei in einer weiteren Software Ihrer Wahl und verwenden Sie die Werte für statistische Analysen und weitere Darstellungen.
HINWEIS: Die Datei enthält das aus jeder Kurve erhaltene E sowie den Mittelwert E und seine SD. Darüber hinaus enthält die Datei Metadaten, die der Analyse zugeordnet sind, einschließlich der Anzahl der analysierten Kurven, R, k und des maximalen Einzugs, der für das Hertz-Modell verwendet wird. - Dieser Schritt ist optional. Klicken Sie auf Durchschnittliche F-Ind , um die durchschnittliche Kraft und den durchschnittlichen Einzug zusammen mit einem SD in der Kraft zu exportieren.
- Für Zellnanoindentationsdaten klicken Sie auf Elastizitätsspektrenanalyse (siehe Repräsentative Ergebnisse und Diskussion). Untersuchen Sie die beiden erzeugten Diagramme, nämlich E als Funktion der Eindringtiefe (E(δ)) für jede Kurve und das durchschnittliche E(δ) mit Fehlerband, das einen SD (durchgezogene rote Linie und schattierte Fläche) zeigt, der von einem Modell angepasst wird (schwarze gestrichelte Linie), das es ermöglicht, den Elastizitätsmodul des Aktincortex der Zelle, den Young-Modul der Zelle zu schätzen. und die Dicke des Aktinkortex. Überprüfen Sie außerdem den Durchschnitt E(δ) im oberen Diagramm in rot.
- Stellen Sie sicher, dass das Kontrollkästchen Interpolieren aktiviert ist, um sicherzustellen, dass die für die Elastizitätsspektrenanalyse erforderliche Ableitung für das interpolierte Signal berechnet wird (siehe Repräsentative Ergebnisse).
- Untersuchen Sie das Feld Ergebnisse und melden Sie den Elastizitätsmodul des Kortex (E 0 ± σ), den Bulk-Elastizitätsmodul der Zelle (Eb ± σ) und die Kortexdicke (d0 ± σ).
HINWEIS: Die durchschnittlichen Elastizitätsspektren können auf den ersten Blick verrauscht erscheinen, mit deutlichen sinusförmigen Schwingungen. Dies hat zur Folge, dass Gleichung (3) möglicherweise nicht korrekt angepasst wird. Wenn dies der Fall ist, löst eine iterative Erhöhung der Fensterlänge des glättenden SAVGOL-Filters34 dieses Problem. - Sobald die Analyse abgeschlossen ist, klicken Sie auf ES im Feld Speichern . Dadurch wird eine TSV-Datei in das angegebene Verzeichnis exportiert, die die durchschnittliche Elastizität als Funktion der durchschnittlichen Eindringtiefe und des Kontaktradius, die dem Experiment zugeordneten Metadaten (siehe oben) und die oben erläuterten geschätzten Modellparameter enthält. Schließlich wird auch die durchschnittliche Elastizität ohne Berücksichtigung der Abhängigkeit von δ und ihrer SD berichtet.
- Schließen Sie die Software und geben Sie die gespeicherten Ergebnisse in eine andere Software Ihrer Wahl ein, um die Daten weiter zu zeichnen und statistische Analysen durchzuführen.
- Dieser Schritt ist optional: Exportieren Sie Diagramme aus der GUI, indem Sie mit der rechten Maustaste auf das Diagramm klicken und Exportieren auswählen. Exportieren Sie das Diagramm in .svg, so dass Parameter wie Schriftart, Schriftgröße, Linienstil usw. kann in einer anderen Software Ihrer Wahl bearbeitet werden.
HINWEIS:Benutzerdefinierte CP-Algorithmen und Filter können programmiert und zu den bereits vorhandenen hinzugefügt werden. Einzelheiten siehe Ergänzende Anmerkung 1 .
Ergebnisse
Nach dem Protokoll wird ein Satz von F-z-Kurven erhalten. Das Dataset enthält höchstwahrscheinlich gute Kurven und Kurven, die verworfen werden müssen, bevor mit der Analyse fortgefahren wird. Im Allgemeinen sollten Kurven verworfen werden, wenn sich ihre Form von der in Abbildung 4A gezeigten unterscheidet. Abbildung 5AI zeigt einen Datensatz von ~100 Kurven, die auf einem weichen PAAm-Hydrogel von erwartetem E 0,8 KPa35
Diskussion
Dieses Protokoll zeigt, wie Kraftspektroskopie-Nanoindentationsdaten mit einem kommerziell erhältlichen Ferrule-Top-Nanoindenter sowohl auf Hydrogelen als auch auf einzelnen Zellen robust erfasst werden können. Darüber hinaus werden Anleitungen zur Verwendung einer in Python programmierten Open-Source-Software bereitgestellt, die einen präzisen Workflow für die Analyse von Nanoindentationsdaten umfasst.
Kritische Schritte im Protokoll
Die folgenden Schritte haben sich ...
Offenlegungen
Die Autoren haben nichts offenzulegen.
Danksagungen
GC und MAGO erkennen alle Mitglieder des CeMi an. MSS würdigt die Unterstützung durch einen EPSRC-Programmzuschuss (EP/P001114/1).
GC: Software (Beitrag zur Softwareentwicklung und Algorithmen), formale Analyse (Analyse von Nanoindentationsdaten), Validierung, Untersuchung (Nanoindentationsexperimente an Polyacrylamidgelen), Datenkuration, Schreiben (Originalentwurf, Review und Bearbeitung), Visualisierung (Abbildungen und Grafiken). MAGO: Untersuchung (Vorbereitung von Gel- und Zellproben, Nanoindentationsexperimente an Zellen), Schreiben (Originalentwurf, Review und Editierung), Visualisierung (Abbildungen und Grafiken). NA: Validierung, Schreiben (Review und Editierung). IL: Software (Beitrag zur Softwareentwicklung und Algorithmen), Validierung, Schreiben (Review und Editing); MV: Konzeptualisierung, Software (Design und Entwicklung von Originalsoftware und Algorithmen), Validierung, Ressourcen, Schreiben (Originalentwurf, Review und Redaktion), Supervision, Projektadministration, Mittelakquise MSS: Ressourcen, Schreiben (Review und Editing), Supervision, Projektadministration, Mittelakquise. Alle Autoren haben das endgültige Manuskript gelesen und genehmigt.
Materialien
| Name | Company | Catalog Number | Comments |
| 12 mm coverslips | VWR | 631-1577P | |
| 35 mm cell treated culture dishes | Greiner CELLSTAR | 627160 | |
| Acrylamide | Sigma-Aldrich | A4058 | |
| Acrylsilane | Alfa Aesar | L16400 | |
| Ammonium Persulfate | Merk | 7727-54-0 | |
| Bisacrylamide | Merk | 110-26-9 | |
| Chiaro nanoindenter | Optics 11 Life | no catologue number | |
| Ethanol | general | ||
| Fetal bovine serum | Gibco | 16140071 | |
| High glucose DMEM | Gibco | 11995065 | |
| Isopropanol | general | ||
| Kimwipe | Kimberly Clark | 21905-026 | |
| Microscope glass slides | VWR | 631-1550P | |
| MilliQ system | Merk Millipore | ZR0Q008WW | |
| OP1550 Interferometer | Optics11 Life | no catalogue number | |
| Optics 11 Life probe (k = 0.02-0.005 N/m, R = 3-3.5 um) | Optics 11 Life | no catologue number | |
| Optics 11 Life probe (k = 0.46-0.5 N/m, R = 50-55 um) | Optics 11 Life | no catologue number | |
| Penicillin/Streptomycin | Gibco | 15140122 | |
| RainX rain repellent | RainX | 26012 | |
| Standard petri dishes (90 mm) | Thermo Scientific | 101RTIRR | |
| Tetramethylethylenediamine | Sigma-Aldrich | 110-18-9 | |
| Vaccum dessicator | Thermo Scientific | 531-0250 | |
| Software | |||
| Data acquisition software (v 3.4.1) | Optics 11 Life | ||
| GitHub Desktop (Optional) | Microsoft | ||
| Python 3 | Python Software Foundation | ||
| Visual Studio Code (Optional) | Microsoft |
Referenzen
- Discher, D. E., Janmey, P. Y. W. Tissue cells feel and respond to the stiffness of their substrate. Science. 310 (5751), 1139-1143 (2005).
- Roca-Cusachs, P., Conte, V., Trepat, X. Quantifying forces in cell biology. Nature Cell Biology. 19 (7), 742-751 (2017).
- Guimarães, C. F., Gasperini, L., Marques, A. P., Reis, R. L. The stiffness of living tissues and its implications for tissue engineering. Nature. Reviews. Materials. 5, 351-370 (2020).
- Moeendarbary, E., Harris, A. R. Cell mechanics: Principles, practices, and prospects. Wiley Interdisciplinary. Reviews. Systems. Biology and Medicine. 6 (5), 371-388 (2014).
- Butcher, D. T., Alliston, T., Weaver, V. M. A tense situation: Forcing tumour progression. Nature Reviews Cancer. 9 (2), 108-122 (2009).
- Kubánková, M., et al. Physical phenotype of blood cells is altered in COVID-19. Biophysical Journal. 120 (14), 2838-2847 (2021).
- Engler, A. J., Sen, S., Sweeney, H. L., Discher, D. E. Matrix elasticity directs stem cell lineage specification. Cell. 126, 677-689 (2006).
- Ciccone, G., et al. What caging force cells feel in 3D hydrogels: A rheological perspective. Advanced Healthcare Materials. 9 (17), 2000517 (2020).
- Wu, P. H., et al. A comparison of methods to assess cell mechanical properties. Nature Methods. 15 (7), 491-498 (2018).
- McKee, C. T., Last, J. A., Russell, P., Murphy, C. J. Indentation versus tensile measurements of young's modulus for soft biological tissues. Tissue Engineering B Reviews. 17 (3), 155-164 (2011).
- Humphrey, J. D., Delange, S. L. . An Introduction to Biomechanics Solids and Fluids, Analysis and Design. , 271-371 (2004).
- Prevedel, R., Diz-Muñoz, A., Ruocco, G., Antonacci, G. Brillouin microscopy: an emerging tool for mechanobiology. Nature Methods. 16 (10), 969-977 (2019).
- Krieg, M., et al. Atomic force microscopy-based mechanobiology. Nature Reviews Physics. 1, 41-57 (2019).
- Norman, M. D. A., Ferreira, S. A., Jowett, G. M., Bozec, L., Gentleman, E. Measuring the elastic modulus of soft culture surfaces and three-dimensional hydrogels using atomic force microscopy. Nature Protocols. 16 (5), 2418-2449 (2021).
- Gavara, N. A beginner's guide to atomic force microscopy probing for cell mechanics. Microscopy Research and Technique. 80 (1), 75-84 (2017).
- Whitehead, A. J., Kirkland, N. J., Engler, A. J. Atomic force microscopy for live-cell and hydrogel measurement. Methods in Molecular Biology. 2299, 217-226 (2021).
- Chavan, D., et al. Ferrule-top nanoindenter: An optomechanical fiber sensor for nanoindentation. The Review of Scientific Instruments. 83 (11), 115110 (2012).
- Van Hoorn, H., Kurniawan, N. A., Koenderink Ac, G. H., Iannuzzi, D. Local dynamic mechanical analysis for heterogeneous soft matter using ferrule-top indentation. Soft Matter. 12 (12), 3066-3073 (2016).
- Baldini, F., et al. Biomechanics of cultured hepatic cells during different steatogenic hits. Journal of the Mechanical Behavior of Biomedical Materials. 97, 296-305 (2019).
- Emig, R., et al. Piezo1 channels contribute to the regulation of human atrial fibroblast mechanical properties and matrix stiffness sensing. Cells. 10 (3), 663 (2021).
- Dobre, O., et al. A hydrogel platform that incorporates laminin isoforms for efficient presentation of growth factors - Neural growth and osteogenesis. Advanced Functional Materials. 31 (21), 2010225 (2021).
- Antonovaite, N., Beekmans, S. V., Hol, E. M., Wadman, W. J., Iannuzzi, D. Regional variations in stiffness in live mouse brain tissue determined by depth-controlled indentation mapping. Scientific Reports. 8 (1), 12517 (2018).
- Wu, G., Gotthardt, M., Gollasch, M. Assessment of nanoindentation in stiffness measurement of soft biomaterials: kidney, liver, spleen and uterus. Scientific Reports. 10 (1), 18784 (2020).
- Lüchtefeld, I., et al. Elasticity spectra as a tool to investigate actin cortex mechanics. Journal of Nanobiotechnology. 18 (1), 147 (2020).
- Caliari, S. R., Burdick, J. A. A practical guide to hydrogels for cell culture. Nature Methods. 13 (5), 405-414 (2016).
- Blumlein, A., Williams, N., McManus, J. J. The mechanical properties of individual cell spheroids. Scientific Reports. 7 (1), 7346 (2017).
- Mirsandi, H., et al. Influence of wetting conditions on bubble formation from a submerged orifice. Experiments in Fluids. 61, 83 (2020).
- Vassalli, M., Ciccone, G. . CellMechLab/NanoPrepare: v0.1.1. Zendo. , (2021).
- Vassalli, M., Ciccone, G., Lüchtefeld, I. CellMechLab/nanoindentation: v1.0.0. Zendo. , (2021).
- Savitzky, A., Golay, M. J. E. Smoothing and differentiation of data by simplified least squares procedures. Analytical Chemistry. 36 (8), 1627-1639 (1964).
- Schafer, R. W. What is a savitzky-golay filter. IEEE Signal Processing Magazine. 28 (4), 111-117 (2011).
- Gavara, N. Combined strategies for optimal detection of the contact point in AFM force-indentation curves obtained on thin samples and adherent cells. Scientific Reports. 6, 21267 (2016).
- Lin, D. C., Dimitriadis, E. K., Horkay, F. Robust strategies for automated AFM force curve analysis-I. Non-adhesive indentation of soft, inhomogeneous materials. Journal of Biomechanical Engineering. 129 (3), 430-440 (2007).
- Virtanen, P., et al. SciPy 1.0: fundamental algorithms for scientific computing in Python. Nature Methods. 17 (3), 261-272 (2020).
- Tse, J. R., Engler, A. J. Preparation of hydrogel substrates with tunable mechanical properties. Current Protocols in Cell Biology. , (2010).
- Pharr, G. M., Oliver, W. C., Brotzen, F. R. On the generality of the relationship among contact stiffness, contact area, and elastic modulus during indentation. Journal of Materials Research. 7 (3), 613-617 (1992).
- Kumar, R., Saha, S., Sinha, B. Cell spread area and traction forces determine myosin-II-based cortex thickness regulation. Biochimica et Biophysica Acta - Molecular Cell Research. 1866 (12), 118516 (2019).
- Kunda, P., Pelling, A. E., Liu, T., Baum, B. Moesin controls cortical rigidity, cell rounding, and spindle morphogenesis during mitosis. Current Biology: CB. 18 (2), 91-101 (2008).
- Allen, M., Poggiali, D., Whitaker, K., Marshall, T. R., Kievit, R. A. Raincloud plots: a multi-platform tool for robust data visualization. Wellcome Open Research. 4, 63 (2019).
- Clark, A. G., Paluch, E. Mechanics and regulation of cell shape during the cell cycle. Results and Problems in Cell Differentiation. 53, 31-73 (2011).
- Langan, T. J., Rodgers, K. R., Chou, R. C. Synchronization of mammalian cell cultures by serum deprivation. Methods in Molecular Biology. 1524, 97-105 (2017).
- Hodgkinson, T., et al. The use of nanovibration to discover specific and potent bioactive metabolites that stimulate osteogenic differentiation in mesenchymal stem cells. Science Advances. 7 (9), 7921 (2021).
- Kloxin, A. M., Kloxin, C. J., Bowman, C. N., Anseth, K. S. Mechanical properties of cellularly responsive hydrogels and their experimental determination. Advanced Materials (Deerfield Beach, Fla). 22 (31), 3484-3494 (2010).
- Loessner, D., et al. Functionalization, preparation and use of cell-laden gelatin methacryloyl-based hydrogels as modular tissue culture platforms. Nature Protocols. 11 (4), 727-746 (2016).
- Yue, K., et al. Synthesis, properties, and biomedical applications of gelatin methacryloyl (GelMA) hydrogels. Biomaterials. 73, 254-271 (2015).
- Sarrigiannidis, S. O., et al. A tough act to follow: collagen hydrogel modifications to improve mechanical and growth factor loading capabilities. Materials Today. Bio. 10, 100098 (2021).
- Karoutas, A., et al. The NSL complex maintains nuclear architecture stability via lamin A/C acetylation. Nature Cell Biology. 21 (10), 1248-1260 (2019).
- Ryu, H., et al. Transparent, compliant 3D mesostructures for precise evaluation of mechanical characteristics of organoids. Advanced Materials (Deerfield Beach, Fla). 33 (25), 2100026 (2021).
- Carvalho, D. T. O., Feijão, T., Neves, M. I., Da Silva, R. M. P., Barrias, C. C. Directed self-assembly of spheroids into modular vascular beds for engineering large tissue constructs Biofabrication Directed self-assembly of spheroids into modular vascular beds for engineering large tissue constructs. Biofabrication. 13 (3), 035008 (2021).
- Chaudhuri, O., Cooper-White, J., Janmey, P. A., Mooney, D. J., Shenoy, V. B. Effects of extracellular matrix viscoelasticity on cellular behaviour. Nature. 584 (7822), 535-546 (2020).
- Cantini, M., Donnelly, H., Dalby, M. J., Salmeron-Sanchez, M. The plot thickens: The emerging role of matrix viscosity in cell mechanotransduction. Advanced Healthcare Materials. 9 (8), 1901259 (2020).
- Elosegui-Artola, A. The extracellular matrix viscoelasticity as a regulator of cell and tissue dynamics. Current Opinion in Cell Biology. 72, 10-18 (2021).
- Chaudhuri, O. Viscoelastic hydrogels for 3D cell culture. Biomaterials Science. 5 (8), 1480-1490 (2017).
- Nguyen, T. D., Gu, Y. Determination of strain-rate-dependent mechanical behavior of living and fixed osteocytes and chondrocytes using atomic force microscopy and inverse finite element analysis. Journal of Biomechanical Engineering. 136 (10), 101004 (2014).
- Mokbel, M., Hosseini, K., Aland, S., Fischer-Friedrich, E. The Poisson Ratio of the Cellular Actin Cortex Is Frequency Dependent. Biophysical Journal. 118 (8), 1968-1976 (2020).
- Javanmardi, Y., Colin-York, H., Szita, N., Fritzsche, M., Moeendarbary, E. Quantifying cell-generated forces: Poisson's ratio matters. Communications Physics. 4, 237 (2021).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten