Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Экспериментальный рабочий процесс и анализ данных для наноиндентации мягкого вещества
В этой статье
Резюме
Протокол представляет собой полный рабочий процесс для экспериментов с наноиндентированием мягких материалов, включая гидрогели и клетки. Во-первых, подробно описаны экспериментальные этапы получения данных силовой спектроскопии; затем анализ таких данных детализируется с помощью недавно разработанного программного обеспечения Python с открытым исходным кодом, которое можно бесплатно загрузить с GitHub.
Аннотация
Наноиндентация относится к классу экспериментальных методов, где микрометрический силовой зонд используется для количественной оценки локальных механических свойств мягких биоматериалов и клеток. Этот подход приобрел центральную роль в области механобиологии, проектирования биоматериалов и тканевой инженерии, чтобы получить надлежащую механическую характеристику мягких материалов с разрешением, сопоставимым с размером одиночных клеток (мкм). Наиболее популярной стратегией получения таких экспериментальных данных является использование атомно-силового микроскопа (АСМ); в то время как этот прибор предлагает беспрецедентное разрешение в силе (вплоть до pN) и пространстве (суб-нм), его удобство использования часто ограничено его сложностью, которая предотвращает рутинные измерения интегральных показателей механических свойств, таких как модуль Юнга (E). Новое поколение наноиндентаторов, таких как те, которые основаны на технологии оптического зондирования волокна, недавно приобрело популярность благодаря своей простоте интеграции, позволяя применять силы sub-nN с пространственным разрешением μm, поэтому они подходят для исследования локальных механических свойств гидрогелей и ячеек.
В этом протоколе представлено пошаговое руководство, подробно описывающее экспериментальную процедуру получения данных наноиндентирования на гидрогелях и клетках с использованием коммерчески доступного наноиндентера с оптическим волоконным зондированием с наконечником. Принимая во внимание, что некоторые этапы являются специфическими для инструмента, используемого в настоящем документе, предлагаемый протокол может быть принят в качестве руководства для других устройств наноиндентирования, при условии, что некоторые этапы адаптированы в соответствии с руководящими принципами производителя. Кроме того, представлено новое программное обеспечение Python с открытым исходным кодом, оснащенное удобным графическим пользовательским интерфейсом для анализа данных наноиндентации, которое позволяет проводить скрининг неправильно полученных кривых, фильтрацию данных, вычисление точки контакта с помощью различных численных процедур, обычное вычисление E, а также более продвинутый анализ, особенно подходящий для одноклеточных наноиндентационных данных.
Введение
Фундаментальная роль механики в биологии в настоящее время установлена 1,2. От целых тканей до одиночных клеток механические свойства могут информировать о патофизиологическом состоянии исследуемого биоматериала 3,4. Например, ткань молочной железы, пораженная раком, жестче, чем здоровая ткань, концепция, которая лежит в основе популярного пальпационного теста5. Примечательно, что недавно было показано, что коронавирусная болезнь 2019 года (COVID-19), вызванная тяжелым острым респираторным синдромом коронавируса 2 (SARS-CoV-2), подчеркивается изменениями механических свойств клеток крови, включая снижение деформируемости эритроцитов и снижение жесткости лимфоцитов и нейтрофилов по сравнению с клетками крови у SARS-CoV-2-наивных лиц6.
В целом, механика клеток и тканей по своей природе переплетена: каждая ткань обладает специфическими механическими свойствами, которые одновременно влияют и зависят от свойств составляющих клеток и внеклеточного матрикса (ECM)5. Из-за этого стратегии изучения механики в биологии часто включают инженерные субстраты с физиологически значимыми механическими стимулами для выяснения поведения клеток в ответ на эти стимулы. Например, основополагающая работа Энглера и его коллег продемонстрировала, что приверженность линии мезенхимальных стволовых клеток контролируется эластичностью матрицы, как это изучалось на мягких и жестких двумерных полиакриламидных (PAAm) гидрогелях7.
Существует множество стратегий для механической характеристики исследуемого биоматериала, варьирующихся по пространственному масштабу (т.е. от локального к объемному) и по способу деформации (например, осевой или сдвиговой), следовательно, давая различную информацию, которая требует тщательной интерпретации 3,8,9,10. Механика мягких биоматериалов обычно выражается в терминах жесткости. Однако жесткость зависит как от свойств материала, так и от геометрии, тогда как модули упругости являются фундаментальными свойствами материала и не зависят от геометрии материала11. Таким образом, различные модули упругости связаны с жесткостью данного образца, и каждый модуль упругости охватывает сопротивление материала определенному режиму деформации (например, осевой против сдвига) при различных граничных условиях (например, свободное расширение против удержания)11,12. Эксперименты с наноиндентированием позволяют количественно оценить механические свойства через Е, что связано с одноосной деформацией (отступом), когда биоматериал не ограничен латерально 10,11,12.
Наиболее популярным методом количественной оценки Е биологических систем на микроуровне является AFM 13,14,15,16. AFM является чрезвычайно мощным инструментом с силовым разрешением вплоть до уровня pN и пространственным разрешением вплоть до суб-нм масштаба. Кроме того, AFM предлагает чрезвычайную гибкость с точки зрения соединения с дополнительными оптическими и механическими инструментами, расширяя свои возможности по извлечению большого количества информации из исследуемого биоматериала13. Эти привлекательные особенности, однако, имеют барьер для входа, представленный сложностью экспериментальной установки. AFM требует обширной подготовки, прежде чем пользователи смогут получить надежные данные, и его использование для повседневной механической характеристики биологических материалов часто неоправданно, особенно когда его уникальная сила и пространственное разрешение не требуются.
Из-за этого новый класс наноиндеттеров недавно приобрел популярность из-за их простоты использования, в то же время предлагая сопоставимые с AFM данные с разрешением силы sub-nN и пространственным разрешением мкм, отражая силы, оказываемые и воспринимаемые клетками по соответствующим масштабам длины2. В частности, наконечники наноиндентации на основе технологии оптического зондированияволокна 17,18 завоевали популярность среди исследователей, работающих в области механобиологии и за ее пределами; и было опубликовано множество работ, сообщающих о механических свойствах биоматериалов с использованием этих устройств, включая клетки 19,20, гидрогели 8,21 и ткани 22,23. Несмотря на возможности этих систем исследовать локальные динамические механические свойства (т.е. модуль хранения и потерь), квазистатические эксперименты, дающие E, остаются наиболее популярным выбором 8,19,20,21. Короче говоря, квазистатические эксперименты с наноиндентированием состоят из отступа образца с постоянной скоростью до заданной точки, определяемой либо максимальным смещением, силой или глубиной отступа, и регистрации как силы, так и вертикального положения консольного аппарата в так называемых кривых расстояния силы (F-z). Затем кривые F-z преобразуются в кривые с отступом по силе (F-δ) путем идентификации точки контакта (CP) и оснащаются соответствующей моделью контактной механики (обычно моделью Герца13) для вычисления E.
Хотя работа наноиндеттеров с наконечниками напоминает измерения AFM, есть особенности, которые стоит учитывать. В этой работе представлено пошаговое руководство по надежному получению кривых F-z из клеток и тканевых гидрогелей с использованием коммерчески доступного наноиндентера с наконечником, чтобы стимулировать стандартизацию экспериментальных процедур между исследовательскими группами, использующими это и другие подобные устройства. Кроме того, даются советы о том, как наилучшим образом подготовить образцы гидрогеля и клетки для проведения экспериментов по наноиндентации, а также советы по устранению неполадок вдоль экспериментального пути.
Кроме того, большая часть изменчивости результатов наноиндентирования (т.е. E и его распределение) зависит от конкретной процедуры, используемой для анализа данных, которая является нетривиальной. Для решения этой проблемы приведены инструкции по использованию недавно разработанного программного обеспечения с открытым исходным кодом, запрограммированного на Python и оснащенного удобным графическим интерфейсом пользователя (GUI) для пакетного анализа кривых F-z . Программное обеспечение позволяет проводить быстрый скрининг данных, фильтрацию данных, вычисление CP с помощью различных численных процедур, обычное вычисление E, а также более продвинутый анализ, называемый спектрами упругости24, позволяющий оценить объемный модуль Юнга клетки, модуль Юнга актиновой коры и толщину коры актина. Программное обеспечение может быть свободно загружено с GitHub и может быть легко адаптировано для анализа данных, поступающих из других систем, путем добавления соответствующего парсера данных. Подчеркивается, что этот протокол может быть использован для других устройств наноиндентации с наконечниками и других наноиндентационных устройств в целом, при условии, что некоторые этапы адаптированы в соответствии с руководящими принципами конкретного инструмента. Протокол схематично обобщен на рисунке 1.
протокол
1. Подготовка субстратов/клеток для измерений наноиндентов
- Следуйте шагам, приведенным в Дополнительном протоколе для получения гидрогелей/клеток PAAm для экспериментов по наноиндентированию. Процедура обобщена на рисунке 2.
ПРИМЕЧАНИЕ: Гидрогели PAAm были выбраны, поскольку они являются наиболее распространенными гидрогелями, используемыми в области механобиологии. Однако протокол в равной степени применим к любому типу гидрогеля25 (см. Обсуждение, модификации метода).
2. Запуск устройства, выбор зонда и калибровка зонда
- Выполните действия, описанные в Дополнительном протоколе для запуска устройства. Для получения технической информации о работе волоконно-оптических наконечников с верхними наноиндентами, проверьте эти ссылки17,18.
- Выберите наноиндентационный зонд, как описано ниже.
ПРИМЕЧАНИЕ: Все коммерчески доступные датчики, такие как те, которые используются в этом протоколе, оснащены сферическим наконечником (рисунок 3A). Поэтому выбор сужается до двух переменных: жесткости консольного аппарата и радиуса наконечника (рисунок 3B).- Выберите жесткость консольного аппарата (k в Н/м), которая соответствует ожидаемой жесткости образца для достижения наилучших результатов15 (см. Рисунок 3C и Обсуждение, критические шаги в протоколе). Для ячеек выберите зонд с k в диапазоне 0,01-0,09 Н/м. Для гидрогелей выберите зонд с k в диапазоне 0,1-0,9 Н/м, который дает оптимальные результаты для гелей с ожидаемым Е от нескольких кПа до 100 кПа (см. Репрезентативные результаты).
- Выберите радиус наконечника (R в мкм) в соответствии с требуемым пространственным разрешением процесса отступа. Для небольших клеток, таких как клетки эмбриональной почки человека 293T (HEK293T) (средний диаметр ~ 10-15 мкм26), выберите сферу с R = 3 мкм. Для гидрогелей выберите сферу с R = 10-250 мкм, чтобы исследовать механические свойства биоматериала на большой площади контакта и избежать локальных неоднородностей.
- Обратитесь к рисунку 3C и любым дополнительным рекомендациям производителя, чтобы выбрать подходящий зонд.
- После того, как зонд выбран, выполните действия, описанные в Дополнительном протоколе , чтобы установить его на наноиндентер.
3. Калибровка зонда
ПРИМЕЧАНИЕ: Следующие шаги специфичны для наконечников наноиндентационных устройств, основанных на технологии оптического зондирования волокна, и они подробно описаны для программного обеспечения версии 3.4.1. Для других устройств наноиндентации выполните действия, рекомендованные производителем устройства.
- В главном окне программного обеспечения нажмите Инициализировать. Появится меню калибровки. Введите данные зонда (можно найти на боковой стороне коробки зонда; k в Н/м, R в мкм и калибровочный коэффициент в воздухе) во входных коробках.
- Приготовьте калибровочную тарелку: толстую стеклянную чашку Петри с плоским дном (см. Обсуждение, критические шаги в протоколе). Наполните блюдо той же средой, что и блюдо с образцом (это также может быть воздух). Сопоставьте температуру среды с температурой образца.
- Поместите калибровочную тарелку под зонд. При необходимости выдвиньте зонд из держателя наноиндентатора и держите его в одной руке, чтобы освободить место для размещения калибровочной тарелки. Сдвиньте зонд обратно на место.
- Выполните следующие два шага для калибровки в жидкости. При измерении на воздухе используйте предоставленную полиэтиленовую подложку из политетрафторэтилена для калибровки и перейдите к шагу 5.
- Добавьте к зонду каплю 70% этанола с помощью пипетки Пастера с концом пипетки в легком контакте со стеклянным наконечником, чтобы капля скользила по консольному и сферическому наконечнику27 (см. Обсуждение, критические шаги в протоколе).
- Вручную сдвиньте руку наноиндентера вниз, пока зонд не будет полностью погружен, но все еще далеко от дна чашки Петри. При необходимости добавьте в калибровочную посуду больше среды. Подождите 5 минут, чтобы в жидкости были достигнуты равновесные условия.
- В меню Инициализация программного обеспечения нажмите кнопку Scan Wavelength(Длина волны сканирования). На экране интерферометра будет показан индикатор выполнения, а в окне Live Signal в программном обеспечении компьютера будет отображаться рисунок, показанный на рисунке S1A, слева. Чтобы проверить, было ли выполнено оптическое сканирование, перейдите на панель «Сканирование по длине волны » на блоке интерферометра. В случае успеха должна быть видна синусоида (рисунок 1A, справа). См. раздел Обсуждение (устранение неполадок метода), если появляется ошибка.
- В меню «Инициализация » нажмите « Найти поверхность», которая будет постепенно опускать зонд до тех пор, пока не будет достигнут установленный порог в изгибе консольного устройства. Зонд перестает двигаться при контакте со стеклянной чашкой Петри.
- Проверьте, находится ли зонд в контакте с поверхностью. Переместите зонд вниз на 1 мкм с помощью кнопки со стрелкой y вниз в главном окне программного обеспечения. Наблюдайте за зеленым сигналом (отклонением консольа) в Интерактивном окне программного обеспечения для изменений базовой линии с каждым шагом, когда консоль находится в контакте с подложкой (рисунок S1B). Если изменений нет, консоль не контактирует (см. следующий шаг).
- Увеличьте пороговое значение в меню Параметры на вкладке Найти Surface со значения по умолчанию 0,01 на шаг 0,01 за раз и повторяйте шаг Найти Surface до тех пор, пока не вступит в контакт. В качестве альтернативы, опустите зонд для контакта небольшими шагами 1 мкм до тех пор, пока зеленая базовая линия не начнет смещаться на каждом шаге вниз.
ПРИМЕЧАНИЕ: Для самых мягких консольных устройств (k = 0,025 Нм-1) увеличьте пороговое значение в меню «Параметры» на вкладке «Найти поверхность» априори выполнения шага 6. Начните с порогового значения 0,06 или 0,07 и при необходимости увеличьте его до 0,1. Это связано с тем, что шум окружающей среды, вероятно, заставит консоль согнуться выше порога перед контактом. Для мягких зондов уменьшение скорости приближения (мкм/с) в том же меню также улучшает процедуру. - В меню Инициализация выберите Калибровать.
- Проверьте окно Live Signal программного обеспечения и убедитесь, что сигналы смещения пьезо и отклонения консольного устройства движутся вверх одновременно (рисунок S1C).
- Если есть несоответствие во времени, зонд не полностью контактирует со стеклом. Двигайтесь вниз по зонду с шагом 1 мкм до тех пор, пока не изменится базовая линия консольного сигнала (см. шаг 7), и повторите шаг 9.
- Если консольный сигнал вообще не изменяется на этапе калибровки , то зонд находится далеко от поверхности. Увеличивайте порог контакта итеративно (см. шаг 8) до тех пор, пока поверхность не будет найдена правильно, и повторяйте калибровку с самого начала, начиная со сканирования длины волны.
- После завершения калибровки проверьте старый и новый коэффициенты калибровки во всплывающем окне. Если новый калибровочный коэффициент находится в правильном диапазоне, нажмите «Использовать новый фактор». Если калибровка не удалась, и новый коэффициент либо NaN, либо не находится в ожидаемом диапазоне, см. Обсуждение (устранение неполадок метода) для разрешения.
ПРИМЕЧАНИЕ: Новый коэффициент должен быть ~n раз ниже, чем тот, который предусмотрен на коробке зонда, если калибровка проводилась в жидкой среде с показателем преломления n (n = 1,33 для воды). Если калибровка проводилась на воздухе, то новый и старый калибровочные коэффициенты должны быть примерно равны. - Проверьте, правильно ли откалиброван круг демодуляции, как показано ниже. Перейдите на вкладку Демодуляция на рабочем столе интерферометра. Осторожно нажмите на оптический стол или на наноиндентатор, чтобы вызвать достаточно шума. Белый круг, состоящий из дискретных точек данных, должен приблизительно закрывать красный круг (рисунок S1D).
- Если белый круг не перекрывается красным или на дисплее интерферометра появляется предупреждение, круг демодуляции нуждается в повторной калибровке. Это можно сделать двумя способами, как описано ниже.
- Непрерывно нажимайте на корпус наноиндентера, чтобы вызвать один полный круг шума, и нажмите кнопку Calibrate на интерферометре.
- Войдите в контакт со стеклянной подложкой и нажмите Calibrate в меню Инициализация главного окна программного обеспечения. Не сохраняйте калибровочный коэффициент. На этом этапе проверьте еще раз и убедитесь, что белый круг перекрывается с красным кругом.
ПРИМЕЧАНИЕ: Если сигнал не просто слегка смещен от круга демодуляции, а стал очень маленьким или вообще не виден, это означает, что консоль приклеена к оптическому волокну. Следуйте инструкциям по устранению этой проблемы (см. Обсуждение, устранение неполадок метода) и повторите шаги 13.1 или 13.2. Как только консоль вернется в горизонтальное положение (рисунок 3A), сигнал восстановится в круг демодуляции.
- Проверьте калибровку, выполнив отступ на стеклянной подложке непосредственно после калибровки, как описано ниже.
- Загрузите или создайте файл эксперимента, щелкнув Настроить эксперимент и добавьте шаг Найти Surface и шаг Отступа . Для этапа отступа используйте настройки режима смещения по умолчанию и измените максимальное смещение на калибровочное расстояние (3000 нм), чтобы сместить зонд против жесткой подложки.
- Нажмите « Запустить эксперимент» и проверьте круг демодуляции в окне интерферометра. Проверьте белый сигнал и убедитесь, что он находится поверх красного круга во время отступа.
- Проверьте результаты в главном окне программного обеспечения на графике данных о времени и убедитесь, что смещение пьезо (синяя линия) равно отклонению консольного аппарата (зеленая линия), поскольку отступ начинает контактировать и не ожидается деформации материала. Если сигналы не параллельны, см. Обсуждение (устранение неполадок метода).
- Измените локальный путь в меню калибровки, установив для параметра Путь сохранения калибровки соответствующий каталог.
- Когда зонд будет успешно откалиброван, переместите пьезо вверх на 500 мкм.
4. Измерение модуля Юнга мягких материалов
- Наноиндентирование гидрогелей
- Загрузите чашку Петри, содержащую образец (образцы), на ступень микроскопа и вручную переместите зонд наноиндентатора в желаемое положение x-y над образцом.
- Вручную сдвиньте зонд в раствор, позаботившись о том, чтобы оставить 1-2 мм между зондом и поверхностью образца на этом этапе. Подождите 5 минут, пока зонд уравновесится в среде.
- Сфокусируйте z-плоскость оптического микроскопа так, чтобы зонд был хорошо виден.
- Выполните один отступ для настройки экспериментальных параметров, как описано ниже.
- Настройте новый эксперимент в главном окне программного обеспечения. Нажмите настроить эксперимент, после чего откроется новое окно. Добавьте шаг Найти Surface . При необходимости все параметры шага Find Surface можно изменить в меню «Параметры» программы.
ПРИМЕЧАНИЕ: Find Surface будет опускать зонд до тех пор, пока поверхность не будет найдена, а затем он втягивает зонд на расстояние, определенное Z Над поверхностью (мкм), над поверхностью образца. Если выбранный консоль слишком жесткий для образца или образец липкий, после стадии зонд, вероятно, все еще будет контактировать с образцом, что приведет к кривой без исходного уровня (рисунок 4C). Чтобы решить эту проблему, увеличьте Z Над поверхностью (мкм). - Добавьте шаг отступа . Выберите вкладку Профиль и нажмите « Контроль перемещения». Оставьте профиль отступа по умолчанию.
- Нажмите «Запустить эксперимент» в главном окне программного обеспечения. Это позволит найти поверхность и выполнить одинарный отступ. Если одиночный отступ выглядит не так, как ожидалось, настройте экспериментальные параметры, как показано на рисунке 4 и Обсуждение (устранение неполадок метода).
- Настройте новый эксперимент в главном окне программного обеспечения. Нажмите настроить эксперимент, после чего откроется новое окно. Добавьте шаг Найти Surface . При необходимости все параметры шага Find Surface можно изменить в меню «Параметры» программы.
- Как только отступ будет выглядеть должным образом, настройте сканирование матрицы таким образом, чтобы достаточная область образца была с отступом. Щелкните Настроить эксперимент, добавьте шаг Find Surface с ранее определенными экспериментальными параметрами и добавьте шаг Matrix Scan .
- Для плоских гидрогелей сконфигурируйте матричное сканирование, содержащее 50-100 точек (т.е. 5 x 10 или 10 x 10 в x и y), расположенных на расстоянии 10-100 мкм (т.е. dx = dy = 10-100 мкм). Нажмите « Использовать положение рабочей области », чтобы начать сканирование матрицы с текущего положения этапа. Убедитесь, что установлен флажок Автоматический поиск поверхности , чтобы найти поверхность при каждом отступе с использованием заданных экспериментальных параметров.
- Чтобы избежать чрезмерной выборки, установите размер шага, по крайней мере, в два раза превышающий радиус контакта (
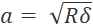 где δ — глубина отступа).
где δ — глубина отступа). - Настройте профиль матричного сканирования в системе управления перемещением. Убедитесь, что профиль не нарушает допущения модели Герца (см. Обсуждение, критические шаги в протоколе).
- Оставьте число сегментов равным 5, что является значением по умолчанию, и используйте профиль смещения по умолчанию. При необходимости измените профиль смещения с точки зрения максимального смещения и времени для каждого наклонного сегмента, что повлияет на максимальную глубину отступа и скорость деформации соответственно. Не превышать скорость деформации > 10 мкм/с (см. Обсуждение, ограничения метода).
- Введите значение скорости приближения, которое определяет, как быстро зонд смещается к образцу перед контактом. Сопоставьте скорость втягивания со скоростью захода на посадку (см. примечание ниже).
ПРИМЕЧАНИЕ: Для мягких консольных аппаратов и шумных сред рекомендуется скорость захода на посадку 1000-2000 нм/с. Для более жестких консольных и контролируемых сред этот показатель может быть увеличен. - Сохраните настроенный эксперимент в нужном пути эксперимента и выберите каталог, в котором будут сохранены данные, в поле Путь сохранения на вкладке Общие окна Настройка эксперимента . Нажмите Кнопка Запустить эксперимент.
- Чтобы избежать чрезмерной выборки, установите размер шага, по крайней мере, в два раза превышающий радиус контакта (
- После завершения матричного сканирования поднимите зонд на 200-500 мкм и переместите зонд в другую область образца, достаточно удаленную от первой области.
- Повторите эксперимент по меньшей мере два раза, чтобы по каждому образцу было получено достаточно данных (т.е. не менее двух матричных сканирований на образец, содержащих по 50-100 кривых каждая).
- Наноиндентация клеток
- Загрузите образец на микроскоп, как описано выше.
- Для одноэлементного отступа сфокусируйте z-плоскость так, чтобы и ячейки, и зонд были видны при 20-кратном или 40-кратном увеличении, в зависимости от размера ячейки и распространения.
- Переместите зонд над ячейкой с отступом.
- Настройте новый эксперимент в главном окне программного обеспечения. Нажмите настроить эксперимент, после чего откроется новое окно. Добавьте шаг Найти поверхность и отступ с параметрами по умолчанию в режиме смещения.
- Нажмите «Запустить эксперимент», который найдет поверхность и выполнит один отступ. Проверьте, был ли отступ успешным. Если кривая выглядит не так, как ожидалось, отрегулируйте экспериментальные параметры (см. Рисунок 4 и Обсуждение, устранение неполадок метода).
- Если отступ выполнен успешно, добавьте в эксперимент сканирование матрицы. Следуйте шагам, приведенным для экспериментов с наноиндентированием гидрогелей; настроить сканирование матрицы таким образом, чтобы размер шага позволял сделать отступ небольшой площади ячейки; 25 точек, расположенных на расстоянии 0,5-5 мкм для клеток HEK293T.
- В зависимости от размера ячейки, адаптируйте сканирование матрицы, чтобы наконечник не отступал за пределы ячейки, т. Е. Выполняя другую геометрию карты или исследуя меньшее количество точек.
- Нажмите «Запустить эксперимент» и дождитесь его завершения.
- Как только сканирование матрицы будет завершено, поднимите зонд вне контакта (50 мкм в плоскости z ).
- Переместите зонд над новой ячейкой и повторите процесс (см. Обсуждение, критические шаги в протоколе).
- Очистка зонда и выключение прибора
- Выполните действия, приведенные в Дополнительном протоколе , чтобы очистить зонд и выключить устройство наноиндентирования.
5. Анализ данных
- Загрузка и установка программного обеспечения
- Выполните действия, приведенные в Дополнительном протоколе, чтобы загрузить и установить программное обеспечение для анализа данных28,29.
- Скрининг кривых F-z и получение очищенного набора данных в формате JSON
- Запустите prepare.py из командной строки на лабораторном компьютере, как описано в шагах 2–3.
- Если вы используете компьютер с Windows, удерживайте клавишу Shift, щелкнув правой кнопкой мыши папку NanoPrepare , и выберите команду Открыть окно PowerShell здесь. Введите команду python prepare.py и нажмите клавишу ENTER . На экране появится графический интерфейс пользователя (рисунок S2).
- Если вы используете компьютер MacOS, щелкните правой кнопкой мыши папку NanoPrepare и выберите Новый терминал в папке. Введите команду python3 prepare.py и нажмите клавишу Enter , которая запустит графический интерфейс пользователя (рисунок S2).
- Выберите формат данных O11NEW из раскрывающегося списка. Если данные загружены неправильно, перезапустите графический интерфейс и выберите O11OLD.
ПРИМЕЧАНИЕ: Формат O11NEW работает для данных, полученных с помощью наконечников оптических волоконно-чувствительных наноиндентационных устройств с программным обеспечением версии 3.4.1. Этот формат также будет работать для предыдущих версий программного обеспечения, по крайней мере, тех, которые принадлежат наноиндентам, установленным в 2019-2020 годах. - Нажмите Загрузить папку. Выберите папку, содержащую анализируемые данные — сканирование одной матрицы или сканирование нескольких матриц. Верхний график (необработанные кривые) будет заполнен загруженным набором данных. Чтобы визуализировать конкретную кривую, нажмите на нее. Это выделит его зеленым цветом и покажет на нижнем графике (Текущая кривая).
- Очистите набор данных с помощью вкладок, присутствующих в правой части графического интерфейса пользователя, как описано ниже.
- Используйте кнопку Сегмент, чтобы выбрать правильный сегмент для анализа, который является прямым сегментом кривых F-z . Конкретное число зависит от количества сегментов, выбранных в программном обеспечении наноиндентирования при выполнении экспериментов.
- Используйте кнопку Обрезать 50 нм , чтобы обрезать кривые на 50 нм в крайнем левом углу (если отмечен галочкой L ), вправо (если отмечен галочкой R) или с обеих сторон (если отмечены галочки R и L). Нажмите эту кнопку несколько раз, чтобы обрезать столько, сколько требуется. Используйте его для удаления артефактов, присутствующих в начале/конце кривых F-z .
- Проверьте вкладку Cantilever на наличие постоянной пружины, геометрии наконечника и радиуса наконечника. Проверьте вкладку, чтобы убедиться, что метаданные прочитаны правильно.
- Вкладка Скрининг используется для установки порогового значения силы, которое отбрасывает все кривые, не достигающие заданной силы. Отброшенные кривые будут выделены красным цветом.
- Используйте переключатель вручную, чтобы вручную удалить неправильно полученные кривые. Удалите все кривые, нажав на конкретную кривую и выбрав OUT, который выделит кривую красным цветом.
- Нажмите сохранить JSON. Введите соответствующее имя для очищенного набора данных, который представляет собой один JSON-файл. Отправьте JSON-файл на компьютер, на котором было установлено программное обеспечение NanoAnalysis.
6. Формальный анализ данных
- Запустите файл nano.py из командной строки, перейдя в папку NanoAnalysis и запустив терминал, как объяснялось ранее. Введите команду python nano.py или python3 nano.py (в зависимости от операционной системы) и нажмите клавишу Enter . На экране появится графический интерфейс пользователя (рисунок S3).
- В левом верхнем углу графического интерфейса нажмите Загрузить эксперимент и выберите файл JSON. Это заполнит список файлов и график необработанных кривых , показывающий набор данных в терминах кривых F-z . Оси F и z отображаются относительно координат CP, которые вычисляются в фоновом режиме при загрузке набора данных (см. следующее примечание). В поле Статистика установите значения трех параметров: Nактивировано, N failed и Nисключено.
ПРИМЕЧАНИЕ:Активированное N представляет собой число кривых, которые будут проанализированы в последующем анализе спектров Герца/упругости и появятся черным цветом на графике raw Curves . Nfailed представляет собой количество кривых, на которых не удалось найти надежный CP и которые отображаются синим цветом на графике. Эти кривые будут автоматически отбрасываться при последующем анализе. При открытии программного обеспечения некоторые кривые могут быть автоматически перемещены в неисправный набор. Это связано с тем, что CP рассчитывается при открытии программного обеспечения с алгоритмом Threshold по умолчанию (см. Ниже). Nисключено представляет кривые, которые вручную выбраны для исключения из анализа и будут отображаться красным цветом на графике (см. ниже). - Проверьте, является ли количество неисправных, исключенных и активированных кривых разумным. Проверьте график необработанных кривых , чтобы визуализировать кривые.
- Чтобы визуализировать конкретную кривую более подробно, нажмите на нее. Это выделит его зеленым цветом и покажет на графике Текущей кривой . После выбора одной кривой параметры R и k (которые должны быть одинаковыми для всех кривых) будут заполнены в поле Статистика графического интерфейса.
- Измените состояние заданной кривой с помощью поля Переключить . Щелкните конкретную кривую, состояние которой вы хотите изменить, а затем выберите Активировано, Не удалось или Исключено. Счетчик в поле Статистика обновляется автоматически.
ПРИМЕЧАНИЕ: Чтобы изменить представление набора данных на графике необработанных кривых, используйте поле Вид. Нажмите «Все», чтобы отобразить все кривые (т. Е. Активированные, неудачные и исключенные в соответствующих цветах). Нажмите «Не удалось», чтобы отобразить активированные и неудачные кривые, и нажмите «Активировано», чтобы отобразить только активированные кривые. Чтобы сбросить состояние всех кривых между активированным и исключенным, нажмите «Активировано» или «Исключено» в поле «Сброс». - После дальнейшей очистки набора данных следуйте описанному ниже конвейеру анализа данных.
- Фильтруйте любые шумы, присутствующие в кривых, используя фильтры, реализованные в графическом интерфейсе (Filtering Box), а именно пользовательский фильтр, называемый фильтром Prominency, фильтр Savitzky Golay 30,31 (SAVGOL) и фильтр сглаживания, основанный на вычислении медианы данных в данном окне (медианный фильтр). Дополнительные сведения о фильтрах см. в разделе Обсуждение (критические шаги в протоколе).
- Проверьте отфильтрованные кривые на графике «Текущая кривая ». Отфильтрованная кривая показана черным цветом, тогда как нефильтрованная версия кривой показана зеленым.
ПРИМЕЧАНИЕ: Рекомендуется фильтровать данные как можно меньше, чтобы сохранить особенности исходного сигнала. Чрезмерная фильтрация может сгладить любые различия, присутствующие в данных. Работы с активированным фильтром проминантности достаточно для герцового анализа данных наноиндентации. Если данные особенно шумные, то дополнительно могут быть применены фильтры SAVGOL или медианы. - Выберите алгоритм, чтобы найти CP. В поле Точка контакта выберите одну из ряда числовых процедур, реализованных в программном обеспечении, а именно: Goodness of Fit (GoF)32, Ratio of Variances (RoV)32, Second derivative33 или Threshold33. Подробности об алгоритмах см. в разделе Обсуждение, критические шаги в протоколе.
ПРИМЕЧАНИЕ: CP - это точка, в которой зонд вступает в контакт с материалом, и его необходимо идентифицировать для преобразования данных F-z в данные F-δ (для мягких материалов δ конечна и должна быть рассчитана). Выбранный алгоритм будет применен ко всем активным кривым набора данных, а кривые, на которых алгоритм не сможет надежно найти CP, будут перемещены в неисправное множество. - Настройте параметры алгоритма в соответствии с вашим набором данных, чтобы CP был расположен правильно, как описано в Обсуждении, критических шагах в протоколе. Чтобы увидеть, где CP был найден на одной кривой, выберите кривую, нажав на нее, и нажмите «Проверить». Проверьте всплывающее окно, которое показывает, где находится CP.
- Проверьте, имеет ли появляющаяся красная линия, которая является параметром, вычисленным алгоритмом в выбранной интересующей области, максимальное или минимальное значение, соответствующее местоположению CP (например, для GoF параметром является R2). При необходимости повторите этот процесс для всех кривых.
ПРИМЕЧАНИЕ: Оси всплывающего окна находятся в абсолютных координатах, так что местоположение CP может быть показано. И наоборот, оси графика Raw Curves и Current Curve показаны относительно CP, т.е. местоположение CP равно (0,0). - Нажмите на Анализ Герца. Это приведет к созданию трех графиков, описанных ниже.
- Проверьте отдельные кривые F-δ в наборе данных вместе со средним соответствием Герца (красная пунктирная линия). Отрегулируйте отступ в нм, до которого установлена модель Hertz, в поле Результаты в разделе Максимальное отступ (нм). Установите максимум ~10% от R , чтобы модель Герца была действительной (см. Обсуждение, критические шаги в протоколе).
- Проверьте среднюю кривую F-δ с полосой ошибок, показывающей одно стандартное отклонение (SD) вместе со средним соответствием Герца (красная пунктирная линия). Визуализируйте среднее соответствие Герца на графике необработанных кривых для справки и соответствие Герца для каждой кривой на текущей кривой.
- Проверьте диаграмму рассеяния E , полученную в результате подгонки модели Герца к каждой отдельной кривой.
- Щелкните имя файла, кривую F-z , кривую F-δ или точку на точечной диаграмме, чтобы выделить кривую на каждом графике. Если точка данных на точечной диаграмме находится за пределами распределения данных, щелкните по ней и проверьте кривую, к которой она принадлежит. Убедитесь, что CP расположен правильно, нажав кнопку Проверить . При необходимости исключите кривую из анализа.
- Проверьте поле Результаты для вычисляемого среднего значения E и его SD (Eγ ± σ) и убедитесь, что они разумны для данного эксперимента.
- В поле Сохранить нажмите на Hertz. Во всплывающем окне введите имя файла и каталог. После этого нажмите «Сохранить». Будет создан файл .tsv. Откройте файл .tsv в любом дополнительном программном обеспечении и используйте значения для статистического анализа и дальнейшего построения графиков.
ПРИМЕЧАНИЕ: Файл содержит E, полученный из каждой кривой, а также среднее значение E и его SD. Кроме того, файл содержит метаданные, связанные с анализом, включая количество анализируемых кривых, R, k и максимальный отступ, используемый для модели Герца. - Этот шаг является необязательным. Нажмите на Average F-Ind , чтобы экспортировать среднюю силу и среднее отступ вместе с одним SD в силе.
- Для получения данных о наноиндентации клеток нажмите « Анализ спектров упругости» (см. Репрезентативные результаты и обсуждение). Осмотрите два полученных графика, а именно: E как функцию глубины отступа (E(δ)) для каждой кривой и средний E(δ) с полосой ошибок, показывающей один SD (сплошная красная линия и затененная область), установленную моделью (черная пунктирная линия), которая позволяет оценить модуль Юнга актиновой коры клетки, модуль Юнга объемной массы клетки, и толщину актиновой коры. Кроме того, проверьте среднее значение E(δ) на верхнем графике красным цветом.
- Убедитесь, что установлен флажок Интерполировать , что гарантирует, что производная, необходимая для выполнения анализа спектров упругости, вычисляется на интерполированном сигнале (см. Репрезентативные результаты).
- Осмотрите поле «Результаты », сообщив о модуле Юнга коры (E0 ± σ), модуле Юнга (Eb ± σ) и толщине коры (d0 ± σ).
ПРИМЕЧАНИЕ: Спектры средней упругости могут сначала казаться шумными, с заметными синусоидальными колебаниями. В результате уравнение (3) может быть установлено неправильно. В этом случае итеративное увеличение длины окна сглаживающего фильтра SAVGOL34 решает эту проблему. - Как только анализ будет завершен, нажмите на ES в поле Сохранить . Это приведет к экспорту файла .tsv в указанном каталоге, содержащего среднюю эластичность в зависимости от средней глубины отступа и радиуса контакта, метаданные, связанные с экспериментом (см. выше), и предполагаемые параметры модели, описанные выше. Наконец, также сообщается о средней эластичности, не учитывающей зависимость от δ и его SD.
- Закройте программное обеспечение и введите сохраненные результаты в любое другое предпочтительное программное обеспечение для дальнейшего построения данных и выполнения статистического анализа.
- Этот шаг является необязательным: экспортируйте графики из графического интерфейса, щелкнув правой кнопкой мыши на графике и выбрав Экспорт. Экспортируйте график в .svg, чтобы такие параметры, как шрифт, размер шрифта, стиль линии и т. Д. может быть отредактирован в другом программном обеспечении по выбору.
ПРИМЕЧАНИЕ: Пользовательские алгоритмы и фильтры CP могут быть запрограммированы и добавлены к уже существующим. Подробную информацию см. в Дополнительном примечании 1 .
Результаты
По протоколу получается набор кривых F-z . Набор данных, скорее всего, будет содержать хорошие кривые и кривые, которые должны быть отброшены, прежде чем продолжить анализ. В общем, кривые должны быть отброшены, если их форма отличается от той, что показана на рисунке 4A...
Обсуждение
Этот протокол показывает, как надежно получать данные наноиндентации силовой спектроскопии с использованием коммерчески доступного наноиндентера с наконечником как на гидрогелях, так и на отдельных клетках. Кроме того, приведены инструкции по использованию программного обеспечения...
Раскрытие информации
Авторам нечего раскрывать.
Благодарности
GC и MAGO признают всех членов CeMi. MSS признает поддержку через грант программы EPSRC (EP/P001114/1).
GC: программное обеспечение (вклад в разработку программного обеспечения и алгоритмов), формальный анализ (анализ данных наноиндентации), валидация, исследование (эксперименты по наноиндентации на полиакриламидных гелях), курирование данных, написание (оригинальный проект, обзор и редактирование), визуализация (рисунки и графики). MAGO: исследование (подготовка гелей и образцов клеток, эксперименты по наноиндентации на клетках), написание (оригинальный черновик, обзор и редактирование), визуализация (рисунки и графики). NA: валидация, написание (рецензирование и редактирование). IL: программное обеспечение (вклад в разработку программного обеспечения и алгоритмов), валидация, написание (рецензирование и редактирование); MV: концептуализация, программное обеспечение (проектирование и разработка оригинального программного обеспечения и алгоритмов), валидация, ресурсы, написание (оригинальный проект, рецензирование и редактирование), надзор, администрирование проекта, приобретение финансирования MSS: ресурсы, написание (обзор и редактирование), надзор, администрирование проекта, приобретение финансирования. Все авторы прочитали и утвердили окончательную рукопись.
Материалы
| Name | Company | Catalog Number | Comments |
| 12 mm coverslips | VWR | 631-1577P | |
| 35 mm cell treated culture dishes | Greiner CELLSTAR | 627160 | |
| Acrylamide | Sigma-Aldrich | A4058 | |
| Acrylsilane | Alfa Aesar | L16400 | |
| Ammonium Persulfate | Merk | 7727-54-0 | |
| Bisacrylamide | Merk | 110-26-9 | |
| Chiaro nanoindenter | Optics 11 Life | no catologue number | |
| Ethanol | general | ||
| Fetal bovine serum | Gibco | 16140071 | |
| High glucose DMEM | Gibco | 11995065 | |
| Isopropanol | general | ||
| Kimwipe | Kimberly Clark | 21905-026 | |
| Microscope glass slides | VWR | 631-1550P | |
| MilliQ system | Merk Millipore | ZR0Q008WW | |
| OP1550 Interferometer | Optics11 Life | no catalogue number | |
| Optics 11 Life probe (k = 0.02-0.005 N/m, R = 3-3.5 um) | Optics 11 Life | no catologue number | |
| Optics 11 Life probe (k = 0.46-0.5 N/m, R = 50-55 um) | Optics 11 Life | no catologue number | |
| Penicillin/Streptomycin | Gibco | 15140122 | |
| RainX rain repellent | RainX | 26012 | |
| Standard petri dishes (90 mm) | Thermo Scientific | 101RTIRR | |
| Tetramethylethylenediamine | Sigma-Aldrich | 110-18-9 | |
| Vaccum dessicator | Thermo Scientific | 531-0250 | |
| Software | |||
| Data acquisition software (v 3.4.1) | Optics 11 Life | ||
| GitHub Desktop (Optional) | Microsoft | ||
| Python 3 | Python Software Foundation | ||
| Visual Studio Code (Optional) | Microsoft |
Ссылки
- Discher, D. E., Janmey, P. Y. W. Tissue cells feel and respond to the stiffness of their substrate. Science. 310 (5751), 1139-1143 (2005).
- Roca-Cusachs, P., Conte, V., Trepat, X. Quantifying forces in cell biology. Nature Cell Biology. 19 (7), 742-751 (2017).
- Guimarães, C. F., Gasperini, L., Marques, A. P., Reis, R. L. The stiffness of living tissues and its implications for tissue engineering. Nature. Reviews. Materials. 5, 351-370 (2020).
- Moeendarbary, E., Harris, A. R. Cell mechanics: Principles, practices, and prospects. Wiley Interdisciplinary. Reviews. Systems. Biology and Medicine. 6 (5), 371-388 (2014).
- Butcher, D. T., Alliston, T., Weaver, V. M. A tense situation: Forcing tumour progression. Nature Reviews Cancer. 9 (2), 108-122 (2009).
- Kubánková, M., et al. Physical phenotype of blood cells is altered in COVID-19. Biophysical Journal. 120 (14), 2838-2847 (2021).
- Engler, A. J., Sen, S., Sweeney, H. L., Discher, D. E. Matrix elasticity directs stem cell lineage specification. Cell. 126, 677-689 (2006).
- Ciccone, G., et al. What caging force cells feel in 3D hydrogels: A rheological perspective. Advanced Healthcare Materials. 9 (17), 2000517 (2020).
- Wu, P. H., et al. A comparison of methods to assess cell mechanical properties. Nature Methods. 15 (7), 491-498 (2018).
- McKee, C. T., Last, J. A., Russell, P., Murphy, C. J. Indentation versus tensile measurements of young's modulus for soft biological tissues. Tissue Engineering B Reviews. 17 (3), 155-164 (2011).
- Humphrey, J. D., Delange, S. L. . An Introduction to Biomechanics Solids and Fluids, Analysis and Design. , 271-371 (2004).
- Prevedel, R., Diz-Muñoz, A., Ruocco, G., Antonacci, G. Brillouin microscopy: an emerging tool for mechanobiology. Nature Methods. 16 (10), 969-977 (2019).
- Krieg, M., et al. Atomic force microscopy-based mechanobiology. Nature Reviews Physics. 1, 41-57 (2019).
- Norman, M. D. A., Ferreira, S. A., Jowett, G. M., Bozec, L., Gentleman, E. Measuring the elastic modulus of soft culture surfaces and three-dimensional hydrogels using atomic force microscopy. Nature Protocols. 16 (5), 2418-2449 (2021).
- Gavara, N. A beginner's guide to atomic force microscopy probing for cell mechanics. Microscopy Research and Technique. 80 (1), 75-84 (2017).
- Whitehead, A. J., Kirkland, N. J., Engler, A. J. Atomic force microscopy for live-cell and hydrogel measurement. Methods in Molecular Biology. 2299, 217-226 (2021).
- Chavan, D., et al. Ferrule-top nanoindenter: An optomechanical fiber sensor for nanoindentation. The Review of Scientific Instruments. 83 (11), 115110 (2012).
- Van Hoorn, H., Kurniawan, N. A., Koenderink Ac, G. H., Iannuzzi, D. Local dynamic mechanical analysis for heterogeneous soft matter using ferrule-top indentation. Soft Matter. 12 (12), 3066-3073 (2016).
- Baldini, F., et al. Biomechanics of cultured hepatic cells during different steatogenic hits. Journal of the Mechanical Behavior of Biomedical Materials. 97, 296-305 (2019).
- Emig, R., et al. Piezo1 channels contribute to the regulation of human atrial fibroblast mechanical properties and matrix stiffness sensing. Cells. 10 (3), 663 (2021).
- Dobre, O., et al. A hydrogel platform that incorporates laminin isoforms for efficient presentation of growth factors - Neural growth and osteogenesis. Advanced Functional Materials. 31 (21), 2010225 (2021).
- Antonovaite, N., Beekmans, S. V., Hol, E. M., Wadman, W. J., Iannuzzi, D. Regional variations in stiffness in live mouse brain tissue determined by depth-controlled indentation mapping. Scientific Reports. 8 (1), 12517 (2018).
- Wu, G., Gotthardt, M., Gollasch, M. Assessment of nanoindentation in stiffness measurement of soft biomaterials: kidney, liver, spleen and uterus. Scientific Reports. 10 (1), 18784 (2020).
- Lüchtefeld, I., et al. Elasticity spectra as a tool to investigate actin cortex mechanics. Journal of Nanobiotechnology. 18 (1), 147 (2020).
- Caliari, S. R., Burdick, J. A. A practical guide to hydrogels for cell culture. Nature Methods. 13 (5), 405-414 (2016).
- Blumlein, A., Williams, N., McManus, J. J. The mechanical properties of individual cell spheroids. Scientific Reports. 7 (1), 7346 (2017).
- Mirsandi, H., et al. Influence of wetting conditions on bubble formation from a submerged orifice. Experiments in Fluids. 61, 83 (2020).
- Vassalli, M., Ciccone, G. . CellMechLab/NanoPrepare: v0.1.1. Zendo. , (2021).
- Vassalli, M., Ciccone, G., Lüchtefeld, I. CellMechLab/nanoindentation: v1.0.0. Zendo. , (2021).
- Savitzky, A., Golay, M. J. E. Smoothing and differentiation of data by simplified least squares procedures. Analytical Chemistry. 36 (8), 1627-1639 (1964).
- Schafer, R. W. What is a savitzky-golay filter. IEEE Signal Processing Magazine. 28 (4), 111-117 (2011).
- Gavara, N. Combined strategies for optimal detection of the contact point in AFM force-indentation curves obtained on thin samples and adherent cells. Scientific Reports. 6, 21267 (2016).
- Lin, D. C., Dimitriadis, E. K., Horkay, F. Robust strategies for automated AFM force curve analysis-I. Non-adhesive indentation of soft, inhomogeneous materials. Journal of Biomechanical Engineering. 129 (3), 430-440 (2007).
- Virtanen, P., et al. SciPy 1.0: fundamental algorithms for scientific computing in Python. Nature Methods. 17 (3), 261-272 (2020).
- Tse, J. R., Engler, A. J. Preparation of hydrogel substrates with tunable mechanical properties. Current Protocols in Cell Biology. , (2010).
- Pharr, G. M., Oliver, W. C., Brotzen, F. R. On the generality of the relationship among contact stiffness, contact area, and elastic modulus during indentation. Journal of Materials Research. 7 (3), 613-617 (1992).
- Kumar, R., Saha, S., Sinha, B. Cell spread area and traction forces determine myosin-II-based cortex thickness regulation. Biochimica et Biophysica Acta - Molecular Cell Research. 1866 (12), 118516 (2019).
- Kunda, P., Pelling, A. E., Liu, T., Baum, B. Moesin controls cortical rigidity, cell rounding, and spindle morphogenesis during mitosis. Current Biology: CB. 18 (2), 91-101 (2008).
- Allen, M., Poggiali, D., Whitaker, K., Marshall, T. R., Kievit, R. A. Raincloud plots: a multi-platform tool for robust data visualization. Wellcome Open Research. 4, 63 (2019).
- Clark, A. G., Paluch, E. Mechanics and regulation of cell shape during the cell cycle. Results and Problems in Cell Differentiation. 53, 31-73 (2011).
- Langan, T. J., Rodgers, K. R., Chou, R. C. Synchronization of mammalian cell cultures by serum deprivation. Methods in Molecular Biology. 1524, 97-105 (2017).
- Hodgkinson, T., et al. The use of nanovibration to discover specific and potent bioactive metabolites that stimulate osteogenic differentiation in mesenchymal stem cells. Science Advances. 7 (9), 7921 (2021).
- Kloxin, A. M., Kloxin, C. J., Bowman, C. N., Anseth, K. S. Mechanical properties of cellularly responsive hydrogels and their experimental determination. Advanced Materials (Deerfield Beach, Fla). 22 (31), 3484-3494 (2010).
- Loessner, D., et al. Functionalization, preparation and use of cell-laden gelatin methacryloyl-based hydrogels as modular tissue culture platforms. Nature Protocols. 11 (4), 727-746 (2016).
- Yue, K., et al. Synthesis, properties, and biomedical applications of gelatin methacryloyl (GelMA) hydrogels. Biomaterials. 73, 254-271 (2015).
- Sarrigiannidis, S. O., et al. A tough act to follow: collagen hydrogel modifications to improve mechanical and growth factor loading capabilities. Materials Today. Bio. 10, 100098 (2021).
- Karoutas, A., et al. The NSL complex maintains nuclear architecture stability via lamin A/C acetylation. Nature Cell Biology. 21 (10), 1248-1260 (2019).
- Ryu, H., et al. Transparent, compliant 3D mesostructures for precise evaluation of mechanical characteristics of organoids. Advanced Materials (Deerfield Beach, Fla). 33 (25), 2100026 (2021).
- Carvalho, D. T. O., Feijão, T., Neves, M. I., Da Silva, R. M. P., Barrias, C. C. Directed self-assembly of spheroids into modular vascular beds for engineering large tissue constructs Biofabrication Directed self-assembly of spheroids into modular vascular beds for engineering large tissue constructs. Biofabrication. 13 (3), 035008 (2021).
- Chaudhuri, O., Cooper-White, J., Janmey, P. A., Mooney, D. J., Shenoy, V. B. Effects of extracellular matrix viscoelasticity on cellular behaviour. Nature. 584 (7822), 535-546 (2020).
- Cantini, M., Donnelly, H., Dalby, M. J., Salmeron-Sanchez, M. The plot thickens: The emerging role of matrix viscosity in cell mechanotransduction. Advanced Healthcare Materials. 9 (8), 1901259 (2020).
- Elosegui-Artola, A. The extracellular matrix viscoelasticity as a regulator of cell and tissue dynamics. Current Opinion in Cell Biology. 72, 10-18 (2021).
- Chaudhuri, O. Viscoelastic hydrogels for 3D cell culture. Biomaterials Science. 5 (8), 1480-1490 (2017).
- Nguyen, T. D., Gu, Y. Determination of strain-rate-dependent mechanical behavior of living and fixed osteocytes and chondrocytes using atomic force microscopy and inverse finite element analysis. Journal of Biomechanical Engineering. 136 (10), 101004 (2014).
- Mokbel, M., Hosseini, K., Aland, S., Fischer-Friedrich, E. The Poisson Ratio of the Cellular Actin Cortex Is Frequency Dependent. Biophysical Journal. 118 (8), 1968-1976 (2020).
- Javanmardi, Y., Colin-York, H., Szita, N., Fritzsche, M., Moeendarbary, E. Quantifying cell-generated forces: Poisson's ratio matters. Communications Physics. 4, 237 (2021).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены