É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Fluxo de trabalho experimental e de análise de dados para nanoindentação de matéria mole
Neste Artigo
Resumo
O protocolo apresenta um fluxo de trabalho completo para experimentos de nanoindentação de materiais moles, incluindo hidrogéis e células. Primeiro, as etapas experimentais para adquirir dados de espectroscopia de força são detalhadas; em seguida, a análise de tais dados é detalhada através de um software Python de código aberto recém-desenvolvido, que é gratuito para download no GitHub.
Resumo
Nanoindentação refere-se a uma classe de técnicas experimentais em que uma sonda de força micrométrica é usada para quantificar as propriedades mecânicas locais de biomateriais e células moles. Esta abordagem ganhou um papel central nos campos da mecanobiologia, design de biomateriais e engenharia de tecidos, para obter uma caracterização mecânica adequada de materiais moles com uma resolução comparável ao tamanho de células individuais (μm). A estratégia mais popular para adquirir tais dados experimentais é empregar um microscópio de força atômica (AFM); enquanto este instrumento oferece uma resolução sem precedentes em força (até pN) e espaço (sub-nm), sua usabilidade é muitas vezes limitada por sua complexidade que impede medições rotineiras de indicadores integrais de propriedades mecânicas, como o Módulo de Young (E). Uma nova geração de nanoindenters, como aqueles baseados na tecnologia de detecção de fibra óptica, ganhou recentemente popularidade por sua facilidade de integração, permitindo aplicar forças sub-nN com resolução espacial μm, portanto, sendo adequado para sondar propriedades mecânicas locais de hidrogéis e células.
Neste protocolo, um guia passo-a-passo detalhando o procedimento experimental para adquirir dados de nanoindentação em hidrogéis e células usando um nanoindenter de detecção de fibra óptica ferrule-top comercialmente disponível é apresentado. Considerando que algumas etapas são específicas para o instrumento aqui utilizado, o protocolo proposto pode ser tomado como um guia para outros dispositivos de nanoindentação, desde que algumas etapas sejam adaptadas de acordo com as diretrizes do fabricante. Além disso, um novo software Python de código aberto equipado com uma interface gráfica de usuário amigável para a análise de dados de nanoindentação é apresentado, o que permite a triagem de curvas incorretamente adquiridas, filtragem de dados, computação do ponto de contato através de diferentes procedimentos numéricos, a computação convencional de E, bem como uma análise mais avançada particularmente adequada para dados de nanoindentação de célula única.
Introdução
O papel fundamental da mecânica na biologia está hoje estabelecido 1,2. De tecidos inteiros a células únicas, as propriedades mecânicas podem informar sobre o estado fisiopatológico do biomaterial investigado 3,4. Por exemplo, o tecido mamário afetado pelo câncer é mais rígido do que o tecido saudável, conceito que é a base do popular teste de palpação5. Notavelmente, foi demonstrado recentemente que a doença do coronavírus 2019 (COVID-19) causada pelo coronavírus 2 da síndrome respiratória aguda grave (SARS-CoV-2) é sublinhada por alterações nas propriedades mecânicas das células sanguíneas, incluindo diminuição da deformabilidade dos eritrócitos e diminuição da rigidez dos linfócitos e neutrófilos em comparação com as células sanguíneas de indivíduos ingênuos SARS-CoV-26.
Em geral, a mecânica das células e dos tecidos está inerentemente entrelaçada: cada tecido possui propriedades mecânicas específicas que influenciam e dependem simultaneamente das células constituintes e da matriz extracelular (MEC)5. Devido a isso, as estratégias para estudar a mecânica em biologia geralmente envolvem substratos de engenharia com estímulos mecânicos fisiologicamente relevantes para elucidar o comportamento celular em resposta a esses estímulos. Por exemplo, o trabalho seminal de Engler e colegas demonstrou que o comprometimento da linhagem de células-tronco mesenquimais é controlado pela elasticidade da matriz, conforme estudado em hidrogéis de poliacrilamida bidimensional (PAAm) macios e rígidos7.
Existem muitas estratégias para caracterizar mecanicamente o biomaterial investigado, variando em escala espacial (ou seja, local a granel) e no modo de deformação (por exemplo, axial vs cisalhamento), consequentemente produzindo diferentes informações, o que necessita de interpretação cuidadosa 3,8,9,10. A mecânica dos biomateriais moles é comumente expressa em termos de rigidez. No entanto, a rigidez depende tanto das propriedades do material quanto da geometria, enquanto os módulos elásticos são propriedades fundamentais de um material e são independentes da geometria do material11. Como tal, diferentes módulos elásticos estão relacionados à rigidez de uma determinada amostra, e cada módulo elástico engloba a resistência do material a um modo específico de deformação (por exemplo, axial versus cisalhamento) sob diferentes condições de contorno (por exemplo, expansão livre vs confinamento)11,12. Experimentos de nanoindentação permitem a quantificação de propriedades mecânicas através do E que está associado à deformação uniaxial (recuo) quando o biomaterial não está confinado lateralmente10,11,12.
O método mais popular para quantificar E de sistemas biológicos em microescala é o AFM13,14,15,16. O AFM é uma ferramenta extremamente poderosa com resolução de força até o nível pN e resolução espacial até a escala sub-nm. Além disso, o AFM oferece extrema flexibilidade em termos de acoplamento com ferramentas ópticas e mecânicas complementares, ampliando suas capacidades de extrair uma riqueza de informações do biomaterial investigado13. Essas características atraentes, no entanto, vêm com uma barreira de entrada representada pela complexidade da configuração experimental. O AFM requer treinamento extensivo antes que os usuários possam adquirir dados robustos, e seu uso para caracterização mecânica diária de materiais biológicos é muitas vezes injustificado, especialmente quando sua força única e resoluções espaciais não são necessárias.
Devido a isso, uma nova classe de nanoindenters ganhou popularidade recentemente devido à sua facilidade de uso, enquanto ainda oferece dados comparáveis ao AFM com resolução de força sub-nN e resolução espacial μm, refletindo forças exercidas e percebidas pelas células em escalas de comprimento relevantes2. Particularmente, os dispositivos de nanoindentação de topo de ferrone baseados na tecnologia de detecção de fibra óptica 17,18 ganharam popularidade entre os pesquisadores ativos no campo da mecanobiologia e além; e uma riqueza de trabalhos relatando as propriedades mecânicas de biomateriais usando esses dispositivos, incluindo células19,20, hidrogéis8,21 e tecidos 22,23 foram publicados. Apesar das capacidades desses sistemas de sondar propriedades mecânicas dinâmicas locais (isto é, módulo de armazenamento e perda), experimentos quase estáticos que produzem E continuam sendo a escolha mais popular 8,19,20,21. Em resumo, os experimentos de nanoindentação quase estática consistem em recuar a amostra com uma velocidade constante até um ponto de ajuste definido por um deslocamento, força ou profundidade máxima de recuo e registrar tanto a força quanto a posição vertical do cantilever nas chamadas curvas força-distância (F-z). As curvas F-z são então convertidas em curvas de indentação de força (F-δ) através da identificação do ponto de contato (CP) e equipadas com um modelo de mecânica de contato apropriado (geralmente o modelo de Hertz13) para calcular E.
Embora a operação de nanoindenters de topo de ferrrule se assemelhe a medições de AFM, existem especificidades que valem a pena considerar. Neste trabalho, um guia passo-a-passo para adquirir robustamente curvas F-z de células e hidrogéis que imitam tecidos usando um nanoindenter de topo de ferrule comercialmente disponível é fornecido, a fim de incentivar a padronização de procedimentos experimentais entre grupos de pesquisa usando este e outros dispositivos similares. Além disso, conselhos sobre a melhor forma de preparar amostras e células de hidrogel para realizar experimentos de nanoindentação são dados, juntamente com dicas de solução de problemas ao longo do caminho experimental.
Além disso, grande parte da variabilidade nos resultados de nanoindentação (ou seja, E e sua distribuição) depende do procedimento específico usado para analisar os dados, que não é trivial. Para resolver esse problema, instruções para o uso de um software de código aberto recém-desenvolvido programado em Python e equipado com uma interface gráfica de usuário (GUI) amigável para análise em lote de curvas F-z são fornecidas. O software permite a triagem rápida de dados, a filtragem de dados, a computação do CP através de diferentes procedimentos numéricos, a computação convencional de E, bem como uma análise mais avançada chamada espectros de elasticidade24, permitindo estimar o módulo de Young em massa da célula, o módulo de Young do córtex de actina e a espessura do córtex de actina. O software pode ser baixado gratuitamente do GitHub e pode ser facilmente adaptado para analisar dados originários de outros sistemas, adicionando um analisador de dados apropriado. Ressalta-se que este protocolo pode ser utilizado para outros dispositivos de nanoindentação de topo de ferrule, e outros dispositivos de nanoindentação em geral, desde que algumas etapas sejam adaptadas de acordo com as diretrizes específicas do instrumento. O protocolo está resumido esquematicamente na Figura 1.
Protocolo
1. Preparação de substratos/células para medições de nanoindentação
- Siga as etapas indicadas no Protocolo Complementar para a preparação de hidrogéis/células PAAm para experimentos de nanoindentação. O procedimento está resumido na Figura 2.
NOTA: Os hidrogéis PAAm foram escolhidos por serem os hidrogéis mais comuns utilizados no campo da mecanobiologia. No entanto, o protocolo é igualmente aplicável a qualquer tipo de hidrogel25 (ver Discussão, modificações do método).
2. Iniciando o dispositivo, a escolha da sonda e a calibração da sonda
- Siga as etapas fornecidas no Protocolo Suplementar para iniciar o dispositivo. Para obter detalhes técnicos sobre o funcionamento dos nanoindenters de fibra óptica com topo de ferrúcula, consulte estas referências17,18.
- Selecione a sonda de nanoindentação conforme descrito abaixo.
NOTA: Todas as sondas comercialmente disponíveis, como as usadas neste protocolo, são equipadas com uma ponta esférica (Figura 3A). Portanto, a escolha se restringe a duas variáveis: rigidez do balanço e raio da ponta (Figura 3B).- Escolha a rigidez de um balanço (k em N/m) que corresponda à rigidez esperada da amostra para obter melhores resultados15 (consulte a Figura 3C e Discussão, etapas críticas no protocolo). Para células, selecione uma sonda com k no intervalo de 0,01-0,09 N/m. Para hidrogéis, selecione uma sonda com k na faixa de 0,1-0,9 N/m, que produz resultados ótimos para géis com E esperado entre alguns kPa e 100 kPa (ver Resultados Representativos).
- Escolha o raio da ponta (R em μm) de acordo com a resolução espacial desejada do processo de recuo. Para células pequenas, como as células do Rim Embrionário Humano 293T (HEK293T) (diâmetro médio de ~10-15 μm26), selecione uma esfera com R = 3 μm. Para hidrogéis, selecione uma esfera com R = 10-250 μm para sondar as propriedades mecânicas do biomaterial em uma grande área de contato e evitar heterogeneidades locais.
- Consulte a Figura 3C e quaisquer diretrizes adicionais do fabricante para selecionar a sonda apropriada.
- Uma vez que a sonda tenha sido selecionada, siga as etapas no Protocolo Suplementar para montá-la no nanoindenter.
3. Calibração da sonda
NOTA: As etapas a seguir são específicas para dispositivos de nanoindentação de topo de ferrule baseados na tecnologia de detecção de fibra óptica e são detalhadas para a versão de software 3.4.1. Para outros dispositivos de nanoindentação, siga as etapas recomendadas pelo fabricante do dispositivo.
- Na janela principal do software, clique em Inicializar. Um menu de calibração será exibido. Insira os detalhes da sonda (podem ser encontrados na lateral da caixa da sonda; k em N/m, R em μm e o fator de calibração no ar) nas caixas de entrada.
- Prepare a placa de calibração: uma placa de Petri de vidro grossa com um fundo plano (consulte Discussão, etapas críticas no protocolo). Encha o prato com o mesmo meio que o prato de amostra (isso também pode ser ar). Combinar a temperatura do meio com a da amostra.
- Coloque a placa de calibração sob a sonda. Se necessário, deslize a sonda do suporte do braço do nanoindenter e segure-a com uma mão para abrir espaço para a colocação do prato de calibração. Deslize a sonda de volta para o lugar.
- Execute as próximas duas etapas para calibração em líquido. Se estiver medindo no ar, use o substrato de politetrafluoroetileno fornecido para calibração e pule para a etapa 5.
- Pré-molhou a sonda com uma gota de etanol a 70% usando uma pipeta Pasteur com a extremidade da pipeta em contato leve com a ferrule de vidro, para que a gota deslize sobre o cantilever e a ponta esférica27 (ver Discussão, etapas críticas no protocolo).
- Deslize manualmente o braço do nanoindenter para baixo até que a sonda esteja totalmente submersa, mas ainda longe do fundo da placa de Petri. Se necessário, adicione mais meio ao prato de calibração. Aguarde 5 min para que as condições de equilíbrio sejam alcançadas no líquido.
- No menu Inicializar do software, clique em Scan Wavelength. A tela do interferômetro mostrará uma barra de progresso e a janela Live Signal no software do computador exibirá o padrão mostrado na Figura S1A, à esquerda. Para verificar se a varredura óptica foi bem-sucedida, navegue até o painel Varredura de comprimento de onda na caixa do interferômetro. Se for bem-sucedida, uma onda senoidal deve ser visível (Figura 1A, à direita). Consulte Discussão (solução de problemas do método) se um erro for exibido.
- No menu Inicializar , clique em Localizar superfície, que abaixará progressivamente a sonda até que um limite definido na flexão do balanço seja atingido. A sonda para de se mover quando o contato com a placa de Petri de vidro é feito.
- Verifique se a sonda está em contato com a superfície. Mova a sonda para baixo em 1 μm usando o botão de seta y para baixo na janela principal do software. Observe o sinal verde (deflexão do balanço) na Janela ao vivo do software para mudanças na linha de base a cada passo, quando o cantilever estiver em contato com o substrato (Figura S1B). Se não houver alteração, o cantilever não está em contato (veja a próxima etapa).
- Aumente o valor Limite no menu Opções na guia Localizar Superfície de seu valor padrão de 0,01 por uma etapa de 0,01 de cada vez e repita a etapa Localizar Superfície até entrar em contato. Alternativamente, desloque a sonda para entrar em contato em pequenos passos de 1 μm até que a linha de base verde comece a se deslocar a cada passo para baixo.
NOTA: Para os balanços mais macios (k = 0,025 Nm-1), aumente o valor do Limiar no menu Opções no separador Localizar Superfície a priori da execução do passo 6. Comece a partir de um valor limite de 0,06 ou 0,07 e aumente-o até 0,1, se necessário. Isso ocorre porque o ruído ambiental provavelmente fará com que o cantilever se dobre acima do limiar antes do contato. Para sondas macias, diminuir a velocidade de aproximação (μm/s) no mesmo menu também melhora o procedimento. - No menu Inicializar , clique em Calibrar.
- Verifique a janela Live Signal do software e certifique-se de que os sinais de deslocamento do piezo e de deflexão do balanço subam ao mesmo tempo (Figura S1C).
- Se houver uma incompatibilidade no tempo, a sonda não está totalmente em contato com o vidro. Desça a sonda em passos de 1 μm até que a linha de base do sinal do balanço do cantilever mude (ver passo 7) e repita o passo 9.
- Se o sinal do cantilever não mudar durante a etapa de calibração , a sonda estará longe da superfície. Aumente o limiar de contato iterativamente (veja a etapa 8) até que a superfície seja encontrada corretamente e repita a calibração desde o início, começando com a varredura do comprimento de onda.
- Quando a calibração estiver concluída, verifique os fatores de calibração antigos e novos na janela pop-up exibida. Se o novo fator de calibração estiver na faixa correta, clique em Usar Novo Fator. Se a calibração falhar e o novo fator for NaN ou não estiver no intervalo esperado, consulte Discussão (solução de problemas do método) para obter a resolução.
NOTA: O novo fator deve ser ~n vezes menor do que o fornecido na caixa da sonda se a calibração foi realizada em um meio líquido com índice de refração n ( n = 1,33 para água). Se a calibração foi realizada no ar, então os fatores de calibração novos e antigos devem ser aproximadamente iguais. - Verifique se o círculo de demodulação foi calibrado corretamente da seguinte forma. Navegue até a guia Demodulação na área de trabalho do interferômetro. Toque suavemente na mesa óptica ou no nanoindenter para induzir ruído suficiente. Um círculo branco composto de pontos de dados discretos deve cobrir aproximadamente o círculo vermelho (Figura S1D).
- Se o círculo branco não se sobrepor ao círculo vermelho, ou se um aviso aparecer no visor do interferômetro, o círculo de demodulação precisará de recalibração. Isso pode ser feito de duas maneiras, conforme descrito abaixo.
- Toque continuamente no corpo do nanoindenter para induzir um círculo completo de ruído e pressione o botão Calibrar no interferômetro.
- Entre em contato com o substrato de vidro e pressione Calibrar no menu Inicializar da janela principal do software. Não salve o fator de calibração. Neste ponto, verifique novamente e certifique-se de que o círculo branco se sobrepõe ao círculo vermelho.
NOTA: Se o sinal não é apenas ligeiramente deslocado do círculo de demodulação, mas se tornou muito pequeno ou não é visível, isso significa que o cantilever está preso à fibra óptica. Siga os conselhos de solução de problemas para esse problema (consulte Discussão, solução de problemas do método) e repita as etapas 13.1 ou 13.2. Uma vez que o cantilever volte à sua posição horizontal (Figura 3A), o sinal se recuperará para o círculo de demodulação.
- Verifique a calibração realizando um recuo no substrato de vidro diretamente após a calibração, conforme descrito abaixo.
- Carregue ou crie um arquivo de experimento clicando em Configurar Experimento e adicione uma etapa Localizar Superfície e uma etapa Recuo . Para a etapa de recuo, use as configurações padrão do modo de deslocamento e altere o deslocamento máximo para a distância de calibração (3.000 nm) para deslocar a sonda contra o substrato rígido.
- Clique em Executar experimento e verifique o círculo de demodulação na janela do interferômetro. Verifique o sinal branco e certifique-se de que está em cima do círculo vermelho durante o recuo.
- Verifique os resultados na janela principal do software no gráfico de dados de tempo e certifique-se de que o deslocamento do piezo (linha azul) seja igual à deflexão do balanço (linha verde), pois o recuo começa em contato e nenhuma deformação de material é esperada. Se os sinais não forem paralelos, consulte Discussão (solução de problemas do método).
- Altere o caminho local no menu de calibração definindo o Caminho de salvamento de calibração para um diretório apropriado.
- Quando a sonda tiver sido calibrada com sucesso, mova o piezo para cima em 500 μm.
4. Medindo o módulo de materiais moles do jovem
- Nanoindentação de hidrogéis
- Carregue a placa de Petri que contém a(s) amostra(s) no estágio do microscópio e mova manualmente a sonda do nanoindenter para uma posição x-y desejada acima da amostra.
- Deslize manualmente a sonda em solução, tomando o cuidado de deixar 1-2 mm entre a sonda e a superfície da amostra nesta fase. Aguarde 5 min para que a sonda se equilibre no meio.
- Foque o plano z do microscópio óptico de modo que a sonda seja claramente visível.
- Execute um único recuo para ajustar os parâmetros experimentais, conforme descrito abaixo.
- Configure um novo experimento na janela principal do software. Clique em Configurar experimento, que abrirá uma nova janela. Adicione uma etapa Localizar Surface . Todos os parâmetros da etapa Localizar superfície podem ser alterados no menu Opções do software, se necessário.
NOTA: A superfície de localização abaixará a sonda até que a superfície seja encontrada e, em seguida, retrairá a sonda para uma distância definida por Z acima da superfície (μm), acima da superfície da amostra. Se o cantilever selecionado estiver muito rígido para a amostra ou a amostra for pegajosa, após a etapa a sonda provavelmente ainda estará em contato com a amostra, o que resultará em uma curva sem linha de base (Figura 4C). Para resolver esse problema, aumente o Z acima da superfície (μm). - Adicione uma etapa de recuo . Selecione a guia Perfil e clique em Controle de deslocamento. Deixe o perfil de recuo padrão.
- Clique em Executar experimento na janela principal do software. Isso localizará a superfície e executará um único recuo. Se o recuo único não tiver a aparência esperada, ajuste os parâmetros experimentais conforme descrito na Figura 4 e Discussão (solução de problemas do método).
- Configure um novo experimento na janela principal do software. Clique em Configurar experimento, que abrirá uma nova janela. Adicione uma etapa Localizar Surface . Todos os parâmetros da etapa Localizar superfície podem ser alterados no menu Opções do software, se necessário.
- Quando o recuo parecer desejado, configure a varredura da matriz para que uma área suficiente da amostra seja recuada. Clique em Configurar Experimento, adicione uma etapa Localizar superfície com os parâmetros experimentais determinados anteriormente e adicione uma etapa Matrix Scan .
- Para hidrogéis planos, configure uma varredura de matriz contendo 50-100 pontos (ou seja, 5 x 10 ou 10 x 10 em x e y) espaçados em 10-100 μm (ou seja, dx = dy = 10-100 μm). Clique em Usar posição do estágio para iniciar a varredura da matriz a partir da posição do estágio atual. Certifique-se de que a caixa Localizar Superfície Automaticamente está marcada para localizar a superfície em cada recuo utilizando os parâmetros experimentais definidos.
- Para evitar a amostragem excessiva, defina o tamanho da etapa para pelo menos o dobro do raio de contato (
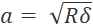 , onde δ é a profundidade de recuo).
, onde δ é a profundidade de recuo). - Configure o perfil de varredura de matriz no controle de deslocamento. Certifique-se de que o perfil não viole as suposições do modelo de Hertz (consulte Discussão, etapas críticas no protocolo).
- Deixe o número de segmentos para 5, que é o valor padrão, e use o perfil de deslocamento padrão. Se necessário, alterar o perfil de deslocamento em termos de deslocamento máximo e tempo para cada segmento inclinado, o que afetará a profundidade máxima de recuo e a taxa de deformação, respectivamente. Não exceda as taxas de deformação > 10 μm/s (ver Discussão, limitações do método).
- Insira um valor para a velocidade de aproximação, que determina a rapidez com que a sonda é deslocada em direção à amostra antes do contato. Combine a velocidade de retração com a velocidade de aproximação (veja a nota abaixo).
NOTA: Para balanços macios e ambientes barulhentos, recomenda-se uma velocidade de aproximação de 1.000-2.000 nm/s. Para balanços mais rígidos e ambientes controlados, isso pode ser aumentado. - Salve o experimento configurado no Caminho do experimento desejado e selecione um diretório onde os dados serão salvos no Caminho de salvamento, na guia Geral da janela Configurar experimento . Clique em Executar experimento.
- Para evitar a amostragem excessiva, defina o tamanho da etapa para pelo menos o dobro do raio de contato (
- Uma vez que a varredura da matriz esteja concluída, levante a sonda em 200-500 μm e mova a sonda para uma área diferente da amostra suficientemente longe da primeira área.
- Repita o experimento pelo menos duas vezes para que dados suficientes sejam adquiridos em cada amostra (ou seja, pelo menos duas varreduras de matriz por amostra, contendo 50-100 curvas cada).
- Nanoindentação de células
- Carregue a amostra no microscópio conforme descrito acima.
- Para recuo de célula única, foque o plano z de modo que as células e a sonda sejam visíveis com ampliação de 20x ou 40x, dependendo do tamanho e da dispersão da célula.
- Mova o teste acima da célula a ser recuada.
- Configure um novo experimento na janela principal do software. Clique em Configurar experimento, que abrirá uma nova janela. Adicione uma etapa Localizar superfície e recuo com parâmetros padrão no modo de deslocamento.
- Clique em Executar experimento, que localizará a superfície e executará um único recuo. Verifique se o recuo foi bem-sucedido. Se a curva não tiver a aparência esperada, ajuste os parâmetros experimentais (consulte a Figura 4 e Discussão, solução de problemas do método).
- Se o recuo foi bem-sucedido, adicione uma varredura de matriz ao experimento. Siga os passos dados para os experimentos de nanoindentação dos hidrogéis; configurar a varredura da matriz para que o tamanho da etapa permita que uma pequena área da célula seja recuada; 25 pontos espaçados em 0,5-5 μm para células HEK293T.
- Dependendo do tamanho da célula, adapte a varredura da matriz para garantir que a ponta não recue fora do limite da célula, ou seja, fazendo uma geometria de mapa diferente ou sondando menos pontos.
- Clique em Executar experimento e aguarde que ele seja concluído.
- Uma vez concluída a varredura da matriz, levante a sonda para fora de contato (50 μm no plano z).
- Mova o teste acima de uma nova célula e repita o processo (consulte Discussão, etapas críticas no protocolo).
- Limpeza da sonda e desligamento do instrumento
- Siga as etapas fornecidas no Protocolo Suplementar para limpar a sonda e desligar o dispositivo de nanoindentação.
5. Análise dos dados
- Baixando e instalando o software
- Siga os passos dados no Protocolo Suplementar para baixar e instalar o software para análise de dados28,29.
- Triagem de curvas F-z e produção de conjunto de dados limpos no formato JSON
- Inicie prepare.py a partir da linha de comando no computador do laboratório, conforme descrito nas etapas 2 a 3.
- Se estiver usando um computador Windows, mantenha pressionada a tecla shift enquanto clica com o botão direito do mouse na pasta NanoPrepare e clique em Abrir Janela do PowerShell Aqui. Digite o comando python prepare.py e pressione a tecla Enter . Uma GUI aparecerá na tela (Figura S2).
- Se estiver usando um computador MacOS, clique com o botão direito do mouse na pasta NanoPrepare e clique em Novo Terminal na Pasta. Digite o comando python3 prepare.py e pressione a tecla Enter , que iniciará a GUI (Figura S2).
- Selecione o formato de dados O11NEW na lista suspensa. Se os dados não forem carregados corretamente, reinicie a GUI e selecione O11OLD.
NOTA: O formato O11NEW funciona para dados obtidos usando dispositivos de nanoindentação de detecção de fibra óptica com ponta de ferrule com a versão de software 3.4.1. Este formato também funcionará para versões anteriores do software, pelo menos aquelas pertencentes a nanoindenters instalados em 2019-2020. - Clique em Carregar pasta. Selecione uma pasta que contenha os dados a serem analisados - varredura de matriz única ou varreduras de matriz múltipla. O gráfico superior (Raw Curves) será preenchido com o conjunto de dados carregado. Para visualizar uma curva específica, clique nela. Isso irá realçá-lo em verde e mostrá-lo no gráfico inferior (Curva Atual).
- Limpe o conjunto de dados usando as guias presentes na parte direita da GUI, conforme descrito abaixo.
- Use o botão Segmento para selecionar o segmento correto a ser analisado, que é o segmento à frente das curvas F-z . O número específico depende do número de segmentos selecionados no software de nanoindentação ao realizar experimentos.
- Use o botão Cortar 50 nm para cortar as curvas em 50 nm na extrema esquerda (se L estiver marcado), à direita (se R estiver marcado) ou em ambos os lados (se R e L estiverem marcados). Clique neste botão várias vezes para cortar o máximo que for necessário. Use isso para remover artefatos presentes no início/fim das curvas F-z.
- Inspecione a guia Cantilever em busca da constante da mola, da geometria da ponta e do raio da ponta. Inspecione a guia para garantir que os metadados tenham sido lidos corretamente.
- Use a guia Triagem para definir um limite de força que descartará todas as curvas que não atingiram a força fornecida. As curvas descartadas serão destacadas em vermelho.
- Use o botão Alternar manual para remover manualmente as curvas que não foram adquiridas corretamente. Remova todas as curvas clicando na curva específica e selecionando OUT, que destacará a curva em vermelho.
- Clique em Salvar JSON. Insira um nome apropriado para o conjunto de dados limpo, que é um único arquivo JSON. Envie o arquivo JSON para o computador onde o software NanoAnalysis foi instalado.
6. Análise formal dos dados
- Inicie o arquivo nano.py a partir da linha de comando navegando até a pasta NanoAnalysis e iniciando um terminal, conforme explicado anteriormente. Digite o comando python nano.py ou python3 nano.py (dependendo do sistema operacional) e pressione a tecla Enter . Uma GUI aparecerá na tela (Figura S3).
- No canto superior esquerdo da GUI, clique em Carregar Experimento e selecione o arquivo JSON. Isso preencherá a lista de arquivos e o gráfico Curvas Brutas mostrando o conjunto de dados em termos de curvas F-z . Os eixos F e z são mostrados em relação às coordenadas CP, que são calculadas em segundo plano ao carregar o conjunto de dados (consulte a nota a seguir). Na caixa Estatísticas , marque os valores dos três parâmetros: Nativado, N falhou e Nexcluído.
NOTA: Nativado representa o número de curvas que serão analisadas na análise subsequente dos espectros de Hertz/elasticidade e aparecem em preto no gráfico Curvas Brutas . Nfalhou representa o número de curvas nas quais um CP confiável não pôde ser encontrado e aparece em azul no gráfico. Essas curvas serão automaticamente descartadas na análise subsequente. Ao abrir o software, algumas curvas podem ser movidas automaticamente para o conjunto com falha. Isso ocorre porque o CP é calculado ao abrir o software com o algoritmo Threshold padrão (veja abaixo). Nexcluído representa as curvas que são selecionadas manualmente para serem excluídas da análise e aparecerão em vermelho no gráfico (veja abaixo). - Verifique se o número de curvas com falha, excluídas e ativadas é razoável. Verifique o gráfico Curvas brutas para visualizar as curvas.
- Para visualizar uma curva específica com mais detalhes, clique nela. Isso irá realçá-lo em verde e mostrá-lo no gráfico Curva Atual . Uma vez que uma única curva tenha sido selecionada, os parâmetros R e k (que devem ser os mesmos para todas as curvas) serão preenchidos na caixa Estatísticas da GUI.
- Altere o status de uma determinada curva usando a caixa Alternar . Clique na curva específica da qual você deseja alterar o status e, em seguida, clique em Ativado, Falhado ou Excluído. A contagem na caixa Estatísticas é atualizada automaticamente.
Observação : para alterar a exibição do conjunto de dados no gráfico de curvas brutas use a caixa Exibir . Clique em Tudo para mostrar todas as curvas (ou seja, ativadas, reprovadas e excluídas nas respectivas cores). Clique em Falha ao mostrar as curvas ativadas e as curvas com falha e clique em Ativado para mostrar apenas as curvas ativadas. Para redefinir o status de todas as curvas entre ativadas e excluídas, clique em Ativado ou Excluído na caixa Redefinir . - Depois que o conjunto de dados tiver sido limpo, siga o pipeline de análise de dados descrito abaixo.
- Filtre qualquer ruído presente nas curvas usando os filtros implementados na GUI (Filtering Box), ou seja, um filtro personalizado chamado filtro de proeminência, o filtro Savitzky Golay 30,31 (SAVGOL) e um filtro de suavização baseado no cálculo da mediana dos dados em uma determinada janela (filtro mediano). Consulte Discussão (etapas críticas no protocolo) para obter detalhes sobre filtros.
- Inspecione as curvas filtradas no gráfico Curva de Corrente . A curva filtrada é mostrada em preto, enquanto a versão não filtrada da curva é mostrada em verde.
NOTA: É aconselhável filtrar os dados o mínimo possível para preservar as características do sinal original. A filtragem excessiva pode suavizar quaisquer diferenças presentes nos dados. Trabalhar com o filtro de proeminência ativado é suficiente para a análise Hertz de dados de nanoindentação. Se os dados forem particularmente ruidosos, um SAVGOL ou filtros medianos podem ser adicionalmente aplicados. - Selecione um algoritmo para encontrar o CP. Na caixa Ponto de Contato, escolha um de uma série de procedimentos numéricos que foram implementados no software, a saber, a Bondade do Ajuste (GoF)32, a Razão de Variâncias (RoV)32, a Segunda derivada33 ou o Limiar 33. Consulte Discussão, etapas críticas no protocolo para obter detalhes sobre os algoritmos.
NOTA: O CP é o ponto em que a sonda entra em contato com o material e precisa ser identificado para converter dados F-z em dados F-δ (para materiais macios, δ é finito e precisa ser calculado). O algoritmo selecionado será aplicado a todas as curvas ativas do conjunto de dados, e as curvas em que o algoritmo não conseguir localizar o CP de forma robusta serão movidas para o conjunto com falha. - Ajuste os parâmetros do algoritmo para se adequar ao seu conjunto de dados para que o CP esteja localizado corretamente, conforme detalhado nas etapas críticas de Discussão do protocolo. Para ver onde o CP foi encontrado em uma única curva, selecione a curva clicando nela e clique em Inspecionar. Verifique a janela pop-up que aparece para identificar onde o CP foi localizado.
- Verifique se a linha vermelha que aparece, que é o parâmetro que o algoritmo calculou na região de interesse selecionada, tem um valor máximo ou mínimo que corresponda à localização do CP (por exemplo, para o GoF, o parâmetro é o R2). Repita esse processo para todas as curvas, se necessário.
NOTA: Os eixos do pop-up estão em coordenadas absolutas para que a localização do CP possa ser mostrada. Por outro lado, os eixos do gráfico Curvas Brutas e Curva Atual são mostrados em relação ao CP, ou seja, a localização do CP é (0,0). - Clique em Análise Hertz. Isso gerará três gráficos descritos abaixo.
- Verifique as curvas F-δ individuais no conjunto de dados juntamente com o ajuste médio de Hertz (linha tracejada vermelha). Ajuste o recuo em nm até o qual o modelo Hertz está instalado na caixa Resultados em Recuo Máximo (nm). Defina-o para um máximo de ~10% de R para que o modelo Hertz seja válido (consulte Discussão, etapas críticas no protocolo).
- Verifique a curva média de δ com a banda de erro mostrando um desvio padrão (DP) juntamente com o ajuste médio de Hertz (linha tracejada vermelha). Visualize o ajuste médio de Hertz no gráfico Curvas Brutas para referência e o ajuste de Hertz para cada curva na Curva de Corrente.
- Verifique o gráfico de dispersão de E originado do ajuste do modelo de Hertz a cada curva individual.
- Clique no nome do arquivo, na curva F-z, na curva F-δ ou em um ponto no gráfico de dispersão para realçar a curva em cada gráfico. Se um ponto de dados no gráfico de dispersão parecer estar fora da distribuição dos dados, clique nele e inspecione a curva à qual ele pertence. Verifique se o CP foi localizado corretamente clicando no botão Inspecionar . Exclua a curva da análise, se necessário.
- Inspecione a caixa Resultados para a média calculada E e seu DP (Eγ ± σ) e certifique-se de que eles são razoáveis para o experimento dado.
- Na caixa Salvar , clique em Hertz. Na janela pop-up, digite o nome do arquivo e o diretório. Uma vez feito, clique em Salvar. Um arquivo .tsv será criado. Abra o arquivo .tsv em qualquer software adicional de preferência e use os valores para análise estatística e plotagem adicional.
Observação : O arquivo contém o E obtido de cada curva e a média E e seu SD. Além disso, o arquivo contém metadados associados à análise, incluindo o número de curvas analisadas, R, k e o recuo máximo usado para o modelo de Hertz. - Esta etapa é opcional. Clique em F-Ind médio para exportar a força média e o recuo médio, juntamente com um DP na força.
- Para obter dados de nanoindentação celular, clique em Análise de espectros de elasticidade (consulte Resultados representativos e discussão). Inspecione os dois gráficos produzidos, a saber, E em função da profundidade de recuo (E(δ)) para cada curva, e o E médio (δ) com banda de erro mostrando um DP (linha vermelha sólida e área sombreada) ajustado por um modelo (linha tracejada preta), que permite estimar o módulo de Young do córtex de actina da célula, o módulo de Young em massa da célula, e a espessura do córtex de actina. Além disso, verifique a média E(δ) no gráfico superior em vermelho.
- Certifique-se de que a caixa Interpolar esteja marcada, o que garante que a derivada necessária para executar a análise de espectros de elasticidade seja calculada no sinal interpolado (consulte Resultados representativos).
- Inspecione a caixa Resultados, relatando o módulo de Young do córtex (E 0 ± σ), o módulo de Young em massa da célula (Eb ± σ) e a espessura do córtex (d 0 ± σ).
NOTA: Os espectros de elasticidade média podem parecer ruidosos no início, com oscilações sinusoidais proeminentes. Como resultado, a equação (3) pode não ser ajustada corretamente. Se este for o caso, aumentar iterativamente o comprimento da janela do filtro SAVGOL de suavização34 resolve esse problema. - Quando a análise estiver concluída, clique em ES na caixa Salvar . Isso exportará um arquivo .tsv no diretório especificado, contendo a elasticidade média em função da profundidade média de recuo e do raio de contato, os metadados associados ao experimento (veja acima) e os parâmetros estimados do modelo explicados acima. Por fim, a elasticidade média, desconsiderando a dependência δ e seu DS, também é relatada.
- Feche o software e insira os resultados salvos em qualquer outro software de preferência para plotar ainda mais os dados e realizar análises estatísticas.
- Esta etapa é opcional: exporte gráficos da GUI clicando com o botão direito do mouse no gráfico e selecionando Exportar. Exporte o gráfico em .svg, para que parâmetros como fonte, tamanho da fonte, estilo de linha, etc. pode ser editado em outro software de escolha.
NOTA:Algoritmos e filtros CP personalizados podem ser programados e adicionados aos já existentes. Ver a Nota Complementar 1 para mais pormenores.
Resultados
Seguindo o protocolo, obtém-se um conjunto de curvas F-z. O conjunto de dados provavelmente conterá boas curvas e curvas a serem descartadas antes de continuar com a análise. Em geral, as curvas devem ser descartadas se sua forma for diferente da mostrada na Figura 4A. A Figura 5AI mostra um conjunto de dados de ~100 curvas obtidas em um hidrogel PAAm macio de E 0,8 KPa35 esperado carregado na GUI do NanoPrepare. A ma...
Discussão
Este protocolo mostra como adquirir de forma robusta dados de nanoindentação por espectroscopia de força usando um nanoindenter de topo de ferrule comercialmente disponível em hidrogéis e células individuais. Além disso, instruções para o uso de um software de código aberto programado em Python compreendendo um fluxo de trabalho preciso para a análise de dados de nanoindentação são fornecidas.
Etapas críticas no protocolo
As etapas a seguir foram identificada...
Divulgações
Os autores não têm nada a revelar.
Agradecimentos
GC e MAGO reconhecem todos os membros do CeMi. O MSS reconhece o apoio através de uma subvenção do programa EPSRC (EP/P001114/1).
GC: software (contribuição para o desenvolvimento de software e algoritmos), análise formal (análise de dados de nanoindentação), validação, Investigação (experimentos de nanoindentação em géis de poliacrilamida), curadoria de dados, redação (rascunho original, revisão e edição), visualização (figuras e gráficos). MAGO: investigação (preparação de amostras de géis e células, experimentos de nanoindentação em células), escrita (rascunho original, revisão e edição), visualização (figuras e gráficos). NA: validação, redação (revisão e edição). IL: software (contribuição para o desenvolvimento de software e algoritmos), validação, escrita (revisão e edição); MV: conceituação, software (design e desenvolvimento de software e algoritmos originais), validação, recursos, redação (rascunho original, revisão e edição), supervisão, administração de projetos, aquisição de financiamento MSS: recursos, redação (revisão e edição), supervisão, administração de projetos, aquisição de financiamento. Todos os autores leram e aprovaram o manuscrito final.
Materiais
| Name | Company | Catalog Number | Comments |
| 12 mm coverslips | VWR | 631-1577P | |
| 35 mm cell treated culture dishes | Greiner CELLSTAR | 627160 | |
| Acrylamide | Sigma-Aldrich | A4058 | |
| Acrylsilane | Alfa Aesar | L16400 | |
| Ammonium Persulfate | Merk | 7727-54-0 | |
| Bisacrylamide | Merk | 110-26-9 | |
| Chiaro nanoindenter | Optics 11 Life | no catologue number | |
| Ethanol | general | ||
| Fetal bovine serum | Gibco | 16140071 | |
| High glucose DMEM | Gibco | 11995065 | |
| Isopropanol | general | ||
| Kimwipe | Kimberly Clark | 21905-026 | |
| Microscope glass slides | VWR | 631-1550P | |
| MilliQ system | Merk Millipore | ZR0Q008WW | |
| OP1550 Interferometer | Optics11 Life | no catalogue number | |
| Optics 11 Life probe (k = 0.02-0.005 N/m, R = 3-3.5 um) | Optics 11 Life | no catologue number | |
| Optics 11 Life probe (k = 0.46-0.5 N/m, R = 50-55 um) | Optics 11 Life | no catologue number | |
| Penicillin/Streptomycin | Gibco | 15140122 | |
| RainX rain repellent | RainX | 26012 | |
| Standard petri dishes (90 mm) | Thermo Scientific | 101RTIRR | |
| Tetramethylethylenediamine | Sigma-Aldrich | 110-18-9 | |
| Vaccum dessicator | Thermo Scientific | 531-0250 | |
| Software | |||
| Data acquisition software (v 3.4.1) | Optics 11 Life | ||
| GitHub Desktop (Optional) | Microsoft | ||
| Python 3 | Python Software Foundation | ||
| Visual Studio Code (Optional) | Microsoft |
Referências
- Discher, D. E., Janmey, P. Y. W. Tissue cells feel and respond to the stiffness of their substrate. Science. 310 (5751), 1139-1143 (2005).
- Roca-Cusachs, P., Conte, V., Trepat, X. Quantifying forces in cell biology. Nature Cell Biology. 19 (7), 742-751 (2017).
- Guimarães, C. F., Gasperini, L., Marques, A. P., Reis, R. L. The stiffness of living tissues and its implications for tissue engineering. Nature. Reviews. Materials. 5, 351-370 (2020).
- Moeendarbary, E., Harris, A. R. Cell mechanics: Principles, practices, and prospects. Wiley Interdisciplinary. Reviews. Systems. Biology and Medicine. 6 (5), 371-388 (2014).
- Butcher, D. T., Alliston, T., Weaver, V. M. A tense situation: Forcing tumour progression. Nature Reviews Cancer. 9 (2), 108-122 (2009).
- Kubánková, M., et al. Physical phenotype of blood cells is altered in COVID-19. Biophysical Journal. 120 (14), 2838-2847 (2021).
- Engler, A. J., Sen, S., Sweeney, H. L., Discher, D. E. Matrix elasticity directs stem cell lineage specification. Cell. 126, 677-689 (2006).
- Ciccone, G., et al. What caging force cells feel in 3D hydrogels: A rheological perspective. Advanced Healthcare Materials. 9 (17), 2000517 (2020).
- Wu, P. H., et al. A comparison of methods to assess cell mechanical properties. Nature Methods. 15 (7), 491-498 (2018).
- McKee, C. T., Last, J. A., Russell, P., Murphy, C. J. Indentation versus tensile measurements of young's modulus for soft biological tissues. Tissue Engineering B Reviews. 17 (3), 155-164 (2011).
- Humphrey, J. D., Delange, S. L. . An Introduction to Biomechanics Solids and Fluids, Analysis and Design. , 271-371 (2004).
- Prevedel, R., Diz-Muñoz, A., Ruocco, G., Antonacci, G. Brillouin microscopy: an emerging tool for mechanobiology. Nature Methods. 16 (10), 969-977 (2019).
- Krieg, M., et al. Atomic force microscopy-based mechanobiology. Nature Reviews Physics. 1, 41-57 (2019).
- Norman, M. D. A., Ferreira, S. A., Jowett, G. M., Bozec, L., Gentleman, E. Measuring the elastic modulus of soft culture surfaces and three-dimensional hydrogels using atomic force microscopy. Nature Protocols. 16 (5), 2418-2449 (2021).
- Gavara, N. A beginner's guide to atomic force microscopy probing for cell mechanics. Microscopy Research and Technique. 80 (1), 75-84 (2017).
- Whitehead, A. J., Kirkland, N. J., Engler, A. J. Atomic force microscopy for live-cell and hydrogel measurement. Methods in Molecular Biology. 2299, 217-226 (2021).
- Chavan, D., et al. Ferrule-top nanoindenter: An optomechanical fiber sensor for nanoindentation. The Review of Scientific Instruments. 83 (11), 115110 (2012).
- Van Hoorn, H., Kurniawan, N. A., Koenderink Ac, G. H., Iannuzzi, D. Local dynamic mechanical analysis for heterogeneous soft matter using ferrule-top indentation. Soft Matter. 12 (12), 3066-3073 (2016).
- Baldini, F., et al. Biomechanics of cultured hepatic cells during different steatogenic hits. Journal of the Mechanical Behavior of Biomedical Materials. 97, 296-305 (2019).
- Emig, R., et al. Piezo1 channels contribute to the regulation of human atrial fibroblast mechanical properties and matrix stiffness sensing. Cells. 10 (3), 663 (2021).
- Dobre, O., et al. A hydrogel platform that incorporates laminin isoforms for efficient presentation of growth factors - Neural growth and osteogenesis. Advanced Functional Materials. 31 (21), 2010225 (2021).
- Antonovaite, N., Beekmans, S. V., Hol, E. M., Wadman, W. J., Iannuzzi, D. Regional variations in stiffness in live mouse brain tissue determined by depth-controlled indentation mapping. Scientific Reports. 8 (1), 12517 (2018).
- Wu, G., Gotthardt, M., Gollasch, M. Assessment of nanoindentation in stiffness measurement of soft biomaterials: kidney, liver, spleen and uterus. Scientific Reports. 10 (1), 18784 (2020).
- Lüchtefeld, I., et al. Elasticity spectra as a tool to investigate actin cortex mechanics. Journal of Nanobiotechnology. 18 (1), 147 (2020).
- Caliari, S. R., Burdick, J. A. A practical guide to hydrogels for cell culture. Nature Methods. 13 (5), 405-414 (2016).
- Blumlein, A., Williams, N., McManus, J. J. The mechanical properties of individual cell spheroids. Scientific Reports. 7 (1), 7346 (2017).
- Mirsandi, H., et al. Influence of wetting conditions on bubble formation from a submerged orifice. Experiments in Fluids. 61, 83 (2020).
- Vassalli, M., Ciccone, G. . CellMechLab/NanoPrepare: v0.1.1. Zendo. , (2021).
- Vassalli, M., Ciccone, G., Lüchtefeld, I. CellMechLab/nanoindentation: v1.0.0. Zendo. , (2021).
- Savitzky, A., Golay, M. J. E. Smoothing and differentiation of data by simplified least squares procedures. Analytical Chemistry. 36 (8), 1627-1639 (1964).
- Schafer, R. W. What is a savitzky-golay filter. IEEE Signal Processing Magazine. 28 (4), 111-117 (2011).
- Gavara, N. Combined strategies for optimal detection of the contact point in AFM force-indentation curves obtained on thin samples and adherent cells. Scientific Reports. 6, 21267 (2016).
- Lin, D. C., Dimitriadis, E. K., Horkay, F. Robust strategies for automated AFM force curve analysis-I. Non-adhesive indentation of soft, inhomogeneous materials. Journal of Biomechanical Engineering. 129 (3), 430-440 (2007).
- Virtanen, P., et al. SciPy 1.0: fundamental algorithms for scientific computing in Python. Nature Methods. 17 (3), 261-272 (2020).
- Tse, J. R., Engler, A. J. Preparation of hydrogel substrates with tunable mechanical properties. Current Protocols in Cell Biology. , (2010).
- Pharr, G. M., Oliver, W. C., Brotzen, F. R. On the generality of the relationship among contact stiffness, contact area, and elastic modulus during indentation. Journal of Materials Research. 7 (3), 613-617 (1992).
- Kumar, R., Saha, S., Sinha, B. Cell spread area and traction forces determine myosin-II-based cortex thickness regulation. Biochimica et Biophysica Acta - Molecular Cell Research. 1866 (12), 118516 (2019).
- Kunda, P., Pelling, A. E., Liu, T., Baum, B. Moesin controls cortical rigidity, cell rounding, and spindle morphogenesis during mitosis. Current Biology: CB. 18 (2), 91-101 (2008).
- Allen, M., Poggiali, D., Whitaker, K., Marshall, T. R., Kievit, R. A. Raincloud plots: a multi-platform tool for robust data visualization. Wellcome Open Research. 4, 63 (2019).
- Clark, A. G., Paluch, E. Mechanics and regulation of cell shape during the cell cycle. Results and Problems in Cell Differentiation. 53, 31-73 (2011).
- Langan, T. J., Rodgers, K. R., Chou, R. C. Synchronization of mammalian cell cultures by serum deprivation. Methods in Molecular Biology. 1524, 97-105 (2017).
- Hodgkinson, T., et al. The use of nanovibration to discover specific and potent bioactive metabolites that stimulate osteogenic differentiation in mesenchymal stem cells. Science Advances. 7 (9), 7921 (2021).
- Kloxin, A. M., Kloxin, C. J., Bowman, C. N., Anseth, K. S. Mechanical properties of cellularly responsive hydrogels and their experimental determination. Advanced Materials (Deerfield Beach, Fla). 22 (31), 3484-3494 (2010).
- Loessner, D., et al. Functionalization, preparation and use of cell-laden gelatin methacryloyl-based hydrogels as modular tissue culture platforms. Nature Protocols. 11 (4), 727-746 (2016).
- Yue, K., et al. Synthesis, properties, and biomedical applications of gelatin methacryloyl (GelMA) hydrogels. Biomaterials. 73, 254-271 (2015).
- Sarrigiannidis, S. O., et al. A tough act to follow: collagen hydrogel modifications to improve mechanical and growth factor loading capabilities. Materials Today. Bio. 10, 100098 (2021).
- Karoutas, A., et al. The NSL complex maintains nuclear architecture stability via lamin A/C acetylation. Nature Cell Biology. 21 (10), 1248-1260 (2019).
- Ryu, H., et al. Transparent, compliant 3D mesostructures for precise evaluation of mechanical characteristics of organoids. Advanced Materials (Deerfield Beach, Fla). 33 (25), 2100026 (2021).
- Carvalho, D. T. O., Feijão, T., Neves, M. I., Da Silva, R. M. P., Barrias, C. C. Directed self-assembly of spheroids into modular vascular beds for engineering large tissue constructs Biofabrication Directed self-assembly of spheroids into modular vascular beds for engineering large tissue constructs. Biofabrication. 13 (3), 035008 (2021).
- Chaudhuri, O., Cooper-White, J., Janmey, P. A., Mooney, D. J., Shenoy, V. B. Effects of extracellular matrix viscoelasticity on cellular behaviour. Nature. 584 (7822), 535-546 (2020).
- Cantini, M., Donnelly, H., Dalby, M. J., Salmeron-Sanchez, M. The plot thickens: The emerging role of matrix viscosity in cell mechanotransduction. Advanced Healthcare Materials. 9 (8), 1901259 (2020).
- Elosegui-Artola, A. The extracellular matrix viscoelasticity as a regulator of cell and tissue dynamics. Current Opinion in Cell Biology. 72, 10-18 (2021).
- Chaudhuri, O. Viscoelastic hydrogels for 3D cell culture. Biomaterials Science. 5 (8), 1480-1490 (2017).
- Nguyen, T. D., Gu, Y. Determination of strain-rate-dependent mechanical behavior of living and fixed osteocytes and chondrocytes using atomic force microscopy and inverse finite element analysis. Journal of Biomechanical Engineering. 136 (10), 101004 (2014).
- Mokbel, M., Hosseini, K., Aland, S., Fischer-Friedrich, E. The Poisson Ratio of the Cellular Actin Cortex Is Frequency Dependent. Biophysical Journal. 118 (8), 1968-1976 (2020).
- Javanmardi, Y., Colin-York, H., Szita, N., Fritzsche, M., Moeendarbary, E. Quantifying cell-generated forces: Poisson's ratio matters. Communications Physics. 4, 237 (2021).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados