È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Flusso di lavoro sperimentale e di analisi dei dati per la nanoindentazione della materia soffice
In questo articolo
Riepilogo
Il protocollo presenta un flusso di lavoro completo per esperimenti di nanoindentazione di materiali morbidi, inclusi idrogel e cellule. In primo luogo, i passaggi sperimentali per acquisire i dati della spettroscopia di forza sono dettagliati; quindi, l'analisi di tali dati è dettagliata attraverso un software Python open source di nuova concezione, che può essere scaricato gratuitamente da GitHub.
Abstract
La nanoindentazione si riferisce a una classe di tecniche sperimentali in cui una sonda di forza micrometrica viene utilizzata per quantificare le proprietà meccaniche locali di biomateriali morbidi e cellule. Questo approccio ha acquisito un ruolo centrale nei campi della meccanobiologia, della progettazione di biomateriali e dell'ingegneria tissutale, per ottenere una corretta caratterizzazione meccanica di materiali morbidi con una risoluzione paragonabile alla dimensione delle singole cellule (μm). La strategia più popolare per acquisire tali dati sperimentali è quella di impiegare un microscopio a forza atomica (AFM); mentre questo strumento offre una risoluzione senza precedenti in forza (fino a pN) e spazio (sub-nm), la sua usabilità è spesso limitata dalla sua complessità che impedisce misure di routine di indicatori integrali di proprietà meccaniche, come il modulo di Young (E). Una nuova generazione di nanoindentatori, come quelli basati sulla tecnologia di rilevamento delle fibre ottiche, ha recentemente guadagnato popolarità per la sua facilità di integrazione mentre consente di applicare forze sub-nN con risoluzione spaziale μm, quindi è adatto a sondare le proprietà meccaniche locali di idrogel e celle.
In questo protocollo, viene presentata una guida passo-passo che descrive in dettaglio la procedura sperimentale per acquisire dati di nanoindentazione su idrogel e cellule utilizzando un nanopenetratore di rilevamento in fibra ottica disponibile in commercio. Mentre alcuni passaggi sono specifici per lo strumento utilizzato nel presente documento, il protocollo proposto può essere preso come guida per altri dispositivi di nanoindentazione, a condizione che alcuni passaggi siano adattati secondo le linee guida del produttore. Inoltre, viene presentato un nuovo software Python open-source dotato di un'interfaccia utente grafica user-friendly per l'analisi dei dati di nanoindentazione, che consente lo screening di curve acquisite in modo errato, il filtraggio dei dati, il calcolo del punto di contatto attraverso diverse procedure numeriche, il calcolo convenzionale di E, nonché un'analisi più avanzata particolarmente adatta per i dati di nanoindentazione a singola cellula.
Introduzione
Il ruolo fondamentale della meccanica in biologia è oggi stabilito 1,2. Dai tessuti interi alle singole cellule, le proprietà meccaniche possono informare sullo stato fisiopatologico del biomateriale in esame 3,4. Ad esempio, il tessuto mammario affetto da cancro è più rigido del tessuto sano, un concetto che è alla base del popolare test di palpazione5. In particolare, è stato recentemente dimostrato che la malattia da coronavirus 2019 (COVID-19) causata dalla sindrome respiratoria acuta grave coronavirus 2 (SARS-CoV-2) è sottolineata da cambiamenti nelle proprietà meccaniche delle cellule del sangue, tra cui una diminuzione della deformabilità eritrocitaria e una diminuzione della rigidità dei linfociti e dei neutrofili rispetto alle cellule del sangue di individui naïve al SARS-CoV-26.
In generale, la meccanica delle cellule e dei tessuti è intrinsecamente intrecciata: ogni tessuto ha specifiche proprietà meccaniche che influenzano e dipendono simultaneamente da quelle delle cellule costituenti e della matrice extracellulare (ECM)5. Per questo motivo, le strategie per studiare la meccanica in biologia spesso coinvolgono substrati ingegneristici con stimoli meccanici fisiologicamente rilevanti per chiarire il comportamento cellulare in risposta a tali stimoli. Ad esempio, il lavoro seminale di Engler e colleghi ha dimostrato che l'impegno del lignaggio delle cellule staminali mesenchimali è controllato dall'elasticità della matrice, come studiato su idrogel di poliacrilammide bidimensionale (PAAm) morbidi e rigidi7.
Esistono molte strategie per caratterizzare meccanicamente il biomateriale in esame, che variano nella scala spaziale (cioè da locale a massa) e nella modalità di deformazione (ad esempio, assiale vs taglio), producendo di conseguenza informazioni diverse, che richiedono un'attenta interpretazione 3,8,9,10. La meccanica dei biomateriali morbidi è comunemente espressa in termini di rigidità. Tuttavia, la rigidità dipende sia dalle proprietà del materiale che dalla geometria, mentre i moduli elastici sono proprietà fondamentali di un materiale e sono indipendenti dalla geometria del materiale11. In quanto tali, diversi moduli elastici sono correlati alla rigidità di un dato campione e ciascun modulo elastico comprende la resistenza del materiale a una specifica modalità di deformazione (ad esempio, assiale vs taglio) in diverse condizioni al contorno (ad esempio, espansione libera vs confinamento)11,12. Gli esperimenti di nanoindentazione consentono la quantificazione delle proprietà meccaniche attraverso l'E che è associata alla deformazione uniassiale (indentazione) quando il biomateriale non è confinato lateralmente10,11,12.
Il metodo più popolare per quantificare E dei sistemi biologici su microscala è AFM13,14,15,16. AFM è uno strumento estremamente potente con risoluzione della forza fino al livello pN e risoluzione spaziale fino alla scala sub-nm. Inoltre, AFM offre un'estrema flessibilità in termini di accoppiamento con strumenti ottici e meccanici complementari, estendendo le sue capacità di estrarre una grande quantità di informazioni dal biomateriale in esame13. Queste caratteristiche interessanti, tuttavia, presentano una barriera all'ingresso rappresentata dalla complessità dell'assetto sperimentale. L'AFM richiede una formazione approfondita prima che gli utenti possano acquisire dati robusti e il suo uso per la caratterizzazione meccanica quotidiana dei materiali biologici è spesso ingiustificato, specialmente quando non sono richieste la sua forza unica e le risoluzioni spaziali.
Per questo motivo, una nuova classe di nanoindentatori ha recentemente guadagnato popolarità grazie alla loro facilità d'uso, pur offrendo dati comparabili con AFM con risoluzione di forza sub-nN e risoluzione spaziale μm, riflettendo le forze esercitate e percepite dalle cellule su scale di lunghezza rilevanti2. In particolare, i dispositivi di nanoindentazione con ghiera basati sulla tecnologia di rilevamento delle fibre ottiche17,18 hanno guadagnato popolarità tra i ricercatori attivi nel campo della meccanobiologia e non solo; e sono stati pubblicati numerosi lavori che riportano le proprietà meccaniche dei biomateriali che utilizzano questi dispositivi, tra cui le cellule19,20, gli idrogel8,21 e i tessuti22,23. Nonostante la capacità di questi sistemi di sondare le proprietà meccaniche dinamiche locali (cioè il modulo di stoccaggio e perdita), gli esperimenti quasi-statici che producono E rimangono la scelta più popolare 8,19,20,21. In breve, gli esperimenti di nanoindentazione quasi-statica consistono nell'indentare il campione con una velocità costante fino a un set-point definito da uno spostamento massimo, forza o profondità di indentazione e registrare sia la forza che la posizione verticale del cantilever nelle cosiddette curve forza-distanza (F-z). Le curve F-z vengono quindi convertite in curve di indentazione della forza (F-δ) attraverso l'identificazione del punto di contatto (CP) e dotate di un modello di meccanica di contatto appropriato (di solito il modello Hertz13) per calcolare E.
Mentre il funzionamento dei nanopenetratori con ghiera assomiglia alle misurazioni AFM, ci sono specificità che vale la pena considerare. In questo lavoro, viene fornita una guida passo-passo per acquisire in modo robusto curve F-z da cellule e idrogel che imitano i tessuti utilizzando un nanopenetratore a ghiera disponibile in commercio, al fine di incoraggiare la standardizzazione delle procedure sperimentali tra gruppi di ricerca che utilizzano questo e altri dispositivi simili. Inoltre, vengono forniti consigli su come preparare al meglio campioni e cellule di idrogel per eseguire esperimenti di nanoindentazione, insieme a suggerimenti per la risoluzione dei problemi lungo il percorso sperimentale.
Inoltre, gran parte della variabilità nei risultati della nanoindentazione (cioè E e la sua distribuzione) dipende dalla procedura specifica utilizzata per analizzare i dati, che non è banale. Per risolvere questo problema, vengono fornite istruzioni per l'uso di un software open source di nuova concezione programmato in Python e dotato di un'interfaccia utente grafica (GUI) di facile utilizzo per l'analisi batch delle curve F-z . Il software consente uno screening rapido dei dati, il filtraggio dei dati, il calcolo del CP attraverso diverse procedure numeriche, il calcolo convenzionale di E, nonché un'analisi più avanzata chiamata spettri di elasticità24, che consente di stimare il modulo di Young della massa della cellula, il modulo di Young della corteccia di actina e lo spessore della corteccia di actina. Il software può essere scaricato liberamente da GitHub e può essere facilmente adattato per analizzare i dati provenienti da altri sistemi aggiungendo un parser di dati appropriato. Si sottolinea che questo protocollo può essere utilizzato per altri dispositivi di nanoindentazione con ghiera e altri dispositivi di nanoindentazione in generale, a condizione che alcuni passaggi siano adattati in base alle linee guida dello strumento specifico. Il protocollo è schematicamente riassunto nella Figura 1.
Protocollo
1. Preparazione di substrati/celle per misure di nanoindentazione
- Seguire i passaggi indicati nel protocollo supplementare per la preparazione di idrogel/cellule PAAm per esperimenti di nanoindentazione. La procedura è riepilogata nella Figura 2.
NOTA: Gli idrogel PAAm sono stati scelti in quanto sono gli idrogel più comuni utilizzati nel campo della meccanobiologia. Tuttavia, il protocollo è ugualmente applicabile a qualsiasi tipo di idrogel25 (vedi Discussione, modifiche del metodo).
2. Avvio del dispositivo, scelta della sonda e calibrazione della sonda
- Seguire i passaggi indicati nel protocollo supplementare per l'avvio del dispositivo. Per i dettagli tecnici sul funzionamento dei nanoindentatori a ghiera in fibra ottica, controllare questi riferimenti17,18.
- Selezionare la sonda di nanoindentazione come descritto di seguito.
NOTA: Tutte le sonde disponibili in commercio, come quelle utilizzate in questo protocollo, sono dotate di una punta sferica (Figura 3A). Pertanto, la scelta si restringe a due variabili: rigidità del cantilever e raggio della punta (Figura 3B).- Scegliere la rigidità di un cantilever (k in N/m) che corrisponda alla rigidità del campione prevista per ottenere i migliori risultati15 (vedere Figura 3C e Discussione, passaggi critici nel protocollo). Per le celle, selezionare una sonda con k nell'intervallo 0,01-0,09 N/m. Per gli idrogel, selezionare una sonda con k nell'intervallo 0,1-0,9 N/m, che produce risultati ottimali per gel con E atteso tra pochi kPa e 100 kPa (vedere Risultati rappresentativi).
- Scegli il raggio della punta (R in μm) in base alla risoluzione spaziale desiderata del processo di indentazione. Per le piccole cellule come le cellule del rene embrionale umano 293T (HEK293T) (diametro medio di ~ 10-15 μm26) selezionare una sfera con R = 3 μm. Per gli idrogel, selezionare una sfera con R = 10-250 μm per sondare le proprietà meccaniche del biomateriale su un'ampia area di contatto ed evitare eterogeneità locali.
- Consultare la Figura 3C e le eventuali linee guida aggiuntive del produttore per selezionare la sonda appropriata.
- Una volta selezionata la sonda, seguire i passaggi del protocollo supplementare per montarla sul nanopenetratore.
3. Calibrazione della sonda
NOTA: i seguenti passaggi sono specifici per i dispositivi di nanoindentazione con ghiera basati sulla tecnologia di rilevamento delle fibre ottiche e sono dettagliati per la versione software 3.4.1. Per altri dispositivi di nanoindentazione, seguire i passaggi consigliati dal produttore del dispositivo.
- Nella finestra principale del software, fare clic su Inizializza. Apparirà un menu di calibrazione. Inserire i dettagli della sonda (si trovano sul lato della scatola della sonda; k in N/m, R in μm e il fattore di taratura in aria) nelle caselle di input.
- Preparare il piatto di calibrazione: una piastra di Petri di vetro spesso con un fondo piatto (vedere Discussione, passaggi critici nel protocollo). Riempi il piatto con lo stesso supporto del piatto campione (questo può anche essere aria). Abbinare la temperatura del fluido con quella del campione.
- Posizionare il piatto di calibrazione sotto la sonda. Se necessario, estrarre la sonda dal supporto del braccio del nanopenetratore e tenerla in una mano per fare spazio per il posizionamento del piatto di calibrazione. Far scorrere indietro la sonda in posizione.
- Eseguire i due passaggi successivi per la calibrazione in liquido. Se si esegue la misurazione in aria, utilizzare il substrato di politetrafluoroetilene fornito per la calibrazione e saltare al passaggio 5.
- Prebagnare la sonda con una goccia di etanolo al 70% utilizzando una pipetta Pasteur con l'estremità della pipetta a contatto leggero con la ghiera di vetro, affinché la goccia scivoli sul cantilever e sulla punta sferica27 (vedere Discussione, passaggi critici nel protocollo).
- Far scorrere manualmente il braccio del nanopenetratore verso il basso fino a quando la sonda è completamente sommersa, ma ancora lontana dal fondo della capsula di Petri. Se necessario, aggiungere altro mezzo al piatto di calibrazione. Attendere 5 minuti in modo da raggiungere le condizioni di equilibrio nel liquido.
- Nel menu Inizializza del software, fare clic su Scan Wavelength. Lo schermo dell'interferometro mostrerà una barra di avanzamento e la finestra Live Signal sul software del computer visualizzerà il modello mostrato nella Figura S1A, a sinistra. Per verificare se la scansione ottica ha avuto esito positivo, accedere al pannello Scansione lunghezza d'onda sulla casella dell'interferometro. In caso di successo, dovrebbe essere visibile un'onda sinusoidale (Figura 1A, a destra). Vedere Discussione (risoluzione dei problemi del metodo) se viene visualizzato un errore.
- Nel menu Inizializza, fate clic su Trova superficie (Find Surface), che abbasserà progressivamente la sonda fino a raggiungere una soglia impostata nella piegatura a sbalzo. La sonda smette di muoversi quando viene effettuato il contatto con la capsula di Petri di vetro.
- Controllare se la sonda è a contatto con la superficie. Spostare la sonda verso il basso di 1 μm utilizzando il pulsante freccia y verso il basso nella finestra principale del software. Osservare il segnale verde (deflessione del cantilever) nella finestra live del software per le modifiche nella linea di base ad ogni passo, quando il cantilever è a contatto con il substrato (Figura S1B). Se non ci sono cambiamenti, il cantilever non è in contatto (vedi passaggio successivo).
- Aumentate il valore Soglia (Threshold) nel menu Opzioni della scheda Trova superficie (Find Surface) dal valore predefinito 0,01 di un passaggio di 0,01 alla volta e ripetete il passaggio Trova superficie fino a quando non vengono contattati. In alternativa, portare la sonda a contatto con piccoli passi di 1 μm fino a quando la linea di base verde inizia a spostarsi ad ogni passo verso il basso.
NOTA: per i cantilever più morbidi (k = 0,025 Nm-1), aumentate il valore Soglia nel menu Opzioni della scheda Trova superficie a priori per eseguire il passaggio 6. Partire da un valore di soglia di 0,06 o 0,07 e aumentarlo fino a 0,1 se necessario. Questo perché il rumore ambientale probabilmente causerà la flessione del cantilever al di sopra della soglia prima del contatto. Per le sonde morbide, la diminuzione della velocità di avvicinamento (μm/s) nello stesso menu migliora anche la procedura. - Nel menu Inizializza, fare clic su Calibra.
- Controllare la finestra Live Signal del software e assicurarsi che sia lo spostamento del piezo che i segnali di deflessione del cantilever si muovano contemporaneamente verso l'alto (Figura S1C).
- Se c'è una mancata corrispondenza nel tempo, la sonda non è completamente a contatto con il vetro. Spostarsi verso il basso della sonda a passi di 1 μm fino a quando la linea di base del segnale a sbalzo cambia (vedere il punto 7) e ripetere il punto 9.
- Se il segnale a sbalzo non cambia affatto durante la fase di calibrazione , la sonda è lontana dalla superficie. Aumentare la soglia di contatto in modo iterativo (vedere il passaggio 8) fino a trovare correttamente la superficie e ripetere la calibrazione dall'inizio iniziando con la scansione della lunghezza d'onda.
- Al termine della calibrazione, controllare i fattori di calibrazione vecchi e nuovi nella finestra pop-up visualizzata. Se il nuovo fattore di calibrazione è nell'intervallo corretto, fare clic su Usa nuovo fattore. Se la calibrazione non riesce e il nuovo fattore è NaN o non rientra nell'intervallo previsto, vedere Discussione (risoluzione dei problemi del metodo) per la risoluzione.
NOTA: Il nuovo fattore dovrebbe essere ~n volte inferiore a quello fornito sulla scatola della sonda se la calibrazione è stata eseguita in un mezzo liquido con indice di rifrazione n ( n = 1,33 per l'acqua). Se la calibrazione è stata eseguita in aria, i nuovi e i vecchi fattori di calibrazione dovrebbero essere approssimativamente uguali. - Verificare se il cerchio di demodulazione è stato calibrato correttamente come segue. Passare alla scheda Demodulazione sul desktop dell'interferometro. Picchiettare delicatamente sul tavolo ottico o sul nanopenetratore per indurre abbastanza rumore. Un cerchio bianco costituito da punti dati discreti dovrebbe coprire approssimativamente il cerchio rosso (Figura S1D).
- Se il cerchio bianco non si sovrappone al cerchio rosso o viene visualizzato un avviso sul display dell'interferometro, il cerchio di demodulazione deve essere ricalibrato. Questo può essere fatto in due modi come descritto di seguito.
- Toccare continuamente il corpo del nanopenetratore per indurre un cerchio completo di rumore e premere il pulsante Calibrate sull'interferometro.
- Entrare in contatto con il substrato di vetro e premere Calibra dal menu Inizializza della finestra principale del software. Non salvare il fattore di calibrazione. A questo punto, controlla di nuovo e assicurati che il cerchio bianco si sovrapponga al cerchio rosso.
NOTA: Se il segnale non è solo leggermente spostato dal cerchio di demodulazione, ma piuttosto è diventato molto piccolo o non è affatto visibile, significa che il cantilever è attaccato alla fibra ottica. Seguire i consigli per la risoluzione dei problemi relativi a questo problema (vedere Discussione, risoluzione dei problemi relativi al metodo) e ripetere i passaggi 13.1 o 13.2. Una volta che il cantilever torna alla sua posizione orizzontale (Figura 3A), il segnale tornerà al cerchio di demodulazione.
- Verificare la calibrazione eseguendo una rientranza sul substrato di vetro subito dopo la calibrazione come descritto di seguito.
- Caricare o creare un file dell'esperimento facendo clic su Configura esperimento e aggiungere un passaggio Trova superficie e un passaggio Rientro . Per la fase di rientro, utilizzare le impostazioni predefinite della modalità di spostamento e modificare lo spostamento massimo alla distanza di calibrazione (3.000 nm) per spostare la sonda contro il substrato rigido.
- Fare clic su Esegui esperimento e controllare il cerchio di demodulazione nella finestra dell'interferometro. Controlla il segnale bianco e assicurati che sia sopra il cerchio rosso durante l'indentazione.
- Controllare i risultati nella finestra principale del software nel grafico Time Data e assicurarsi che lo spostamento del piezo (linea blu) sia uguale alla deflessione del cantilever (linea verde) poiché la rientranza inizia a contatto e non è prevista alcuna deformazione del materiale. Se i segnali non sono paralleli, vedere Discussione (risoluzione dei problemi relativi al metodo).
- Modificare il percorso locale nel menu di calibrazione impostando il percorso di salvataggio della calibrazione su una directory appropriata.
- Quando la sonda è stata calibrata correttamente, spostare la piezoelettrica verso l'alto di 500 μm.
4. Misurare il modulo di Young dei materiali morbidi
- Nanoindentazione di idrogel
- Caricare la capsula di Petri contenente i campioni sullo stadio del microscopio e spostare manualmente la sonda del nanopenetratore nella posizione x-y desiderata sopra il campione.
- Far scorrere manualmente la sonda in soluzione, avendo cura di lasciare 1-2 mm tra la sonda e la superficie del campione in questa fase. Attendere 5 minuti affinché la sonda si bilanci nel mezzo.
- Focalizzare il piano z del microscopio ottico in modo che la sonda sia chiaramente visibile.
- Eseguire una singola rientranza per regolare i parametri sperimentali come descritto di seguito.
- Configura un nuovo esperimento nella finestra principale del software. Fai clic su Configura esperimento, che aprirà una nuova finestra. Aggiungete un passo Trova superficie . Se necessario, è possibile modificare tutti i parametri del passaggio Trova superficie nel menu Opzioni del software.
NOTA: la superficie Trova abbassa la sonda fino a quando non viene rilevata la superficie, quindi la ritrae a una distanza definita da Z sopra la superficie (μm), sopra la superficie del campione. Se il cantilever selezionato è troppo rigido per il campione o il campione è appiccicoso, dopo la fase è probabile che la sonda sia ancora in contatto con il campione, il che si tradurrà in una curva senza una linea di base (Figura 4C). Per risolvere questo problema, aumentare la superficie Z sopra (μm). - Aggiungere un passaggio di rientro . Selezionare la scheda Profilo e fare clic su Controllo spostamento. Lasciare il profilo di rientro predefinito.
- Fare clic su Esegui esperimento nella finestra principale del software. Questo troverà la superficie ed eseguirà una singola rientranza. Se il singolo rientro non appare come previsto, modificare i parametri sperimentali come descritto nella Figura 4 e nella Discussione (risoluzione dei problemi del metodo).
- Configura un nuovo esperimento nella finestra principale del software. Fai clic su Configura esperimento, che aprirà una nuova finestra. Aggiungete un passo Trova superficie . Se necessario, è possibile modificare tutti i parametri del passaggio Trova superficie nel menu Opzioni del software.
- Una volta che il rientro appare come desiderato, configurare la scansione della matrice in modo che venga rientrata un'area sufficiente del campione. Fare clic su Configura esperimento, aggiungere un passo Trova superficie con i parametri sperimentali determinati in precedenza e aggiungere un passaggio Scansione matrice .
- Per gli idrogel piatti, configurare una scansione a matrice contenente 50-100 punti (cioè 5 x 10 o 10 x 10 in x e y) distanziati a 10-100 μm (cioè dx = dy = 10-100 μm). Fare clic su Usa posizione stage per avviare la scansione della matrice dalla posizione dello stage corrente. Assicuratevi che la casella Trova automaticamente superficie sia selezionata per trovare la superficie a ogni rientro utilizzando i parametri sperimentali impostati.
- Per evitare il sovracampionamento, impostate la dimensione del passo su almeno il doppio del raggio di contatto (
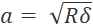 , dove δ è la profondità di rientro).
, dove δ è la profondità di rientro). - Impostare il profilo di scansione della matrice nel controllo dello spostamento. Assicurarsi che il profilo non violi i presupposti del modello Hertz (vedere Discussione, passaggi critici nel protocollo).
- Lasciate il numero di segmenti su 5, che è il valore di default, e utilizzate il profilo di spostamento di default. Se necessario, modificare il profilo di spostamento in termini di spostamento massimo e tempo per ciascun segmento inclinato, che influirà rispettivamente sulla profondità massima di rientro e sulla velocità di deformazione. Non superare le velocità di deformazione > 10 μm/s (vedere Discussione, limitazioni del metodo).
- Immettete un valore per la velocità di avvicinamento, che determina la velocità con cui la sonda viene spostata verso il campione prima del contatto. Abbinare la velocità di retrazione alla velocità di avvicinamento (vedere la nota seguente).
NOTA: Per cantilever morbidi e ambienti rumorosi, si consiglia una velocità di avvicinamento di 1.000-2.000 nm/s. Per cantilever più rigidi e ambienti controllati, questo può essere aumentato. - Salvare l'esperimento configurato nel percorso dell'esperimento desiderato e selezionare una directory in cui salvare i dati nel percorso di salvataggio, nella scheda Generale della finestra Configura esperimento . Fai clic su Esegui esperimento.
- Per evitare il sovracampionamento, impostate la dimensione del passo su almeno il doppio del raggio di contatto (
- Una volta completata la scansione della matrice, sollevare la sonda di 200-500 μm e spostare la sonda in un'area diversa del campione sufficientemente lontana dalla prima area.
- Ripetere l'esperimento almeno due volte in modo da acquisire dati sufficienti su ciascun campione (cioè almeno due scansioni a matrice per campione, contenenti 50-100 curve ciascuna).
- Nanoindentazione delle cellule
- Caricare il campione sul microscopio come descritto sopra.
- Per l'indentazione a cella singola, focalizzare il piano z in modo che sia le celle che la sonda siano visibili con un ingrandimento 20x o 40x, a seconda delle dimensioni e della diffusione della cella.
- Spostare la sonda sopra la cella da rientrare.
- Configura un nuovo esperimento nella finestra principale del software. Fai clic su Configura esperimento, che aprirà una nuova finestra. Aggiungete un passo Trova superficie e rientro con i parametri di default in modalità di spostamento.
- Fai clic su Esegui esperimento, che troverà la superficie ed eseguirà un singolo rientro. Verificare se il rientro è andato a buon fine. Se la curva non appare come previsto, regolare i parametri sperimentali (vedere Figura 4 e Discussione, risoluzione dei problemi del metodo).
- Se il rientro ha avuto esito positivo, aggiungere una scansione a matrice all'esperimento. Seguire i passaggi indicati per gli esperimenti di nanoindentazione degli idrogel; configurare la scansione della matrice in modo che la dimensione del passo consenta di rientrare una piccola area della cella; 25 punti distanziati a 0,5-5 μm per le celle HEK293T.
- A seconda delle dimensioni della cella, adattare la scansione della matrice per garantire che la punta non rientri al di fuori del limite della cella, ad esempio, eseguendo una geometria della mappa diversa o sondando meno punti.
- Fai clic su Esegui esperimento e attendi che sia completato.
- Una volta completata la scansione della matrice, sollevare la sonda fuori contatto (50 μm nel piano z ).
- Spostare la sonda sopra una nuova cella e ripetere il processo (vedere Discussione, passaggi critici nel protocollo).
- Pulizia della sonda e spegnimento dello strumento
- Seguire i passaggi indicati nel protocollo supplementare per pulire la sonda e spegnere il dispositivo di nanoindentazione.
5. Analisi dei dati
- Download e installazione del software
- Seguire i passaggi indicati nel protocollo supplementare per scaricare e installare il software per l'analisi dei dati28,29.
- Screening delle curve F-z e produzione di set di dati puliti in formato JSON
- Avviare prepare.py dalla riga di comando nel computer lab, come descritto nei passaggi da 2 a 3.
- Se si utilizza un computer Windows, tenere premuto il tasto Maiusc mentre si fa clic con il pulsante destro del mouse sulla cartella NanoPrepare e fare clic su Apri finestra PowerShell qui. Digitare il comando python prepare.py e premere il tasto Invio . Una GUI apparirà sullo schermo (Figura S2).
- Se si utilizza un computer MacOS, fare clic con il pulsante destro del mouse sulla cartella NanoPrepare e fare clic su Nuovo terminale nella cartella. Digitare il comando python3 prepare.py e premere il tasto Invio , che avvierà la GUI (Figura S2).
- Selezionare il formato dati O11NEW dall'elenco a discesa. Se i dati non vengono caricati correttamente, riavviare la GUI e selezionare O11OLD.
NOTA: Il formato O11NEW funziona per i dati ottenuti utilizzando dispositivi di nanoindentazione con rilevamento della fibra ottica con ghiera con versione software 3.4.1. Questo formato funzionerà anche per le versioni precedenti del software, almeno quelle appartenenti ai nanopenetratori installati nel 2019-2020. - Fare clic su Carica cartella. Selezionare una cartella contenente i dati da analizzare: scansione a matrice singola o scansione a matrice multipla. Il grafico superiore (Curve non elaborate) verrà popolato con il set di dati caricato. Per visualizzare una curva specifica, fare clic su di essa. Questo lo evidenzierà in verde e lo mostrerà sul grafico in basso (Curva corrente).
- Pulire il set di dati utilizzando le schede presenti nella parte destra della GUI come descritto di seguito.
- Utilizzare il pulsante Segmento per selezionare il segmento corretto da analizzare, ovvero il segmento in avanti delle curve F-z . Il numero specifico dipende dal numero di segmenti selezionati nel software di nanoindentazione durante l'esecuzione degli esperimenti.
- Usate il pulsante Ritaglia 50 nm per ritagliare le curve di 50 nm all'estrema sinistra (se L è selezionata), a destra (se R è selezionata) o su entrambi i lati (se R e L sono selezionati). Fare clic su questo pulsante più volte per ritagliare quanto necessario. Utilizzate questa opzione per rimuovere gli artefatti presenti all'inizio/alla fine delle curve F-z.
- Esaminate la linguetta Cantilever per verificare la costante della molla, la geometria della punta e il raggio della punta. Esaminare la scheda per assicurarsi che i metadati siano stati letti correttamente.
- Utilizzare la scheda Screening per impostare una soglia di forza che eliminerà tutte le curve che non hanno raggiunto la forza specificata. Le curve scartate verranno evidenziate in rosso.
- Utilizzate il pulsante Attiva/disattiva manualmente per rimuovere manualmente le curve che non sono state acquisite correttamente. Rimuovete eventuali curve facendo clic sulla curva specifica e selezionando OUT, che evidenzierà la curva in rosso.
- Fare clic su Salva JSON. Immettere un nome appropriato per il set di dati pulito, ovvero un singolo file JSON. Inviare il file JSON al computer in cui è stato installato il software NanoAnalysis.
6. Analisi formale dei dati
- Avvia il file nano.py dalla riga di comando accedendo alla cartella NanoAnalysis e avviando un terminale come spiegato in precedenza. Digitare il comando python nano.py o python3 nano.py (a seconda del sistema operativo) e premere il tasto Invio . Una GUI apparirà sullo schermo (Figura S3).
- In alto a sinistra della GUI, fai clic su Carica esperimento e seleziona il file JSON. Questo popolerà l'elenco dei file e il grafico Curve grezze che mostra il set di dati in termini di curve F-z . Entrambi gli assi F e z sono mostrati rispetto alle coordinate CP, che vengono calcolate in background durante il caricamento del set di dati (vedere la nota seguente). Nella casella Statistiche selezionare i valori dei tre parametri: Nattivato, N non riuscito e Nescluso.
NOTA: Nattivato rappresenta il numero di curve che verranno analizzate nella successiva analisi degli spettri di Hertz/elasticità e appariranno in nero nel grafico delle curve grezze . Nfallito rappresenta il numero di curve su cui non è stato possibile trovare un CP affidabile e appaiono in blu nel grafico. Queste curve verranno automaticamente scartate nell'analisi successiva. All'apertura del software, alcune curve possono essere spostate automaticamente sull'insieme fallito. Questo perché il CP viene calcolato all'apertura del software con l'algoritmo Threshold predefinito (vedi sotto). N escluso rappresenta le curve selezionate manualmente per essereescluse dall'analisi e verranno visualizzate in rosso nel grafico (vedere di seguito). - Verificare se il numero di curve fallite, escluse e attivate è ragionevole. Controllate il grafico Curve grezze per visualizzare le curve.
- Per visualizzare una curva specifica in modo più dettagliato, fare clic su di essa. Questo lo evidenzierà in verde e lo mostrerà sul grafico Curva corrente . Una volta selezionata una singola curva, i parametri R e k (che devono essere gli stessi per tutte le curve) verranno popolati nella casella Statistiche della GUI.
- Modificate lo stato di una determinata curva utilizzando la casella Attiva/disattiva (Toggle ). Fare clic sulla curva specifica di cui si desidera modificare lo stato, quindi fare clic su Attivato, Non riuscito o Escluso. Il conteggio nella casella Statistiche viene aggiornato automaticamente.
Nota : per modificare la visualizzazione del set di dati nel grafico Curve non elaborate , utilizzare la casella Visualizza ( View ). Fare clic su Tutte per visualizzare tutte le curve (ad esempio, attivate, fallite ed escluse nei rispettivi colori). Fare clic su Non riuscito per visualizzare le curve attivate e non riuscite e fare clic su Attivato per visualizzare solo le curve attivate. Per ripristinare lo stato di tutte le curve tra attivato ed escluso, fare clic su Attivato o Escluso nella casella Reset . - Una volta che il set di dati è stato ulteriormente pulito, seguire la pipeline di analisi dei dati descritta di seguito.
- Filtrare qualsiasi rumore presente nelle curve utilizzando i filtri implementati nella GUI (Filtering Box), ovvero un filtro personalizzato chiamato Prominency filter, il filtro Savitzky Golay30,31 (SAVGOL) e un filtro di smoothing basato sul calcolo della mediana dei dati in una determinata finestra (filtro mediano). Vedere Discussione (passaggi critici nel protocollo) per i dettagli sui filtri.
- Esaminate le curve filtrate nel grafico Curva corrente . La curva filtrata è mostrata in nero, mentre la versione non filtrata della curva è mostrata in verde.
NOTA: Si consiglia di filtrare i dati il meno possibile per preservare le caratteristiche del segnale originale. Il filtraggio eccessivo può appianare eventuali differenze presenti nei dati. Lavorare con il filtro di prominenza attivato è sufficiente per l'analisi Hertz dei dati di nanoindentazione. Se i dati sono particolarmente rumorosi, è possibile applicare anche un filtro SAVGOL o mediano. - Selezionare un algoritmo per trovare il CP. Nella casella Punto di contatto, scegliere una di una serie di procedure numeriche implementate nel software, ovvero la bontà di adattamento (GoF)32, il rapporto delle varianze (RoV)32, la derivata seconda 33 o la soglia 33. Vedere Discussione, passaggi critici nel protocollo per i dettagli sugli algoritmi.
NOTA: Il CP è il punto in cui la sonda entra in contatto con il materiale e deve essere identificato per convertire i dati F-z in dati F-δ (per i materiali morbidi, δ è finita e deve essere calcolata). L'algoritmo selezionato verrà applicato a tutte le curve attive del set di dati e le curve in cui l'algoritmo non riesce a individuare il CP in modo affidabile verranno spostate nell'insieme fallito. - Regolare i parametri dell'algoritmo in base al set di dati in modo che il CP sia posizionato correttamente come descritto nella discussione, passaggi critici nel protocollo. Per visualizzare dove è stato trovato il CP su una singola curva, selezionare la curva facendo clic su di essa e fare clic su Ispeziona. Controlla la finestra pop-up che appare per identificare dove si trova il CP.
- Controlla se la linea rossa che appare, che è il parametro calcolato dall'algoritmo nella regione di interesse selezionata, ha un valore massimo o minimo che corrisponde alla posizione del CP (ad esempio, per il GoF, il parametro è l'R2). Se necessario, ripetete questo processo per tutte le curve.
NOTA: gli assi del pop-up sono in coordinate assolute in modo che sia possibile visualizzare la posizione del CP. Al contrario, gli assi del grafico Curve grezze e Curva corrente sono mostrati rispetto al CP, cioè la posizione del CP è (0,0). - Fare clic su Analisi Hertz. Questo genererà tre grafici descritti di seguito.
- Controlla le singole curve F-δ nel set di dati insieme all'adattamento medio di Hertz (linea tratteggiata rossa). Regolare l'indentazione in nm fino alla quale è montato il modello Hertz nella casella Risultati sotto Rientro massimo (nm). Impostarlo su un massimo di ~10% di R affinché il modello Hertz sia valido (vedere Discussione, passaggi critici nel protocollo).
- Controllare la curva F-δ media con banda di errore che mostra una deviazione standard (SD) insieme all'adattamento medio di Hertz (linea tratteggiata rossa). Visualizzate l'adattamento medio di Hertz sul grafico Curve grezze come riferimento e l'adattamento Hertz per ogni curva sulla curva corrente.
- Controllate il grafico a dispersione di E originato dall'adattamento del modello di Hertz a ogni singola curva.
- Fare clic sul nome del file, sulla curva F-z, sulla curva F-δ o su un punto del grafico a dispersione per evidenziare la curva in ciascun grafico. Se un punto dati nel grafico a dispersione sembra trovarsi al di fuori della distribuzione dei dati, fare clic su di esso e ispezionare la curva a cui appartiene. Verificare che il CP sia stato posizionato correttamente facendo clic sul pulsante Ispeziona . Se necessario, escludete la curva dall'analisi.
- Ispezionare la casella Risultati per la media calcolata E e la sua SD (Eγ ± σ) e assicurarsi che siano ragionevoli per l'esperimento dato.
- Nella casella Salva , fai clic su Hertz. Nella finestra pop-up, inserisci il nome e la directory del file. Una volta fatto, fai clic su Salva. Verrà creato un file .tsv. Aprire il file .tsv in qualsiasi software aggiuntivo di preferenza e utilizzare i valori per l'analisi statistica e ulteriori grafici.
NOTA: il file contiene la E ottenuta da ogni curva e la media E e la relativa SD. Inoltre, il file contiene metadati associati all'analisi, incluso il numero di curve analizzate, R, k e il rientro massimo utilizzato per il modello di Hertz. - Questo passaggio è facoltativo. Fare clic su Media F-Ind per esportare la forza media e l'indentazione media, insieme a una SD nella forza.
- Per i dati di nanoindentazione cellulare, fare clic su Analisi spettri di elasticità (vedere Risultati rappresentativi e discussione). Ispezionare i due grafici prodotti, vale a dire, E in funzione della profondità di indentazione (E(δ)) per ogni curva, e la media E(δ) con banda di errore che mostra una SD (linea rossa continua e area ombreggiata) dotata di un modello (linea tratteggiata nera), che consente di stimare il modulo di Young della corteccia di actina della cellula, il modulo di Young della massa della cellula, e lo spessore della corteccia di actina. Inoltre, controlla la media E(δ) nel grafico in alto in rosso.
- Assicurarsi che la casella Interpolazione sia selezionata, il che garantisce che la derivata necessaria per eseguire l'analisi degli spettri di elasticità venga calcolata sul segnale interpolato (vedere Risultati rappresentativi).
- Ispezionare la casella Risultati, riportando il modulo di Young della corteccia (E 0 ± σ), il modulo di Young della massa della cellula (Eb ± σ) e lo spessore della corteccia (d 0 ± σ).
NOTA: Gli spettri di elasticità media possono apparire rumorosi all'inizio, con oscillazioni sinusoidali prominenti. Di conseguenza, l'equazione (3) potrebbe non essere adattata correttamente. In questo caso, l'aumento iterativo della lunghezza della finestra del filtro SAVGOL34 risolve questo problema. - Una volta terminata l'analisi, fare clic su ES nella casella Salva . Questo esporterà un file .tsv nella directory specificata, contenente l'elasticità media in funzione della profondità di rientro media e del raggio di contatto, i metadati associati all'esperimento (vedi sopra) e i parametri stimati del modello spiegati sopra. Infine, viene riportata anche l'elasticità media che non tiene conto della dipendenza da δ e dalla sua SD.
- Chiudere il software e inserire i risultati salvati in qualsiasi altro software di preferenza per tracciare ulteriormente i dati ed eseguire analisi statistiche.
- Questo passaggio è facoltativo: esportare i grafici dalla GUI facendo clic con il pulsante destro del mouse sul grafico e selezionando Esporta. Esporta il grafico in .svg, in modo che parametri come carattere, dimensione del carattere, stile della linea, ecc. può essere modificato in un altro software di scelta.
NOTA: gli algoritmi e i filtri CP personalizzati possono essere programmati e aggiunti a quelli già esistenti. Cfr. nota supplementare 1 per i dettagli.
Risultati
Seguendo il protocollo, si ottiene una serie di curve F-z . Il set di dati conterrà molto probabilmente curve valide e curve da scartare prima di continuare con l'analisi. In generale, le curve devono essere scartate se la loro forma è diversa da quella mostrata nella Figura 4A. La Figura 5AI mostra un set di dati di ~100 curve ottenute su un idrogel PAAm morbido di E 0,8 KPa35 previsto caricato nella GUI di NanoPrepar...
Discussione
Questo protocollo mostra come acquisire in modo robusto i dati di nanoindentazione della spettroscopia di forza utilizzando un nanopenetratore a ghiera disponibile in commercio sia su idrogel che su singole cellule. Inoltre, vengono fornite istruzioni per l'uso di un software open-source programmato in Python comprendente un preciso flusso di lavoro per l'analisi dei dati di nanoindentazione.
Passaggi critici nel protocollo
I seguenti passaggi sono stati identificati per es...
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
GC e MAGO riconoscono tutti i membri del CeMi. Gli Stati membri riconoscono il sostegno tramite una sovvenzione del programma EPSRC (EP/P001114/1).
GC: software (contributo allo sviluppo del software e degli algoritmi), analisi formale (analisi dei dati di nanoindentazione), validazione, Indagine (esperimenti di nanoindentazione su gel di poliacrilammide), data curation, scrittura (bozza originale, revisione e editing), visualizzazione (figure e grafici). MAGO: indagine (preparazione di gel e campioni di cellule, esperimenti di nanoindentazione su cellule), scrittura (bozza originale, revisione e modifica), visualizzazione (figure e grafici). NA: validazione, scrittura (revisione e modifica). IL: software (contributo allo sviluppo del software e degli algoritmi), validazione, scrittura (revisione e modifica); MV: concettualizzazione, software (progettazione e sviluppo di software e algoritmi originali), convalida, risorse, scrittura (bozza originale, revisione e modifica), supervisione, amministrazione del progetto, acquisizione di finanziamenti MSS: risorse, scrittura (revisione e modifica), supervisione, amministrazione del progetto, acquisizione di finanziamenti. Tutti gli autori hanno letto e approvato il manoscritto finale.
Materiali
| Name | Company | Catalog Number | Comments |
| 12 mm coverslips | VWR | 631-1577P | |
| 35 mm cell treated culture dishes | Greiner CELLSTAR | 627160 | |
| Acrylamide | Sigma-Aldrich | A4058 | |
| Acrylsilane | Alfa Aesar | L16400 | |
| Ammonium Persulfate | Merk | 7727-54-0 | |
| Bisacrylamide | Merk | 110-26-9 | |
| Chiaro nanoindenter | Optics 11 Life | no catologue number | |
| Ethanol | general | ||
| Fetal bovine serum | Gibco | 16140071 | |
| High glucose DMEM | Gibco | 11995065 | |
| Isopropanol | general | ||
| Kimwipe | Kimberly Clark | 21905-026 | |
| Microscope glass slides | VWR | 631-1550P | |
| MilliQ system | Merk Millipore | ZR0Q008WW | |
| OP1550 Interferometer | Optics11 Life | no catalogue number | |
| Optics 11 Life probe (k = 0.02-0.005 N/m, R = 3-3.5 um) | Optics 11 Life | no catologue number | |
| Optics 11 Life probe (k = 0.46-0.5 N/m, R = 50-55 um) | Optics 11 Life | no catologue number | |
| Penicillin/Streptomycin | Gibco | 15140122 | |
| RainX rain repellent | RainX | 26012 | |
| Standard petri dishes (90 mm) | Thermo Scientific | 101RTIRR | |
| Tetramethylethylenediamine | Sigma-Aldrich | 110-18-9 | |
| Vaccum dessicator | Thermo Scientific | 531-0250 | |
| Software | |||
| Data acquisition software (v 3.4.1) | Optics 11 Life | ||
| GitHub Desktop (Optional) | Microsoft | ||
| Python 3 | Python Software Foundation | ||
| Visual Studio Code (Optional) | Microsoft |
Riferimenti
- Discher, D. E., Janmey, P. Y. W. Tissue cells feel and respond to the stiffness of their substrate. Science. 310 (5751), 1139-1143 (2005).
- Roca-Cusachs, P., Conte, V., Trepat, X. Quantifying forces in cell biology. Nature Cell Biology. 19 (7), 742-751 (2017).
- Guimarães, C. F., Gasperini, L., Marques, A. P., Reis, R. L. The stiffness of living tissues and its implications for tissue engineering. Nature. Reviews. Materials. 5, 351-370 (2020).
- Moeendarbary, E., Harris, A. R. Cell mechanics: Principles, practices, and prospects. Wiley Interdisciplinary. Reviews. Systems. Biology and Medicine. 6 (5), 371-388 (2014).
- Butcher, D. T., Alliston, T., Weaver, V. M. A tense situation: Forcing tumour progression. Nature Reviews Cancer. 9 (2), 108-122 (2009).
- Kubánková, M., et al. Physical phenotype of blood cells is altered in COVID-19. Biophysical Journal. 120 (14), 2838-2847 (2021).
- Engler, A. J., Sen, S., Sweeney, H. L., Discher, D. E. Matrix elasticity directs stem cell lineage specification. Cell. 126, 677-689 (2006).
- Ciccone, G., et al. What caging force cells feel in 3D hydrogels: A rheological perspective. Advanced Healthcare Materials. 9 (17), 2000517 (2020).
- Wu, P. H., et al. A comparison of methods to assess cell mechanical properties. Nature Methods. 15 (7), 491-498 (2018).
- McKee, C. T., Last, J. A., Russell, P., Murphy, C. J. Indentation versus tensile measurements of young's modulus for soft biological tissues. Tissue Engineering B Reviews. 17 (3), 155-164 (2011).
- Humphrey, J. D., Delange, S. L. . An Introduction to Biomechanics Solids and Fluids, Analysis and Design. , 271-371 (2004).
- Prevedel, R., Diz-Muñoz, A., Ruocco, G., Antonacci, G. Brillouin microscopy: an emerging tool for mechanobiology. Nature Methods. 16 (10), 969-977 (2019).
- Krieg, M., et al. Atomic force microscopy-based mechanobiology. Nature Reviews Physics. 1, 41-57 (2019).
- Norman, M. D. A., Ferreira, S. A., Jowett, G. M., Bozec, L., Gentleman, E. Measuring the elastic modulus of soft culture surfaces and three-dimensional hydrogels using atomic force microscopy. Nature Protocols. 16 (5), 2418-2449 (2021).
- Gavara, N. A beginner's guide to atomic force microscopy probing for cell mechanics. Microscopy Research and Technique. 80 (1), 75-84 (2017).
- Whitehead, A. J., Kirkland, N. J., Engler, A. J. Atomic force microscopy for live-cell and hydrogel measurement. Methods in Molecular Biology. 2299, 217-226 (2021).
- Chavan, D., et al. Ferrule-top nanoindenter: An optomechanical fiber sensor for nanoindentation. The Review of Scientific Instruments. 83 (11), 115110 (2012).
- Van Hoorn, H., Kurniawan, N. A., Koenderink Ac, G. H., Iannuzzi, D. Local dynamic mechanical analysis for heterogeneous soft matter using ferrule-top indentation. Soft Matter. 12 (12), 3066-3073 (2016).
- Baldini, F., et al. Biomechanics of cultured hepatic cells during different steatogenic hits. Journal of the Mechanical Behavior of Biomedical Materials. 97, 296-305 (2019).
- Emig, R., et al. Piezo1 channels contribute to the regulation of human atrial fibroblast mechanical properties and matrix stiffness sensing. Cells. 10 (3), 663 (2021).
- Dobre, O., et al. A hydrogel platform that incorporates laminin isoforms for efficient presentation of growth factors - Neural growth and osteogenesis. Advanced Functional Materials. 31 (21), 2010225 (2021).
- Antonovaite, N., Beekmans, S. V., Hol, E. M., Wadman, W. J., Iannuzzi, D. Regional variations in stiffness in live mouse brain tissue determined by depth-controlled indentation mapping. Scientific Reports. 8 (1), 12517 (2018).
- Wu, G., Gotthardt, M., Gollasch, M. Assessment of nanoindentation in stiffness measurement of soft biomaterials: kidney, liver, spleen and uterus. Scientific Reports. 10 (1), 18784 (2020).
- Lüchtefeld, I., et al. Elasticity spectra as a tool to investigate actin cortex mechanics. Journal of Nanobiotechnology. 18 (1), 147 (2020).
- Caliari, S. R., Burdick, J. A. A practical guide to hydrogels for cell culture. Nature Methods. 13 (5), 405-414 (2016).
- Blumlein, A., Williams, N., McManus, J. J. The mechanical properties of individual cell spheroids. Scientific Reports. 7 (1), 7346 (2017).
- Mirsandi, H., et al. Influence of wetting conditions on bubble formation from a submerged orifice. Experiments in Fluids. 61, 83 (2020).
- Vassalli, M., Ciccone, G. . CellMechLab/NanoPrepare: v0.1.1. Zendo. , (2021).
- Vassalli, M., Ciccone, G., Lüchtefeld, I. CellMechLab/nanoindentation: v1.0.0. Zendo. , (2021).
- Savitzky, A., Golay, M. J. E. Smoothing and differentiation of data by simplified least squares procedures. Analytical Chemistry. 36 (8), 1627-1639 (1964).
- Schafer, R. W. What is a savitzky-golay filter. IEEE Signal Processing Magazine. 28 (4), 111-117 (2011).
- Gavara, N. Combined strategies for optimal detection of the contact point in AFM force-indentation curves obtained on thin samples and adherent cells. Scientific Reports. 6, 21267 (2016).
- Lin, D. C., Dimitriadis, E. K., Horkay, F. Robust strategies for automated AFM force curve analysis-I. Non-adhesive indentation of soft, inhomogeneous materials. Journal of Biomechanical Engineering. 129 (3), 430-440 (2007).
- Virtanen, P., et al. SciPy 1.0: fundamental algorithms for scientific computing in Python. Nature Methods. 17 (3), 261-272 (2020).
- Tse, J. R., Engler, A. J. Preparation of hydrogel substrates with tunable mechanical properties. Current Protocols in Cell Biology. , (2010).
- Pharr, G. M., Oliver, W. C., Brotzen, F. R. On the generality of the relationship among contact stiffness, contact area, and elastic modulus during indentation. Journal of Materials Research. 7 (3), 613-617 (1992).
- Kumar, R., Saha, S., Sinha, B. Cell spread area and traction forces determine myosin-II-based cortex thickness regulation. Biochimica et Biophysica Acta - Molecular Cell Research. 1866 (12), 118516 (2019).
- Kunda, P., Pelling, A. E., Liu, T., Baum, B. Moesin controls cortical rigidity, cell rounding, and spindle morphogenesis during mitosis. Current Biology: CB. 18 (2), 91-101 (2008).
- Allen, M., Poggiali, D., Whitaker, K., Marshall, T. R., Kievit, R. A. Raincloud plots: a multi-platform tool for robust data visualization. Wellcome Open Research. 4, 63 (2019).
- Clark, A. G., Paluch, E. Mechanics and regulation of cell shape during the cell cycle. Results and Problems in Cell Differentiation. 53, 31-73 (2011).
- Langan, T. J., Rodgers, K. R., Chou, R. C. Synchronization of mammalian cell cultures by serum deprivation. Methods in Molecular Biology. 1524, 97-105 (2017).
- Hodgkinson, T., et al. The use of nanovibration to discover specific and potent bioactive metabolites that stimulate osteogenic differentiation in mesenchymal stem cells. Science Advances. 7 (9), 7921 (2021).
- Kloxin, A. M., Kloxin, C. J., Bowman, C. N., Anseth, K. S. Mechanical properties of cellularly responsive hydrogels and their experimental determination. Advanced Materials (Deerfield Beach, Fla). 22 (31), 3484-3494 (2010).
- Loessner, D., et al. Functionalization, preparation and use of cell-laden gelatin methacryloyl-based hydrogels as modular tissue culture platforms. Nature Protocols. 11 (4), 727-746 (2016).
- Yue, K., et al. Synthesis, properties, and biomedical applications of gelatin methacryloyl (GelMA) hydrogels. Biomaterials. 73, 254-271 (2015).
- Sarrigiannidis, S. O., et al. A tough act to follow: collagen hydrogel modifications to improve mechanical and growth factor loading capabilities. Materials Today. Bio. 10, 100098 (2021).
- Karoutas, A., et al. The NSL complex maintains nuclear architecture stability via lamin A/C acetylation. Nature Cell Biology. 21 (10), 1248-1260 (2019).
- Ryu, H., et al. Transparent, compliant 3D mesostructures for precise evaluation of mechanical characteristics of organoids. Advanced Materials (Deerfield Beach, Fla). 33 (25), 2100026 (2021).
- Carvalho, D. T. O., Feijão, T., Neves, M. I., Da Silva, R. M. P., Barrias, C. C. Directed self-assembly of spheroids into modular vascular beds for engineering large tissue constructs Biofabrication Directed self-assembly of spheroids into modular vascular beds for engineering large tissue constructs. Biofabrication. 13 (3), 035008 (2021).
- Chaudhuri, O., Cooper-White, J., Janmey, P. A., Mooney, D. J., Shenoy, V. B. Effects of extracellular matrix viscoelasticity on cellular behaviour. Nature. 584 (7822), 535-546 (2020).
- Cantini, M., Donnelly, H., Dalby, M. J., Salmeron-Sanchez, M. The plot thickens: The emerging role of matrix viscosity in cell mechanotransduction. Advanced Healthcare Materials. 9 (8), 1901259 (2020).
- Elosegui-Artola, A. The extracellular matrix viscoelasticity as a regulator of cell and tissue dynamics. Current Opinion in Cell Biology. 72, 10-18 (2021).
- Chaudhuri, O. Viscoelastic hydrogels for 3D cell culture. Biomaterials Science. 5 (8), 1480-1490 (2017).
- Nguyen, T. D., Gu, Y. Determination of strain-rate-dependent mechanical behavior of living and fixed osteocytes and chondrocytes using atomic force microscopy and inverse finite element analysis. Journal of Biomechanical Engineering. 136 (10), 101004 (2014).
- Mokbel, M., Hosseini, K., Aland, S., Fischer-Friedrich, E. The Poisson Ratio of the Cellular Actin Cortex Is Frequency Dependent. Biophysical Journal. 118 (8), 1968-1976 (2020).
- Javanmardi, Y., Colin-York, H., Szita, N., Fritzsche, M., Moeendarbary, E. Quantifying cell-generated forces: Poisson's ratio matters. Communications Physics. 4, 237 (2021).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon