JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
연질 물질 나노인덴테이션을 위한 실험 및 데이터 분석 워크플로우
요약
이 프로토콜은 하이드로겔 및 세포를 포함한 연질 물질 나노인덴테이션 실험을 위한 완전한 워크플로우를 제공합니다. 첫째, 힘 분광법 데이터를 획득하기 위한 실험 단계가 자세히 설명되어 있습니다. 그런 다음 이러한 데이터의 분석은 GitHub에서 무료로 다운로드할 수 있는 새로 개발된 오픈 소스 Python 소프트웨어를 통해 자세히 설명됩니다.
초록
나노인덴테이션은 마이크로메트릭 힘 프로브를 사용하여 연질 생체 재료 및 세포의 국부적 기계적 특성을 정량화하는 실험 기술의 한 종류를 말합니다. 이 접근법은 기계 생물학, 생체 재료 설계 및 조직 공학 분야에서 중심적인 역할을 수행하여 단일 세포 크기 (μm)에 필적하는 해상도로 연질 재료의 적절한 기계적 특성을 얻습니다. 이러한 실험 데이터를 얻는 가장 인기있는 전략은 원자력 현미경 (AFM)을 사용하는 것입니다. 이 기기는 힘(pN까지) 및 공간(sub-nm)에서 전례 없는 분해능을 제공하지만 영률(E)과 같은 기계적 특성의 적분 지표의 일상적인 측정을 방해하는 복잡성으로 인해 유용성이 제한되는 경우가 많습니다. 광섬유 감지 기술을 기반으로 하는 것과 같은 차세대 나노인덴터는 최근 μm 공간 분해능으로 nN 이하 힘을 적용할 수 있는 동시에 통합이 용이하여 하이드로겔 및 세포의 국부적 기계적 특성을 조사하는 데 적합하다는 점에서 인기를 얻고 있습니다.
이 프로토콜에서는 상용 페룰 탑 광섬유 감지 나노인덴터를 사용하여 하이드로겔 및 세포에 대한 나노인덴테이션 데이터를 획득하는 실험 절차를 자세히 설명하는 단계별 가이드를 제공합니다. 일부 단계는 본원에 사용된 기기에 특정되는 반면, 제안된 프로토콜은 다른 나노인덴테이션 장치에 대한 가이드로서 취해질 수 있으며, 일부 단계는 제조업체의 가이드라인에 따라 적응될 수 있습니다. 또한 나노인덴테이션 데이터 분석을 위한 사용자 친화적인 그래픽 사용자 인터페이스를 갖춘 새로운 오픈 소스 Python 소프트웨어가 제공되어 잘못 획득된 곡선의 스크리닝, 데이터 필터링, 다양한 수치 절차를 통한 접점 계산, 기존의 E 계산, 특히 단일 세포 나노인덴테이션 데이터에 적합한 고급 분석이 가능합니다.
서문
생물학에서 역학의 근본적인 역할은 오늘날 확립되어 있습니다 1,2. 전체 조직에서 단일 세포에 이르기까지 기계적 특성은 조사중인 생체 재료의 병태 생리 학적 상태를 알릴 수 있습니다 3,4. 예를 들어, 암에 의해 영향을받는 유방 조직은 건강한 조직보다 뻣뻣하며, 이는 인기있는 촉진 테스트5의 기초가되는 개념입니다. 특히, 최근 중증 급성 호흡기 증후군 코로나바이러스 2(SARS-CoV-2)로 인한 코로나바이러스 질병 2019(COVID-19)는 SARS-CoV-2-순진한 개인의 혈액 세포와 비교하여 적혈구 변형성 감소, 림프구 및 호중구 경직 감소를 포함한 혈액 세포의 기계적 특성 변화로 강조되는 것으로 나타났습니다6.
일반적으로 세포와 조직의 역학은 본질적으로 얽혀 있습니다 : 각 조직은 구성 세포와 세포 외 기질 (ECM)5의 특성에 동시에 영향을 미치고 의존하는 특정 기계적 특성을 가지고 있습니다. 이 때문에 생물학에서 역학을 연구하는 전략에는 종종 이러한 자극에 대한 반응으로 세포 행동을 설명하기 위해 생리학적으로 관련된 기계적 자극을 가진 엔지니어링 기판이 포함됩니다. 예를 들어, Engler와 동료들의 정액 연구는 부드럽고 뻣뻣한 2차원 폴리아크릴아미드(PAAm) 하이드로겔에 대한 연구와 같이 중간엽 줄기 세포 혈통 헌신이 매트릭스 탄성에 의해 제어된다는 것을보여주었습니다7.
조사중인 생체 재료를 기계적으로 특성화하기위한 많은 전략이 존재하며, 공간 규모 (즉, 국부적 인 대형) 및 변형 모드 (예 : 축 방향 대 전단)가 다양하여 결과적으로 신중한 해석이 필요한 다른 정보를 산출합니다 3,8,9,10. 부드러운 생체 재료의 역학은 일반적으로 강성으로 표현됩니다. 그러나 강성은 재료 특성과 형상 모두에 의존하는 반면 탄성 계수는 재료의 기본 특성이며 재료의 기하학적(11)과 독립적입니다. 따라서 서로 다른 탄성 계수는 주어진 샘플의 강성과 관련이 있으며 각 탄성 계수는 서로 다른 경계 조건 (예 : 자유 팽창 대 감금)에서 특정 변형 모드 (예 : 축 방향 대 전단)에 대한 재료의 저항을 포함합니다 11,12. 나노인덴테이션 실험은 생체 재료가 측면으로 제한되지 않을 때 단축 변형(indentation)과 관련된 E를 통한 기계적 특성의 정량화를 가능하게 한다(10,11,12).
마이크로 스케일에서 생물학적 시스템의 E를 정량화하는 가장 보편적 인 방법은 AFM13,14,15,16입니다. AFM은 pN 수준까지의 힘 분해능과 서브 nm 규모의 공간 분해능을 갖춘 매우 강력한 도구입니다. 또한, AFM은 보완적인 광학 및 기계 도구와의 결합 측면에서 극도의 유연성을 제공하여 조사 중인 생체 재료로부터 풍부한 정보를 추출하는 기능을 확장합니다13. 그러나 이러한 매력적인 기능에는 실험 설정의 복잡성으로 대표되는 진입 장벽이 있습니다. AFM은 사용자가 강력한 데이터를 획득하기 전에 광범위한 교육이 필요하며, 특히 고유한 힘과 공간 분해능이 필요하지 않은 경우 생물학적 물질의 일상적인 기계적 특성화에 AFM을 사용하는 것이 정당화되지 않는 경우가 많습니다.
이 때문에 새로운 종류의 나노인덴터는 사용 편의성으로 인해 최근 인기를 얻었으며, 관련 길이척도에서 세포가 가하고 감지하는 힘을 반영하여 nN 이하 힘 분해능 및 μm 공간 분해능으로 AFM과 비교할 수 있는 데이터를 계속 제공합니다2. 특히, 광섬유 감지 기술17,18을 기반으로 한 페룰 탑 나노 압흔 장치는 기계 생물학 및 그 이상의 분야에서 활동하는 연구자들 사이에서 인기를 얻고 있습니다. 그리고 세포19,20, 하이드로겔8,21 및 조직22,23을 포함하여 이러한 장치를 사용하여 생체 재료의 기계적 특성을 보고하는 풍부한 연구가 출판되었습니다. 국부적 동적 기계적 특성(즉, 저장 및 손실 계수)을 조사하는 이러한 시스템의 기능에도 불구하고, E를 산출하는 준정적 실험은 여전히 가장 인기 있는 선택 8,19,20,21로 남아 있습니다. 간단히 말해서, 준 정적 나노인덴테이션 실험은 최대 변위, 힘 또는 압입 깊이로 정의된 설정점까지 일정한 속도로 샘플을 들여쓰기하고 캔틸레버의 힘과 수직 위치를 소위 힘-거리(F-z) 곡선으로 기록하는 것으로 구성됩니다. 그런 다음 F-z 곡선은 접촉점(CP)의 식별을 통해 힘-압입(F-δ) 곡선으로 변환되고 적절한 접촉 역학 모델(일반적으로 Hertz 모델13)을 장착하여 E를 계산합니다.
페룰 탑 나노인덴터의 작동은 AFM 측정과 유사하지만 고려해야 할 특수성이 있습니다. 이 연구에서는 상용 페룰 탑 나노인덴터를 사용하여 세포와 조직 모방 하이드로겔에서 F-z 곡선을 강력하게 획득하기 위한 단계별 가이드가 제공되어 이 장치와 다른 유사한 장치를 사용하는 연구 그룹 간의 실험 절차 표준화를 장려합니다. 또한 나노인덴테이션 실험을 수행하기 위해 하이드로겔 샘플과 세포를 가장 잘 준비하는 방법에 대한 조언과 실험 경로를 따른 문제 해결 팁이 제공됩니다.
또한, 나노인덴테이션 결과(즉, E 및 그 분포)의 많은 변동성은 데이터를 분석하는 데 사용되는 특정 절차에 따라 달라지며, 이는 사소하지 않습니다. 이 문제를 해결하기 위해 Python으로 프로그래밍되고 F-z 곡선의 배치 분석을 위한 사용자 친화적인 그래픽 사용자 인터페이스(GUI)가 장착된 새로 개발된 오픈 소스 소프트웨어 사용 지침이 제공됩니다. 이 소프트웨어는 빠른 데이터 스크리닝, 데이터 필터링, 다양한 수치 절차를 통한 CP 계산, E의 기존 계산 및 탄성 스펙트럼24라는 고급 분석을 허용하여 세포의 벌크 영률, 액틴 피질의 영률 및 액틴 피질의 두께를 추정 할 수 있습니다. 이 소프트웨어는 GitHub에서 자유롭게 다운로드 할 수 있으며 적절한 데이터 파서를 추가하여 다른 시스템에서 발생하는 데이터를 분석하는 데 쉽게 적용 할 수 있습니다. 이 프로토콜은 다른 페룰 탑 나노인덴테이션 장치 및 일반적으로 다른 나노인덴테이션 장치에 사용될 수 있으며 일부 단계는 특정 기기의 지침에 따라 조정됩니다. 프로토콜은 그림 1에 개략적으로 요약되어 있습니다.
프로토콜
1. 나노인덴테이션 측정을 위한 기질/세포의 준비
- 나노인덴테이션 실험을 위한 PAAm 하이드로겔/세포 준비를 위한 보충 프로토콜 에 제공된 단계를 따르십시오. 절차는 그림 2에 요약되어 있습니다.
참고: PAAm 하이드로겔은 기계생물학 분야에서 사용되는 가장 일반적인 하이드로겔이기 때문에 선택되었습니다. 그러나, 상기 프로토콜은 모든 유형의 히드로겔(25 )에 동일하게 적용가능하다( 방법의 논의, 변형 참조).
2. 장치 시작, 프로브 선택 및 프로브 교정
- 장치를 시작하기 위한 보충 프로토콜에 제공된 단계를 따릅니다. 광섬유 페룰 탑 나노인덴터의 작동에 대한 기술적 세부 정보는 다음 참조17,18을 확인하십시오.
- 아래 설명된 대로 나노인덴테이션 프로브를 선택합니다.
참고: 이 프로토콜에 사용되는 것과 같이 상업적으로 이용 가능한 모든 프로브에는 구형 팁이 장착되어 있습니다(그림 3A). 따라서 캔틸레버의 강성과 팁 반경이라는 두 가지 변수로 선택 범위가 좁혀집니다(그림 3B).- 최상의결과를 위해 예상되는 샘플 강성과 일치하는 캔틸레버의 강성(k in N/m)을 선택하십시오(그림 3C 및 토론, 프로토콜의 중요한 단계 참조). 세포의 경우 k가 0.01-0.09 N / m 범위 인 프로브를 선택하십시오. 하이드로겔의 경우 0.1-0.9N/m 범위의 k를 가진 프로브를 선택하면 몇 kPa에서 100kPa 사이의 예상 E를 가진 젤에 대해 최적의 결과를 얻을 수 있습니다(대표 결과 참조).
- 압입 공정의 원하는 공간 분해능에 따라 팁 반경(R in μm)을 선택합니다. 인간 배아 신장 293T (HEK293T) 세포 (평균 직경 ~ 10-15 μm26)와 같은 작은 세포의 경우 R = 3 μm의 구를 선택하십시오. 하이드로겔의 경우 R = 10-250 μm인 구를 선택하여 넓은 접촉 영역에서 생체 재료의 기계적 특성을 조사하고 국소 이질성을 피하십시오.
- 그림 3C 및 추가 제조업체의 지침을 참조하여 적절한 프로브를 선택하십시오.
- 프로브가 선택되면 보충 프로토콜 의 단계에 따라 나노인덴터에 장착합니다.
3. 프로브 교정
참고: 다음 단계는 광섬유 감지 기술을 기반으로 하는 페룰 탑 나노인덴테이션 장치에만 해당되며 소프트웨어 버전 3.4.1에 대해 자세히 설명되어 있습니다. 다른 나노인덴테이션 장치의 경우 장치 제조업체에서 권장하는 단계를 따르십시오.
- 소프트웨어의 기본 창에서 초기화를 클릭합니다. 보정 메뉴가 나타납니다. 프로브 세부 정보를 입력하십시오 (프로브 상자 측면에서 찾을 수 있음). n /m의 k, μm의 R 및 공기의 교정 계수)를 입력 상자에 입력합니다.
- 교정 접시를 준비하십시오 : 평평한 바닥이있는 두꺼운 유리 페트리 접시 ( 토론, 프로토콜의 중요한 단계 참조). 샘플 접시와 동일한 매체로 접시를 채 웁니다 (공기 일 수도 있음). 매체의 온도를 샘플의 온도와 일치시킵니다.
- 프로브 아래에 교정 접시를 놓습니다. 필요한 경우 나노인덴터의 암 홀더에서 프로브를 밀어내고 한 손으로 잡고 교정 접시를 놓을 공간을 만듭니다. 프로브를 제자리에 밀어 넣습니다.
- 액체 교정을 위해 다음 두 단계를 수행하십시오. 공기 중에서 측정하는 경우 제공된 폴리테트라플루오로에틸렌 기판을 사용하여 교정하고 5단계로 건너뜁니다.
- 피펫의 끝이 유리 페룰과 가볍게 접촉하도록 파스퇴르 피펫을 사용하여 70% 에탄올 방울로 프로브를 미리 적셔서 방울이 캔틸레버 및 구형 팁27 위로 미끄러지도록 합니다( 토론, 프로토콜의 중요한 단계 참조).
- 프로브가 완전히 잠길 때까지 나노인덴터의 팔을 수동으로 아래쪽으로 밀지만 페트리 접시의 바닥에서 멀리 떨어져 있습니다. 필요한 경우 보정 접시에 유체를 더 추가합니다. 액체에서 평형 조건에 도달하도록 5분 동안 기다립니다.
- 소프트웨어의 초기화 메뉴에서 스캔 파장을 클릭합니다. 간섭계 화면에는 진행률 표시줄이 표시되고 컴퓨터 소프트웨어의 라이브 신호 창에는 그림 S1A(왼쪽)와 같은 패턴이 표시됩니다. 광학 스캔이 성공했는지 확인하려면 간섭계 상자의 파장 스캔 패널로 이동합니다. 성공하면 사인파가 표시되어야 합니다(그림 1A, 오른쪽). 오류가 나타나는 경우 토론 (방법 문제 해결)을 참조하십시오.
- 초기화 메뉴에서 표면 찾기를 클릭하면 캔틸레버 굽힘의 설정된 임계값에 도달할 때까지 프로브가 점진적으로 낮아집니다. 프로브는 유리 페트리 접시와 접촉 할 때 움직임을 멈 춥니 다.
- 프로브가 표면과 접촉했는지 확인하십시오. 소프트웨어의 메인 창에 있는 y 아래쪽 화살표 버튼을 사용하여 프로브를 1μm 아래로 이동합니다. 캔틸레버가 기판과 접촉할 때 각 단계에 따른 기준선의 변화에 대해 소프트웨어의 라이브 창에서 녹색 신호(캔틸레버 편향)를 관찰합니다(그림 S1B). 변경 사항이 없으면 캔틸레버가 접촉하지 않습니다(다음 단계 참조).
- 지표면 찾기 탭의 옵션 메뉴에서 임계값을 기본값인 0.01에서 한 번에 0.01씩 늘리고 접촉할 때까지 지표면 찾기 단계를 반복합니다. 또는 녹색 기준선이 각 아래쪽 단계에서 이동하기 시작할 때까지 작은 1μm 단계로 접촉할 프로브를 내립니다.
참고: 가장 부드러운 캔틸레버(k = 0.025 Nm-1)의 경우 6단계를 수행하기 전에 표면 찾기 탭 아래의 옵션 메뉴에서 임계값을 늘립니다. 임계값 0.06 또는 0.07에서 시작하여 필요한 경우 최대 0.1까지 늘립니다. 이는 환경 소음으로 인해 캔틸레버가 접촉하기 전에 임계 값 이상으로 구부러 질 수 있기 때문입니다. 소프트 프로브의 경우 동일한 메뉴에서 접근 속도(μm/s)를 낮추면 절차도 향상됩니다. - 초기화 메뉴에서 보정을 클릭합니다.
- 소프트웨어의 라이브 신호 창을 확인하고 피에조의 변위와 캔틸레버의 편향 신호가 동시에 위로 이동하는지 확인하십시오 (그림 S1C).
- 시간이 일치하지 않으면 프로브가 유리와 완전히 접촉하지 않습니다. 캔틸레버 신호의 기준선이 변경될 때까지 프로브를 1μm 간격으로 아래로 이동하고(7단계 참조) 9단계를 반복합니다.
- 보정 단계에서 캔틸레버 신호가 전혀 변경되지 않으면 프로브가 표면에서 멀리 떨어져 있습니다. 표면이 올바르게 발견될 때까지 접촉 임계값을 반복적으로 늘리고(8단계 참조) 파장 스캔부터 시작하여 처음부터 보정을 반복합니다.
- 보정이 완료되면 표시된 팝업 창에서 이전 보정 계수와 새 보정 계수를 확인합니다. 새 보정 계수가 올바른 범위에 있으면 새 계수 사용을 클릭합니다. 보정이 실패하고 새 계수가 NaN이거나 예상 범위에 없는 경우 해결 방법에 대한 논의(방법 문제 해결)를 참조하십시오.
알림: 새로운 계수는 굴절률이 n (물의 경우 n = 1.33)의 액체 매체에서 교정을 수행 한 경우 프로브 상자에 제공된 계수보다 ~ n 배 낮아야합니다. 공기 중에서 교정을 수행 한 경우 새 교정 계수와 기존 교정 계수는 거의 동일해야합니다. - 복조 원이 다음과 같이 올바르게 보정되었는지 확인하십시오. 간섭계 데스크탑의 복조 탭으로 이동합니다. 광학 테이블이나 나노인덴터를 부드럽게 두드려 충분한 소음을 유도합니다. 불연속 데이터 요소로 구성된 흰색 원은 대략 빨간색 원을 덮어야 합니다(그림 S1D).
- 흰색 원이 빨간색 원과 겹치지 않거나 간섭계의 디스플레이에 경고가 나타나면 복조 원을 다시 보정해야 합니다. 이것은 아래에 설명 된대로 두 가지 방법으로 수행 할 수 있습니다.
- 나노인덴터의 본체를 계속 탭하여 하나의 완전한 노이즈 원을 유도하고 간섭계의 보정 버튼을 누릅니다.
- 유리 기판에 접촉하여 입력하고 보정 소프트웨어 기본 창의 초기화 메뉴에서. 보정 계수를 저장하지 마십시오. 이 시점에서 다시 확인하고 흰색 원이 빨간색 원과 겹치는지 확인하십시오.
알림: 신호가 복조 원에서 약간 변위된 것이 아니라 매우 작아지거나 전혀 보이지 않으면 캔틸레버가 광섬유에 붙어 있음을 의미합니다. 이 문제점에 대한 문제점 해결 조언( 방법 논의, 문제점 해결 참조)을 따르고 13.1 또는 13.2단계를 반복하십시오. 캔틸레버가 수평 위치로 돌아 오면 (그림 3A) 신호가 복조 원으로 복구됩니다.
- 아래 설명된 대로 교정 직후 유리 기판에 압입(indentation)을 수행하여 교정을 확인합니다.
- 실험 구성을 클릭하여 실험 파일을 로드하거나 만들고 표면 찾기 단계 및 들여쓰기 단계를 추가합니다. 압입 단계의 경우 기본 변위 모드 설정을 사용하고 최대 변위를 교정 거리(3,000nm)로 변경하여 프로브를 뻣뻣한 기판에 대해 변위시킵니다.
- 실험 실행을 클릭하고 간섭계 창에서 복조 원을 확인합니다. 흰색 신호를 확인하고 들여 쓰기 중에 빨간색 원 위에 있는지 확인하십시오.
- 시간 데이터 그래프에서 소프트웨어의 기본 창에서 결과를 확인하고 압입자의 변위와 같은지 확인하십시오 (파란색 선) 캔틸레버의 처짐 (녹색 선) 압입이 접촉하기 시작하고 재료 변형이 예상되지 않음. 신호가 병렬이 아닌 경우 토론(방법 문제 해결)을 참조하십시오.
- 보정 메뉴에서 로컬 경로를 변경하려면 보정 저장 경로를 적절한 디렉터리로 설정합니다.
- 프로브가 성공적으로 보정되면, 피에조를 위로 이동 500 μm.
4. 부드러운 재료의 영률 측정
- 하이드로겔의 나노인덴테이션
- 샘플이 들어 있는 페트리 접시를 현미경 스테이지에 놓고 나노인덴터의 프로브를 샘플 위의 원하는 xy 위치로 수동으로 이동합니다.
- 이 단계에서 프로브와 샘플 표면 사이에 1-2mm를 남겨 두면서 용액에 프로브를 수동으로 밀어 넣습니다. 프로브가 매체에서 평형을 이룰 때까지 5분 동안 기다립니다.
- 프로브가 명확하게 보이도록 광학 현미경의 z 평면에 초점을 맞춥니다.
- 단일 들여쓰기를 수행하여 아래에 설명된 대로 실험 매개변수를 조정합니다.
- 소프트웨어의 기본 창에서 새 실험을 구성합니다. 실험 구성을 클릭하면 새 창이 열립니다. 지표면 찾기 단계를 추가합니다. 필요한 경우 지표면 찾기 단계의 모든 매개변수를 소프트웨어의 옵션 메뉴에서 변경할 수 있습니다.
알림: 표면 찾기 는 표면이 발견 될 때까지 프로브를 내린 다음 샘플 표면 위의 Z 표면 위 (μm)로 정의 된 거리로 프로브를 수축시킵니다. 선택한 캔틸레버가 샘플에 비해 너무 뻣뻣하거나 샘플이 끈적 거리면 단계 후에도 프로브가 여전히 샘플과 접촉 할 가능성이 높으며 이로 인해 기준선이없는 곡선이 생성됩니다 (그림 4C). 이 문제를 해결하려면 표면 위의 Z (μm)를 늘리십시오. - 들여쓰기 단계를 추가합니다. 프로파일 탭을 선택하고 변위 제어를 클릭합니다. 기본 들여쓰기 프로필을 그대로 둡니다.
- 기본 소프트웨어 창에서 실험 실행을 클릭합니다. 그러면 표면을 찾고 단일 들여 쓰기가 수행됩니다. 단일 압흔이 예상대로 보이지 않으면 그림 4 및 토론 (방법 문제 해결)에 설명된 대로 실험 매개변수를 조정합니다.
- 소프트웨어의 기본 창에서 새 실험을 구성합니다. 실험 구성을 클릭하면 새 창이 열립니다. 지표면 찾기 단계를 추가합니다. 필요한 경우 지표면 찾기 단계의 모든 매개변수를 소프트웨어의 옵션 메뉴에서 변경할 수 있습니다.
- 들여쓰기가 원하는 대로 보이면 샘플의 충분한 영역이 들여쓰기되도록 매트릭스 스캔을 구성합니다. 실험 구성을 클릭하고 이전에 결정된 실험 매개변수가 있는 표면 찾기 단계를 추가하고 매트릭스 스캔 단계를 추가합니다.
- 평평한 하이드로겔의 경우 10-100μm(즉, dx = dy = 10-100μm)에 간격을 둔 50-100점(즉, x 10 x 10 또는 10 x 10 x y)을 포함하는 매트릭스 스캔을 구성합니다. 스테이지 위치 사용을 클릭하여 현재 스테이지 위치에서 매트릭스 스캔을 시작합니다. 설정된 실험 매개변수를 사용하여 각 들여쓰기에서 표면을 찾으려면 표면 자동 찾기 상자가 선택되어 있는지 확인합니다.
- 과잉 샘플링을 방지하려면 스텝 크기를 접촉 반경의 두 배 이상으로 설정합니다(
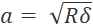 여기서 δ 는 압입 깊이임).
여기서 δ 는 압입 깊이임). - 변위 제어에서 매트릭스 스캔 프로파일을 설정합니다. 프로파일이 Hertz 모델의 가정을 위반하지 않는지 확인합니다( 토론, 프로토콜의 중요한 단계 참조).
- 세그먼트 수를 기본값인 5로 두고 기본 변위 프로파일을 사용합니다. 필요한 경우 각 경사 세그먼트의 최대 변위 및 시간 측면에서 변위 프로파일을 변경하면 각각 최대 압입 깊이와 변형률에 영향을 미칩니다. 변형률 속도가 10 μm/s > 초과하지 마십시오( 방법의 논의, 한계 참조).
- 접촉하기 전에 프로브가 샘플 쪽으로 얼마나 빨리 변위되는지를 결정하는 접근 속도 값을 입력합니다. 후퇴 속도를 접근 속도와 일치시키십시오(아래 참고 참조).
알림: 부드러운 캔틸레버 및 시끄러운 환경의 경우 1,000-2,000nm/s의 접근 속도가 권장됩니다. 더 단단한 캔틸레버와 통제된 환경의 경우 이를 증가시킬 수 있습니다. - 구성된 실험을 원하는 실험 경로에 저장하고 실험 구성 창의 일반 탭에 있는 경로 저장에서 데이터를 저장할 디렉터리를 선택합니다. 실험 실행을 클릭합니다.
- 과잉 샘플링을 방지하려면 스텝 크기를 접촉 반경의 두 배 이상으로 설정합니다(
- 매트릭스 스캔이 완료되면 프로브를 200-500 μm 올리고 프로브를 첫 번째 영역에서 충분히 멀리 떨어진 샘플의 다른 영역으로 이동합니다.
- 각 샘플에서 충분한 데이터가 수집되도록 실험을 최소 2회 반복합니다(즉, 샘플당 최소 2개의 매트릭스 스캔, 각각 50-100개의 곡선 포함).
- 세포의 나노인덴테이션
- 위에서 설명한 대로 현미경에 샘플을 로드합니다.
- 단일 셀 압입의 경우 셀 크기 및 확산에 따라 셀과 프로브가 모두 20x 또는 40x 배율로 보이도록 z 평면에 초점을 맞춥니다.
- 들여쓸 셀 위로 프로브를 이동합니다.
- 소프트웨어의 기본 창에서 새 실험을 구성합니다. 실험 구성을 클릭하면 새 창이 열립니다. 지표면 찾기 및 들여쓰기 단계를 변위 모드에서 기본 매개변수가 있는 단계로 추가합니다.
- Run Experiment(실험 실행)를 클릭하면 표면을 찾고 단일 들여쓰기를 수행합니다. 들여쓰기가 성공했는지 확인합니다. 곡선이 예상대로 보이지 않으면 실험 매개 변수를 조정하십시오 (그림 4 및 토론, 방법 문제 해결 참조).
- 들여쓰기에 성공하면 실험에 매트릭스 스캔을 추가합니다. 하이드로겔의 나노인덴테이션 실험에 주어진 단계를 따르십시오. 단계 크기가 셀의 작은 영역을 들여 쓸 수 있도록 매트릭스 스캔을 구성하십시오. HEK293T 세포의 경우 0.5-5 μm에서 25 점 간격.
- 셀 크기에 따라 매트릭스 스캔을 조정하여 팁이 셀 한계를 벗어나지 않도록 합니다(예: 다른 맵 지오메트리를 수행하거나 더 적은 수의 점을 프로빙).
- 실험 실행을 클릭하고 완료 될 때까지 기다립니다.
- 매트릭스 스캔이 완료되면 프로브를 접촉에서 들어 올립니다( z 평면에서 50μm).
- 프로브를 새 셀 위로 이동하고 프로세스를 반복합니다( 토론, 프로토콜의 중요한 단계 참조).
- 프로브 청소 및 기기 끄기
- 보충 프로토콜에 제공된 단계에 따라 프로브를 청소하고 나노인덴테이션 장치를 끕니다.
5. 데이터 분석
- 소프트웨어 다운로드 및 설치
- 보충 프로토콜에 제공된 단계에 따라 데이터 분석용 소프트웨어를 다운로드하고 설치합니다28,29.
- F-z 곡선 스크리닝 및 정리된 데이터 세트를 JSON 형식으로 생성
- 랩 컴퓨터의 명령줄에서 2-3 단계에 설명된 대로 prepare.py 시작합니다.
- Windows 컴퓨터를 사용하는 경우 Shift 키를 누른 상태에서 NanoPrepare 폴더를 마우스 오른쪽 버튼으로 클릭하고 여기에서 PowerShell 창 열기를 클릭합니다. 파이썬 prepare.py 명령을 입력하고 엔터 버튼 키. 화면에 GUI가 나타납니다 (그림 S2).
- MacOS 컴퓨터를 사용하는 경우 NanoPrepare 폴더를 마우스 오른쪽 버튼으로 클릭하고 폴더에서 새 터미널을 클릭합니다. python3 prepare.py 명령을 입력하고 Enter 키를 누르면 GUI가 시작됩니다(그림 S2).
- 드롭다운 목록에서 O11NEW 데이터 형식을 선택합니다. 데이터가 올바르게 로드되지 않은 경우 GUI를 다시 시작하고 O11OLD를 선택합니다.
참고: O11NEW 형식은 소프트웨어 버전 3.4.1의 페룰 탑 광섬유 감지 나노인덴테이션 장치를 사용하여 얻은 데이터에 대해 작동합니다. 이 형식은 이전 소프트웨어 버전, 적어도 2019-2020 년에 설치된 나노인덴터에 속하는 버전에서도 작동합니다. - 폴더 로드를 클릭합니다. 분석할 데이터가 포함된 폴더(단일 매트릭스 스캔 또는 다중 매트릭스 스캔)를 선택합니다. 상단 그래프(원시 곡선)는 업로드된 데이터 세트로 채워집니다. 특정 곡선을 시각화하려면 클릭하십시오. 그러면 녹색으로 강조 표시되고 하단 그래프 (현재 곡선)에 표시됩니다.
- 아래에 설명된 대로 GUI 오른쪽에 있는 탭을 사용하여 데이터 세트를 정리합니다.
- 세그먼트 버튼을 사용하여 분석할 올바른 세그먼트( F-z 곡선의 순방향 세그먼트)를 선택합니다. 구체적인 숫자는 실험을 수행할 때 나노인덴테이션 소프트웨어에서 선택한 세그먼트 수에 따라 다릅니다.
- 50nm 자르기 버튼을 사용하여 맨 왼쪽(L을 선택한 경우), 오른쪽(R을 선택한 경우) 또는 양쪽(R과 L을 모두 선택한 경우)에서 곡선을 50nm씩 자릅니다. 이 버튼을 여러 번 클릭하여 필요한 만큼 자를 수 있습니다. F-z 커브의 시작/끝에 있는 아티팩트를 제거하려면 이 옵션을 사용합니다.
- 캔틸레버 탭에서 스프링 상수, 팁 형상 및 팁 반지름을 검사합니다. 탭을 검사하여 메타데이터를 올바르게 읽었는지 확인합니다.
- 스크리닝(Screening) 탭을 사용하여 지정된 힘에 도달하지 않은 모든 커브를 버릴 힘 임계값을 설정합니다. 버려진 곡선은 빨간색으로 강조 표시됩니다.
- 수동 토글 버튼을 사용하여 올바르게 획득되지 않은 곡선을 수동으로 제거합니다. 특정 곡선을 클릭하고 OUT을 선택하여 곡선을 제거하면 곡선이 빨간색으로 강조 표시됩니다.
- JSON 저장을 클릭합니다. 정리된 데이터 세트의 적절한 이름(단일 JSON 파일)을 입력합니다. JSON 파일을 나노분석 소프트웨어가 설치된 컴퓨터로 보냅니다.
6. 공식 데이터 분석
- 앞에서 설명한 대로 NanoAnalysis 폴더로 이동하고 터미널을 시작하여 명령줄에서 nano.py 파일을 실행합니다. python nano.py 또는 python3 nano.py 명령(운영 체제에 따라 다름)을 입력하고 Enter 키를 누릅니다. 화면에 GUI가 나타납니다 (그림 S3).
- GUI의 왼쪽 상단에서 실험 로드를 클릭하고 JSON 파일을 선택합니다. 그러면 파일 목록과 F-z 곡선으로 데이터 세트를 보여주는 원시 곡선 그래프가 채워집니다. F축과 z축은 모두 CP 좌표를 기준으로 표시되며, 이는 데이터 세트를 로드할 때 백그라운드에서 계산됩니다(다음 참고 참조). 통계 상자에서 세 매개 변수의 값(N활성화, N 실패 및 N제외)을 선택합니다.
참고:활성화된 N은 후속 Hertz/탄성 스펙트럼 분석에서 분석될 곡선의 수를 나타내며 원시 곡선 그래프에서 검은색으로 표시됩니다. Nfailed 는 신뢰할 수 있는 CP를 찾을 수 없고 그래프에서 파란색으로 표시되는 곡선의 수를 나타냅니다. 이러한 곡선은 후속 분석에서 자동으로 삭제됩니다. 소프트웨어를 열면 일부 곡선이 자동으로 실패한 세트로 이동할 수 있습니다. 이는 CP가 기본 임계값 알고리즘으로 소프트웨어를 열 때 계산되기 때문입니다(아래 참조). N 제외는 분석에서제외 되도록 수동으로 선택한 곡선을 나타내며 그래프에서 빨간색으로 표시됩니다(아래 참조). - 실패, 제외 및 활성화된 곡선의 수가 적절한지 확인합니다. 원시 곡선 그래프를 확인하여 곡선 을 시각화합니다.
- 특정 곡선을 더 자세히 시각화하려면 클릭하십시오. 그러면 녹색으로 강조 표시되고 현재 곡선 그래프에 표시됩니다. 단일 커브가 선택되면 R 및 k 매개변수(모든 커브에 대해 동일해야 함)가 GUI의 통계 상자에 채워집니다.
- 토글 상자를 사용하여 지정된 곡선의 상태를 변경합니다. 상태를 변경하려는 특정 곡선을 클릭한 다음 활성화됨, 실패 또는 제외됨을 클릭합니다. 통계 상자의 개수가 자동으로 업데이트됩니다.
참고: 원시 곡선 그래프에서 데이터 세트의 보기를 변경하려면 보기 상자를 사용합니다. 모두를 클릭하여 모든 곡선을 표시합니다(즉, 활성화됨, 실패됨, 해당 색상에서 제외됨). Failed(실패)를 클릭하여 활성화된 커브와 실패한 커브를 표시하고 Activated 커브를 클릭하여 활성화된 커브만 표시합니다. 활성화된 커브와 제외된 커브 사이의 모든 커브 상태를 재설정하려면 재설정 상자에서 활성화 또는 제외됨을 클릭합니다. - 데이터 세트가 추가로 정리되면 아래에 설명된 데이터 분석 파이프라인을 따릅니다.
- GUI(필터링 상자)에 구현된 필터, 즉 돌출 필터라는 사용자 지정 필터, Savitzky Golay30,31(SAVGOL) 필터 및 주어진 창에서 데이터의 중앙값 계산을 기반으로 하는 평활화 필터(중앙값 필터)를 사용하여 곡선에 존재하는 노이즈를 필터링합니다. 필터에 대한 자세한 내용은 토론(프로토콜의 중요한 단계)을 참조하십시오.
- 현재 곡선 그래프에서 필터링된 곡선 을 검사합니다. 필터링된 곡선은 검은색으로 표시되고 필터링되지 않은 버전의 곡선은 녹색으로 표시됩니다.
참고: 원래 신호의 특징을 보존하기 위해 데이터를 가능한 한 적게 필터링하는 것이 좋습니다. 과도한 필터링은 데이터에 존재하는 차이를 완화할 수 있습니다. 돌출 필터를 활성화한 상태에서 작업하면 나노인덴테이션 데이터의 Hertz 분석에 충분합니다. 데이터가 특히 잡음이 있는 경우 SAVGOL 또는 중앙값 필터를 추가로 적용할 수 있습니다. - CP를 찾을 알고리즘을 선택합니다. 접점 상자에서 소프트웨어에서 구현된 일련의 수치 절차, 즉 적합도(GoF)32, 분산 비율(RoV)32, 2차 도함수(33) 또는 임계값 33 중 하나를 선택합니다. 알고리즘에 대한 자세한 내용은 토론, 프로토콜의 중요한 단계를 참조하십시오.
알림: CP는 프로브가 재료와 접촉하는 지점이며 F-z 데이터를 F-δ 데이터로 변환하기 위해 식별해야 합니다(연질 재료의 경우 δ는 유한하며 계산해야 함). 선택한 알고리즘은 데이터 세트의 모든 활성 곡선에 적용되며 알고리즘이 CP를 확실하게 찾지 못한 곡선은 실패한 세트로 이동합니다. - CP가 토론, 프로토콜의 중요한 단계에 자세히 설명된 대로 올바르게 배치되도록 알고리즘의 매개변수를 데이터 세트에 맞게 조정합니다. 단일 커브에서 CP가 발견된 위치를 보려면 커브를 클릭하여 선택하고 검사(Inspect)를 클릭합니다. 나타나는 팝업 창을 확인하여 CP가 있는 위치를 식별합니다.
- 알고리즘이 선택된 관심 영역에서 계산한 파라미터인 적색 선이 CP의 위치에 해당하는 최대값 또는 최소값을 갖는지 검사한다(예를 들어, GoF의 경우, 파라미터는 R2이다). 필요한 경우 모든 곡선에 대해 이 과정을 반복합니다.
참고: 팝업의 축은 CP의 위치를 표시할 수 있도록 절대 좌표에 있습니다. 반대로, 원시 곡선 및 전류 곡선 그래프의 축은 CP에 상대적으로 표시되며, 즉 CP의 위치는 (0,0)입니다. - 헤르츠 해석을 클릭합니다. 그러면 아래에 설명된 세 개의 그래프가 생성됩니다.
- 데이터셋의 개별 F-δ 곡선을 평균 헤르츠 피팅(빨간색 파선)과 함께 확인합니다. Hertz 모델이 장착되는 들여쓰기를 최대 압입(nm) 아래의 결과 상자에 조정합니다. Hertz 모델이 유효하려면 R의 최대 ~10%로 설정합니다(토론, 프로토콜의 중요한 단계 참조).
- 평균 헤르츠 피팅(빨간색 점선)과 함께 하나의 표준 편차(SD)를 보여주는 오차 대역이 있는 평균 F-δ 곡선을 확인합니다. 참조를 위해 원시 곡선 그래프의 평균 Hertz 피팅과 현재 곡선의 각 커브에 대한 Hertz 피팅을 시각화합니다.
- Hertz 모델을 각 개별 곡선에 피팅한 데서 발생하는 E 의 산점도를 확인합니다.
- 파일 이름, F-z 곡선, F-δ 곡선 또는 산점도의 점을 클릭하여 각 플롯의 곡선을 강조 표시합니다. 산점도의 데이터 점이 데이터 분포 외부에 있는 것처럼 보이면 해당 점을 클릭하고 해당 요소가 속한 곡선을 검사합니다. 검사 버튼을 클릭하여 CP가 올바르게 배치되었는지 확인합니다. 필요한 경우 분석에서 곡선을 제외합니다.
- 결과 상자에서 계산된 평균 E와 해당 SD(E γ ± σ)를 검사하고 지정된 실험에 적합한지 확인합니다.
- 저장 상자에서 Hertz를 클릭합니다. 팝업 창에서 파일 이름과 디렉토리를 입력합니다. 완료되면 저장을 클릭하십시오. .tsv 파일이 생성됩니다. 기본 설정 추가 소프트웨어에서 .tsv 파일을 열고 통계 분석 및 추가 플로팅에 값을 사용합니다.
참고: 이 파일에는 각 곡선 에서 얻은 E와 평균 E 및 SD가 포함되어 있습니다. 또한 이 파일에는 분석된 곡선 수, R, k 및 Hertz 모델에 사용된 최대 압입 등 해석과 연관된 메타데이터가 포함되어 있습니다. - 이 단계는 선택 사항입니다. 평균 F-Ind 를 클릭하여 평균 힘과 평균 압입과 함께 힘의 SD 1개를 내보냅니다.
- 세포 나노인덴테이션 데이터의 경우 탄성 스펙트럼 분석을 클릭합니다(대표 결과 및 토론 참조). 생성 된 두 개의 플롯, 즉 각 곡선에 대한 압입 깊이 (E (δ))의 함수로서의 E와 모델의 장착 된 하나의 SD (빨간색 실선 및 음영 영역)를 보여주는 오차 대역이있는 평균 E (δ)를 검사하여 세포의 액틴 피질의 영률, 세포의 벌크 영률을 추정 할 수 있습니다. 그리고 액틴 피질의 두께. 또한 상단 그래프의 평균 E(δ)를 빨간색으로 확인합니다.
- 보간(Interpolate) 상자가 선택되어 있는지 확인하여 탄성 스펙트럼 분석을 수행하는 데 필요한 도함수가 보간된 신호에서 계산되도록 합니다(대표 결과 참조).
- 결과 상자를 검사하여 피질의 영률(E 0 ± σ), 세포의 벌크 영률(Eb ± σ) 및 피질 두께(d0 ± σ)를 보고합니다.
참고: 평균 탄성 스펙트럼은 처음에는 시끄럽게 보일 수 있으며 현사인파 진동이 두드러집니다. 그 결과, 식 (3)이 정확하게 피팅되지 않을 수 있다. 이 경우, 평활 SAVGOL 필터(34 )의 윈도우 길이를 반복적으로 증가시키면 이 문제가 해결된다. - 분석이 완료되면 저장 상자에서 ES를 클릭합니다. 그러면 평균 압입 깊이 및 접촉 반경의 함수로 평균 탄력성, 실험과 관련된 메타데이터(위 참조) 및 위에서 설명한 예상 모델 매개 변수를 포함하는 지정된 디렉터리의 .tsv 파일이 내보내집니다. 마지막으로, δ 및 SD에 대한 의존성을 무시한 평균 탄력성도보고됩니다.
- 소프트웨어를 닫고 저장된 결과를 선호하는 다른 소프트웨어에 입력하여 데이터를 추가로 플롯하고 통계 분석을 수행합니다.
- 이 단계는 선택 사항입니다: 그래프를 마우스 오른쪽 버튼으로 클릭하고 내보내기를 선택하여 GUI에서 그래프를 내보냅니다. 그래프를 .svg로 내보내 글꼴, 글꼴 크기, 선 스타일 등과 같은 매개 변수를 만듭니다. 선택한 다른 소프트웨어에서 편집할 수 있습니다.
알림: 사용자 지정 CP 알고리즘 및 필터를 프로그래밍하여 기존 알고리즘에 추가할 수 있습니다. 자세한 내용은 보충 참고 1 을 참조하십시오.
결과
프로토콜에 따라 F-z 곡선 세트가 얻어집니다. 데이터 세트에는 양호한 곡선과 분석을 계속하기 전에 폐기해야 할 곡선이 포함될 가능성이 큽니다. 일반적으로 곡선의 모양이 그림 4A에 표시된 것과 다른 경우 버려야 합니다. 그림 5AI는 NanoPrepare GUI에 업로드된 예상 E 0.8 KPa35의 소프트 PAAm 하이드로겔에서 얻은 ~100개의 곡?...
토론
이 프로토콜은 하이드로겔과 단일 세포 모두에서 상업적으로 이용 가능한 페룰 탑 나노인덴터를 사용하여 힘 분광법 나노인덴테이션 데이터를 강력하게 획득하는 방법을 보여줍니다. 또한 나노인덴테이션 데이터 분석을 위한 정확한 워크플로를 포함하는 Python으로 프로그래밍된 오픈 소스 소프트웨어의 사용에 대한 지침이 제공됩니다.
프로토콜의 중요한 단계<...
공개
저자는 공개 할 것이 없습니다.
감사의 말
GC와 MAGO는 CeMi의 모든 회원을 인정합니다. MSS는 EPSRC 프로그램 보조금(EP/P001114/1)을 통해 지원을 인정합니다.
GC : 소프트웨어 (소프트웨어 개발 및 알고리즘에 대한 기여), 공식 분석 (나노인덴테이션 데이터 분석), 검증, 조사 (폴리 아크릴 아미드 겔에 대한 나노 압흔 실험), 데이터 큐레이션, 작성 (원본 초안, 검토 및 편집), 시각화 (그림 및 그래프). MAGO : 조사 (젤 및 세포 샘플 준비, 세포에 대한 나노인덴 테이션 실험), 쓰기 (원본 초안, 검토 및 편집), 시각화 (그림 및 그래프). NA : 검증, 작성 (검토 및 편집). IL : 소프트웨어 (소프트웨어 개발 및 알고리즘에 대한 기여), 검증, 작성 (검토 및 편집); MV : 개념화, 소프트웨어 (원본 소프트웨어 및 알고리즘의 설계 및 개발), 검증, 리소스, 작성 (원본 초안, 검토 및 편집), 감독, 프로젝트 관리, 자금 조달 MSS : 리소스, 작성 (검토 및 편집), 감독, 프로젝트 관리, 자금 조달. 모든 저자는 최종 원고를 읽고 승인했습니다.
자료
| Name | Company | Catalog Number | Comments |
| 12 mm coverslips | VWR | 631-1577P | |
| 35 mm cell treated culture dishes | Greiner CELLSTAR | 627160 | |
| Acrylamide | Sigma-Aldrich | A4058 | |
| Acrylsilane | Alfa Aesar | L16400 | |
| Ammonium Persulfate | Merk | 7727-54-0 | |
| Bisacrylamide | Merk | 110-26-9 | |
| Chiaro nanoindenter | Optics 11 Life | no catologue number | |
| Ethanol | general | ||
| Fetal bovine serum | Gibco | 16140071 | |
| High glucose DMEM | Gibco | 11995065 | |
| Isopropanol | general | ||
| Kimwipe | Kimberly Clark | 21905-026 | |
| Microscope glass slides | VWR | 631-1550P | |
| MilliQ system | Merk Millipore | ZR0Q008WW | |
| OP1550 Interferometer | Optics11 Life | no catalogue number | |
| Optics 11 Life probe (k = 0.02-0.005 N/m, R = 3-3.5 um) | Optics 11 Life | no catologue number | |
| Optics 11 Life probe (k = 0.46-0.5 N/m, R = 50-55 um) | Optics 11 Life | no catologue number | |
| Penicillin/Streptomycin | Gibco | 15140122 | |
| RainX rain repellent | RainX | 26012 | |
| Standard petri dishes (90 mm) | Thermo Scientific | 101RTIRR | |
| Tetramethylethylenediamine | Sigma-Aldrich | 110-18-9 | |
| Vaccum dessicator | Thermo Scientific | 531-0250 | |
| Software | |||
| Data acquisition software (v 3.4.1) | Optics 11 Life | ||
| GitHub Desktop (Optional) | Microsoft | ||
| Python 3 | Python Software Foundation | ||
| Visual Studio Code (Optional) | Microsoft |
참고문헌
- Discher, D. E., Janmey, P. Y. W. Tissue cells feel and respond to the stiffness of their substrate. Science. 310 (5751), 1139-1143 (2005).
- Roca-Cusachs, P., Conte, V., Trepat, X. Quantifying forces in cell biology. Nature Cell Biology. 19 (7), 742-751 (2017).
- Guimarães, C. F., Gasperini, L., Marques, A. P., Reis, R. L. The stiffness of living tissues and its implications for tissue engineering. Nature. Reviews. Materials. 5, 351-370 (2020).
- Moeendarbary, E., Harris, A. R. Cell mechanics: Principles, practices, and prospects. Wiley Interdisciplinary. Reviews. Systems. Biology and Medicine. 6 (5), 371-388 (2014).
- Butcher, D. T., Alliston, T., Weaver, V. M. A tense situation: Forcing tumour progression. Nature Reviews Cancer. 9 (2), 108-122 (2009).
- Kubánková, M., et al. Physical phenotype of blood cells is altered in COVID-19. Biophysical Journal. 120 (14), 2838-2847 (2021).
- Engler, A. J., Sen, S., Sweeney, H. L., Discher, D. E. Matrix elasticity directs stem cell lineage specification. Cell. 126, 677-689 (2006).
- Ciccone, G., et al. What caging force cells feel in 3D hydrogels: A rheological perspective. Advanced Healthcare Materials. 9 (17), 2000517 (2020).
- Wu, P. H., et al. A comparison of methods to assess cell mechanical properties. Nature Methods. 15 (7), 491-498 (2018).
- McKee, C. T., Last, J. A., Russell, P., Murphy, C. J. Indentation versus tensile measurements of young's modulus for soft biological tissues. Tissue Engineering B Reviews. 17 (3), 155-164 (2011).
- Humphrey, J. D., Delange, S. L. . An Introduction to Biomechanics Solids and Fluids, Analysis and Design. , 271-371 (2004).
- Prevedel, R., Diz-Muñoz, A., Ruocco, G., Antonacci, G. Brillouin microscopy: an emerging tool for mechanobiology. Nature Methods. 16 (10), 969-977 (2019).
- Krieg, M., et al. Atomic force microscopy-based mechanobiology. Nature Reviews Physics. 1, 41-57 (2019).
- Norman, M. D. A., Ferreira, S. A., Jowett, G. M., Bozec, L., Gentleman, E. Measuring the elastic modulus of soft culture surfaces and three-dimensional hydrogels using atomic force microscopy. Nature Protocols. 16 (5), 2418-2449 (2021).
- Gavara, N. A beginner's guide to atomic force microscopy probing for cell mechanics. Microscopy Research and Technique. 80 (1), 75-84 (2017).
- Whitehead, A. J., Kirkland, N. J., Engler, A. J. Atomic force microscopy for live-cell and hydrogel measurement. Methods in Molecular Biology. 2299, 217-226 (2021).
- Chavan, D., et al. Ferrule-top nanoindenter: An optomechanical fiber sensor for nanoindentation. The Review of Scientific Instruments. 83 (11), 115110 (2012).
- Van Hoorn, H., Kurniawan, N. A., Koenderink Ac, G. H., Iannuzzi, D. Local dynamic mechanical analysis for heterogeneous soft matter using ferrule-top indentation. Soft Matter. 12 (12), 3066-3073 (2016).
- Baldini, F., et al. Biomechanics of cultured hepatic cells during different steatogenic hits. Journal of the Mechanical Behavior of Biomedical Materials. 97, 296-305 (2019).
- Emig, R., et al. Piezo1 channels contribute to the regulation of human atrial fibroblast mechanical properties and matrix stiffness sensing. Cells. 10 (3), 663 (2021).
- Dobre, O., et al. A hydrogel platform that incorporates laminin isoforms for efficient presentation of growth factors - Neural growth and osteogenesis. Advanced Functional Materials. 31 (21), 2010225 (2021).
- Antonovaite, N., Beekmans, S. V., Hol, E. M., Wadman, W. J., Iannuzzi, D. Regional variations in stiffness in live mouse brain tissue determined by depth-controlled indentation mapping. Scientific Reports. 8 (1), 12517 (2018).
- Wu, G., Gotthardt, M., Gollasch, M. Assessment of nanoindentation in stiffness measurement of soft biomaterials: kidney, liver, spleen and uterus. Scientific Reports. 10 (1), 18784 (2020).
- Lüchtefeld, I., et al. Elasticity spectra as a tool to investigate actin cortex mechanics. Journal of Nanobiotechnology. 18 (1), 147 (2020).
- Caliari, S. R., Burdick, J. A. A practical guide to hydrogels for cell culture. Nature Methods. 13 (5), 405-414 (2016).
- Blumlein, A., Williams, N., McManus, J. J. The mechanical properties of individual cell spheroids. Scientific Reports. 7 (1), 7346 (2017).
- Mirsandi, H., et al. Influence of wetting conditions on bubble formation from a submerged orifice. Experiments in Fluids. 61, 83 (2020).
- Vassalli, M., Ciccone, G. . CellMechLab/NanoPrepare: v0.1.1. Zendo. , (2021).
- Vassalli, M., Ciccone, G., Lüchtefeld, I. CellMechLab/nanoindentation: v1.0.0. Zendo. , (2021).
- Savitzky, A., Golay, M. J. E. Smoothing and differentiation of data by simplified least squares procedures. Analytical Chemistry. 36 (8), 1627-1639 (1964).
- Schafer, R. W. What is a savitzky-golay filter. IEEE Signal Processing Magazine. 28 (4), 111-117 (2011).
- Gavara, N. Combined strategies for optimal detection of the contact point in AFM force-indentation curves obtained on thin samples and adherent cells. Scientific Reports. 6, 21267 (2016).
- Lin, D. C., Dimitriadis, E. K., Horkay, F. Robust strategies for automated AFM force curve analysis-I. Non-adhesive indentation of soft, inhomogeneous materials. Journal of Biomechanical Engineering. 129 (3), 430-440 (2007).
- Virtanen, P., et al. SciPy 1.0: fundamental algorithms for scientific computing in Python. Nature Methods. 17 (3), 261-272 (2020).
- Tse, J. R., Engler, A. J. Preparation of hydrogel substrates with tunable mechanical properties. Current Protocols in Cell Biology. , (2010).
- Pharr, G. M., Oliver, W. C., Brotzen, F. R. On the generality of the relationship among contact stiffness, contact area, and elastic modulus during indentation. Journal of Materials Research. 7 (3), 613-617 (1992).
- Kumar, R., Saha, S., Sinha, B. Cell spread area and traction forces determine myosin-II-based cortex thickness regulation. Biochimica et Biophysica Acta - Molecular Cell Research. 1866 (12), 118516 (2019).
- Kunda, P., Pelling, A. E., Liu, T., Baum, B. Moesin controls cortical rigidity, cell rounding, and spindle morphogenesis during mitosis. Current Biology: CB. 18 (2), 91-101 (2008).
- Allen, M., Poggiali, D., Whitaker, K., Marshall, T. R., Kievit, R. A. Raincloud plots: a multi-platform tool for robust data visualization. Wellcome Open Research. 4, 63 (2019).
- Clark, A. G., Paluch, E. Mechanics and regulation of cell shape during the cell cycle. Results and Problems in Cell Differentiation. 53, 31-73 (2011).
- Langan, T. J., Rodgers, K. R., Chou, R. C. Synchronization of mammalian cell cultures by serum deprivation. Methods in Molecular Biology. 1524, 97-105 (2017).
- Hodgkinson, T., et al. The use of nanovibration to discover specific and potent bioactive metabolites that stimulate osteogenic differentiation in mesenchymal stem cells. Science Advances. 7 (9), 7921 (2021).
- Kloxin, A. M., Kloxin, C. J., Bowman, C. N., Anseth, K. S. Mechanical properties of cellularly responsive hydrogels and their experimental determination. Advanced Materials (Deerfield Beach, Fla). 22 (31), 3484-3494 (2010).
- Loessner, D., et al. Functionalization, preparation and use of cell-laden gelatin methacryloyl-based hydrogels as modular tissue culture platforms. Nature Protocols. 11 (4), 727-746 (2016).
- Yue, K., et al. Synthesis, properties, and biomedical applications of gelatin methacryloyl (GelMA) hydrogels. Biomaterials. 73, 254-271 (2015).
- Sarrigiannidis, S. O., et al. A tough act to follow: collagen hydrogel modifications to improve mechanical and growth factor loading capabilities. Materials Today. Bio. 10, 100098 (2021).
- Karoutas, A., et al. The NSL complex maintains nuclear architecture stability via lamin A/C acetylation. Nature Cell Biology. 21 (10), 1248-1260 (2019).
- Ryu, H., et al. Transparent, compliant 3D mesostructures for precise evaluation of mechanical characteristics of organoids. Advanced Materials (Deerfield Beach, Fla). 33 (25), 2100026 (2021).
- Carvalho, D. T. O., Feijão, T., Neves, M. I., Da Silva, R. M. P., Barrias, C. C. Directed self-assembly of spheroids into modular vascular beds for engineering large tissue constructs Biofabrication Directed self-assembly of spheroids into modular vascular beds for engineering large tissue constructs. Biofabrication. 13 (3), 035008 (2021).
- Chaudhuri, O., Cooper-White, J., Janmey, P. A., Mooney, D. J., Shenoy, V. B. Effects of extracellular matrix viscoelasticity on cellular behaviour. Nature. 584 (7822), 535-546 (2020).
- Cantini, M., Donnelly, H., Dalby, M. J., Salmeron-Sanchez, M. The plot thickens: The emerging role of matrix viscosity in cell mechanotransduction. Advanced Healthcare Materials. 9 (8), 1901259 (2020).
- Elosegui-Artola, A. The extracellular matrix viscoelasticity as a regulator of cell and tissue dynamics. Current Opinion in Cell Biology. 72, 10-18 (2021).
- Chaudhuri, O. Viscoelastic hydrogels for 3D cell culture. Biomaterials Science. 5 (8), 1480-1490 (2017).
- Nguyen, T. D., Gu, Y. Determination of strain-rate-dependent mechanical behavior of living and fixed osteocytes and chondrocytes using atomic force microscopy and inverse finite element analysis. Journal of Biomechanical Engineering. 136 (10), 101004 (2014).
- Mokbel, M., Hosseini, K., Aland, S., Fischer-Friedrich, E. The Poisson Ratio of the Cellular Actin Cortex Is Frequency Dependent. Biophysical Journal. 118 (8), 1968-1976 (2020).
- Javanmardi, Y., Colin-York, H., Szita, N., Fritzsche, M., Moeendarbary, E. Quantifying cell-generated forces: Poisson's ratio matters. Communications Physics. 4, 237 (2021).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유