このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
ソフトマターナノインデンテーションの実験・データ解析ワークフロー
要約
このプロトコルは、ヒドロゲルや細胞を含むソフトマテリアルナノインデンテーション実験のための完全なワークフローを提供します。まず、力分光法データを取得するための実験手順が詳述されています。次に、そのようなデータの分析は、GitHubから無料でダウンロードできる新しく開発されたオープンソースのPythonソフトウェアを通じて詳細に説明されます。
要約
ナノインデンテーションとは、マイクロメトリックフォースプローブを使用してソフトバイオマテリアルや細胞の局所的な機械的特性を定量化する実験技術のクラスを指します。このアプローチは、メカノバイオロジー、生体材料設計、組織工学の分野で中心的な役割を果たし、単一細胞サイズ(μm)に匹敵する分解能でソフトマテリアルの適切な機械的特性評価を取得しています。このような実験データを取得するための最も一般的な戦略は、原子間力顕微鏡(AFM)を使用することです。この装置は、力(pNまで)と空間(サブnmまで)において前例のない分解能を提供しますが、その使いやすさは、ヤング率(E)などの機械的特性の積分指標の日常的な測定を妨げる複雑さによって制限されることがよくあります。光ファイバセンシング技術に基づくものなどの新世代のナノインデンターは、μmの空間分解能でサブnN力を加えることを可能にしながら、統合が容易であることから最近人気を博しているため、ヒドロゲルや細胞の局所的な機械的特性を調べるのに適しています。
このプロトコルでは、市販のフェルールトップ光ファイバセンシングナノインデンターを使用してヒドロゲルおよびセルに関するナノインデンテーションデータを取得するための実験手順を詳述するステップバイステップガイドが提示されます。いくつかのステップは本明細書で使用される器具に固有であるが、提案されたプロトコルは、他のナノインデンテーションデバイスのためのガイドとしてとらえることができ、いくつかのステップが製造業者のガイドラインに従って適合されることを前提とする。さらに、ナノインデンテーションデータを分析するためのユーザーフレンドリーなグラフィカルユーザーインターフェイスを備えた新しいオープンソースのPythonソフトウェアが提示され、誤って取得された曲線のスクリーニング、データのフィルタリング、さまざまな数値手順による接触点の計算、 従来のEの計算、および特にシングルセルナノインデンテーションデータに適したより高度な分析が可能になります。
概要
生物学における力学の基本的な役割は、今日確立されています1,2。全組織から単一細胞まで、機械的特性は、調査中の生体材料の病態生理学的状態について知らせることができます3,4。例えば、癌の影響を受けた乳房組織は健康な組織よりも硬い、これは一般的な触診検査5の基礎となる概念である。特に、重症急性呼吸器症候群コロナウイルス2(SARS-CoV-2)によって引き起こされるコロナウイルス病2019(COVID-19)は、赤血球変形能の低下、リンパ球および好中球のこわばりの減少など、血球の機械的特性の変化によって強調されていることが最近示されていますSARS-CoV-2ナイーブな個人の血球と比較して6。
一般に、細胞と組織の力学は本質的に絡み合っており、各組織は構成細胞と細胞外マトリックス(ECM)のそれらに同時に影響を及ぼし、依存する特定の機械的特性を持っています5。このため、生物学の力学を研究するための戦略には、生理学的に関連する機械的刺激を備えた基板を操作して、それらの刺激に応答した細胞の挙動を解明することがよくあります。例えば、Englerらによる独創的な研究は、柔らかくて硬い2次元ポリアクリルアミド(PAAm)ヒドロゲルで研究されたように、間葉系幹細胞の系統の関与がマトリックスの弾力性によって制御されることを実証しました7。
調査中の生体材料を機械的に特徴付けるための多くの戦略が存在し、空間スケール(すなわち、局所からバルク)および変形のモード(例えば、軸方向対せん断)において異なり、その結果、慎重な解釈を必要とする異なる情報が得られる3,8,9,10。ソフトバイオマテリアルの力学は、一般的に剛性の観点から表されます。ただし、剛性は材料特性と形状の両方に依存しますが、弾性率は材料の基本的な特性であり、材料のジオメトリとは無関係です11。このように、異なる弾性率は特定のサンプルの剛性に関連しており、各弾性率は、異なる境界条件(例えば、自由膨張対閉じ込め)における特定の変形モード(例えば、軸方向対せん断)に対する材料の抵抗を包含する11,12。ナノインデンテーション実験は、生体材料が横方向に閉じ込められていない場合の一軸変形(インデンテーション)に関連するEを介した機械的特性の定量化を可能にする10,11,12。
マイクロスケールで生物学的システムのEを定量化する最も一般的な方法は、AFM13,14,15,16です。AFMは、pNレベルまでの力分解能とサブnmスケールまでの空間分解能を備えた非常に強力なツールです。さらに、AFMは、補完的な光学的および機械的ツールとの結合に関して非常に柔軟性があり、調査中の生体材料から豊富な情報を抽出する機能を拡張します13。ただし、これらの魅力的な機能には、実験セットアップの複雑さに代表される参入障壁があります。AFMは、ユーザーが堅牢なデータを取得する前に広範なトレーニングが必要であり、特にその独自の力と空間分解能が必要ない場合、生体材料の日常的な機械的特性評価に使用することは不当であることがよくあります。
このため、新しいクラスのナノインデンターは、その使いやすさから最近人気を博していますが、サブnNの力分解能とμmの空間分解能を備えたAFMに匹敵するデータを提供し、関連する長さスケールにわたって細胞によって加えられ、知覚される力を反映しています2。特に、光ファイバセンシング技術17,18に基づくフェルールトップナノインデンテーションデバイスは、メカノバイオロジーの分野で活躍する研究者の間で人気を博しています。そして、細胞19,20、ヒドロゲル8,21、および組織22,23を含む、これらのデバイスを使用した生体材料の機械的特性を報告する豊富な研究が発表されている。局所的な動的機械的特性(すなわち、貯蔵弾性率および損失弾性率)を調査するこれらのシステムの能力にもかかわらず、Eを生成する準静的実験は依然として最も一般的な選択肢である8,19,20,21。簡単に言えば、準静的ナノインデンテーション実験は、最大変位、力、または押し込み深さのいずれかによって定義される設定値まで一定の速度でサンプルをインデントし、カンチレバーの力と垂直位置の両方をいわゆる力-距離(F-z)曲線で記録することで構成されます。次に、F-z曲線は、接触点(CP)の識別によって力-インデンテーション(F-δ)曲線に変換され、適切な接触力学モデル(通常はヘルツモデル13)を取り付けてEを計算します。
フェルールトップナノインデンターの動作はAFM測定に似ていますが、検討する価値のある特異性があります。本研究では、市販のフェルールトップナノインデンターを用いて細胞や組織模倣ハイドロゲルから F-z 曲線をロバストに取得するためのステップバイステップガイドを提供し、本デバイスと他の同様のデバイスを使用した研究グループ間の実験手順の標準化を促進します。さらに、ナノインデンテーション実験を行うためのヒドロゲルサンプルと細胞の最適な調製方法に関するアドバイスと、実験経路に沿ったトラブルシューティングのヒントが提供されます。
さらに、ナノインデンテーション結果(すなわち、 E およびその分布)の変動性の多くは、データの分析に使用される特定の手順に依存し、これは自明ではない。この問題に対処するために、Pythonでプログラムされ、 F-z 曲線のバッチ分析のためのユーザーフレンドリーなグラフィカルユーザーインターフェイス(GUI)を備えた新しく開発されたオープンソースソフトウェアの使用手順が提供されています。このソフトウェアは、高速データスクリーニング、データのフィルタリング、さまざまな数値手順によるCPの計算、 Eの従来の計算、および弾性スペクトル24と呼ばれるより高度な分析を可能にし、細胞のバルクヤング率、アクチン皮質ヤング率、およびアクチン皮質の厚さを推定することができます。ソフトウェアはGitHubから自由にダウンロードでき、適切なデータパーサーを追加することで、他のシステムから発信されたデータを分析するように簡単に適合させることができます。このプロトコルは、他のフェルールトップナノインデンテーションデバイス、および他のナノインデンテーションデバイス全般に使用できることを強調し、いくつかのステップは特定の機器のガイドラインに従って適合されます。プロトコルを概略的に図 1にまとめます。
プロトコル
1. ナノインデンテーション測定用基板/セルの作製
- ナノインデンテーション実験用のPAAmヒドロゲル/セルの調製に関する 補足プロトコル に記載されている手順に従ってください。この手順を 図 2 にまとめます。
注:PAAmヒドロゲルは、メカノバイオロジーの分野で使用される最も一般的なヒドロゲルであるため、選択されています。しかしながら、プロトコルは、任意のタイプのヒドロゲル25 に等しく適用可能である( 考察、方法の修正を参照されたい)。
2.デバイスの起動、プローブの選択、およびプローブのキャリブレーション
- 補足プロトコルに記載されている手順に従って、デバイスを起動します。光ファイバフェルールトップナノインデンターの動作に関する技術的な詳細については、これらの参考文献17,18を確認してください。
- 以下の説明に従ってナノインデンテーションプローブを選択します。
注意: このプロトコルで使用されているようなすべての市販のプローブには、球形の先端が装備されています(図3A)。したがって、選択肢はカンチレバーの剛性と先端半径の2つの変数に絞り込まれます(図3B)。- 最良の結果を得るには、予想されるサンプル剛性に一致するカンチレバーの剛性(N / mのk)を選択します15(図3Cおよびディスカッション、プロトコルの重要なステップを参照)。細胞の場合、0.01〜0.09 N / mの範囲のkを持つプローブを選択します。ヒドロゲルの場合、0.1〜0.9 N / mの範囲のkプローブを選択すると、予想されるEが数kPa〜100 kPaのゲルで最適な結果が得られます(代表的な結果を参照)。
- 先端半径(R(μm)は、インデンテーションプロセスの所望の空間分解能に応じて選択します。ヒト胚性腎臓293T(HEK293T)細胞(平均直径~10-15μm26)などの小細胞の場合は、R = 3μmの球体を選択します。ヒドロゲルの場合、R = 10〜250μmの球を選択して、広い接触面積にわたって生体材料の機械的特性を調査し、局所的な不均一性を回避します。
- 図3Cおよびその他の製造元のガイドラインを参照して、適切なプローブを選択してください。
- プローブを選択したら、 補足プロトコル の手順に従ってナノインデンターに取り付けます。
3.プローブキャリブレーション
注意: 次の手順は、光ファイバセンシング技術に基づくフェルールトップナノインデンテーションデバイスに固有であり、ソフトウェアバージョン3.4.1で詳しく説明されています。その他のナノインデンテーションデバイスの場合は、デバイスの製造元が推奨する手順に従ってください。
- ソフトウェアのメインウィンドウで、[ 初期化]をクリックします。キャリブレーションメニューが表示されます。プローブの詳細を入力します(プローブのボックスの側面にあります。 入力 ボックス内のN / mの k、μmのR 、および空気中のキャリブレーション係数)。
- キャリブレーションディッシュを準備します:底が平らな厚いガラスのペトリ皿( ディスカッション、プロトコルの重要なステップを参照)。サンプル皿と同じ媒体で皿を満たします(これは空気でもかまいません)。媒体の温度をサンプルの温度と一致させます。
- キャリブレーション皿をプローブの下に置きます。必要に応じて、ナノインデンターのアームホルダーからプローブを引き出し、片手で持ってキャリブレーションディッシュを配置するスペースを確保します。プローブを元の位置にスライドさせて戻します。
- 液体中でのキャリブレーションのために次の2つのステップを実行します。空気中で測定する場合は、付属のポリテトラフルオロエチレン基板をキャリブレーションに使用し、手順5に進みます。
- ピペットの端をガラスフェルールに軽く接触させた状態でパスツールピペットを使用してプローブを70%エタノールの滴で予湿し、滴がカンチレバーと球形の先端27 の上を滑るようにします( ディスカッション、プロトコルの重要なステップを参照)。
- プローブが完全に水没するまでナノインデンターのアームを手動で下にスライドさせますが、それでもペトリ皿の底から遠く離れています。必要に応じて、キャリブレーションディッシュに培地を追加します。液体中の平衡状態に達するまで5分間待ちます。
- ソフトウェアの [初期化 ]メニューで、[ スキャン波長]をクリックします。干渉計の画面にはプログレスバーが表示され、コンピュータのソフトウェアの ライブ信号 ウィンドウには 図S1A(左)に示すパターンが表示されます。光学スキャンが成功したかどうかを確認するには、干渉計ボックスの 波長スキャン パネルに移動します。成功すると、正弦波が表示されます(図1A、右)。エラーが表示された場合は、 ディスカッション (メソッドのトラブルシューティング) を参照してください。
- [初期化]メニューで[サーフェスの検索]をクリックすると、カンチレバーの曲げで設定されたしきい値に達するまでプローブが徐々に下がります。ガラスのペトリ皿と接触すると、プローブの動きが止まります。
- プローブが表面に接触しているかどうかを確認します。ソフトウェアのメインウィンドウにある y 下向き矢印ボタンを使用して、プローブを1μm下に移動します。カンチレバーが基板に接触しているときに、各ステップでのベースラインの変化について、ソフトウェアの ライブウィンドウ の緑色の信号(カンチレバーのたわみ)を観察します(図S1B)。変化がない場合、カンチレバーは接触していません(次の手順を参照)。
- [サーフェスを検索]タブの[オプション]メニューの[しきい値]の値をデフォルト値の 0.01 から一度に 0.01 ずつ増やし、接触するまで[サーフェスの検索]ステップを繰り返します。または、緑色のベースラインが各下向きのステップでシフトし始めるまで、プローブを1μmの小さなステップで接触させます。
注: 最も柔らかいカンチレバー(k = 0.025 Nm-1)の場合は、[サーフェスの検索]タブの[オプション]メニューの[しきい値]の値を、手順 6 を事前に実行して大きくします。しきい値 0.06 または 0.07 から開始し、必要に応じて 0.1 まで増やします。これは、環境ノイズにより、接触前にカンチレバーがしきい値を超えて曲がる可能性があるためです。ソフトプローブの場合、同じメニューでアプローチ速度(μm/s)を下げると、手順も改善されます。 - [初期化]メニューで、[調整]をクリックします。
- ソフトウェアのライブ信号ウィンドウをチェックし、ピエゾの変位信号とカンチレバーの偏向 信号 の両方が同時に上昇することを確認します(図S1C)。
- 時間に不一致がある場合、プローブはガラスと完全に接触していません。カンチレバー信号のベースラインが変わるまでプローブを1μmのステップで下に移動し(ステップ7を参照)、ステップ9を繰り返します。
- キャリブレーションステップ中にカンチレバー信号がまったく変化しない場合、プローブは表面から遠く離れています。表面が正しく見つかるまで接触しきい値を繰り返し上げ(手順8を参照)、波長スキャンから最初からキャリブレーションを繰り返します。
- キャリブレーションが完了したら、表示されるポップアップウィンドウで新旧のキャリブレーション係数を確認します。新しいキャリブレーション係数が正しい範囲内にある場合は、[新しい係数を使用]をクリックします。キャリブレーションに失敗し、新しい係数がNaNであるか、予想される範囲内にない場合は、解決策についてディスカッション(メソッドのトラブルシューティング)を参照してください。
注:屈折率n(水の場合はn = 1.33)の液体媒体でキャリブレーションを実行した場合、新しい係数はプローブのボックスに提供されている係数よりも~n倍低くする必要があります。キャリブレーションが空気中で実行された場合、新しいキャリブレーション係数と古いキャリブレーション係数はほぼ等しくなります。 - 復調円が次のように正しく校正されているかどうかを確認します。干渉計デスクトップの復 調 タブに移動します。光学テーブルまたはナノインデンターを軽くたたいて、十分なノイズを誘発します。離散データポイントで構成される白い円は、赤い円をほぼ覆う必要があります(図S1D)。
- 白い円が赤い円と重なっていない場合、または干渉計のディスプレイに警告が表示される場合は、復調円を再校正する必要があります。これは、以下に説明するように2つの方法で実行できます。
- ナノインデンターの本体を連続してタップして、1つの完全なノイズを誘導し、干渉計の キャリブレーション ボタンを押します。
- ガラス基板に接触して入力し、ソフトウェアのメインウィンドウの[初期化]メニューから[キャリブレーション]を押します。キャリブレーション係数は保存しないでください。この時点で、もう一度確認し、白い円が赤い円と重なっていることを確認します。
注意: 信号が復調サークルからわずかにずれているだけでなく、非常に小さくなっているか、まったく見えない場合は、カンチレバーが光ファイバに付着していることを意味します。この問題に関するトラブルシューティングのアドバイスに従い ( 説明、方法のトラブルシューティングを参照)、手順 13.1 または 13.2 を繰り返します。カンチレバーが水平位置に戻ると(図3A)、信号は復調円に回復します。
- 下記のように、キャリブレーション直後にガラス基板にくぼみを入れてキャリブレーションを検証します。
- [ 実験の構成 ] をクリックして実験ファイルを読み込むか作成し、[ サーフェスの検索 ] ステップと [インデント ] ステップを追加します。圧痕ステップでは、デフォルトの変位モード設定を使用し、最大変位をキャリブレーション距離(3,000 nm)に変更して、プローブを硬い基板に対して変位させます。
- [実験の実行]をクリックし、干渉計ウィンドウで復調円を確認します。白い信号を確認し、インデント中に赤い円の上にあることを確認してください。
- Time Data グラフのソフトウェアのメイン ウィンドウで結果を確認し、圧痕が接触し始め、材料の変形が予想されないため、ピエゾの変位(青い線)がカンチレバーのたわみ(緑の線)と等しいことを確認します。信号が並列でない場合は、ディスカッション(メソッドのトラブルシューティング)を参照してください。
- キャリブレーションメニューのローカルパスを変更するには、 キャリブレーション保存パス を適切なディレクトリに設定します。
- プローブが正常に校正されたら、ピエゾを500μm上に移動します。
4. 軟質材料のヤング率の測定
- ヒドロゲルのナノインデンテーション
- サンプルを含むペトリ皿を顕微鏡のステージにロードし、ナノインデンターのプローブをサンプルの上の目的の x-y 位置に手動で移動します。
- この段階でプローブとサンプルの表面の間に1〜2 mmを残すように注意しながら、プローブを溶液中で手動でスライドさせます。プローブが培地内で平衡化するまで5分間待ちます。
- プローブがはっきりと見えるように光学顕微鏡の z 面に焦点を合わせます。
- 以下に説明するように、単一のインデントを実行して実験パラメータを調整します。
- ソフトウェアのメインウィンドウで新しい実験を構成します。[ 実験の構成] をクリックすると、新しいウィンドウが開きます。[ サーフェスの検索 ] ステップを追加します。[ サーフェスを検索 ]ステップのすべてのパラメータは、必要に応じてソフトウェアの [オプション ]メニューで変更できます。
注意: 検索サーフェス は、表面が見つかるまでプローブを下げてから、サンプルの表面 上のZ(μm)で定義された距離までプローブを引っ込めます。選択したカンチレバーがサンプルに対して硬すぎる場合、またはサンプルが粘着性がある場合、ステップ後もプローブはサンプルと接触している可能性があり、ベースラインのない曲線になります(図4C)。この問題を解決するには、 表面上のZ(μm)を増やします。 - インデントステップを追加します。[プロファイル]タブを選択し、[変位制御]をクリックします。デフォルトのインデントプロファイルのままにします。
- ソフトウェアのメインウィンドウで [実験の実行 ]をクリックします。これにより、サーフェスが検索され、単一のインデントが実行されます。単一のインデントが期待どおりに表示されない場合は、 図4 と ディスカッション( メソッドのトラブルシューティング)で説明されているように実験パラメーターを調整します。
- ソフトウェアのメインウィンドウで新しい実験を構成します。[ 実験の構成] をクリックすると、新しいウィンドウが開きます。[ サーフェスの検索 ] ステップを追加します。[ サーフェスを検索 ]ステップのすべてのパラメータは、必要に応じてソフトウェアの [オプション ]メニューで変更できます。
- インデントが希望どおりに表示されたら、サンプルの十分な領域がインデントされるようにマトリックススキャンを構成します。[実験 の構成]をクリックし、以前に決定した実験パラメータを使用して [サーフェス検索 ]ステップを追加し、[ マトリックススキャン ]ステップを追加します。
- フラットヒドロゲルの場合、10〜100μm(すなわち、dx = dy = 10〜100μm)の間隔で50〜100点(すなわち、xおよびyで5 x 10または10 x 10)を含むマトリックススキャンを構成する。[ステージ位置を使用]をクリックして、現在のステージ位置からマトリックススキャンを開始します。[サーフェスの自動検索]ボックスにチェックマークを付けて、設定された実験パラメータを使用して各インデントのサーフェスを検索します。
- オーバーサンプリングを回避するには、ステップ サイズを接触半径の少なくとも 2 倍に設定します (
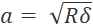 δ はくぼみの深さ)。
δ はくぼみの深さ)。 - 変位制御でマトリックススキャンプロファイルを設定します。プロファイルがヘルツモデルの仮定に違反していないことを確認します( ディスカッション、プロトコルの重要なステップを参照)。
- セグメント数はデフォルト値である 5 のままにして、デフォルトの変位プロファイルを使用します。必要に応じて、各傾斜セグメントの最大変位と時間の観点から変位プロファイルを変更すると、それぞれ最大押し込み深さとひずみ速度に影響します。ひずみ速度>10 μm/sを超えないようにしてください( 説明、方法の制限を参照)。
- アプローチ速度の値を入力すると、接触前にプローブがサンプルに向かって移動する速度が決まります。格納速度をアプローチ速度に合わせます(下記の注を参照)。
注意: ソフトカンチレバーやノイズの多い環境では、1,000〜2,000 nm / sのアプローチ速度が推奨されます。より硬いカンチレバーと制御された環境では、これを増やすことができます。 - 構成済みの実験を目的の [実験パス] に保存し、[実験の構成] ウィンドウの [全般] タブで、データを保存するディレクトリを [パスの保存] で選択します。[実験を実行] をクリックします。
- オーバーサンプリングを回避するには、ステップ サイズを接触半径の少なくとも 2 倍に設定します (
- マトリックススキャンが完了したら、プローブを200〜500μm上げ、プローブを最初の領域から十分に離れたサンプルの別の領域に移動します。
- 各サンプルで十分なデータが取得されるように、実験を少なくとも2回繰り返します(つまり、サンプルごとに少なくとも2つのマトリックススキャン、それぞれ50〜100の曲線を含む)。
- 細胞のナノインデンテーション
- 上記のようにサンプルを顕微鏡にロードします。
- 単一セルのくぼみの場合は、セルのサイズと広がりに応じて、セルとプローブの両方が20倍または40倍の倍率で見えるように z 平面に焦点を合わせます。
- インデントするセルの上にプローブを移動します。
- ソフトウェアのメインウィンドウで新しい実験を構成します。[ 実験の構成] をクリックすると、新しいウィンドウが開きます。変位モードのデフォルトパラメータを持つサーフェス 検索 および インデンテーション ステップを追加します。
- [ 実験を実行]をクリックすると、サーフェスが検索され、1つのインデントが実行されます。インデントが成功したかどうかを確認します。曲線が期待どおりに表示されない場合は、実験パラメータを調整します( 図4 および ディスカッション、メソッドのトラブルシューティングを参照)。
- インデントが成功した場合は、マトリックス スキャンを実験に追加します。ヒドロゲルのナノインデンテーション実験に与えられた手順に従ってください。ステップサイズでセルの小さな領域をインデントできるようにマトリックススキャンを構成します。HEK293T細胞については、0.5〜5μmの間隔で25点。
- セルサイズに応じて、マトリックススキャンを調整して、チップがセル制限の外側にインデントしないようにします(つまり、異なるマップジオメトリを実行したり、より少ないポイントをプローブしたりします)。
- [ 実験の実行] をクリックし、完了するのを待ちます。
- マトリックススキャンが完了したら、プローブを接触から外します( z 平面で50 μm)。
- プローブを新しいセルの上に移動し、プロセスを繰り返します( ディスカッション、プロトコルの重要なステップを参照)。
- プローブの清掃と機器の電源オフ
- 補足プロトコルに記載されている手順に従って、プローブをクリーニングし、ナノインデンテーションデバイスのスイッチを切ります。
5.データ分析
- ソフトウェアのダウンロードとインストール
- 補足プロトコルに記載されている手順に従って、データ分析用のソフトウェアをダウンロードしてインストールします28,29。
- F-z曲線のスクリーニングとJSON形式のクリーニングデータセットの作成
- 手順 2 から 3 で説明されているように、ラボ コンピューターのコマンド ラインから prepare.py を起動します。
- Windows コンピューターを使用している場合は、 NanoPrepare フォルダーを Shift キーを押しながら右クリックし、[ PowerShell ウィンドウをここで開く] をクリックします。 python prepare.py コマンドを入力し、 Enter キーを押します。GUIが画面にポップアップ表示されます(図S2)。
- MacOSコンピュータを使用している場合は、 NanoPrepare フォルダを右クリックし、[ フォルダの新しいターミナル]をクリックします。 python3 prepare.py コマンドを入力して Enter キーを押すと、GUI が起動します (図 S2)。
- ドロップダウン リストから O11NEW データ形式を選択します。データが正しくロードされていない場合は、GUIを再起動して O11OLDを選択します。
注: O11NEW 形式は、ソフトウェアバージョン3.4.1のフェルールトップ光ファイバセンシングナノインデンテーションデバイスを使用して取得したデータに対して機能します。この形式は、以前のソフトウェアバージョン、少なくとも2019年から2020年にインストールされたナノインデンターに属するソフトウェアバージョンでも機能します。 - [ フォルダの読み込み]をクリックします。分析するデータを含むフォルダーを選択します-シングルマトリックススキャンまたはマルチマトリックススキャン。一番上のグラフ(生の曲線)には、アップロードされたデータセットが入力されます。特定の曲線を視覚化するには、その曲線をクリックします。これにより、緑色で強調表示され、下のグラフ(現在の曲線)に表示されます。
- 以下に概説するように、GUIの右側にあるタブを使用してデータセットをクリーンアップします。
- [セグメント]ボタンを使用して、解析する正しいセグメント( F-z カーブの前方セグメント)を選択します。具体的な数は、実験を行う際にナノインデンテーションソフトウェアで選択したセグメントの数によって異なります。
- [50 nmの切り抜き]ボタンを使用して、左端(Lがチェックされている場合)、右端(Rがチェックされている場合)、または両側(RとLの両方がチェックされている場合)で曲線を50nmトリミングします。このボタンを数回クリックすると、必要なだけトリミングされます。これを使用して、F-zカーブの開始/終了に存在するアーチファクトを除去します。
- [カンチレバー]タブで、ばね定数、先端形状、先端半径を調べます。タブを調べて、メタデータが正しく読み取られていることを確認します。
- [スクリーニング]タブを使用して、特定の力に達しなかったすべてのカーブを破棄する力のしきい値を設定します。破棄されたカーブは赤で強調表示されます。
- 手動切り替えボタンを使用して、正しく取り込まれていないカーブを手動で削除します。特定の曲線をクリックしてOUTを選択すると、曲線が赤で強調表示されます。
- [ JSON の保存] をクリックします。クリーニングされたデータ・セットの適切な名前 (単一の JSON ファイル) を入力します。ナノアナリシス ソフトウェアがインストールされているコンピューターに JSON ファイルを送信します。
6.正式なデータ分析
- 前述のように、NanoAnalysisフォルダーに移動し、ターミナルを起動して、コマンドラインから nano.py ファイルを起動します。python nano.py または python3 nano.py コマンド (オペレーティング・システムによって異なります) を入力し、Enter キーを押します。GUIが画面にポップアップ表示されます(図S3)。
- GUI の左上にある [実験の読み込み] をクリックし、JSON ファイルを選択します。これにより、ファイルリストと、F-zカーブでデータセットを示す生のカーブグラフが入力されます。F 軸と z 軸の両方が、データセットの読み込み時にバックグラウンドで計算される CP 座標を基準にして表示されます (次の注を参照)。[統計] ボックスで、3 つのパラメーター (アクティブ化された N、失敗した N 、除外された N の値) を確認します。
注:アクティブ化 されたNは、後続のヘルツ/弾性スペクトル分析で分析され、 生の 曲線グラフに黒で表示される曲線の数を表します。Nfailed は、信頼できる CP が見つからなかった曲線の数を表し、グラフに青色で表示されます。これらの曲線は、後続の解析で自動的に破棄されます。ソフトウェアを開くと、一部のカーブが自動的に故障したセットに移動される場合があります。これは、CPがデフォルトのしきい値アルゴリズム(以下を参照)でソフトウェアを開いたときに計算されるためです。Nexcluded は、解析から除外するために手動で選択されたカーブを表し、グラフに赤で表示されます(以下を参照)。 - 破損カーブ、除外カーブ、およびアクティブ化カーブの数が妥当かどうかを確認します。 生の 曲線グラフをチェックして、曲線を視覚化します。
- 特定の曲線をより詳細に視覚化するには、その曲線をクリックします。これにより、緑色で強調表示され、 現在の曲線 グラフに表示されます。単一のカーブを選択すると、 R と k のパラメータ(すべてのカーブで同じである必要があります)がGUIの Stats ボックスに入力されます。
- トグルボックスを使用して、特定のカーブのステータスを変更します。ステータスを変更する特定のカーブをクリックし、[アクティブ化]、[失敗]、または[除外]のいずれかをクリックします。[統計] ボックスのカウントは自動的に更新されます。
注: 生の曲線グラフのデータ セットのビューを変更するには、[表示] ボックスを使用します。すべてをクリックすると、すべてのカーブが表示されます(つまり、アクティブ化、失敗、およびそれぞれの色で除外されます)。アクティブ化されたカーブと失敗したカーブを表示するには 失敗 をクリックし、アクティブ化された曲線のみを表示するには アクティブ化 をクリックします。アクティブ化と除外の間のすべてのカーブのステータスをリセットするには、リセットボックスでアクティブ化または除外をクリックします。 - データセットをさらにクリーンアップしたら、以下に概説するデータ分析パイプラインに従います。
- GUI(フィルタリングボックス)に実装されたフィルター、つまりプロミネンシーフィルターと呼ばれるカスタムフィルター、Savitzky Golay30,31(SAVGOL)フィルター、および特定のウィンドウ内のデータの中央値の計算に基づく平滑化フィルター(中央値フィルター)を使用して、曲線に存在するノイズをフィルタリングします。フィルターの詳細については、「説明 (プロトコルの重要な手順)」を参照してください。
- 現在の曲線グラフでフィルタリングされた 曲線 を調べます。フィルタリングされた曲線は黒で表示され、フィルタリングされていないバージョンの曲線は緑色で表示されます。
注:元の信号の特徴を保持するために、データをできるだけフィルタリングしないことをお勧めします。過剰フィルタリングは、データに存在する違いを滑らかにする可能性があります。プロミネンシーフィルターをアクティブにして作業すれば、ナノインデンテーションデータのヘルツ分析には十分です。データが特にノイズが多い場合は、SAVGOLまたはメディアンフィルタを追加で適用できます。 - CPを見つけるアルゴリズムを選択します。[接触点]ボックスで、ソフトウェアに実装されている一連の数値手順、つまり適合度(GoF)32、分散比(RoV)32、二次導関数33、またはしきい値33のいずれかを選択します。アルゴリズムの詳細については、「ディスカッション、プロトコルの重要な手順」を参照してください。
注:CPは、プローブが材料と接触するポイントであり、 F-z データを F-δ データに変換するために識別する必要があります(ソフトマテリアルの場合、 δ は有限であり、計算する必要があります)。選択したアルゴリズムはデータセットのすべてのアクティブな曲線に適用され、アルゴリズムがCPをロバストに見つけることができない曲線は失敗したセットに移動されます。 - データセットに合わせてアルゴリズムのパラメーターを調整して、プロトコルの重要なステップである ディスカッションで詳しく説明されているようにCPが正しく配置されるようにします。CPが単一のカーブのどこに見つかったかを表示するには、カーブをクリックして選択し、 検査をクリックします。表示されるポップアップ・ウィンドウをチェックして、CPが配置されている場所を特定します。
- 表示される赤い線(選択された関心領域においてアルゴリズムが計算したパラメータ)が、CPの位置に対応する最大値または最小値を有するかどうかを確認する(例えば、GoFの場合、パラメータはR2である)。必要に応じて、すべてのカーブに対してこのプロセスを繰り返します。
注: ポップアップの軸は絶対座標であるため、CP の位置を表示できます。逆に、 生の曲線 と 現在の曲線 のグラフの軸はCPを基準にして表示され、CPの位置は(0,0)です。 - ヘルツ解析をクリックします。これにより、以下に説明する3つのグラフが生成されます。
- データセット内の個々のF δ曲線を平均ヘルツ適合値(赤い破線)とともに確認します。[最大インデンテーション (nm)] の下の [結果] ボックスで、ヘルツ モデルが適合されるインデントを nm 単位で調整します。ヘルツモデルを有効にするには、Rの最大~10%に設定します(ディスカッション、プロトコルの重要なステップを参照)。
- 平均F δ曲線と平均ヘルツフィット(赤い破線)とともに1標準偏差(SD)を示す誤差バンドを確認します。参考のために、生の曲線グラフの平均ヘルツ適合度と、現在の曲線の各曲線のヘルツ適合を視覚化します。
- ヘルツモデルを個々の曲線に当てはめることから生じる E の散布図を確認してください。
- ファイル名、 F-z 曲線、 F-δ 曲線、または散布図上の点のいずれかをクリックして、各プロットの曲線を強調表示します。散布図のデータポイントがデータの分布の外側にあるように見える場合は、それをクリックして、それが属する曲線を調べます。「 検査 」ボタンをクリックして、CP が正しく検出されたことを確認します。必要に応じて、解析から曲線を除外します。
- 結果ボックスで、計算された平均EとそのSD(Eγ ± σ)を調べ、それらが特定の実験に対して妥当であることを確認します。
- [ 保存 ] ボックスで、[ Hertz] をクリックします。ポップアップウィンドウで、ファイル名とディレクトリを入力します。完了したら、[ 保存]をクリックします。.tsv ファイルが作成されます。任意の追加のソフトウェアで.tsvファイルを開き、統計分析とさらなるプロットに値を使用します。
注: このファイルには、各曲線から得られた E と平均 E とその SD が含まれています。 さらに、このファイルには、解析された曲線の数、R、k、ヘルツ モデルに使用される最大インデントなど、解析に関連付けられたメタデータが含まれています。 - この手順は省略可能です。 [平均 F-Ind ] をクリックして、平均力と平均くぼみを、力の 1 つの SD と共にエクスポートします。
- 細胞ナノインデンテーションデータについては、弾性スペクトル分析をクリックしてください(代表的な結果と考察を参照)。生成された2つのプロット、すなわち、各曲線のくぼみの深さ(E(δ))の関数としてのEと、モデルの(黒い破線)によって適合された1つのSD(赤の実線と影付きの領域)を示す誤差帯のある平均E(δ)を調べ、細胞のアクチン皮質のヤング率、細胞のバルクヤング率を推定できます。 そしてアクチン皮質の厚さ。さらに、上のグラフの平均E(δ)を赤で確認してください。
- 補間ボックスがオンになっていることを確認し、弾性スペクトル解析の実行に必要な導関数が補間された信号で計算されるようにします(代表的な結果を参照)。
- [結果] ボックスを調べて、皮質のヤング率 (E 0 ± σ)、細胞のバルク ヤング率 (Eb ± σ)、および皮質の厚さ (d0 ± σ) を報告します。
注:平均弾性スペクトルは、最初はノイズが多く、正弦波振動が目立つように見える場合があります。その結果、式(3)が正しくはめ込まれないことがある。この場合、平滑化SAVGOLフィルタ34 のウィンドウ長を繰り返し増加させることで、この問題が解決する。 - 分析が終了したら、[保存] ボックスの [ES] をクリックします。これにより、指定したディレクトリに、平均くぼみの深さと接触半径の関数としての平均弾性、実験に関連付けられたメタデータ(上記を参照)、および上記で説明した推定モデルパラメータを含む.tsvファイルがエクスポートされます。最後に、δとそのSDへの依存性を無視した平均弾性も報告されています。
- ソフトウェアを閉じ、保存した結果を他の任意のソフトウェアに入力して、データをさらにプロットし、統計分析を実行します。
- この手順はオプションです: グラフを右クリックして [エクスポート] を選択して、GUI からグラフをエクスポートします。グラフを.svgでエクスポートして、フォント、フォントサイズ、線のスタイルなどのパラメータを作成します。選択した別のソフトウェアで編集できます。
メモ:カスタムCPアルゴリズムとフィルタをプログラムして、既存のものに追加することができます。詳細については、 補足注 1 を参照してください。
結果
プロトコルに従って、F-z曲線のセットが得られます。データセットには、適切な曲線と、分析を続行する前に破棄する曲線が含まれている可能性があります。一般に、曲線の形状が図 4A に示すものと異なる場合は、曲線を破棄する必要があります。図5AIは、NanoPrepare GUIにアップロードされた予想されるE 0.8 KPa35のソ?...
ディスカッション
このプロトコルは、市販のフェルールトップナノインデンターを使用して、ハイドロゲルとシングルセルの両方で力分光ナノインデンテーションデータを堅牢に取得する方法を示しています。さらに、ナノインデンテーションデータの分析のための正確なワークフローを含むPythonでプログラムされたオープンソースソフトウェアの使用手順が提供されます。
プロト...
開示事項
著者は開示するものは何もありません。
謝辞
GCとMAGOは、CeMiのすべてのメンバーを認めています。MSSは、EPSRCプログラム助成金(EP/P001114/1 )による 支援を認めています。
GC:ソフトウェア(ソフトウェア開発とアルゴリズムへの貢献)、形式解析(ナノインデンテーションデータの解析)、検証、調査(ポリアクリルアミドゲルのナノインデンテーション実験)、データキュレーション、ライティング(原案、レビュー、編集)、可視化(図とグラフ)。 MAGO:調査(ゲルと細胞サンプルの準備、細胞のナノインデンテーション実験)、書き込み(元のドラフト、レビューと編集)、視覚化(図とグラフ)。 NA:検証、書き込み(レビューと編集)。 IL:ソフトウェア(ソフトウェア開発とアルゴリズムへの貢献)、検証、執筆(レビューと編集)。 MV:概念化、ソフトウェア(オリジナルソフトウェアとアルゴリズムの設計と開発)、検証、リソース、執筆(原案、レビューと編集)、監督、プロジェクト管理、資金調達 MSS:リソース、ライティング(レビューと編集)、監督、プロジェクト管理、資金調達。すべての著者が最終原稿を読み、承認しました。
資料
| Name | Company | Catalog Number | Comments |
| 12 mm coverslips | VWR | 631-1577P | |
| 35 mm cell treated culture dishes | Greiner CELLSTAR | 627160 | |
| Acrylamide | Sigma-Aldrich | A4058 | |
| Acrylsilane | Alfa Aesar | L16400 | |
| Ammonium Persulfate | Merk | 7727-54-0 | |
| Bisacrylamide | Merk | 110-26-9 | |
| Chiaro nanoindenter | Optics 11 Life | no catologue number | |
| Ethanol | general | ||
| Fetal bovine serum | Gibco | 16140071 | |
| High glucose DMEM | Gibco | 11995065 | |
| Isopropanol | general | ||
| Kimwipe | Kimberly Clark | 21905-026 | |
| Microscope glass slides | VWR | 631-1550P | |
| MilliQ system | Merk Millipore | ZR0Q008WW | |
| OP1550 Interferometer | Optics11 Life | no catalogue number | |
| Optics 11 Life probe (k = 0.02-0.005 N/m, R = 3-3.5 um) | Optics 11 Life | no catologue number | |
| Optics 11 Life probe (k = 0.46-0.5 N/m, R = 50-55 um) | Optics 11 Life | no catologue number | |
| Penicillin/Streptomycin | Gibco | 15140122 | |
| RainX rain repellent | RainX | 26012 | |
| Standard petri dishes (90 mm) | Thermo Scientific | 101RTIRR | |
| Tetramethylethylenediamine | Sigma-Aldrich | 110-18-9 | |
| Vaccum dessicator | Thermo Scientific | 531-0250 | |
| Software | |||
| Data acquisition software (v 3.4.1) | Optics 11 Life | ||
| GitHub Desktop (Optional) | Microsoft | ||
| Python 3 | Python Software Foundation | ||
| Visual Studio Code (Optional) | Microsoft |
参考文献
- Discher, D. E., Janmey, P. Y. W. Tissue cells feel and respond to the stiffness of their substrate. Science. 310 (5751), 1139-1143 (2005).
- Roca-Cusachs, P., Conte, V., Trepat, X. Quantifying forces in cell biology. Nature Cell Biology. 19 (7), 742-751 (2017).
- Guimarães, C. F., Gasperini, L., Marques, A. P., Reis, R. L. The stiffness of living tissues and its implications for tissue engineering. Nature. Reviews. Materials. 5, 351-370 (2020).
- Moeendarbary, E., Harris, A. R. Cell mechanics: Principles, practices, and prospects. Wiley Interdisciplinary. Reviews. Systems. Biology and Medicine. 6 (5), 371-388 (2014).
- Butcher, D. T., Alliston, T., Weaver, V. M. A tense situation: Forcing tumour progression. Nature Reviews Cancer. 9 (2), 108-122 (2009).
- Kubánková, M., et al. Physical phenotype of blood cells is altered in COVID-19. Biophysical Journal. 120 (14), 2838-2847 (2021).
- Engler, A. J., Sen, S., Sweeney, H. L., Discher, D. E. Matrix elasticity directs stem cell lineage specification. Cell. 126, 677-689 (2006).
- Ciccone, G., et al. What caging force cells feel in 3D hydrogels: A rheological perspective. Advanced Healthcare Materials. 9 (17), 2000517 (2020).
- Wu, P. H., et al. A comparison of methods to assess cell mechanical properties. Nature Methods. 15 (7), 491-498 (2018).
- McKee, C. T., Last, J. A., Russell, P., Murphy, C. J. Indentation versus tensile measurements of young's modulus for soft biological tissues. Tissue Engineering B Reviews. 17 (3), 155-164 (2011).
- Humphrey, J. D., Delange, S. L. . An Introduction to Biomechanics Solids and Fluids, Analysis and Design. , 271-371 (2004).
- Prevedel, R., Diz-Muñoz, A., Ruocco, G., Antonacci, G. Brillouin microscopy: an emerging tool for mechanobiology. Nature Methods. 16 (10), 969-977 (2019).
- Krieg, M., et al. Atomic force microscopy-based mechanobiology. Nature Reviews Physics. 1, 41-57 (2019).
- Norman, M. D. A., Ferreira, S. A., Jowett, G. M., Bozec, L., Gentleman, E. Measuring the elastic modulus of soft culture surfaces and three-dimensional hydrogels using atomic force microscopy. Nature Protocols. 16 (5), 2418-2449 (2021).
- Gavara, N. A beginner's guide to atomic force microscopy probing for cell mechanics. Microscopy Research and Technique. 80 (1), 75-84 (2017).
- Whitehead, A. J., Kirkland, N. J., Engler, A. J. Atomic force microscopy for live-cell and hydrogel measurement. Methods in Molecular Biology. 2299, 217-226 (2021).
- Chavan, D., et al. Ferrule-top nanoindenter: An optomechanical fiber sensor for nanoindentation. The Review of Scientific Instruments. 83 (11), 115110 (2012).
- Van Hoorn, H., Kurniawan, N. A., Koenderink Ac, G. H., Iannuzzi, D. Local dynamic mechanical analysis for heterogeneous soft matter using ferrule-top indentation. Soft Matter. 12 (12), 3066-3073 (2016).
- Baldini, F., et al. Biomechanics of cultured hepatic cells during different steatogenic hits. Journal of the Mechanical Behavior of Biomedical Materials. 97, 296-305 (2019).
- Emig, R., et al. Piezo1 channels contribute to the regulation of human atrial fibroblast mechanical properties and matrix stiffness sensing. Cells. 10 (3), 663 (2021).
- Dobre, O., et al. A hydrogel platform that incorporates laminin isoforms for efficient presentation of growth factors - Neural growth and osteogenesis. Advanced Functional Materials. 31 (21), 2010225 (2021).
- Antonovaite, N., Beekmans, S. V., Hol, E. M., Wadman, W. J., Iannuzzi, D. Regional variations in stiffness in live mouse brain tissue determined by depth-controlled indentation mapping. Scientific Reports. 8 (1), 12517 (2018).
- Wu, G., Gotthardt, M., Gollasch, M. Assessment of nanoindentation in stiffness measurement of soft biomaterials: kidney, liver, spleen and uterus. Scientific Reports. 10 (1), 18784 (2020).
- Lüchtefeld, I., et al. Elasticity spectra as a tool to investigate actin cortex mechanics. Journal of Nanobiotechnology. 18 (1), 147 (2020).
- Caliari, S. R., Burdick, J. A. A practical guide to hydrogels for cell culture. Nature Methods. 13 (5), 405-414 (2016).
- Blumlein, A., Williams, N., McManus, J. J. The mechanical properties of individual cell spheroids. Scientific Reports. 7 (1), 7346 (2017).
- Mirsandi, H., et al. Influence of wetting conditions on bubble formation from a submerged orifice. Experiments in Fluids. 61, 83 (2020).
- Vassalli, M., Ciccone, G. . CellMechLab/NanoPrepare: v0.1.1. Zendo. , (2021).
- Vassalli, M., Ciccone, G., Lüchtefeld, I. CellMechLab/nanoindentation: v1.0.0. Zendo. , (2021).
- Savitzky, A., Golay, M. J. E. Smoothing and differentiation of data by simplified least squares procedures. Analytical Chemistry. 36 (8), 1627-1639 (1964).
- Schafer, R. W. What is a savitzky-golay filter. IEEE Signal Processing Magazine. 28 (4), 111-117 (2011).
- Gavara, N. Combined strategies for optimal detection of the contact point in AFM force-indentation curves obtained on thin samples and adherent cells. Scientific Reports. 6, 21267 (2016).
- Lin, D. C., Dimitriadis, E. K., Horkay, F. Robust strategies for automated AFM force curve analysis-I. Non-adhesive indentation of soft, inhomogeneous materials. Journal of Biomechanical Engineering. 129 (3), 430-440 (2007).
- Virtanen, P., et al. SciPy 1.0: fundamental algorithms for scientific computing in Python. Nature Methods. 17 (3), 261-272 (2020).
- Tse, J. R., Engler, A. J. Preparation of hydrogel substrates with tunable mechanical properties. Current Protocols in Cell Biology. , (2010).
- Pharr, G. M., Oliver, W. C., Brotzen, F. R. On the generality of the relationship among contact stiffness, contact area, and elastic modulus during indentation. Journal of Materials Research. 7 (3), 613-617 (1992).
- Kumar, R., Saha, S., Sinha, B. Cell spread area and traction forces determine myosin-II-based cortex thickness regulation. Biochimica et Biophysica Acta - Molecular Cell Research. 1866 (12), 118516 (2019).
- Kunda, P., Pelling, A. E., Liu, T., Baum, B. Moesin controls cortical rigidity, cell rounding, and spindle morphogenesis during mitosis. Current Biology: CB. 18 (2), 91-101 (2008).
- Allen, M., Poggiali, D., Whitaker, K., Marshall, T. R., Kievit, R. A. Raincloud plots: a multi-platform tool for robust data visualization. Wellcome Open Research. 4, 63 (2019).
- Clark, A. G., Paluch, E. Mechanics and regulation of cell shape during the cell cycle. Results and Problems in Cell Differentiation. 53, 31-73 (2011).
- Langan, T. J., Rodgers, K. R., Chou, R. C. Synchronization of mammalian cell cultures by serum deprivation. Methods in Molecular Biology. 1524, 97-105 (2017).
- Hodgkinson, T., et al. The use of nanovibration to discover specific and potent bioactive metabolites that stimulate osteogenic differentiation in mesenchymal stem cells. Science Advances. 7 (9), 7921 (2021).
- Kloxin, A. M., Kloxin, C. J., Bowman, C. N., Anseth, K. S. Mechanical properties of cellularly responsive hydrogels and their experimental determination. Advanced Materials (Deerfield Beach, Fla). 22 (31), 3484-3494 (2010).
- Loessner, D., et al. Functionalization, preparation and use of cell-laden gelatin methacryloyl-based hydrogels as modular tissue culture platforms. Nature Protocols. 11 (4), 727-746 (2016).
- Yue, K., et al. Synthesis, properties, and biomedical applications of gelatin methacryloyl (GelMA) hydrogels. Biomaterials. 73, 254-271 (2015).
- Sarrigiannidis, S. O., et al. A tough act to follow: collagen hydrogel modifications to improve mechanical and growth factor loading capabilities. Materials Today. Bio. 10, 100098 (2021).
- Karoutas, A., et al. The NSL complex maintains nuclear architecture stability via lamin A/C acetylation. Nature Cell Biology. 21 (10), 1248-1260 (2019).
- Ryu, H., et al. Transparent, compliant 3D mesostructures for precise evaluation of mechanical characteristics of organoids. Advanced Materials (Deerfield Beach, Fla). 33 (25), 2100026 (2021).
- Carvalho, D. T. O., Feijão, T., Neves, M. I., Da Silva, R. M. P., Barrias, C. C. Directed self-assembly of spheroids into modular vascular beds for engineering large tissue constructs Biofabrication Directed self-assembly of spheroids into modular vascular beds for engineering large tissue constructs. Biofabrication. 13 (3), 035008 (2021).
- Chaudhuri, O., Cooper-White, J., Janmey, P. A., Mooney, D. J., Shenoy, V. B. Effects of extracellular matrix viscoelasticity on cellular behaviour. Nature. 584 (7822), 535-546 (2020).
- Cantini, M., Donnelly, H., Dalby, M. J., Salmeron-Sanchez, M. The plot thickens: The emerging role of matrix viscosity in cell mechanotransduction. Advanced Healthcare Materials. 9 (8), 1901259 (2020).
- Elosegui-Artola, A. The extracellular matrix viscoelasticity as a regulator of cell and tissue dynamics. Current Opinion in Cell Biology. 72, 10-18 (2021).
- Chaudhuri, O. Viscoelastic hydrogels for 3D cell culture. Biomaterials Science. 5 (8), 1480-1490 (2017).
- Nguyen, T. D., Gu, Y. Determination of strain-rate-dependent mechanical behavior of living and fixed osteocytes and chondrocytes using atomic force microscopy and inverse finite element analysis. Journal of Biomechanical Engineering. 136 (10), 101004 (2014).
- Mokbel, M., Hosseini, K., Aland, S., Fischer-Friedrich, E. The Poisson Ratio of the Cellular Actin Cortex Is Frequency Dependent. Biophysical Journal. 118 (8), 1968-1976 (2020).
- Javanmardi, Y., Colin-York, H., Szita, N., Fritzsche, M., Moeendarbary, E. Quantifying cell-generated forces: Poisson's ratio matters. Communications Physics. 4, 237 (2021).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved