A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
فحص الأدوية القائم على النسخ الفعال من حيث التكلفة
In This Article
Summary
يصف هذا البروتوكول سير العمل من مزارع الخلايا خارج الجسم الحي أو في المختبر إلى المعالجة المسبقة للبيانات النسخية لفحص الأدوية القائم على النسخ الفعال من حيث التكلفة.
Abstract
يسمح Transcriptomics بالحصول على رؤى شاملة حول البرامج الخلوية واستجاباتها للاضطرابات. على الرغم من الانخفاض الكبير في تكاليف إنتاج المكتبات وتسلسلها في العقد الماضي ، فإن تطبيق هذه التقنيات على النطاق اللازم لفحص المخدرات لا يزال مكلفا للغاية ، مما يعيق الإمكانات الهائلة لهذه الأساليب. تقدم دراستنا نظاما فعالا من حيث التكلفة لفحص الأدوية القائم على النسخ ، ويجمع بين ثقافات الاضطراب المصغرة والنسخ السائبة الصغيرة. يوفر بروتوكول السائبة الصغيرة المحسن إشارات بيولوجية إعلامية على عمق تسلسل فعال من حيث التكلفة ، مما يتيح فحصا مكثفا للأدوية المعروفة والجزيئات الجديدة. اعتمادا على العلاج المختار ووقت الحضانة ، سيؤدي هذا البروتوكول إلى تسلسل المكتبات في غضون أيام 2 تقريبا. نظرا للعديد من نقاط التوقف داخل هذا البروتوكول ، يمكن إجراء إعداد المكتبة ، وكذلك التسلسل ، بشكل مستقل عن الوقت. معالجة عدد كبير من العينات في وقت واحد ممكن ؛ تم اختبار قياس ما يصل إلى 384 عينة دون فقدان جودة البيانات. لا توجد أيضا قيود معروفة على عدد الحالات و / أو الأدوية ، على الرغم من مراعاة التباين في أوقات حضانة الدواء المثلى.
Introduction
يعد تطوير عقاقير جديدة عملية معقدة وتستغرق وقتا طويلا وتتضمن تحديد الأدوية المحتملة وأهدافها ، وتحسين وتوليف الأدوية المرشحة ، واختبار فعاليتها وسلامتها في التجارب قبل السريرية والسريرية1. تتضمن الطرق التقليدية لفحص الأدوية ، أي التقييم المنهجي لمكتبات المركبات المرشحة للأغراض العلاجية ، استخدام نماذج حيوانية أو مقايسات قائمة على الخلايا لاختبار التأثيرات على أهداف أو مسارات محددة. في حين أن هذه الطرق كانت ناجحة في تحديد الأدوية المرشحة ، إلا أنها في كثير من الأحيان لم تقدم رؤى كافية حول الآليات الجزيئية المعقدة الكامنة وراء فعالية الدواء وكذلك السمية وآليات الآثار الجانبية المحتملة.
يقدم تقييم حالات النسخ على مستوى الجينوم نهجا قويا للتغلب على القيود الحالية في فحص الأدوية ، لأنه يتيح إجراء تقييمات شاملة للتعبير الجيني استجابة للعلاجات الدوائية2. من خلال قياس نسخ الحمض النووي الريبي بطريقة على مستوى الجينوم يتم التعبير عنها في وقت معين ، يهدف علم النسخ إلى توفير نظرة شاملة للتغيرات النسخية التي تحدث استجابة للأدوية ، بما في ذلك التغيرات في أنماط التعبير الجيني ، والربط البديل ، وتعبير الحمض النووي الريبي غير المشفر3. يمكن استخدام هذه المعلومات لتحديد أهداف الأدوية ، والتنبؤ بفعالية الدواء وسميته ، وتحسين جرعات الأدوية وأنظمة العلاج.
تتمثل إحدى الفوائد الرئيسية للجمع بين علم النسخ وفحص الأدوية غير المتحيز في إمكانية تحديد أهداف دوائية جديدة لم يتم النظر فيها من قبل. غالبا ما تركز مناهج فحص الأدوية التقليدية على الجزيئات أو المسارات المستهدفة المحددة ، مما يعيق تحديد أهداف جديدة ويحتمل أن يؤدي إلى عقاقير ذات آثار جانبية غير متوقعة وفعالية محدودة. يمكن لعلم النسخ التغلب على هذه القيود من خلال تقديم نظرة ثاقبة للتغيرات الجزيئية التي تحدث استجابة للعلاج الدوائي ، والكشف عن الأهداف أو المسارات المحتملة التي ربما لم يتم النظر فيها سابقا2.
بالإضافة إلى تحديد أهداف دوائية جديدة ، يمكن أيضا استخدام علم النسخ للتنبؤ بفعالية الدواء وسميته. من خلال تحليل أنماط التعبير الجيني المرتبطة بالاستجابات الدوائية ، يمكن تطوير المؤشرات الحيوية التي يمكن استخدامها للتنبؤ باستجابة المريض لدواء معين أو نظام علاج. يمكن أن يساعد هذا أيضا في تحسين جرعات الدواء وتقليل مخاطر الآثار الجانبية الضارة4.
على الرغم من فوائدها المحتملة ، لا تزال تكلفة النسخ تشكل عائقا كبيرا أمام تطبيقها على نطاق واسع في فحص المخدرات. يتطلب تحليل النسخ معدات متخصصة وخبرة فنية وتحليل البيانات ، مما قد يجعل من الصعب على فرق البحث الأصغر أو المنظمات ذات التمويل المحدود استخدام علم النسخ في فحص الأدوية. ومع ذلك ، فإن تكلفة النسخ آخذة في الانخفاض بشكل مطرد ، مما يجعلها في متناول مجتمعات البحث. بالإضافة إلى ذلك ، جعلت التطورات في التكنولوجيا وطرق تحليل البيانات النسخ أكثر كفاءة وفعالية من حيث التكلفة ، مما زاد من إمكانية الوصول إليها2.
في هذا البروتوكول ، نصف نظاما استكشافيا عالي الأبعاد لفحص الأدوية القائم على النسخ ، ويجمع بين ثقافات الاضطراب المصغرة وتحليل النسخ السائبالمصغر 5,6. باستخدام هذا البروتوكول ، من الممكن تقليل تكلفة العينة إلى 1/6 من التكلفة الحالية للحلول التجارية لتسلسل mRNA كامل الطول. ولا يتطلب البروتوكول سوى معدات مختبرية قياسية، والاستثناء الوحيد هو استخدام تكنولوجيات تسلسل القراءة القصيرة، التي يمكن الاستعانة بمصادر خارجية إذا لم تكن أدوات التسلسل متاحة داخليا. يوفر بروتوكول الحجم الصغير المحسن إشارات بيولوجية غنية بالمعلومات على عمق تسلسل فعال من حيث التكلفة ، مما يتيح فحصا مكثفا للأدوية المعروفة والجزيئات الجديدة.
الهدف من التجربة هو فحص نشاط الدواء على PBMCs في سياقات بيولوجية مختلفة. يمكن تطبيق هذا البروتوكول على أي سؤال بيولوجي حيث يجب اختبار العديد من الأدوية باستخدام قراءة نسخية ، مما يعطي رؤية واسعة للنسخ للتأثير الخلوي للعلاج.
Protocol
يتبع هذا البروتوكول المبادئ التوجيهية للجان الأخلاقيات المحلية في جامعة بون.
1. إعداد المخازن المؤقتة والحلول والمعدات
- إعداد الحلول وجمع المواد الموضحة في جدول المواد.
- قم بتسخين الحمام المائي إلى 37 درجة مئوية وقم بتسخين وسط النمو الكامل (RPMI-1640 + 10٪ مصل عجل الجنين (FCS) + 1٪ بنسلين / ستربتومايسين).
- لحصاد الخلايا ، استخدم محلول ملحي مخزن بالفوسفات المثلج (PBS).
ملاحظة: حافظ على بيئة نظيفة أثناء العمل مع الخلايا للحفاظ على العقم.
2. التعامل مع الخلايا
ملاحظة: يمكن العثور على بروتوكول مفصل للحفظ بالتبريد لخلايا الدم أحادية النواة المحيطية (PBMC) من دم الإنسان في7.
- ذوبان الخلايا والعد
- قم بإزالة الكريوفيالات من النيتروجين السائل وقم بإذابتها في حمام مائي عند 37 درجة مئوية لمدة 2-3 دقائق مع التقليب برفق.
- انقل الخلايا المذابة إلى أنبوب مخروطي سعة 50 مل.
- شطف cryovial مع 1 مل دافئ وسط النمو الكامل وإضافة هذا الحل قطرة إلى الخلايا في الأنبوب (1ش خطوة التخفيف).
- كرر التخفيف 1: 1 حتى تصل إلى حجم 32 مل (إجمالي 5 تخفيفات مع 1 و 2 و 4 و 8 و 16 مل على التوالي من وسط النمو الكامل الدافئ). أضف الوسط بالتنقيط لتقليل اضطراب الخلايا مع تحريك الأنبوب المخروطي قليلا.
- قم بطرد مركزي تعليق الخلية لمدة 5 دقائق (300 × جم ، 20 درجة مئوية) وقم بإزالة المادة الطافية عن طريق قلب الأنبوب المخروطي برفق بحركة واحدة بطلاقة.
- أعد تعليق الخلايا في 3 مل من وسط النمو الكامل الدافئ واستمر في عد الخلايا.
- للعد ، امزج 10 ميكرولتر من معلق الخلية مع Trypan Blue (تخفيف 1: 2 إلى 1:10 اعتمادا على كثافة تعليق الخلية) لتمييز الخلايا الحية عن الخلايا الميتة أثناء العد. ستظهر الخلايا الميتة أو التالفة باللون الأزرق بسبب امتصاص الصبغة ، في حين أن الخلايا الحيوية غير ملطخة.
ملاحظة: Trypan Blue سام للخلايا قليلا ، ويجب عدم تخزين الخلايا الملطخة لأكثر من 5 دقائق. - عد الخلايا إما باستخدام عداد خلية آلي أو غرفة عد باستخدام 10 ميكرولتر من محلول الخلايا الملطخة.
- عند استخدام حجرة الخلية المحسنة بواسطة نيوباور ، عد جميع الخلايا داخل المربعات الأربعة الكبيرة الموجودة في الزوايا واستخدم المعادلة التالية لحساب تركيز تعليق الخلية.
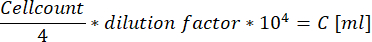
- عند استخدام حجرة الخلية المحسنة بواسطة نيوباور ، عد جميع الخلايا داخل المربعات الأربعة الكبيرة الموجودة في الزوايا واستخدم المعادلة التالية لحساب تركيز تعليق الخلية.
- تمييع الخلايا إلى تركيز نهائي قدره 1 × 106 خلايا / مل مع وسط نمو كامل دافئ. أجهزة الطرد المركزي فقط إذا كان الحجم المطلوب أقل من 3 مل وإعادة تعليق بيليه الخلية إلى الحجم الصحيح.
- بذر الخلايا وعلاجها
- بذرة 100 ميكرولتر من تعليق الخلية لكل بئر في لوحة زراعة الخلايا 96 بئرا (1 × 105 خلايا / بئر).
ملاحظة: تم تحسين رقم الخلية للبذر لمزارع PBMC. في حين أن تركيزات الخلايا المنخفضة لن تنتج كمية كافية من الحمض النووي الريبي للتسلسل ، فإن العدد المفرط من الخلايا سيزيد من خطر تحلل الخلايا دون المستوى الأمثل وتثبيط تفاعل النسخ العكسي. - تحضير تخفيفات الدواء بتركيز مرتين من التركيز المستخدم للعلاج (2x). اختر المذيب وفقا لقابلية ذوبان المركب ، ويفضل أن يكون متوسط النمو الكامل.
ملاحظة: يمكن استخدام PBS أو ثنائي ميثيل سلفوكسيد (DMSO) إذا تعذر الوصول إلى قابلية ذوبان كافية بخلاف ذلك. يجب ألا يتجاوز الحد الأقصى لتركيز DMSO في حجم الحضانة الناتج 0.5٪ لمنع السمية الخلوية التي يسببها المذيب. - أضف 100 ميكرولتر من تخفيف الدواء 2x (التركيز النهائي في البئر 1X) واحتضانه عند 37 درجة مئوية للوقت المحدد.
ملاحظة: ينبغي إجراء تحقيقات تكميلية من أجل تحديد الوقت الأمثل لحضانة الدواء واستبعاد الآليات المحتملة للسمية الخلوية.
- بذرة 100 ميكرولتر من تعليق الخلية لكل بئر في لوحة زراعة الخلايا 96 بئرا (1 × 105 خلايا / بئر).
- حصاد الخلايا والتحلل
- جهاز طرد مركزي لوحة زراعة الخلايا لمدة 10 دقائق (300 × جم ، 20 درجة مئوية). قم بإزالة المادة الطافية برفق باستخدام مضخة تفريغ تحافظ على اللوحة بزاوية (30 درجة -45 درجة) مع توجيه الطرف في الزاوية المنخفضة من البئر لإزالة أقل قدر ممكن من الخلايا.
- اغسل الخلايا بإضافة 200 ميكرولتر من برنامج تلفزيوني بارد إلى كل بئر وطرد مركزي للوحة لمدة 5 دقائق (300 × جم ، 4 درجات مئوية). قم بإزالة المادة الطافية بمضخة تفريغ ، مرة أخرى ، مع الحفاظ على اللوحة بزاوية حادة لإزالة أكبر قدر ممكن من PBS.
- قم بإعداد المخزن المؤقت للتحلل كما هو موضح في الجدول 1 لعدد التفاعلات (rxn) المطلوبة ، مضيفا زيادة بنسبة 10٪.
- أضف 15 ميكرولتر من محلول التحلل إلى كل بئر وأغلق اللوحة بغشاء مانع للتسرب لاصق لحماية العينات من التلوث. دوامة اللوحة قبل الطرد المركزي لمدة 1 دقيقة (1000 × جم ، 4 درجات مئوية) واحتضانها لمدة 5 دقائق على الجليد.
- اجمع 6 ميكرولتر من محلول الخلية المحللة في لوحة تفاعل البوليميراز المتسلسل وقم بتجميد محللة الخلية لفترة وجيزة عند -80 درجة مئوية لضمان تحلل الخلية الأمثل. تأكد من تجنب دورات التجميد المتعددة للألواح.
ملاحظة: نقطة التوقف: إذا لم يتم إنتاج cDNA لاحقا ، فيمكن تخزين تحلل الخلية عند -80 درجة مئوية لمدة تصل إلى عدة أشهر دون انخفاض كبير في جودة الحمض النووي الريبي.
3. إعداد المكتبة للتسلسل
- تفاعل النسخ العكسي (RT)
ملاحظة: عند العمل مع الحمض النووي الريبي ، ضع جميع العينات على الثلج وتأكد من استخدام معدات خالية من النيوكلياز (أدوات بلاستيكية معقمة يمكن التخلص منها) والماء.- قم بإعداد مزيج تفاعل RT كما هو موضح في الجدول 2 لعدد التفاعلات المطلوبة ، مع إضافة 10٪ زيادة. دوامة لفترة وجيزة المزيج وتدور لفترة وجيزة. احتفظ بالمزيج على الثلج حتى الاستخدام.
- قم بإذابة الخلية المحللة في درجة حرارة الغرفة (RT) وقم بتدويرها لفترة وجيزة لجمع كل محللات الخلايا في أسفل اللوحة.
- قم بإجراء تمسخ mRNA على جهاز تدوير حراري كما هو موضح في الجدول 3.
- أخرج اللوحة من جهاز الدورة الحرارية وقم بتدويرها لأسفل لفترة وجيزة لجمع المكثفات المحتملة. أضف 6 ميكرولتر من مزيج تفاعل RT في كل بئر للحصول على حجم نهائي يبلغ 12 ميكرولتر. أغلق اللوحة لحماية اللوحة ولتجنب التبخر والدوامة وقم بتدويرها لأسفل.
- ضع اللوحة على جهاز التدوير الحراري وابدأ برنامج تفاعل RT كما هو موضح في الجدول 3.
- التضخيم المسبق
- قم بإذابة بوليميراز الحمض النووي عالي الدقة وبرايمر تفاعل البوليميراز المتسلسل في الموقع (ISPCR) في درجة حرارة الغرفة.
- قم بإعداد مزيج التضخيم المسبق كما هو موضح في الجدول 4 لعدد التفاعلات المطلوبة ، مع إضافة 10٪ من الزيادة. دوامة لفترة وجيزة المزيج وتدويره.
ملاحظة: مزيج الإنزيم مستقر في درجة حرارة الغرفة لساعات. - قم بتدوير لوحة PCR ، وأضف 15 ميكرولتر من مزيج التضخيم المسبق إلى كل بئر وأغلق اللوحة.
- ضعه على جهاز التدوير الحراري وابدأ تفاعل التضخيم المسبق كما هو موضح في الجدول 5.
- اضبط عدد الدورات بسبب محتوى الحمض النووي الريبي. ابدأ برقم أقل وقم بزيادته إذا لم يكن إنتاج cDNA كافيا.
ملاحظة: في تجربتنا ، يجب تحديد العدد الأمثل للدورات لكل نوع من أنواع الخلايا ، ولن تؤثر الحالة التجريبية والعلاج على العدد الأمثل للدورات. لذلك ، نوصي بإنشاء العدد الأمثل للدورات تجريبيا عند إنشاء هذا البروتوكول لنوع خلية جديد. بشكل عام ، ستتطلب الخلايا الأولية عددا أكبر من الدورات مقارنة بخطوط الخلايا. نقطة التوقف: يمكن تخزين منتج التضخيم المسبق عند - 20 درجة مئوية.
- اضبط عدد الدورات بسبب محتوى الحمض النووي الريبي. ابدأ برقم أقل وقم بزيادته إذا لم يكن إنتاج cDNA كافيا.
- تنظيف cDNA ومراقبة الجودة (QC)
ملاحظة: يمكن إجراء تنظيف العينة بالتتابع لكل لوحة لأن العينات مستقرة إلى حد ما في هذه المرحلة. ستؤدي زيادة عدد العينات إلى مدة أطول للبروتوكول ولكنها لن تحد من عدد العينات التي يمكن معالجتها في وقت واحد.- قبل البدء ، أحضر حبات التنقية المغناطيسية إلى درجة حرارة الغرفة والدوامة على سرعة عالية لمدة 1 دقيقة لإعادة تعليق الخرز بالكامل.
- أضف 0.8x v / v (20 μL) حبات تنقية مغناطيسية إلى كل بئر واحتضانها في درجة حرارة الغرفة لمدة 5 دقائق.
- ضع لوحة PCR على رف مغناطيسي لمدة 5 دقائق حتى يتم فصل الحبيبات تماما. قم بإزالة المادة الطافية بعناية.
- احتفظ باللوحة على الرف المغناطيسي ، أضف برفق 100 ميكرولتر من الإيثانول الطازج بنسبة 80٪ لغسل الخرز واحتضانه لمدة 30 ثانية. استخدم ماصة صغيرة الحجم لإزالة المادة الطافية. كرر لخطوة غسيل إضافية. تأكد من إزالة أكبر قدر ممكن من الإيثانول.
- جفف الخرزات الموجودة على الرف المغناطيسي في الهواء في درجة حرارة الغرفة لمدة تصل إلى 5 دقائق حتى يتبخر الإيثانول بالكامل ولم تعد الحبيبات تبدو لامعة.
- أخرج اللوحة من الرف المغناطيسي ، وأعد تعليق الخرزات في 20 ميكرولتر من الماء الخالي من النيوكلياز واحتضانها لمدة دقيقتين.
- ضع اللوحة مرة أخرى على الرف المغناطيسي حتى يتم فصل الخرزات.
- استعادة الشطف في لوحة PCR جديدة ل cDNA QC ووضع العلامات.
- قم بإجراء مقايسة TapeStation أو FragmentAnalyzer لتقييم توزيع حجم وتركيز مكتبة cDNA (موصى به: مقايسة TapeStation D5000). للحصول على التفاصيل، راجع تعليمات الشركة المصنعة. من المتوقع حدوث عائد نموذجي يبلغ 20 نانوغرام.
- وضع العلامات مع عدة
ملاحظة: يمكن استخدام بروتوكولات وضع العلامات الأخرى إذا تم إنشاؤها بالفعل.- قم بتخفيف cDNA إلى تركيز نهائي يتراوح بين 150 و 300 بيكوغرام / ميكرولتر باستخدام الماء الخالي من النيوكلياز.
- قم ببرمجة جهاز التدوير الحراري مسبقا وفقا للجدول 6 لضمان البدء الفوري لتفاعل وضع العلامات بعد إضافة cDNA إلى مزيج الإنزيم.
- قم بإعداد مزيج العلامات كما هو موضح في الجدول 7 لعدد التفاعلات المطلوبة ، مع إضافة 10٪ زيادة.
- قم بتوزيع 3 ميكرولتر من مزيج العلامات لكل تفاعل على لوحة PCR جديدة وأضف 1 ميكرولتر من cDNA إلى كل تفاعل.
- ابدأ تفاعل وضع العلامات مباشرة بعد إضافة الحمض النووي الكيميائي.
- قم بتعطيل التفاعل عن طريق إضافة 1 ميكرولتر من المخزن المؤقت لتحييد العلامات (NT ؛ بدلا من ذلك ، يمكن استخدام 0.2٪ كبريتات دوديسيل الصوديوم (SDS)).
- إثراء PCR
- تحضير مزيج PCR التخصيب الجدول 7 لعدد التفاعلات ، مضيفا 10 ٪ زيادة. (موصى به: تم تعيين Nextera UDI لمنع قفز الفهرس على خلايا التدفق المنقوشة (على سبيل المثال ، Illumina NovaSeq 6000)).
- أضف 9 ميكرولتر من مزيج PCR للتخصيب إلى كل تفاعل لحجم إجمالي قدره 14 ميكرولتر.
- قم بتشغيل برنامج PCR الإثرائي كما هو موضح في الجدول 8.
ملاحظة: بالنسبة ل PCR للتخصيب ، هناك 16 دورة قياسية. إذا كانت جودة cDNA رديئة ، فيمكن إضافة دورات إضافية.
- التنظيف ومراقبة الجودة
- قبل البدء ، أحضر حبات التنقية المغناطيسية إلى درجة حرارة الغرفة والدوامة على سرعة عالية لمدة 1 دقيقة لإعادة تعليق الخرز بالكامل.
- أضف حبات تنقية مغناطيسية 1.0x v / v (14 ميكرولتر) واحتضانها في درجة حرارة الغرفة لمدة 5 دقائق.
- ضع اللوحة على رف مغناطيسي وانتظر لمدة 5 دقائق حتى يتم فصل الخرزات تماما. قم بإزالة المادة الطافية بعناية.
- احتفظ باللوحة على الرف المغناطيسي ، أضف برفق 100 ميكرولتر من الإيثانول الطازج بنسبة 80٪ لغسل الخرز واحتضانه لمدة 30 ثانية. استخدم ماصة صغيرة الحجم لإزالة المادة الطافية. كرر لخطوة غسيل إضافية. تأكد من إزالة أكبر قدر ممكن من الإيثانول.
- جفف الخرزات في الهواء في درجة حرارة الغرفة لمدة 3 دقائق أو حتى تختفي من اللمعان.
- أخرج اللوحة من الرف المغناطيسي ، وأعد تعليق الخرزات في 20 ميكرولتر من الماء الخالي من النيوكلياز واحتضانها لمدة دقيقتين.
- ضع اللوحة مرة أخرى على الرف المغناطيسي حتى تنفصل الحبيبات وتستعيد التجويف في لوحة PCR جديدة لمراقبة الجودة في المكتبة.
- قم بإجراء مقايسة TapeStation أو محلل الأجزاء لتقييم توزيع حجم وتركيز مكتبة cDNA (موصى به: مقايسة TapeStation D1000 عالية الحساسية). للحصول على التفاصيل، راجع تعليمات الشركة المصنعة. في المتوسط ، من المتوقع تحقيق عائد قدره 10 نانوغرام.
ملاحظة: نقطة التوقف: يمكن تخزين منتج PCR في - 20 درجة مئوية.
4. التسلسل والمعالجة المسبقة للبيانات
- التسلسل
ملاحظة: سيتم تطبيق المبدأ التوجيهي التالي على جميع أدوات Illumina لتسلسل القراءة القصيرة. إذا لم تكن الأجهزة متوفرة ، يمكن إجراء التسلسل بواسطة مرفق تسلسل خارجي. ويمكن أيضا استخدام نهج أخرى للتسلسل. من أجل البساطة ، اخترنا الإبلاغ عن تقنية التسلسل الأكثر استخداما فقط.
ملاحظة: تصف الخطوات التالية المتعلقة باستخدام البرنامج الإجراء على جهاز التسلسل Illumina NovaSeq6000.- تجميع المكتبات المفهرسة بشكل فريد بنسبة متساوية وفقا للنتائج التي تم الحصول عليها في الخطوة 3.6.8.
- قم بقياس تركيز التجمع النهائي باستخدام مقايسة عالية الحساسية لحساب مولارية العينة على النحو التالي:
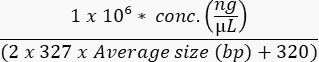
- قم بتحميل خلية التدفق وفقا لمواصفات الجهاز والتحسين التجريبي. وترد في الجدول 9 أمثلة لتركيزات تحميل الأدوات الشائعة.
- عن طريق لمس الشاشة ، حدد تسلسل لبدء إعداد التشغيل.
- اتبع الإرشادات التي تظهر على الشاشة وخلية تدفق التحميل وخرطوشة التسلسل حسب التركيب وخرطوشة التجميع وخرطوشة المخزن المؤقت وتأكد من أن حاويات النفايات فارغة.
- بمجرد التعرف على جميع الكواشف بواسطة الأداة ، انقر فوق تشغيل الإعداد. حدد هنا اسم التشغيل ومجلد الإخراج لتخزين البيانات.
- حدد تفاصيل التسلسل على أنها نهاية مزدوجة مع كلتا القراءتين 51 نقطة أساس. كما يتم تسلسل قراءتين للمؤشر ب 8 نقاط أساس لكل منهما.
- اضغط على مراجعة وبعد التحقق مما إذا كانت جميع تفاصيل التسلسل صحيحة ، اضغط على Start Run.
ملاحظة: عمق التسلسل الموصى به هو 5 × 106 قراءات / عينة ، بحد أدنى 1 × 106 قراءات / عينة.
- المعالجة المسبقة للبيانات
- قم بتحويل بيانات التسلسل الخام إلى تنسيق FASTQ وإلغاء تعدد الإرسال وفقا لفهارس العينة باستخدام الأداة Bcl2Fastq2. قم بإجراء إلغاء تعدد الإرسال باستخدام الإعدادات الافتراضية. للحصول على إرشادات مفصلة حول Bcl2Fastq2 ، راجع الدليل المرجعي.
ملاحظة: عادة ما يتم إجراء تحويل FASTQ وإزالة تعدد الإرسال بواسطة مرفق التسلسل إذا لم يتم إجراء التسلسل داخل الشركة. عادة ما توفر مرافق التسلسل FASTQ غير المضاعف لمزيد من المعالجة. - تتوفر العديد من الخيارات لمحاذاة البيانات والقياس الكمي الوفير لقراءات التسلسل (موصى به: خط أنابيب nf-core RNA-seq (https://nf-co.re/rnaseq)). يوفر خط الأنابيب العديد من الخيارات ، استخدم الإعداد الافتراضي مع محاذاة STAR8 و Salmon9 لتحديد وفرة النص.
- ملاحظة: لمزيد من تحليل المعلوماتية الحيوية ، تتوفر عدة طرق. ليس في نطاق هذا البروتوكول تغطية الكل (موصى به: خط أنابيب DEseq210). يمكن العثور على برنامج نصي قياسي يعتمد على سير عمل DEseq2 الذي طورناه على GitHub (https://github.com/jsschrepping/RNA-DESeq2).
- قم بتحويل بيانات التسلسل الخام إلى تنسيق FASTQ وإلغاء تعدد الإرسال وفقا لفهارس العينة باستخدام الأداة Bcl2Fastq2. قم بإجراء إلغاء تعدد الإرسال باستخدام الإعدادات الافتراضية. للحصول على إرشادات مفصلة حول Bcl2Fastq2 ، راجع الدليل المرجعي.
النتائج
باتباع البروتوكول المبلغ عنه ، تم زرع PBMCs البشرية ، ومعالجتها بأدوية مناعية مختلفة ، وبعد أوقات حضانة مختلفة ، تم حصادها لتحليل النسخ السائب باستخدام بروتوكول التسلسل (الشكل 1).
يجب تحديد تركيزات الدواء المثالية وأوقات الحضانة لمركبات الاختبار قبل هذا البرو...
Discussion
يمكن أن يستفيد اكتشاف الأدوية وتطوير الأدوية بشكل كبير من النظرة الشاملة للعمليات الخلوية التي يمكن أن توفرها النسخ السائبة. ومع ذلك ، غالبا ما يكون هذا النهج محدودا بسبب التكلفة العالية للتجربة مع بروتوكول RNA-seq القياسي السائب ، مما يحظر تطبيقه في البيئات الأكاديمية بالإضافة إلى إمكانية ...
Disclosures
يعلن المؤلفون عدم وجود مصالح متنافسة.
Acknowledgements
يتم دعم JLS من قبل مؤسسة الأبحاث الألمانية (DFG) في إطار استراتيجية التميز الألمانية (EXC2151-390873048) ، وكذلك بموجب SCHU 950 / 8-1 ؛ GRK 2168 ، TP11 ؛ CRC SFB 1454 Metaflammation ، IRTG GRK 2168 ، WGGC INST 216/981-1 ، CCU INST 217/988-1 ، مشروع التميز الممول من BMBF النظام الغذائي - الجسم - الدماغ (DietBB) ؛ ومشروع الاتحاد الأوروبي SYSCID بموجب المنحة رقم 733100. MBمدعوم من DFG (IRTG2168-272482170 ، SFB1454-432325352). L.B. مدعوم من DFG (ImmuDiet BO 6228 / 2-1 - رقم المشروع 513977171) واستراتيجية التميز الألمانية (EXC2151-390873048). الصور التي تم إنشاؤها باستخدام BioRender.com.
Materials
| Name | Company | Catalog Number | Comments |
| 50 mL conical tube | fisher scientific | 10203001 | |
| Adhesive PCR Plate Seals | Thermo Fisher Scientific | AB0558 | |
| Amplicon Tagment Mix (ATM) | Illumina | FC-131-1096 | Nextera XT DNA Library Prep Kit (96 samples) |
| AMPure XP beads | Beckman Coulter | A 63881 | |
| Betaine | Sigma-Aldrich | 61962 | |
| Cell culture grade 96-well plates | Thermo Fisher Scientific | 260860 | |
| Cell culture vacuum pump (VACUSAFE) | Integra Bioscience | 158300 | |
| Deoxynucleotide triphosphates (dNTPs) mix 10 mM each | Fermentas | R0192 | |
| DMSO | Sigma-Aldrich | 276855 | |
| DTT (100 mM) | Invitrogen | 18064-014 | |
| EDTA | Sigma-Aldrich | 798681 | for adherent cells |
| Ethanol | Sigma-Aldrich | 51976 | |
| Fetal Bovine Serum | Thermo Fisher Scientific | 26140079 | |
| Filter tips (10 µL) | Gilson | F171203 | |
| Filter tips (100 µL) | Gilson | F171403 | |
| Filter tips (20 µL) | Gilson | F171303 | |
| Filter tips (200 µL) | Gilson | F171503 | |
| Guanidine Hydrochloride | Sigma-Aldrich | G3272 | |
| ISPCR primer (10 µM) | Biomers.net GmbH | SP10006 | 5′-AAGCAGTGGTATCAACGCAGAG T-3′ |
| KAPA HiFi HotStart ReadyMix (2X) | KAPA Biosystems | KK2601 | |
| Magnesium chloride (MgCl2) | Sigma-Aldrich | M8266 | |
| Magnetic stand 96 | Ambion | AM10027 | |
| Neutralize Tagment (NT) Buffer | Illumina | FC-131-1096 | Nextera XT DNA Library Prep Kit (96 samples), alternatively 0.2 % SDS |
| Nextera-compatible indexing primer | Illumina | ||
| Nuclease-free water | Invitrogen | 10977049 | |
| PBS | Thermo Fisher Scientific | AM9624 | |
| PCR 96-well plates | Thermo Fisher Scientific | AB0600 | |
| PCR plate sealer | Thermo Fisher Scientific | HSF0031 | |
| Penicillin / Streptomycin | Thermo Fisher Scientific | 15070063 | |
| Qubit 4 fluorometer | Invitrogen | 15723679 | |
| Recombinant RNase inhibitor (40 U/ul) | TAKARA | 2313A | |
| RPMI-1640 cell culture medium | Gibco | 61870036 | If not working with PBMCs, adjust to cell type |
| SMART dT30VN primer | Sigma-Aldrich | 5' Bio-AAGCAGTGGTATCAACGCAGAG TACT30VN-3 | |
| Standard lab equipment | various | various | e.g. centrifuge, ice machine, ice bucket, distilled water, water bath |
| SuperScript II Reverse Transcriptase (SSRT II) | Thermo Fisher Scientific | 18064-014 | |
| SuperScript II Reverse Transcriptase (SSRT II) buffer (5x) | Thermo Fisher Scientific | 18064-014 | |
| Tagment DNA Buffer (TD) | Illumina | FC-131-1096 | Nextera XT DNA Library Prep Kit (96 samples) |
| TapeStation system 4200 | Agilent | G2991BA | |
| Thermocycler (S1000) | Bio-Rad | 1852148 | |
| TSO-LNA (100 uM) | Eurogentec | 5' Biotin AAGCAGTGGTATCAACGCAGAG TACAT(G)(G){G | |
| Vortex-Genie 2 Mixer | Sigma-Aldrich | Z258415 |
References
- Hughes, J. P., Rees, S., Kalindjian, S. B., Philpott, K. L. Principles of early drug discovery. Br J Pharmacol. 162 (6), 1239-1249 (2011).
- Yang, X., et al. High-throughput transcriptome profiling in drug and biomarker discovery. Front Genet. 11, 19 (2020).
- Bonaguro, L., et al. A guide to systems-level immunomics. Nat Immunol. 23 (10), 1412-1423 (2022).
- Carraro, C., et al. Decoding mechanism of action and sensitivity to drug candidates from integrated transcriptome and chromatin state. ELife. 11, 78012 (2022).
- Picelli, S., et al. Smart-seq2 for sensitive full-length transcriptome profiling in single cells. Nat Methods. 10 (11), 1096-1098 (2013).
- Picelli, S., et al. Full-length RNA-seq from single cells using Smart-seq2. Nat Protoc. 9 (1), 171-181 (2014).
- De Domenico, E., et al. Optimized workflow for single-cell transcriptomics on infectious diseases including COVID-19. STAR Protoc. 1 (3), 100233 (2020).
- Dobin, A., et al. ultrafast universal RNA-seq aligner. Bioinformatics. 29 (1), 15-21 (2013).
- Patro, R., et al. Salmon provides fast and bias-aware quantification of transcript expression. Nat Methods. 14 (4), 417-419 (2017).
- Love, M. I., Huber, W., Anders, S. Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2. Genome Biol. 15 (12), 550 (2014).
- Frankish, A., et al. GENCODE reference annotation for the human and mouse genomes. Nucleic Acids Res. 47, D766-D773 (2019).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved