Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Cribado de fármacos basado en transcriptómica rentable
En este artículo
Resumen
Este protocolo describe un flujo de trabajo desde los cultivos celulares ex vivo o in vitro hasta el preprocesamiento de datos transcriptómicos para el cribado rentable de fármacos basado en transcriptomas.
Resumen
La transcriptómica permite obtener información completa sobre los programas celulares y sus respuestas a las perturbaciones. A pesar de una disminución significativa en los costos de producción y secuenciación de bibliotecas en la última década, la aplicación de estas tecnologías a la escala necesaria para el cribado de drogas sigue siendo prohibitivamente costosa, lo que obstruye el inmenso potencial de estos métodos. Nuestro estudio presenta un sistema rentable para el cribado de fármacos basado en transcriptomas, que combina cultivos de perturbaciones miniaturizados con transcriptómica minimasiva. El protocolo minimasivo optimizado proporciona señales biológicas informativas a una profundidad de secuenciación rentable, lo que permite un cribado exhaustivo de fármacos conocidos y nuevas moléculas. Dependiendo del tratamiento elegido y del tiempo de incubación, este protocolo dará como resultado la secuenciación de las bibliotecas en aproximadamente 2 días. Debido a varios puntos de parada dentro de este protocolo, la preparación de la biblioteca, así como la secuenciación, se pueden realizar independientemente del tiempo. Es posible procesar simultáneamente un gran número de muestras; Se probó la medición de hasta 384 muestras sin pérdida de calidad de los datos. Tampoco se conocen limitaciones en cuanto al número de afecciones y/o fármacos, a pesar de tener en cuenta la variabilidad en los tiempos óptimos de incubación de fármacos.
Introducción
El desarrollo de nuevos fármacos es un proceso complejo y lento que implica la identificación de fármacos potenciales y sus dianas, la optimización y síntesis de candidatos a fármacos, y la comprobación de su eficacia y seguridad en ensayos preclínicos y clínicos1. Los métodos tradicionales para el cribado de fármacos, es decir, la evaluación sistemática de bibliotecas de compuestos candidatos con fines terapéuticos, implican el uso de modelos animales o ensayos basados en células para probar los efectos sobre dianas o vías específicas. Si bien estos métodos han tenido éxito en la identificación de candidatos a fármacos, a menudo no proporcionaron información suficiente sobre los complejos mecanismos moleculares que subyacen a la eficacia de los medicamentos y también sobre la toxicidad y los mecanismos de los posibles efectos secundarios.
La evaluación de los estados transcripcionales de todo el genoma presenta un enfoque poderoso para superar las limitaciones actuales en el cribado de fármacos, ya que permite realizar evaluaciones exhaustivas de la expresión génica en respuesta a los tratamientos farmacológicos2. Al medir las transcripciones de ARN de manera genómica expresada en un momento dado, la transcriptómica tiene como objetivo proporcionar una visión holística de los cambios transcripcionales que ocurren en respuesta a los fármacos, incluidos los cambios en los patrones de expresión génica, el empalme alternativo y la expresión de ARN no codificante3. Esta información se puede utilizar para determinar los objetivos farmacológicos, predecir la eficacia y toxicidad de los fármacos, y optimizar la dosificación de los fármacos y los regímenes de tratamiento.
Uno de los beneficios clave de combinar la transcriptómica con el cribado imparcial de fármacos es la posibilidad de identificar nuevas dianas farmacológicas que no se han considerado previamente. Los enfoques convencionales de cribado de fármacos a menudo se centran en moléculas o vías diana establecidas, lo que dificulta la identificación de nuevas dianas y puede dar lugar a fármacos con efectos secundarios imprevistos y eficacia restringida. La transcriptómica puede superar estas limitaciones al proporcionar información sobre los cambios moleculares que se producen en respuesta al tratamiento farmacológico, descubriendo posibles dianas o vías que pueden no haberse considerado previamente2.
Además de la identificación de nuevas dianas farmacológicas, la transcriptómica también puede utilizarse para predecir la eficacia y la toxicidad de los fármacos. Mediante el análisis de los patrones de expresión génica asociados con las respuestas a los fármacos, se pueden desarrollar biomarcadores que pueden utilizarse para predecir la respuesta de un paciente a un fármaco o régimen de tratamiento en particular. Esto también puede ayudar a optimizar la dosificación de medicamentos y reducir el riesgo de efectos secundarios adversos4.
A pesar de sus beneficios potenciales, el costo de la transcriptómica sigue siendo un obstáculo importante para su aplicación generalizada en el cribado de fármacos. El análisis transcriptómico requiere equipos especializados, conocimientos técnicos y análisis de datos, lo que puede dificultar la utilización de la transcriptómica en la detección de fármacos para equipos de investigación más pequeños u organizaciones con fondos limitados. Sin embargo, el costo de la transcriptómica ha ido disminuyendo constantemente, haciéndola más accesible para las comunidades de investigación. Además, los avances en la tecnología y los métodos de análisis de datos han hecho que la transcriptómica sea más eficiente y rentable, aumentando aún más su accesibilidad2.
En este protocolo, describimos un sistema exploratorio y de alta dimensión para el cribado de fármacos basado en transcriptoma, combinando cultivos de perturbaciones miniaturizados con análisis transcriptómicos minimasivos 5,6. Con este protocolo, es posible reducir el coste por muestra a 1/6 del coste actual de las soluciones comerciales para la secuenciación completa de ARNm. El protocolo solo requiere equipos de laboratorio estándar, con la única excepción del uso de tecnologías de secuenciación de lectura corta, que pueden subcontratarse si los instrumentos de secuenciación no están disponibles internamente. El protocolo minimasivo optimizado proporciona señales biológicas ricas en información a una profundidad de secuenciación rentable, lo que permite un cribado exhaustivo de fármacos conocidos y nuevas moléculas.
El objetivo del experimento es detectar la actividad farmacológica de las PBMC en diferentes contextos biológicos. Este protocolo se puede aplicar a cualquier cuestión biológica en la que se deban probar varios fármacos con una lectura transcriptómica, lo que proporciona una visión de todo el transcriptoma del efecto celular del tratamiento.
Protocolo
Este protocolo sigue las directrices de los comités locales de ética de la Universidad de Bonn.
1. Preparación de tampones, soluciones y equipos
- Prepare las soluciones y reúna los materiales descritos en la Tabla de materiales.
- Calentar el baño de agua a 37 °C y calentar todo el medio de cultivo (RPMI-1640 + 10% de suero fetal de ternero (FCS) + 1% de penicilina/estreptomicina).
- Para la recolección de células, use solución salina tamponada con fosfato (PBS) helada.
NOTA: Mantenga un ambiente limpio mientras trabaja con las células para mantener la esterilidad.
2. Manejo de células
NOTA: Un protocolo detallado para la criopreservación de células mononucleares de sangre periférica (PBMC) de sangre humana se puede encontrar en7.
- Descongelación y recuento de células
- Retire los crioviales del nitrógeno líquido y descongélelos en un baño de agua a 37 °C durante 2 - 3 minutos mientras invierte suavemente.
- Transfiera las células descongeladas a un tubo cónico de 50 ml.
- Enjuague el criovial con 1 ml de medio de crecimiento completo tibio y agregue esta solución gota a gota a las células en el tubo (1er paso de dilución).
- Repita la dilución 1:1 hasta alcanzar un volumen de 32 ml (5 diluciones en total con 1, 2, 4, 8 y 16 ml respectivamente de medio de crecimiento completo caliente). Agregue el medio gota a gota para reducir la perturbación celular mientras agita ligeramente el tubo cónico.
- Centrifugar la suspensión celular durante 5 min (300 x g, 20 °C) y eliminar el sobrenadante inclinando suavemente el tubo cónico con un movimiento fluido.
- Vuelva a suspender las células en 3 ml de medio de crecimiento completo tibio y proceda con el recuento celular.
- Para el recuento, mezcle 10 μL de suspensión celular con azul de tripano (dilución de 1:2 a 1:10 dependiendo de la densidad de la suspensión celular) para distinguir las células vivas de las muertas durante el conteo. Las células muertas o dañadas aparecerán azules debido a la absorción del tinte, mientras que las células vitales no se tiñen.
NOTA: El azul de tripano es ligeramente citotóxico, las células teñidas no deben almacenarse durante más de 5 minutos. - Cuente las células con un contador celular automatizado o una cámara de recuento utilizando 10 μL de solución celular teñida.
- Cuando utilice la cámara celular mejorada por Neubauer, cuente todas las células dentro de los cuatro cuadrados grandes ubicados en las esquinas y use la siguiente ecuación para calcular la concentración de la suspensión celular.
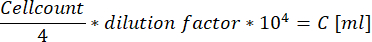
- Cuando utilice la cámara celular mejorada por Neubauer, cuente todas las células dentro de los cuatro cuadrados grandes ubicados en las esquinas y use la siguiente ecuación para calcular la concentración de la suspensión celular.
- Diluir las células hasta una concentración final de 1 x 106 células/ml con medio de crecimiento completo tibio. Centrifugar solo si el volumen requerido es inferior a 3 ml y volver a suspender el gránulo celular al volumen correcto.
- Siembra y tratamiento celular
- Siembre 100 μL de suspensión celular por pocillo en una placa de cultivo celular de 96 pocillos (1 x 105 células/pocillo).
NOTA: El número de celdas para la siembra se optimizó para cultivos PBMC. Si bien las concentraciones celulares bajas no producirán una cantidad suficiente de ARN para la secuenciación, un número excesivo de células aumentará el riesgo de lisis celular subóptima y la inhibición de la reacción de transcripción inversa. - Preparar diluciones de fármacos a una concentración dos veces superior a la utilizada para el tratamiento (2x). Elija el disolvente de acuerdo con la solubilidad del compuesto, preferiblemente medio de crecimiento completo.
NOTA: Se puede usar PBS o dimetilsulfóxido (DMSO) si no se puede alcanzar una solubilidad suficiente de otra manera. La concentración máxima de DMSO en el volumen de incubación resultante no debe superar el 0,5 % para evitar la citotoxicidad inducida por el disolvente. - Añadir 100 μL de la dilución del fármaco 2x (concentración final en pocillo 1X) e incubar a 37 °C durante el tiempo definido.
NOTA: Se deben realizar investigaciones complementarias para establecer el tiempo óptimo de incubación del fármaco y excluir posibles mecanismos de citotoxicidad.
- Siembre 100 μL de suspensión celular por pocillo en una placa de cultivo celular de 96 pocillos (1 x 105 células/pocillo).
- Recolección y lisis celular
- Centrifugar la placa de cultivo celular durante 10 min (300 x g, 20 °C). Retire suavemente el sobrenadante con una bomba de vacío manteniendo la placa en ángulo (30°-45°) mientras apunta la punta hacia la esquina inferior del pocillo para eliminar la menor cantidad posible de células.
- Lave las celdas añadiendo 200 μL de PBS helado a cada pocillo y centrifugue la placa durante 5 min (300 x g, 4 °C). Retire el sobrenadante con una bomba de vacío, nuevamente, manteniendo la placa en un ángulo agudo para eliminar la mayor cantidad posible de PBS.
- Prepare el tampón de lisis como se describe en la Tabla 1 para el número de reacciones (rxn) necesarias, agregando un 10% de excedente.
- Agregue 15 μL de tampón de lisis a cada pocillo y selle la placa con una película adhesiva de sellado para proteger las muestras contra la contaminación. Agitar la placa antes de centrifugar durante 1 min (1000 x g, 4 °C) e incubar durante 5 min en hielo.
- Recoja 6 μL de solución celular lisada en una placa de PCR y congele brevemente el lisado celular a -80 °C para garantizar una lisis celular óptima. Asegúrese de evitar múltiples ciclos de congelación de las placas.
NOTA: PUNTO DE PARADA: Si la producción de ADNc no se lleva a cabo posteriormente, el lisado celular puede almacenarse a -80 °C durante varios meses sin una disminución considerable de la calidad del ARN.
3. Preparación de la biblioteca para la secuenciación
- Reacción de transcriptasa inversa (RT)
NOTA: Cuando trabaje con ARN, coloque todas las muestras en hielo y asegúrese de usar equipo libre de nucleasas (estéril, plástico desechable) y agua.- Prepare la mezcla de reacción RT como se describe en la Tabla 2 para el número de reacciones necesarias, agregando un 10% de excedente. Agita brevemente la mezcla y gírala brevemente. Mantenga la mezcla en hielo hasta su uso.
- Descongele el lisado celular a temperatura ambiente (RT) y gírelo brevemente hacia abajo para recolectar todo el lisado celular en el fondo de la placa.
- Realice la desnaturalización del ARNm en un termociclador según la Tabla 3.
- Saque la placa del termociclador y gírela brevemente hacia abajo para recoger el condensado potencial. Agregue 6 μL de mezcla de reacción RT en cada pocillo para obtener un volumen final de 12 μL. Selle la placa para protegerla y evitar la evaporación, el vórtice y gírela hacia abajo.
- Coloque la placa en el termociclador e inicie el programa de reacción RT según la Tabla 3.
- Preamplificación
- Descongele la ADN polimerasa de alta fidelidad y el cebador de PCR in situ (ISPCR) a temperatura ambiente.
- Prepare la mezcla de preamplificación según la Tabla 4 para el número de reacciones necesarias, agregando un 10% de excedente. Agita brevemente la mezcla y gírala hacia abajo.
NOTA: La mezcla de enzimas es estable a temperatura ambiente durante horas. - Gire hacia abajo la placa de PCR, agregue 15 μL de la mezcla de preamplificación a cada pocillo y selle la placa.
- Colóquelo en el termociclador e inicie la reacción de preamplificación según la Tabla 5.
- Ajuste el número de ciclos debido al contenido de ARN. Comience con un número más bajo y aumente si el rendimiento de ADNc no es suficiente.
NOTA: En nuestra experiencia, se debe establecer un número óptimo de ciclos para cada tipo de célula, la condición experimental y el tratamiento no influirán en el número óptimo de ciclos. Por lo tanto, recomendamos establecer experimentalmente el número óptimo de ciclos al establecer este protocolo para un nuevo tipo de célula. En general, las células primarias requerirán un mayor número de ciclos en comparación con las líneas celulares. PUNTO DE PARADA: El producto de preamplificación se puede almacenar a -20 °C.
- Ajuste el número de ciclos debido al contenido de ARN. Comience con un número más bajo y aumente si el rendimiento de ADNc no es suficiente.
- Limpieza de ADNc y control de calidad (QC)
NOTA: La limpieza de la muestra se puede realizar secuencialmente para cada placa, ya que las muestras son bastante estables en esta etapa. El aumento del número de muestras dará lugar a una mayor duración del protocolo, pero no limitará el número de muestras que se pueden procesar simultáneamente.- Antes de comenzar, lleve las perlas de purificación magnética a temperatura ambiente y en vórtice a alta velocidad durante 1 minuto para resuspender completamente las perlas.
- Agregue 0,8x v/v (20 μL) de perlas de purificación magnética a cada pocillo e incube a temperatura ambiente durante 5 min.
- Coloque la placa de PCR en una rejilla magnética durante 5 minutos hasta que las perlas estén completamente separadas. Retire el sobrenadante con cuidado.
- Manteniendo la placa en la rejilla magnética, agregue suavemente 100 μL de etanol al 80% recién preparado para lavar las perlas e incubar durante 30 s. Utilice una pipeta de pequeño volumen para extraer el sobrenadante. Repita para un paso de lavado adicional. Asegúrese de eliminar la mayor cantidad de etanol posible.
- Seque al aire las perlas en la rejilla magnética a temperatura ambiente durante un máximo de 5 minutos hasta que el etanol se evapore por completo y las perlas ya no se vean brillantes.
- Retire la placa de la rejilla magnética, vuelva a suspender las perlas en 20 μL de agua sin nucleasas e incube durante 2 min.
- Vuelva a colocar la placa en la rejilla magnética hasta que las cuentas se separen.
- Recuperar el eluido en una nueva placa de PCR para el control de calidad y el etiquetado del ADNc.
- Realice un ensayo TapeStation o FragmentAnalyser para evaluar la distribución del tamaño y la concentración de la biblioteca de ADNc (recomendado: ensayo TapeStation D5000). Para obtener más información, consulte las instrucciones del fabricante. Se espera un rendimiento típico de 20 ng.
- Etiquetado con kit
NOTA: Se pueden utilizar otros protocolos de etiquetado si ya están establecidos.- Diluir el ADNc hasta una concentración final de 150 - 300 pg/μL utilizando agua libre de nucleasas.
- Programe previamente el termociclador según la Tabla 6 para garantizar un inicio inmediato de la reacción de etiquetado después de agregar el ADNc a la mezcla de enzimas.
- Prepare la mezcla de etiquetado según la Tabla 7 para el número de reacciones necesarias, agregando un 10% de excedente.
- Dispense 3 μL de la mezcla de etiquetado por reacción en una nueva placa de PCR y agregue 1 μL de ADNc a cada reacción.
- Inicie la reacción de etiquetado inmediatamente después de agregar el ADNc.
- Inactive la reacción añadiendo 1 μL de tampón de marcado neutralizante (NT; alternativamente, se puede utilizar dodecil sulfato de sodio (SDS) al 0,2%).
- PCR de enriquecimiento
- Prepare la mezcla de PCR de enriquecimiento Tabla 7 para el número de reacciones, añadiendo un 10% de excedente. (Recomendado: Nextera UDI configurado para evitar el salto de índice en celdas de flujo con patrón (por ejemplo, Illumina NovaSeq 6000)).
- Añadir 9 μL de la mezcla de PCR de enriquecimiento a cada reacción para obtener un volumen total de 14 μL.
- Ejecute el programa de PCR de enriquecimiento según la Tabla 8.
NOTA: Para la PCR de enriquecimiento, 16 ciclos son estándar. Si la calidad del ADNc es mala, se pueden agregar ciclos adicionales.
- Limpieza y control de calidad
- Antes de comenzar, lleve las perlas de purificación magnética a temperatura ambiente y haga vórtice a alta velocidad durante 1 minuto para resuspender completamente las perlas.
- Agregue 1,0x v/v (14 μL) perlas de purificación magnética e incube a temperatura ambiente durante 5 min.
- Coloque el plato en una rejilla magnética y espere 5 minutos hasta que las cuentas estén completamente separadas. Retire el sobrenadante con cuidado.
- Manteniendo la placa en la rejilla magnética, agregue suavemente 100 μL de etanol al 80% recién preparado para lavar las perlas e incubar durante 30 s. Utilice una pipeta de pequeño volumen para extraer el sobrenadante. Repita para un paso de lavado adicional. Asegúrese de eliminar la mayor cantidad de etanol posible.
- Seca las cuentas al aire a temperatura ambiente durante 3 minutos o hasta que ya no se vean brillantes.
- Retire la placa de la rejilla magnética, vuelva a suspender las perlas en 20 μL de agua sin nucleasas e incube durante 2 min.
- Vuelva a colocar la placa en la rejilla magnética hasta que las perlas se separen y recupere el eluido en una nueva placa de PCR para el control de calidad de la biblioteca.
- Realice un ensayo TapeStation o Fragment Analyzer para evaluar la distribución del tamaño y la concentración de la biblioteca de ADNc (recomendado: ensayo de alta sensibilidad TapeStation D1000). Para obtener más información, consulte las instrucciones del fabricante. En promedio, se espera un rendimiento de 10 ng.
NOTA: PUNTO DE PARADA: El producto PCR se puede almacenar a - 20 °C.
4. Secuenciación y preprocesamiento de datos
- Secuenciación
NOTA: La siguiente directriz será aplicable a todos los instrumentos de Illumina para la secuenciación de lectura corta. Si la instrumentación no está disponible, la secuenciación puede ser realizada por una instalación de secuenciación externa. También podrían utilizarse otros métodos de secuenciación. Para simplificar, elegimos informar solo sobre la tecnología de secuenciación más utilizada.
NOTA: Los siguientes pasos relacionados con el uso del software describen el procedimiento en un secuenciador Illumina NovaSeq6000.- Agrupe bibliotecas indexadas de forma única en una proporción equimolar de acuerdo con los resultados obtenidos en el paso 3.6.8.
- Mida la concentración del grupo final con un ensayo de alta sensibilidad para calcular la molaridad de la muestra de la siguiente manera:
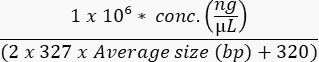
- Cargue la celda de flujo de acuerdo con las especificaciones del instrumento y la optimización experimental. En el Cuadro 9 se muestran ejemplos de concentraciones de carga de instrumentos comunes.
- Al tocar la pantalla, seleccione Secuencia para iniciar la configuración de la ejecución.
- Siga las instrucciones de la pantalla y cargue la celda de flujo, el cartucho de secuencia por síntesis, el cartucho de agrupación, el cartucho tampón y asegúrese de que los contenedores de residuos estén vacíos.
- Una vez que el instrumento haya reconocido todos los reactivos, haga clic en Ejecutar configuración. Defina aquí el nombre de ejecución y la carpeta de salida para almacenar los datos.
- Defina los detalles de secuenciación como paired-end con ambas lecturas de 51 pb. También se secuencian dos lecturas de índice con 8 pb cada una.
- Presione Revisar y, después de verificar si todos los detalles de la secuencia son correctos, presione Iniciar ejecución.
NOTA: La profundidad de secuenciación recomendada es de 5 x 106 lecturas/muestra, con un mínimo de 1 x 106 lecturas/muestra.
- Preprocesamiento de datos
- Transforme los datos de secuenciación sin procesar al formato FASTQ y demultiplexe de acuerdo con los índices de muestra con la herramienta Bcl2Fastq2. Realice la demultiplexación con la configuración predeterminada. Para obtener instrucciones detalladas sobre Bcl2Fastq2, consulte el manual de referencia.
NOTA: La conversión y la demultiplexación de FASTQ generalmente las realiza la instalación de secuenciación si la secuenciación no se realiza internamente. Las instalaciones de secuenciación suelen proporcionar FASTQ demultiplexado para su posterior procesamiento. - Hay varias opciones disponibles para la alineación de datos y la cuantificación de la abundancia de lecturas de secuenciación (recomendado: canalización de secuenciación de ARN de núcleo nf (https://nf-co.re/rnaseq)). La canalización proporciona varias opciones, use la configuración predeterminada con STAR8 alineado y Salmon9 para cuantificar la abundancia de la transcripción.
- NOTA: Para un análisis bioinformático adicional, hay varios métodos disponibles. No está en el ámbito de este protocolo abarcar todos (recomendado: canalización DEseq210). Un script estándar basado en el flujo de trabajo DEseq2 desarrollado por nosotros se puede encontrar en GitHub (https://github.com/jsschrepping/RNA-DESeq2).
- Transforme los datos de secuenciación sin procesar al formato FASTQ y demultiplexe de acuerdo con los índices de muestra con la herramienta Bcl2Fastq2. Realice la demultiplexación con la configuración predeterminada. Para obtener instrucciones detalladas sobre Bcl2Fastq2, consulte el manual de referencia.
Resultados
Siguiendo el protocolo informado, las PBMC humanas se sembraron, se trataron con diferentes fármacos inmunomoduladores y, después de diferentes tiempos de incubación, se cosecharon para su análisis transcriptómico masivo utilizando el protocolo de secuenciación (Figura 1).
Las concentraciones ideales del fármaco y los tiempos de incubación de los compuestos problema deben identificarse antes de este protocolo con la ayuda de estrategias experimentales comp...
Discusión
El descubrimiento y el desarrollo de fármacos pueden beneficiarse enormemente de la visión holística de los procesos celulares que puede proporcionar la transcriptómica masiva. Sin embargo, este enfoque a menudo se ve limitado por el alto costo del experimento con el protocolo estándar de secuenciación de ARN a granel, lo que prohíbe su aplicación en entornos académicos, así como su potencial de escalabilidad industrial.
Los pasos más críticos del protocolo son la descongelación c...
Divulgaciones
Los autores declaran no tener intereses contrapuestos.
Agradecimientos
J.L.S. cuenta con el apoyo de la Fundación Alemana de Investigación (DFG) en el marco de la Estrategia de Excelencia de Alemania (EXC2151-390873048), así como en el marco de la SCHU 950/8-1; GRK 2168, TP11; CRC SFB 1454 Metaflammation, IRTG GRK 2168, WGGC INST 216/981-1, CCU INST 217/988-1, el proyecto de excelencia financiado por BMBF Diet-Body-Brain (DietBB); y el proyecto europeo SYSCID con el número de subvención 733100. M.B. cuenta con el apoyo de DFG (IRTG2168-272482170, SFB1454-432325352). L.B. cuenta con el apoyo de DFG (ImmuDiet BO 6228/2-1 - Proyecto número 513977171) y de la Estrategia de Excelencia de Alemania (EXC2151-390873048). Imágenes creadas con BioRender.com.
Materiales
| Name | Company | Catalog Number | Comments |
| 50 mL conical tube | fisher scientific | 10203001 | |
| Adhesive PCR Plate Seals | Thermo Fisher Scientific | AB0558 | |
| Amplicon Tagment Mix (ATM) | Illumina | FC-131-1096 | Nextera XT DNA Library Prep Kit (96 samples) |
| AMPure XP beads | Beckman Coulter | A 63881 | |
| Betaine | Sigma-Aldrich | 61962 | |
| Cell culture grade 96-well plates | Thermo Fisher Scientific | 260860 | |
| Cell culture vacuum pump (VACUSAFE) | Integra Bioscience | 158300 | |
| Deoxynucleotide triphosphates (dNTPs) mix 10 mM each | Fermentas | R0192 | |
| DMSO | Sigma-Aldrich | 276855 | |
| DTT (100 mM) | Invitrogen | 18064-014 | |
| EDTA | Sigma-Aldrich | 798681 | for adherent cells |
| Ethanol | Sigma-Aldrich | 51976 | |
| Fetal Bovine Serum | Thermo Fisher Scientific | 26140079 | |
| Filter tips (10 µL) | Gilson | F171203 | |
| Filter tips (100 µL) | Gilson | F171403 | |
| Filter tips (20 µL) | Gilson | F171303 | |
| Filter tips (200 µL) | Gilson | F171503 | |
| Guanidine Hydrochloride | Sigma-Aldrich | G3272 | |
| ISPCR primer (10 µM) | Biomers.net GmbH | SP10006 | 5′-AAGCAGTGGTATCAACGCAGAG T-3′ |
| KAPA HiFi HotStart ReadyMix (2X) | KAPA Biosystems | KK2601 | |
| Magnesium chloride (MgCl2) | Sigma-Aldrich | M8266 | |
| Magnetic stand 96 | Ambion | AM10027 | |
| Neutralize Tagment (NT) Buffer | Illumina | FC-131-1096 | Nextera XT DNA Library Prep Kit (96 samples), alternatively 0.2 % SDS |
| Nextera-compatible indexing primer | Illumina | ||
| Nuclease-free water | Invitrogen | 10977049 | |
| PBS | Thermo Fisher Scientific | AM9624 | |
| PCR 96-well plates | Thermo Fisher Scientific | AB0600 | |
| PCR plate sealer | Thermo Fisher Scientific | HSF0031 | |
| Penicillin / Streptomycin | Thermo Fisher Scientific | 15070063 | |
| Qubit 4 fluorometer | Invitrogen | 15723679 | |
| Recombinant RNase inhibitor (40 U/ul) | TAKARA | 2313A | |
| RPMI-1640 cell culture medium | Gibco | 61870036 | If not working with PBMCs, adjust to cell type |
| SMART dT30VN primer | Sigma-Aldrich | 5' Bio-AAGCAGTGGTATCAACGCAGAG TACT30VN-3 | |
| Standard lab equipment | various | various | e.g. centrifuge, ice machine, ice bucket, distilled water, water bath |
| SuperScript II Reverse Transcriptase (SSRT II) | Thermo Fisher Scientific | 18064-014 | |
| SuperScript II Reverse Transcriptase (SSRT II) buffer (5x) | Thermo Fisher Scientific | 18064-014 | |
| Tagment DNA Buffer (TD) | Illumina | FC-131-1096 | Nextera XT DNA Library Prep Kit (96 samples) |
| TapeStation system 4200 | Agilent | G2991BA | |
| Thermocycler (S1000) | Bio-Rad | 1852148 | |
| TSO-LNA (100 uM) | Eurogentec | 5' Biotin AAGCAGTGGTATCAACGCAGAG TACAT(G)(G){G | |
| Vortex-Genie 2 Mixer | Sigma-Aldrich | Z258415 |
Referencias
- Hughes, J. P., Rees, S., Kalindjian, S. B., Philpott, K. L. Principles of early drug discovery. Br J Pharmacol. 162 (6), 1239-1249 (2011).
- Yang, X., et al. High-throughput transcriptome profiling in drug and biomarker discovery. Front Genet. 11, 19 (2020).
- Bonaguro, L., et al. A guide to systems-level immunomics. Nat Immunol. 23 (10), 1412-1423 (2022).
- Carraro, C., et al. Decoding mechanism of action and sensitivity to drug candidates from integrated transcriptome and chromatin state. ELife. 11, 78012 (2022).
- Picelli, S., et al. Smart-seq2 for sensitive full-length transcriptome profiling in single cells. Nat Methods. 10 (11), 1096-1098 (2013).
- Picelli, S., et al. Full-length RNA-seq from single cells using Smart-seq2. Nat Protoc. 9 (1), 171-181 (2014).
- De Domenico, E., et al. Optimized workflow for single-cell transcriptomics on infectious diseases including COVID-19. STAR Protoc. 1 (3), 100233 (2020).
- Dobin, A., et al. ultrafast universal RNA-seq aligner. Bioinformatics. 29 (1), 15-21 (2013).
- Patro, R., et al. Salmon provides fast and bias-aware quantification of transcript expression. Nat Methods. 14 (4), 417-419 (2017).
- Love, M. I., Huber, W., Anders, S. Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2. Genome Biol. 15 (12), 550 (2014).
- Frankish, A., et al. GENCODE reference annotation for the human and mouse genomes. Nucleic Acids Res. 47, D766-D773 (2019).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados