É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Triagem Transcriptomic-Based Drug Screening Custo-Eficiente
Neste Artigo
Resumo
Este protocolo descreve um fluxo de trabalho de culturas de células ex vivo ou in vitro para pré-processamento de dados transcriptômicos para triagem de drogas baseada em transcriptoma custo-efetivo.
Resumo
A transcriptômica permite obter informações abrangentes sobre programas celulares e suas respostas a perturbações. Apesar da diminuição significativa dos custos de produção e sequenciamento das bibliotecas na última década, a aplicação dessas tecnologias na escala necessária para a triagem de drogas continua sendo proibitivamente cara, obstruindo o imenso potencial desses métodos. Nosso estudo apresenta um sistema custo-efetivo para triagem de drogas baseada em transcriptomas, combinando culturas de perturbação miniaturizadas com transcriptômica mini-massil. O protocolo otimizado de mini-bulk fornece sinais biológicos informativos em profundidade de sequenciamento econômica, permitindo uma extensa triagem de drogas conhecidas e novas moléculas. Dependendo do tratamento escolhido e do tempo de incubação, este protocolo resultará em bibliotecas de sequenciamento dentro de aproximadamente 2 dias. Devido a vários pontos de parada dentro deste protocolo, a preparação da biblioteca, bem como o sequenciamento, pode ser realizado de forma independente do tempo. É possível processar simultaneamente um número elevado de amostras; A medição de até 384 amostras foi testada sem perda da qualidade dos dados. Também não há limitações conhecidas para o número de condições e/ou drogas, apesar de considerar a variabilidade nos tempos ideais de incubação dos fármacos.
Introdução
O desenvolvimento de novos fármacos é um processo complexo e demorado que envolve a identificação de potenciais fármacos e seus alvos, a otimização e síntese de candidatos a fármacos e o teste de sua eficácia e segurança em ensaios pré-clínicos e clínicos1. Os métodos tradicionais de triagem de drogas, ou seja, a avaliação sistemática de bibliotecas de compostos candidatos para fins terapêuticos, envolvem o uso de modelos animais ou ensaios baseados em células para testar os efeitos em alvos ou vias específicas. Embora esses métodos tenham sido bem-sucedidos na identificação de candidatos a drogas, eles muitas vezes não forneceram informações suficientes sobre os complexos mecanismos moleculares subjacentes à eficácia da droga e também toxicidade e mecanismos de efeitos colaterais potenciais.
A avaliação de estados transcricionais genômicos apresenta uma abordagem poderosa para superar as limitações atuais na triagem de drogas, pois permite avaliações abrangentes da expressão gênica em resposta a tratamentos medicamentosos2. Ao medir transcritos de RNA de uma forma genômica ampla expressa em um determinado momento, a transcriptômica visa fornecer uma visão holística das mudanças transcricionais que ocorrem em resposta a drogas, incluindo mudanças nos padrões de expressão gênica, splicing alternativo e expressão de RNA não codificante3. Essas informações podem ser usadas para determinar alvos de drogas, prever eficácia e toxicidade de drogas e otimizar esquemas de dosagem e tratamento de medicamentos.
Um dos principais benefícios da combinação de transcriptômica com triagem imparcial de drogas é o potencial para identificar novos alvos de drogas que não foram considerados anteriormente. As abordagens convencionais de triagem de drogas geralmente se concentram em moléculas ou vias alvo estabelecidas, dificultando a identificação de novos alvos e potencialmente resultando em drogas com efeitos colaterais imprevistos e eficácia restrita. A transcriptômica pode superar essas limitações, fornecendo informações sobre as mudanças moleculares que ocorrem em resposta ao tratamento medicamentoso, descobrindo potenciais alvos ou vias que podem não ter sido consideradas anteriormente2.
Além da identificação de novos alvos de fármacos, a transcriptômica também pode ser utilizada para predizer eficácia e toxicidade de fármacos. Ao analisar os padrões de expressão gênica associados às respostas a drogas, biomarcadores podem ser desenvolvidos para prever a resposta de um paciente a um determinado medicamento ou regime de tratamento. Isso também pode ajudar a otimizar a dosagem de medicamentos e reduzir o risco de efeitos adversos4.
Apesar de seus potenciais benefícios, o custo da transcriptômica permanece uma barreira significativa para sua ampla aplicação na triagem de drogas. A análise transcriptômica requer equipamentos especializados, conhecimento técnico e análise de dados, o que pode tornar desafiador para equipes de pesquisa menores ou organizações com financiamento limitado utilizar transcriptômica na triagem de drogas. No entanto, o custo da transcriptômica tem diminuído constantemente, tornando-a mais acessível às comunidades de pesquisa. Além disso, os avanços na tecnologia e nos métodos de análise de dados tornaram a transcriptômica mais eficiente e econômica, aumentando ainda mais sua acessibilidade2.
Neste protocolo, descrevemos um sistema exploratório e de alta dimensão para triagem de drogas baseado em transcriptomas, combinando culturas de perturbação miniaturizadas com análise transcriptômica mini-bulk 5,6. Com este protocolo, é possível reduzir o custo por amostra para 1/6 do custo atual de soluções comerciais para sequenciamento total de mRNA. O protocolo requer apenas equipamentos laboratoriais padrão, sendo a única exceção o uso de tecnologias de sequenciamento de leitura curta, que podem ser terceirizadas se os instrumentos de sequenciamento não estiverem disponíveis internamente. O protocolo otimizado de mini-bulk fornece sinais biológicos ricos em informações em profundidade de sequenciamento econômica, permitindo uma extensa triagem de drogas conhecidas e novas moléculas.
O objetivo do experimento é rastrear a atividade de drogas em PBMCs em diferentes contextos biológicos. Este protocolo pode ser aplicado a qualquer questão biológica onde várias drogas devem ser testadas com uma leitura transcriptômica, dando uma visão ampla do transcriptoma do efeito celular do tratamento.
Protocolo
Este protocolo segue as diretrizes dos comitês de ética locais da Universidade de Bonn.
1. Preparação de tampões, soluções e equipamentos
- Preparar as soluções e reunir os materiais descritos na Tabela de Materiais.
- Aquecer o banho-maria a 37 °C e aquecer o meio de crescimento completo (RPMI-1640 + 10% de soro fetal de bezerro (SFB) + 1% de penicilina/estreptomicina).
- Para a colheita celular, use solução salina tamponada com fosfato (PBS) gelada.
NOTA: Mantenha um ambiente limpo enquanto trabalha com células para manter a esterilidade.
2. Manuseio celular
NOTA: Um protocolo detalhado para a criopreservação de células mononucleares do sangue periférico (CMSP) do sangue humano pode ser encontrado em7.
- Descongelamento e contagem de células
- Retire os criósio-frascos do azoto líquido e descongele-os em banho-maria a 37 °C durante 2 - 3 minutos, invertendo suavemente.
- Transfira as células descongeladas para um tubo cônico de 50 mL.
- Enxaguar o criovial com 1 mL de meio de crescimento completo morno e adicionar esta solução gota a gota às células do tubo (1ª etapa de diluição).
- Repetir a diluição 1:1 até atingir um volume de 32 mL (5 diluições no total com, respectivamente, 1, 2, 4, 8 e 16 mL de meio de crescimento completo aquecido). Adicione o meio gota a gota para reduzir a perturbação celular enquanto agita ligeiramente o tubo cônico.
- Centrifugar a suspensão celular por 5 min (300 x g, 20 °C) e remover o sobrenadante inclinando suavemente o tubo cônico em um movimento fluente.
- Ressuspender as células em 3 mL de meio de crescimento completo aquecido e prosseguir com a contagem celular.
- Para a contagem, misture 10 μL de suspensão celular com Azul de Tripano (diluição de 1:2 a 1:10, dependendo da densidade da suspensão celular) para distinguir células vivas de mortas durante a contagem. As células mortas ou danificadas aparecerão azuis devido à absorção do corante, enquanto as células vitais não são coradas.
NOTA: Azul de Tripano é ligeiramente citotóxico, as células coradas não devem ser armazenadas por mais de 5 min. - Conte as células com um contador de células automatizado ou câmara de contagem usando 10 μL de solução de células coradas.
- Ao usar a câmara celular melhorada de Neubauer, conte todas as células dentro dos quatro grandes quadrados localizados nos cantos e use a seguinte equação para calcular a concentração da suspensão celular.
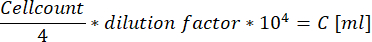
- Ao usar a câmara celular melhorada de Neubauer, conte todas as células dentro dos quatro grandes quadrados localizados nos cantos e use a seguinte equação para calcular a concentração da suspensão celular.
- Diluir as células para uma concentração final de 1 x 106 células/mL com meio de crescimento completo aquecido. Centrifugar somente se o volume necessário for inferior a 3 mL e ressuspender a pastilha de células para o volume certo.
- Semeadura e tratamento celular
- Semente de 100 μL de suspensão celular por poço em placa de cultura celular de 96 poços (1 x 105 células / poço).
NOTA: O número de célula para semeadura foi otimizado para culturas PBMC. Enquanto baixas concentrações celulares não produzirão uma quantidade suficiente de RNA para sequenciamento, um número excessivo de células aumentará o risco de lise celular subótima e inibição da reação de transcrição reversa. - Preparar diluições do fármaco numa concentração duas vezes superior à utilizada para o tratamento (2x). Escolha o solvente de acordo com a solubilidade do composto, preferencialmente meio de crescimento completo.
NOTA: PBS ou dimetilsulfóxido (DMSO) podem ser usados se solubilidade suficiente não puder ser alcançada de outra forma. A concentração máxima de DMSO no volume de incubação resultante não deve exceder 0,5 % para evitar a citotoxicidade induzida pelo solvente. - Adicionar 100 μL da diluição 2x do fármaco (concentração final no poço 1X) e incubar a 37 °C pelo tempo definido.
NOTA: Investigações complementares devem ser realizadas para estabelecer o tempo ideal de incubação do fármaco e excluir potenciais mecanismos de citotoxicidade.
- Semente de 100 μL de suspensão celular por poço em placa de cultura celular de 96 poços (1 x 105 células / poço).
- Colheita e lise de células
- Centrifugar a placa de cultura celular durante 10 minutos (300 x g, 20 °C). Remova suavemente o sobrenadante com uma bomba de vácuo mantendo a placa em um ângulo (30°-45°) enquanto aponta a ponta para o canto inferior do poço para remover o mínimo possível de células.
- Lave as células adicionando 200 μL de PBS gelado a cada poço e centrifugar a placa por 5 min (300 x g, 4 °C). Remova o sobrenadante com uma bomba de vácuo, novamente, mantendo a placa em um ângulo acentuado para remover o máximo de PBS possível.
- Prepare o tampão de lise conforme descrito na Tabela 1 para o número de reações (rxn) necessárias, adicionando 10% de sobrecarga.
- Adicionar 15 μL de tampão de lise a cada poço e selar a placa com película de vedação adesiva para proteger as amostras contra contaminação. Vórtice a placa antes de centrifugar por 1 min (1000 x g, 4 °C) e incubar por 5 min no gelo.
- Recolher 6 μL de solução celular lisada numa placa de PCR e congelar brevemente o lisado celular a -80 °C para garantir a lise celular ideal. Certifique-se de evitar vários ciclos de congelamento das placas.
NOTA: PONTO DE PARADA: Se a produção de cDNA não for realizada posteriormente, o lisado celular pode ser armazenado a -80 °C por até vários meses sem diminuição considerável da qualidade do RNA.
3. Preparação da biblioteca para sequenciamento
- Reação da transcriptase reversa (RT)
NOTA: Ao trabalhar com RNA, coloque todas as amostras no gelo e certifique-se de usar equipamentos livres de nuclease (plásticos estéreis e descartáveis) e água.- Preparar a mistura de reação RT conforme descrito na Tabela 2 para o número de reações necessárias, adicionando 10% de excesso. Brevemente vórtice a mistura e logo gire-a para baixo. Mantenha a mistura no gelo até o uso.
- Descongelar o lisado celular à temperatura ambiente (TR) e girá-lo brevemente para baixo para coletar todo o lisado celular no fundo da placa.
- Realizar a desnaturação do RNAm em um termociclador conforme Tabela 3.
- Retire a placa do termociclador e gire-a rapidamente para baixo para coletar o condensado potencial. Adicione 6 μL de mistura de reação RT em cada poço para um volume final de 12 μL. Sele a placa para proteger a placa e evitar evaporação, vórtice e girá-la para baixo.
- Coloque a placa no termociclador e inicie o programa de reação de RT conforme Tabela 3.
- Pré-amplificação
- Descongelar o primer DNA polimerase de alta fidelidade e PCR in situ (ISPCR) à temperatura ambiente.
- Preparar a mistura de pré-amplificação conforme Tabela 4 para o número de reações necessárias, adicionando 10% de sobrecarga. Brevemente vórtice a mistura e gire-a para baixo.
NOTA: A mistura enzimática é estável à temperatura ambiente durante horas. - Gire a placa de PCR, adicione 15 μL da mistura de pré-amplificação a cada poço e sele a placa.
- Coloque no termociclador e inicie a reação de pré-amplificação conforme Tabela 5.
- Ajuste o número de ciclos devido ao conteúdo de RNA. Comece com um número menor e aumente se o rendimento de cDNA não for suficiente.
NOTA: Em nossa experiência, o número ideal de ciclos deve ser estabelecido para cada tipo celular, a condição experimental e o tratamento não influenciarão o número ótimo de ciclos. Recomenda-se, portanto, estabelecer experimentalmente o número ótimo de ciclos ao estabelecer este protocolo para um novo tipo celular. Em geral, as células primárias exigirão um número maior de ciclos em comparação com as linhagens celulares. PONTO DE PARADA: O produto de pré-amplificação pode ser armazenado a - 20 °C.
- Ajuste o número de ciclos devido ao conteúdo de RNA. Comece com um número menor e aumente se o rendimento de cDNA não for suficiente.
- limpeza de cDNA e controle de qualidade (QC)
NOTA: A limpeza da amostra pode ser realizada sequencialmente para cada placa, pois as amostras são bastante estáveis nesta fase. O aumento do número de amostras levará a uma maior duração do protocolo, mas não limitará o número de amostras que podem ser processadas simultaneamente.- Antes de começar, leve as contas de purificação magnética à temperatura ambiente e vórtice em alta velocidade por 1 min para ressuspender totalmente as contas.
- Adicionar 0,8x v/v (20 μL) esferas de purificação magnética a cada poço e incubar à temperatura ambiente durante 5 minutos.
- Coloque a placa de PCR em um rack magnético por 5 min até que as contas estejam totalmente separadas. Retire o sobrenadante com cuidado.
- Mantendo a placa no rack magnético, adicione suavemente 100 μL de etanol 80% recém-preparado para lavar as contas e incubar por 30 s. Use uma pipeta de pequeno volume para remover o sobrenadante. Repita para uma etapa adicional de lavagem. Certifique-se de remover o máximo de etanol possível.
- Seque ao ar as contas no rack magnético à temperatura ambiente por até 5 minutos até que o etanol seja totalmente evaporado e as contas não pareçam mais brilhantes.
- Retire a placa do rack magnético, ressuspenda as esferas em 20 μL de água livre de nuclease e incube por 2 min.
- Coloque a placa novamente no rack magnético até que as contas estejam separadas.
- Recuperar o eluato em uma nova placa de PCR para cDNA QC e tagmentação.
- Execute um ensaio TapeStation ou FragmentAnalyser para avaliar a distribuição de tamanho e concentração da biblioteca de cDNA (Recomendado: ensaio TapeStation D5000). Para obter detalhes, consulte as instruções do fabricante. Um rendimento típico de 20 ng é esperado.
- Etiquetagem com kit
Observação : outros protocolos de marcação podem ser usados se já estabelecido.- Diluir o cDNA até uma concentração final de 150 - 300 pg/μL usando água livre de nucleases.
- Pré-programe o termociclador conforme Tabela 6 para garantir um início imediato da reação de tagmentação após a adição do cDNA à mistura enzimática.
- Prepare a mistura de marcação conforme Tabela 7 para o número de reações necessárias, adicionando 10% de excesso.
- Distribuir 3 μL da mistura de marcação por reação em uma nova placa de PCR e adicionar 1 μL de cDNA a cada reação.
- Inicie a reação de marcação imediatamente após a adição do cDNA.
- Inative a reação adicionando 1 μL de tampão neutralize tagment (NT; alternativamente, pode-se usar dodecil sulfato de sódio (SDS) a 0,2%).
- PCR de enriquecimento
- Preparar a mistura de PCR de enriquecimento Tabela 7 para o número de reações, adicionando 10% de sobreidade. (Recomendado: Nextera UDI definido para evitar o salto de índice em células de fluxo padronizadas (por exemplo, Illumina NovaSeq 6000)).
- Adicionar 9 μL da mistura de PCR de enriquecimento a cada reacção para um volume total de 14 μL.
- Execute o programa PCR de enriquecimento conforme Tabela 8.
NOTA: Para o PCR de enriquecimento, 16 ciclos são padrão. Se a qualidade do cDNA for ruim, ciclos adicionais podem ser adicionados.
- Limpeza e CQ
- Antes de começar, leve as contas de purificação magnética à temperatura ambiente e ao vórtice em alta velocidade por 1 min para ressuspender totalmente as contas.
- Adicionar 1,0x v/v (14 μL) esferas de purificação magnética e incubar à temperatura ambiente durante 5 minutos.
- Coloque a placa em um rack magnético e aguarde 5 min até que as contas estejam totalmente separadas. Retire o sobrenadante com cuidado.
- Mantendo a placa no rack magnético, adicione suavemente 100 μL de etanol 80% recém-preparado para lavar as contas e incubar por 30 s. Use uma pipeta de pequeno volume para remover o sobrenadante. Repita para uma etapa adicional de lavagem. Certifique-se de remover o máximo de etanol possível.
- Seque ao ar livre as contas à temperatura ambiente por 3 min ou até que não pareçam mais brilhantes.
- Retire a placa do rack magnético, ressuspenda as esferas em 20 μL de água livre de nuclease e incube por 2 min.
- Coloque a placa novamente no rack magnético até que as contas se separem e recupere o eluato em uma nova placa de PCR para biblioteca QC.
- Execute um ensaio TapeStation ou Fragment Analyzer para avaliar a distribuição de tamanho e a concentração da biblioteca de cDNA (Recomendado: ensaio de alta sensibilidade TapeStation D1000). Para obter detalhes, consulte as instruções do fabricante. Em média, um rendimento de 10 ng é esperado.
NOTA: PONTO DE PARADA: O produto PCR pode ser armazenado a - 20 °C.
4. Sequenciamento e pré-processamento de dados
- Seqüenciamento
NOTA: A diretriz a seguir será aplicável a todos os instrumentos Illumina para sequenciamento de leitura curta. Se a instrumentação não estiver disponível, o sequenciamento pode ser realizado por uma instalação de sequenciamento externa. Outras abordagens de sequenciamento também poderiam ser usadas. Para simplificar, optamos por relatar apenas a tecnologia de sequenciamento mais utilizada.
Observação : as etapas a seguir relacionadas ao uso de software descrevem o procedimento em um sequenciador Illumina NovaSeq6000.- Agrupe bibliotecas indexadas exclusivamente em uma razão equimolar de acordo com os resultados obtidos na etapa 3.6.8.
- Medir a concentração do pool final com um ensaio de alta sensibilidade para calcular a molaridade da amostra da seguinte forma:
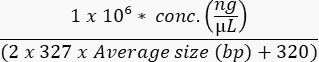
- Carregar a célula de fluxo de acordo com a especificação do instrumento e para otimização experimental. Exemplos de concentrações de carga de instrumentos comuns são mostrados na Tabela 9.
- Ao tocar na tela, selecione Sequência para iniciar a configuração de execução.
- Siga as instruções na tela e na célula de fluxo de carga, cartucho de síntese sequencial, cartucho de clustering, cartucho tampão e verifique se os recipientes de resíduos estão vazios.
- Depois que todos os reagentes tiverem sido reconhecidos pelo instrumento, clique em Executar instalação. Defina aqui o nome da execução e a pasta de saída para armazenar os dados.
- Defina os detalhes de sequenciamento como emparelhados com ambas as leituras de 51 pb. Duas leituras de índice também são sequenciadas com 8 pb cada.
- Pressione Review e, depois de verificar se todos os detalhes do sequenciamento estão corretos, pressione Start Run.
NOTA: A profundidade de sequenciamento recomendada é de 5 x 106 leituras/amostra, com um mínimo de 1 x 106 leituras/amostra.
- Pré-processamento de dados
- Transforme dados brutos de sequenciamento para o formato FASTQ e demultiplex de acordo com os índices de amostra com a ferramenta Bcl2Fastq2. Execute a desmultiplexação com as configurações padrão. Para obter instruções detalhadas sobre Bcl2Fastq2, consulte o manual de referência.
NOTA: A conversão e a desmultiplexação do FASTQ geralmente são realizadas pelo recurso de sequenciamento se o sequenciamento não for realizado internamente. As instalações de sequenciamento geralmente fornecem FASTQ desmultiplexado para processamento posterior. - Várias opções estão disponíveis para alinhamento de dados e quantificação de abundância de leituras de sequenciamento (Recomendado: nf-core RNA-seq pipeline (https://nf-co.re/rnaseq)). O pipeline fornece várias opções, use a configuração padrão com STAR8 como alinhado e Salmon9 para quantificar a abundância de transcrição.
- NOTA: Para análises adicionais de bioinformática, vários métodos estão disponíveis. Não está no escopo deste protocolo cobrir todos (Recomendado: pipeline DEseq210). Um script padrão baseado no fluxo de trabalho DEseq2 desenvolvido por nós pode ser encontrado no GitHub (https://github.com/jsschrepping/RNA-DESeq2).
- Transforme dados brutos de sequenciamento para o formato FASTQ e demultiplex de acordo com os índices de amostra com a ferramenta Bcl2Fastq2. Execute a desmultiplexação com as configurações padrão. Para obter instruções detalhadas sobre Bcl2Fastq2, consulte o manual de referência.
Resultados
Seguindo o protocolo relatado, as CMSP humanas foram semeadas, tratadas com diferentes drogas imunomoduladoras e, após diferentes tempos de incubação, colhidas para análise transcriptômica em massa usando o protocolo de sequenciamento (Figura 1).
As concentrações ideais dos fármacos e os tempos de incubação dos compostos teste devem ser identificados a montante deste protocolo com o auxílio de estratégias experimentais complementares e com base na ques...
Discussão
A descoberta de drogas e o desenvolvimento de drogas podem se beneficiar muito da visão holística dos processos celulares que a transcriptômica em massa pode fornecer. No entanto, essa abordagem é frequentemente limitada pelo alto custo do experimento com o protocolo RNA-seq a granel padrão, proibindo sua aplicação em ambientes acadêmicos, bem como seu potencial para escalabilidade industrial.
As etapas mais críticas do protocolo são o descongelamento celular e as etapas iniciais da ...
Divulgações
Os autores declaram não haver interesses concorrentes.
Agradecimentos
A J.L.S. é apoiada pela Fundação Alemã de Pesquisa (DFG) no âmbito da Estratégia de Excelência da Alemanha (EXC2151-390873048), bem como sob o SCHU 950/8-1; GRK 2168, TP11; CRC SFB 1454 Metaflammation, IRTG GRK 2168, WGGC INST 216/981-1, CCU INST 217/988-1, o projeto de excelência financiado pelo BMBF Diet-Body-Brain (DietBB); e o projeto da UE SYSCID sob o número de subvenção 733100. M.B. é suportado pela DFG (IRTG2168-272482170, SFB1454-432325352). A L.B. é apoiada pela DFG (ImmuDiet BO 6228/2-1 - Projeto número 513977171) e pela Estratégia de Excelência da Alemanha (EXC2151-390873048). Imagens criadas com BioRender.com.
Materiais
| Name | Company | Catalog Number | Comments |
| 50 mL conical tube | fisher scientific | 10203001 | |
| Adhesive PCR Plate Seals | Thermo Fisher Scientific | AB0558 | |
| Amplicon Tagment Mix (ATM) | Illumina | FC-131-1096 | Nextera XT DNA Library Prep Kit (96 samples) |
| AMPure XP beads | Beckman Coulter | A 63881 | |
| Betaine | Sigma-Aldrich | 61962 | |
| Cell culture grade 96-well plates | Thermo Fisher Scientific | 260860 | |
| Cell culture vacuum pump (VACUSAFE) | Integra Bioscience | 158300 | |
| Deoxynucleotide triphosphates (dNTPs) mix 10 mM each | Fermentas | R0192 | |
| DMSO | Sigma-Aldrich | 276855 | |
| DTT (100 mM) | Invitrogen | 18064-014 | |
| EDTA | Sigma-Aldrich | 798681 | for adherent cells |
| Ethanol | Sigma-Aldrich | 51976 | |
| Fetal Bovine Serum | Thermo Fisher Scientific | 26140079 | |
| Filter tips (10 µL) | Gilson | F171203 | |
| Filter tips (100 µL) | Gilson | F171403 | |
| Filter tips (20 µL) | Gilson | F171303 | |
| Filter tips (200 µL) | Gilson | F171503 | |
| Guanidine Hydrochloride | Sigma-Aldrich | G3272 | |
| ISPCR primer (10 µM) | Biomers.net GmbH | SP10006 | 5′-AAGCAGTGGTATCAACGCAGAG T-3′ |
| KAPA HiFi HotStart ReadyMix (2X) | KAPA Biosystems | KK2601 | |
| Magnesium chloride (MgCl2) | Sigma-Aldrich | M8266 | |
| Magnetic stand 96 | Ambion | AM10027 | |
| Neutralize Tagment (NT) Buffer | Illumina | FC-131-1096 | Nextera XT DNA Library Prep Kit (96 samples), alternatively 0.2 % SDS |
| Nextera-compatible indexing primer | Illumina | ||
| Nuclease-free water | Invitrogen | 10977049 | |
| PBS | Thermo Fisher Scientific | AM9624 | |
| PCR 96-well plates | Thermo Fisher Scientific | AB0600 | |
| PCR plate sealer | Thermo Fisher Scientific | HSF0031 | |
| Penicillin / Streptomycin | Thermo Fisher Scientific | 15070063 | |
| Qubit 4 fluorometer | Invitrogen | 15723679 | |
| Recombinant RNase inhibitor (40 U/ul) | TAKARA | 2313A | |
| RPMI-1640 cell culture medium | Gibco | 61870036 | If not working with PBMCs, adjust to cell type |
| SMART dT30VN primer | Sigma-Aldrich | 5' Bio-AAGCAGTGGTATCAACGCAGAG TACT30VN-3 | |
| Standard lab equipment | various | various | e.g. centrifuge, ice machine, ice bucket, distilled water, water bath |
| SuperScript II Reverse Transcriptase (SSRT II) | Thermo Fisher Scientific | 18064-014 | |
| SuperScript II Reverse Transcriptase (SSRT II) buffer (5x) | Thermo Fisher Scientific | 18064-014 | |
| Tagment DNA Buffer (TD) | Illumina | FC-131-1096 | Nextera XT DNA Library Prep Kit (96 samples) |
| TapeStation system 4200 | Agilent | G2991BA | |
| Thermocycler (S1000) | Bio-Rad | 1852148 | |
| TSO-LNA (100 uM) | Eurogentec | 5' Biotin AAGCAGTGGTATCAACGCAGAG TACAT(G)(G){G | |
| Vortex-Genie 2 Mixer | Sigma-Aldrich | Z258415 |
Referências
- Hughes, J. P., Rees, S., Kalindjian, S. B., Philpott, K. L. Principles of early drug discovery. Br J Pharmacol. 162 (6), 1239-1249 (2011).
- Yang, X., et al. High-throughput transcriptome profiling in drug and biomarker discovery. Front Genet. 11, 19 (2020).
- Bonaguro, L., et al. A guide to systems-level immunomics. Nat Immunol. 23 (10), 1412-1423 (2022).
- Carraro, C., et al. Decoding mechanism of action and sensitivity to drug candidates from integrated transcriptome and chromatin state. ELife. 11, 78012 (2022).
- Picelli, S., et al. Smart-seq2 for sensitive full-length transcriptome profiling in single cells. Nat Methods. 10 (11), 1096-1098 (2013).
- Picelli, S., et al. Full-length RNA-seq from single cells using Smart-seq2. Nat Protoc. 9 (1), 171-181 (2014).
- De Domenico, E., et al. Optimized workflow for single-cell transcriptomics on infectious diseases including COVID-19. STAR Protoc. 1 (3), 100233 (2020).
- Dobin, A., et al. ultrafast universal RNA-seq aligner. Bioinformatics. 29 (1), 15-21 (2013).
- Patro, R., et al. Salmon provides fast and bias-aware quantification of transcript expression. Nat Methods. 14 (4), 417-419 (2017).
- Love, M. I., Huber, W., Anders, S. Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2. Genome Biol. 15 (12), 550 (2014).
- Frankish, A., et al. GENCODE reference annotation for the human and mouse genomes. Nucleic Acids Res. 47, D766-D773 (2019).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados