需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
基于转录组学的经济高效的药物筛选
摘要
该协议描述了从 离体 或 体外 细胞培养到转录组数据预处理的工作流程,以实现具有成本效益的基于转录组的药物筛选。
摘要
转录组学可以全面了解细胞程序及其对扰动的反应。尽管在过去十年中,文库生产和测序的成本显著降低,但以药物筛选所需的规模应用这些技术仍然非常昂贵,阻碍了这些方法的巨大潜力。我们的研究提出了一种具有成本效益的基于转录组的药物筛选系统,将小型扰动培养物与小型批量转录组学相结合。优化的小型批量实验方案以经济高效的测序深度提供信息丰富的生物信号,从而能够对已知药物和新分子进行广泛筛选。根据所选的处理和孵育时间,该方案将在大约 2 天内产生测序文库。由于该协议中有多个停止点,因此文库制备和测序可以不受时间限制地进行。可以同时处理大量样品;对多达 384 个样品的测量进行了测试,而不会降低数据质量。尽管考虑了最佳药物孵育时间的可变性,但对条件和/或药物的数量也没有已知的限制。
引言
新药开发是一个复杂且耗时的过程,涉及识别潜在药物及其靶点、优化和合成候选药物,以及在临床前和临床试验中测试其有效性和安全性1。传统的药物筛选方法,即用于治疗目的的候选化合物库的系统评估,涉及使用动物模型或基于细胞的测定来测试对特定靶标或通路的影响。虽然这些方法在识别候选药物方面取得了成功,但它们往往不能对药物疗效背后的复杂分子机制以及潜在副作用的毒性和机制提供足够的见解。
评估全基因组转录状态是克服当前药物筛选局限性的有力方法,因为它可以全面评估响应药物治疗的基因表达2。通过以全基因组方式测量在给定时间表达的 RNA 转录本,转录组学旨在提供响应药物时发生的转录变化的整体视图,包括基因表达模式、选择性剪接和非编码 RNA 表达的变化3。这些信息可用于确定药物靶点,预测药物疗效和毒性,并优化药物剂量和治疗方案。
将转录组学与无偏倚药物筛选相结合的主要好处之一是有可能识别以前未考虑过的新药物靶点。传统的药物筛选方法通常侧重于已建立的靶点分子或通路,阻碍了新靶点的识别,并可能导致药物具有不可预见的副作用和有限的有效性。转录组学可以通过深入了解药物治疗后发生的分子变化来克服这些局限性,发现以前可能未考虑过的潜在靶点或途径2.
除了鉴定新的药物靶点外,转录组学还可用于预测药物疗效和毒性。通过分析与药物反应相关的基因表达模式,可以开发可用于预测患者对特定药物或治疗方案的反应的生物标志物。这也有助于优化药物剂量并降低不良副作用的风险4.
尽管转录组学具有潜在的好处,但其成本仍然是其在药物筛选中广泛应用的重大障碍。转录组学分析需要专门的设备、技术专长和数据分析,这可能使小型研究团队或资金有限的组织难以在药物筛选中使用转录组学。然而,转录组学的成本一直在稳步下降,使其更容易被研究界所接受。此外,技术和数据分析方法的进步使转录组学更加高效和具有成本效益,进一步提高了其可及性2。
在该方案中,我们描述了一种用于基于转录组的药物筛选的高维和探索性系统,将小型化扰动培养物与小型批量转录组学分析相结合5,6。使用该方案,可以将每个样品的成本降低到目前全长mRNA测序商业解决方案成本的1/6。该协议仅需要标准实验室设备,唯一的例外是使用短读长测序技术,如果内部没有测序仪器,则可以将其外包。优化的小型批量实验方案以具有成本效益的测序深度提供信息丰富的生物信号,从而能够对已知药物和新分子进行广泛筛选。
该实验的目的是筛选不同生物学背景下 PBMC 的药物活性。该方案可以应用于任何生物学问题,其中几种药物应该用转录组读数进行测试,从而提供治疗的细胞效应的转录组范围视图。
研究方案
该协议遵循波恩大学当地伦理委员会的指导方针。
1. 缓冲液、溶液和设备的制备
- 准备解决方案并收集 材料表中描述的材料。
- 将水浴加热至37°C,并加热完整的生长培养基(RPMI-1640 + 10%胎牛血清(FCS)+ 1%青霉素/链霉素)。
- 对于细胞收获,使用冰冷的磷酸盐缓冲盐水 (PBS)。
注意:在使用细胞时保持清洁环境以保持无菌。
2. 细胞处理
注意:从人血中冷冻保存外周血单核细胞 (PBMC) 的详细方案可在7 中找到。
- 细胞解冻和计数
- 从液氮中取出冷冻管,并在37°C的水浴中解冻2-3分钟,同时轻轻倒置。
- 将解冻的细胞转移到 50 mL 锥形管中。
- 用 1 mL 温热的完全生长培养基冲洗冷冻管,并将该溶液滴加到管中的细胞中(第 1 个 稀释步骤)。
- 以 1:1 重复稀释,直至达到 32 mL 的体积(总共 5 次稀释,分别使用 1、2、4、8 和 16 mL 温热的完全生长培养基)。滴加培养基以减少细胞干扰,同时稍微搅动锥形管。
- 将细胞悬液离心5分钟(300×g,20°C),并通过轻轻倾斜锥形管以一个流畅的运动除去上清液。
- 将细胞重悬于 3 mL 温暖的完全生长培养基中,然后进行细胞计数。
- 计数时,将 10 μL 细胞悬液与台盼蓝混合(稀释度为 1:2 至 1:10,具体取决于细胞悬液的密度),以在计数时区分活细胞和死细胞。由于染料的吸收,死亡或受损的细胞会呈现蓝色,而重要的细胞则不会被染色。
注意:台盼蓝具有轻微的细胞毒性,染色的细胞不应储存超过5分钟。 - 使用 10 μL 染色细胞溶液使用自动细胞计数器或计数室对细胞进行计数。
- 当使用Neubauer改进的细胞室时,计算位于角落的四个大方块内的所有细胞,并使用以下公式计算细胞悬液的浓度。
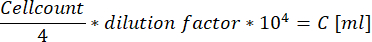
- 当使用Neubauer改进的细胞室时,计算位于角落的四个大方块内的所有细胞,并使用以下公式计算细胞悬液的浓度。
- 用温热的完全生长培养基将细胞稀释至终浓度为 1 x 106 个细胞/mL。仅当所需体积低于 3 mL 时离心,并将细胞沉淀重悬至正确体积。
- 细胞接种和处理
- 在96孔细胞培养板(1×105 个细胞/孔)中每孔接种100μL细胞悬浮液。
注意:用于接种的细胞数针对PBMC培养物进行了优化。虽然低细胞浓度不会产生足够量的RNA进行测序,但过多的细胞会增加细胞裂解次优和逆转录反应抑制的风险。 - 制备浓度为治疗浓度(2x)的两倍的药物稀释液。根据化合物的溶解度选择溶剂,优选完全生长培养基。
注意: 如果无法达到足够的溶解度,则可以使用 PBS 或二甲基亚砜 (DMSO)。所得孵育体积中DMSO的最大浓度不应超过0.5%,以防止溶剂引起的细胞毒性。 - 加入100μL的2x药物稀释液(孔1X中的终浓度),并在37°C孵育规定的时间。
注意:应进行补充研究,以确定最佳药物孵育时间并排除细胞毒性的潜在机制。
- 在96孔细胞培养板(1×105 个细胞/孔)中每孔接种100μL细胞悬浮液。
- 细胞收获和裂解
- 将细胞培养板离心10分钟(300×g,20°C)。用真空泵轻轻除去上清液,使板保持一定角度(30°-45°),同时将尖端对准孔的低角以去除尽可能少的细胞。
- 通过向每个孔中加入200μL冰冷的PBS来洗涤细胞,并将板离心5分钟(300× g,4°C)。再次用真空泵除去上清液,将板保持在锐角以去除尽可能多的PBS。
- 如 表1 所述,根据所需的反应次数(rxn)制备裂解缓冲液,添加10%的过量。
- 向每个孔中加入 15 μL 裂解缓冲液,并用粘性密封膜密封板,以保护样品免受污染。在离心1分钟(1000× g,4°C)之前涡旋板,并在冰上孵育5分钟。
- 在PCR板中收集6μL裂解的细胞溶液,并在-80°C下快速冷冻细胞裂解物,以确保最佳的细胞裂解。确保避免板的多次冷冻循环。
注意:停止点:如果随后不进行cDNA生产,细胞裂解物可以在-80°C下储存长达数月,而不会显着降低RNA质量。
3. 用于测序的文库制备
- 逆转录酶(RT)反应
注意:使用 RNA 时,将所有样品放在冰上,并确保使用无核酸酶设备(无菌、一次性塑料器皿)和水。- 如 表2 所述制备RT反应混合物,以获得所需的反应次数,增加10%的过量。短暂地涡旋混合物,然后快速旋转。将混合物放在冰上直到使用。
- 在室温 (RT) 下解冻细胞裂解物并短暂旋转以收集板底部的所有细胞裂解物。
- 如 表3所示,在热循环仪上进行mRNA变性。
- 将板从热循环仪中取出,并快速旋转以收集潜在的冷凝水。向每个孔中加入 6 μL RT 反应混合物,最终体积为 12 μL。 密封板以保护板并避免蒸发、涡旋和旋转。
- 将板放在热循环仪上,并按照 表3启动RT反应程序。
- 预放大
- 在室温下解冻高保真DNA聚合酶和 原位 PCR(ISPCR)引物。
- 根据 表4 制备所需反应次数的预扩增混合物,添加10%过量。短暂地涡旋混合物并将其旋转下来。
注意:酶混合物在室温下可稳定数小时。 - 旋转PCR板,向每个孔中加入15μL预扩增混合物并密封板。
- 将其放在热循环仪上,并按照 表5开始预扩增反应。
- 根据RNA含量调整循环数。从较低的数字开始,如果 cDNA 产量不足,则增加。
注意:根据我们的经验,应为每种细胞类型建立最佳循环数,实验条件和处理不会影响最佳循环数。因此,我们建议在为新细胞类型建立该方案时通过实验建立最佳循环数。一般来说,与细胞系相比,原代细胞需要更多的循环数。停止点:预扩增产物可储存在-20°C。
- 根据RNA含量调整循环数。从较低的数字开始,如果 cDNA 产量不足,则增加。
- cDNA纯化和质量控制(QC)
注意:可以按顺序对每个板进行样品净化,因为样品在此阶段相当稳定。增加样本数量将导致协议的持续时间更长,但不会限制可以同时处理的样本数量。- 开始前,将磁性纯化珠置于室温,高速涡旋1分钟,使珠子完全重悬。
- 向每个孔中加入 0.8x v/v (20 μL) 磁性纯化微珠,并在室温下孵育 5 分钟。
- 将PCR板放在磁性架上5分钟,直到珠子完全分离。小心地取出上清液。
- 将板放在磁架上,轻轻加入 100 μL 新鲜制备的 80% 乙醇以洗涤珠子并孵育 30 秒。使用小体积移液器除去上清液。重复额外的洗涤步骤。一定要去除尽可能多的乙醇。
- 在室温下将磁性架上的珠子风干长达 5 分钟,直到乙醇完全蒸发并且珠子不再有光泽。
- 从磁性架上取下板,将珠子重悬于20μL无核酸酶水中并孵育2分钟。
- 将板再次放在磁架上,直到珠子分离。
- 在新的PCR板中回收洗脱液,用于cDNA QC和标记。
- 执行 TapeStation 或 FragmentAnalyser 检测以评估 cDNA 文库的大小分布和浓度(推荐:TapeStation D5000 检测)。有关详细信息,请参阅制造商说明。预计产量为 20 ng。
- 使用试剂盒进行标记
注意:如果已经建立,可以使用其他标记协议。- 使用不含核酸酶的水将cDNA稀释至终浓度为150-300pg/μL。
- 按照 表6 对热循环仪进行预编程,以确保在将cDNA添加到酶混合物中后立即开始标记反应。
- 根据 表7 准备所需的反应次数的标记混合物,添加10%的过量。
- 将每个反应的 3 μL 标记混合物分配到新的 PCR 板上,并向每个反应中加入 1 μL cDNA。
- 加入cDNA后立即开始标记反应。
- 通过加入1μL中和标记缓冲液(NT;或者,可以使用0.2%十二烷基硫酸钠(SDS))来灭活反应。
- 富集PCR
- 制备 表7 中用于反应次数的富集PCR混合物,添加10%超量。(推荐:Nextera UDI设置,以防止图案化流通池(例如,Illumina NovaSeq 6000)上的索引跳跃)。
- 向每个反应中加入 9 μL 富集 PCR 混合物,总体积为 14 μL。
- 如 表8所示运行富集PCR程序。
注意:对于富集PCR,标准为16个循环。如果cDNA质量较差,可以添加额外的循环。
- 清理和质量控制
- 开始前,将磁性纯化珠置于室温,高速涡旋1分钟,使珠子完全重悬。
- 加入 1.0x v/v (14 μL) 磁性纯化珠并在室温下孵育 5 分钟。
- 将板放在磁性架上,等待5分钟,直到珠子完全分离。小心地取出上清液。
- 将板放在磁架上,轻轻加入 100 μL 新鲜制备的 80% 乙醇以洗涤珠子并孵育 30 秒。使用小体积移液器除去上清液。重复额外的洗涤步骤。一定要去除尽可能多的乙醇。
- 在室温下将珠子风干 3 分钟或直到它们看起来不再有光泽。
- 从磁性架上取下板,将珠子重悬于20μL无核酸酶水中并孵育2分钟。
- 将板再次放在磁架上,直到磁珠分离,并在新的PCR板中回收洗脱液进行文库QC。
- 执行 TapeStation 或片段分析仪检测,以评估 cDNA 文库的大小分布和浓度(推荐:TapeStation D1000 高灵敏度检测)。有关详细信息,请参阅制造商说明。平均而言,预计产量为 10 ng。
注意:停止点:PCR产物可以储存在-20°C。
4. 测序和数据预处理
- 测 序
注:以下指南将适用于所有用于短读长测序的Illumina仪器。如果没有仪器,则可以通过外部测序设备进行测序。也可以使用其他测序方法。为简单起见,我们选择仅报告使用最广泛的测序技术。
注:以下与软件使用相关的步骤描述了Illumina NovaSeq6000测序仪的操作步骤。- 根据步骤 3.6.8 中获得的结果,以等摩尔比汇集唯一索引的文库。
- 使用高灵敏度测定法测量最终池的浓度,以计算样品摩尔浓度,如下所示:
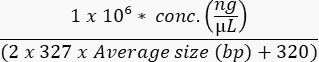
- 根据仪器的规格和实验优化加载流通池。常用仪器的上样浓度示例如 表9所示。
- 通过触摸屏幕,选择 "序列 "以启动运行设置。
- 按照屏幕上的说明进行操作,并加载流通池、逐个合成的滤芯、聚簇滤芯、缓冲滤芯,并确保废物容器是空的。
- 仪器识别出所有试剂后,单击 "运行设置"。在此处定义运行名称和用于存储数据的输出文件夹。
- 将测序细节定义为双端,两个读长均为 51 bp。还对两个索引读长进行测序,每个读长 8 bp。
- 按 Review 并在检查所有排序详细信息是否正确后按 Start Run。
注:推荐的测序深度为 5 x 106 个读段/样本,最小值为 1 x 106 个读段/样本。
- 数据预处理
- 使用Bcl2Fastq2工具将原始测序数据转换为FASTQ格式,并根据样本索引进行复用解复用。使用默认设置执行解复用。有关 Bcl2Fastq2 的详细说明,请参阅参考手册。
注意:如果测序不是在内部进行的,则通常由测序设备执行FASTQ转换和解复用。测序设备通常会提供解复用的 FASTQ 以进行进一步处理。 - 有多种选项可用于测序读长的数据比对和丰度定量(推荐:nf-core RNA-seq 管路 (https://nf-co.re/rnaseq))。该管道提供了多个选项,使用默认设置,其中 STAR8 对齐,Salmon9 用于量化转录本丰度。
- 注意:对于进一步的生物信息学分析,有几种方法可用。它不在此协议的范围内涵盖所有(推荐:DEseq2 管道10)。可以在 GitHub (https://github.com/jsschrepping/RNA-DESeq2) 上找到基于我们开发的 DEseq2 工作流的标准脚本。
- 使用Bcl2Fastq2工具将原始测序数据转换为FASTQ格式,并根据样本索引进行复用解复用。使用默认设置执行解复用。有关 Bcl2Fastq2 的详细说明,请参阅参考手册。
结果
按照报告的方案,接种人PBMC,用不同的免疫调节药物处理,并在不同的孵育时间后,收获使用测序方案进行批量转录组分析(图1)。
测试化合物的理想药物浓度和孵育时间应在补充实验策略的帮助下,根据具体的科学问题确定本协议的上游。在大多数情况下,2-4小时和24小时孵育应提供对治疗的早期和晚期转录反应的代表。
评估?...
讨论
药物发现和药物开发可以从批量转录组学提供的细胞过程的整体视图中受益匪浅。然而,这种方法往往受到标准批量RNA-seq实验方案的高成本的限制,阻碍了其在学术环境中的应用以及其工业可扩展性的潜力。
该方案最关键的步骤是细胞解冻和文库制备的初始步骤。确保解冻后细胞的高活力对于成功的治疗和转录组学分析至关重要。细胞收获和文库制备的第一步,直到cDNA合成...
披露声明
作者声明没有竞争利益。
致谢
J.L.S. 由德国研究基金会 (DFG) 根据德国卓越战略 (EXC2151-390873048) 以及 SCHU 950/8-1 提供支持;GRK 2168,TP11;CRC SFB 1454 Metaflammation、IRTG GRK 2168、WGGC INST 216/981-1、CCU INST 217/988-1、BMBF 资助的卓越项目 Diet-Body-Brain (DietBB);以及欧盟项目 SYSCID,资助号为 733100。M.B. 由 DFG (IRTG2168-272482170, SFB1454-432325352) 提供支持。L.B.得到了DFG(ImmuDiet BO 6228/2-1 - 项目编号513977171)和德国卓越战略(EXC2151-390873048)的支持。使用 BioRender.com 创建的图像。
材料
| Name | Company | Catalog Number | Comments |
| 50 mL conical tube | fisher scientific | 10203001 | |
| Adhesive PCR Plate Seals | Thermo Fisher Scientific | AB0558 | |
| Amplicon Tagment Mix (ATM) | Illumina | FC-131-1096 | Nextera XT DNA Library Prep Kit (96 samples) |
| AMPure XP beads | Beckman Coulter | A 63881 | |
| Betaine | Sigma-Aldrich | 61962 | |
| Cell culture grade 96-well plates | Thermo Fisher Scientific | 260860 | |
| Cell culture vacuum pump (VACUSAFE) | Integra Bioscience | 158300 | |
| Deoxynucleotide triphosphates (dNTPs) mix 10 mM each | Fermentas | R0192 | |
| DMSO | Sigma-Aldrich | 276855 | |
| DTT (100 mM) | Invitrogen | 18064-014 | |
| EDTA | Sigma-Aldrich | 798681 | for adherent cells |
| Ethanol | Sigma-Aldrich | 51976 | |
| Fetal Bovine Serum | Thermo Fisher Scientific | 26140079 | |
| Filter tips (10 µL) | Gilson | F171203 | |
| Filter tips (100 µL) | Gilson | F171403 | |
| Filter tips (20 µL) | Gilson | F171303 | |
| Filter tips (200 µL) | Gilson | F171503 | |
| Guanidine Hydrochloride | Sigma-Aldrich | G3272 | |
| ISPCR primer (10 µM) | Biomers.net GmbH | SP10006 | 5′-AAGCAGTGGTATCAACGCAGAG T-3′ |
| KAPA HiFi HotStart ReadyMix (2X) | KAPA Biosystems | KK2601 | |
| Magnesium chloride (MgCl2) | Sigma-Aldrich | M8266 | |
| Magnetic stand 96 | Ambion | AM10027 | |
| Neutralize Tagment (NT) Buffer | Illumina | FC-131-1096 | Nextera XT DNA Library Prep Kit (96 samples), alternatively 0.2 % SDS |
| Nextera-compatible indexing primer | Illumina | ||
| Nuclease-free water | Invitrogen | 10977049 | |
| PBS | Thermo Fisher Scientific | AM9624 | |
| PCR 96-well plates | Thermo Fisher Scientific | AB0600 | |
| PCR plate sealer | Thermo Fisher Scientific | HSF0031 | |
| Penicillin / Streptomycin | Thermo Fisher Scientific | 15070063 | |
| Qubit 4 fluorometer | Invitrogen | 15723679 | |
| Recombinant RNase inhibitor (40 U/ul) | TAKARA | 2313A | |
| RPMI-1640 cell culture medium | Gibco | 61870036 | If not working with PBMCs, adjust to cell type |
| SMART dT30VN primer | Sigma-Aldrich | 5' Bio-AAGCAGTGGTATCAACGCAGAG TACT30VN-3 | |
| Standard lab equipment | various | various | e.g. centrifuge, ice machine, ice bucket, distilled water, water bath |
| SuperScript II Reverse Transcriptase (SSRT II) | Thermo Fisher Scientific | 18064-014 | |
| SuperScript II Reverse Transcriptase (SSRT II) buffer (5x) | Thermo Fisher Scientific | 18064-014 | |
| Tagment DNA Buffer (TD) | Illumina | FC-131-1096 | Nextera XT DNA Library Prep Kit (96 samples) |
| TapeStation system 4200 | Agilent | G2991BA | |
| Thermocycler (S1000) | Bio-Rad | 1852148 | |
| TSO-LNA (100 uM) | Eurogentec | 5' Biotin AAGCAGTGGTATCAACGCAGAG TACAT(G)(G){G | |
| Vortex-Genie 2 Mixer | Sigma-Aldrich | Z258415 |
参考文献
- Hughes, J. P., Rees, S., Kalindjian, S. B., Philpott, K. L. Principles of early drug discovery. Br J Pharmacol. 162 (6), 1239-1249 (2011).
- Yang, X., et al. High-throughput transcriptome profiling in drug and biomarker discovery. Front Genet. 11, 19 (2020).
- Bonaguro, L., et al. A guide to systems-level immunomics. Nat Immunol. 23 (10), 1412-1423 (2022).
- Carraro, C., et al. Decoding mechanism of action and sensitivity to drug candidates from integrated transcriptome and chromatin state. ELife. 11, 78012 (2022).
- Picelli, S., et al. Smart-seq2 for sensitive full-length transcriptome profiling in single cells. Nat Methods. 10 (11), 1096-1098 (2013).
- Picelli, S., et al. Full-length RNA-seq from single cells using Smart-seq2. Nat Protoc. 9 (1), 171-181 (2014).
- De Domenico, E., et al. Optimized workflow for single-cell transcriptomics on infectious diseases including COVID-19. STAR Protoc. 1 (3), 100233 (2020).
- Dobin, A., et al. ultrafast universal RNA-seq aligner. Bioinformatics. 29 (1), 15-21 (2013).
- Patro, R., et al. Salmon provides fast and bias-aware quantification of transcript expression. Nat Methods. 14 (4), 417-419 (2017).
- Love, M. I., Huber, W., Anders, S. Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2. Genome Biol. 15 (12), 550 (2014).
- Frankish, A., et al. GENCODE reference annotation for the human and mouse genomes. Nucleic Acids Res. 47, D766-D773 (2019).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。