È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Screening farmacologico basato sulla trascrittomica efficiente in termini di costi
In questo articolo
Riepilogo
Questo protocollo descrive un flusso di lavoro che va dalle colture cellulari ex vivo o in vitro alla pre-elaborazione dei dati trascrittomici per uno screening farmacologico basato sul trascrittoma economicamente vantaggioso.
Abstract
La trascrittomica consente di ottenere informazioni complete sui programmi cellulari e sulle loro risposte alle perturbazioni. Nonostante una significativa diminuzione dei costi di produzione e sequenziamento delle librerie nell'ultimo decennio, l'applicazione di queste tecnologie alla scala necessaria per lo screening dei farmaci rimane proibitivamente costosa, ostacolando l'immenso potenziale di questi metodi. Il nostro studio presenta un sistema economicamente vantaggioso per lo screening farmacologico basato sul trascrittoma, che combina colture di perturbazione miniaturizzate con trascrittomica mini-bulk. Il protocollo mini-bulk ottimizzato fornisce segnali biologici informativi a una profondità di sequenziamento conveniente, consentendo uno screening approfondito di farmaci noti e nuove molecole. A seconda del trattamento scelto e del tempo di incubazione, questo protocollo si tradurrà in librerie di sequenziamento entro circa 2 giorni. A causa dei diversi punti di arresto all'interno di questo protocollo, la preparazione della libreria, così come il sequenziamento, possono essere eseguiti indipendentemente dal tempo. È possibile processare contemporaneamente un numero elevato di campioni; La misurazione di un massimo di 384 campioni è stata testata senza perdita di qualità dei dati. Inoltre, non sono note limitazioni al numero di condizioni e/o farmaci, nonostante si consideri la variabilità nei tempi ottimali di incubazione dei farmaci.
Introduzione
Lo sviluppo di nuovi farmaci è un processo complesso e dispendioso in termini di tempo che comporta l'identificazione di potenziali farmaci e dei loro bersagli, l'ottimizzazione e la sintesi di farmaci candidati e la verifica della loro efficacia e sicurezza in studi preclinici e clinici1. I metodi tradizionali per lo screening dei farmaci, cioè la valutazione sistematica di librerie di composti candidati per scopi terapeutici, prevedono l'uso di modelli animali o saggi basati su cellule per testare gli effetti su bersagli o percorsi specifici. Sebbene questi metodi abbiano avuto successo nell'identificare i farmaci candidati, spesso non hanno fornito informazioni sufficienti sui complessi meccanismi molecolari alla base dell'efficacia dei farmaci e anche della tossicità e dei meccanismi dei potenziali effetti collaterali.
La valutazione degli stati trascrizionali dell'intero genoma rappresenta un approccio potente per superare gli attuali limiti nello screening farmacologico, in quanto consente valutazioni complete dell'espressione genica in risposta ai trattamenti farmacologici2. Misurando i trascritti dell'RNA in modo genome-wide espressi in un dato momento, la trascrittomica mira a fornire una visione olistica dei cambiamenti trascrizionali che si verificano in risposta ai farmaci, compresi i cambiamenti nei modelli di espressione genica, lo splicing alternativo e l'espressione dell'RNA non codificante3. Queste informazioni possono essere utilizzate per determinare i bersagli farmacologici, prevedere l'efficacia e la tossicità dei farmaci e ottimizzare il dosaggio dei farmaci e i regimi di trattamento.
Uno dei principali vantaggi della combinazione della trascrittomica con uno screening imparziale dei farmaci è la possibilità di identificare nuovi bersagli farmacologici che non sono stati precedentemente considerati. Gli approcci convenzionali di screening dei farmaci spesso si concentrano su molecole o percorsi bersaglio stabiliti, ostacolando l'identificazione di nuovi bersagli e potenzialmente dando luogo a farmaci con effetti collaterali imprevisti e un'efficacia limitata. La trascrittomica può superare queste limitazioni fornendo informazioni sui cambiamenti molecolari che si verificano in risposta al trattamento farmacologico, scoprendo potenziali bersagli o percorsi che potrebbero non essere stati considerati in precedenza2.
Oltre all'identificazione di nuovi bersagli farmacologici, la trascrittomica può essere utilizzata anche per prevedere l'efficacia e la tossicità dei farmaci. Analizzando i modelli di espressione genica associati alle risposte ai farmaci, è possibile sviluppare biomarcatori che possono essere utilizzati per prevedere la risposta di un paziente a un particolare farmaco o regime di trattamento. Questo può anche aiutare a ottimizzare il dosaggio del farmaco e ridurre il rischio di effetti collaterali avversi4.
Nonostante i suoi potenziali benefici, il costo della trascrittomica rimane un ostacolo significativo alla sua applicazione diffusa nello screening farmacologico. L'analisi trascrittomica richiede attrezzature specializzate, competenze tecniche e analisi dei dati, il che può rendere difficile per i team di ricerca più piccoli o le organizzazioni con finanziamenti limitati utilizzare la trascrittomica nello screening dei farmaci. Tuttavia, il costo della trascrittomica è in costante diminuzione, rendendola più accessibile alle comunità di ricerca. Inoltre, i progressi nella tecnologia e nei metodi di analisi dei dati hanno reso la trascrittomica più efficiente ed economica, aumentandone ulteriormente l'accessibilità2.
In questo protocollo, descriviamo un sistema altamente dimensionale ed esplorativo per lo screening di farmaci basato sul trascrittoma, combinando colture di perturbazione miniaturizzate con analisi trascrittomica mini-bulk 5,6. Con questo protocollo, è possibile ridurre il costo per campione a 1/6 del costo attuale delle soluzioni commerciali per il sequenziamento dell'mRNA a lunghezza intera. Il protocollo richiede solo attrezzature di laboratorio standard, con l'unica eccezione dell'uso di tecnologie di sequenziamento a lettura breve, che possono essere esternalizzate se gli strumenti di sequenziamento non sono disponibili internamente. Il protocollo mini-bulk ottimizzato fornisce segnali biologici ricchi di informazioni a una profondità di sequenziamento conveniente, consentendo uno screening approfondito di farmaci noti e nuove molecole.
Lo scopo dell'esperimento è quello di effettuare lo screening dell'attività dei farmaci sulle PBMC in diversi contesti biologici. Questo protocollo può essere applicato a qualsiasi questione biologica in cui diversi farmaci devono essere testati con una lettura trascrittomica, fornendo una visione a livello di trascrittoma dell'effetto cellulare del trattamento.
Protocollo
Questo protocollo segue le linee guida dei comitati etici locali dell'Università di Bonn.
1. Preparazione di tamponi, soluzioni e attrezzature
- Preparare le soluzioni e raccogliere i materiali descritti nell'Indice dei materiali.
- Riscaldare il bagnomaria a 37 °C e riscaldare il terreno di coltura completo (RPMI-1640 + 10% siero fetale di vitello (FCS) + 1% penicillina/streptomicina).
- Per la raccolta delle cellule, utilizzare soluzione salina tamponata con fosfato ghiacciato (PBS).
NOTA: Mantenere un ambiente pulito mentre si lavora con le cellule per mantenere la sterilità.
2. Manipolazione delle celle
NOTA: Un protocollo dettagliato per la crioconservazione delle cellule mononucleate del sangue periferico (PBMC) dal sangue umano può essere trovato in7.
- Scongelamento e conteggio delle cellule
- Rimuovere i crioviali dall'azoto liquido e scongelarli a bagnomaria a 37 °C per 2 - 3 minuti capovolgendo delicatamente.
- Trasferire le cellule scongelate in una provetta conica da 50 ml.
- Sciacquare il crioviale con 1 mL di terreno di coltura completo caldo e aggiungere questa soluzione goccia a goccia alle cellule della provetta (1a fasedi diluizione ).
- Ripetere la diluizione 1:1 fino a raggiungere un volume di 32 mL (5 diluizioni totali rispettivamente con 1, 2, 4, 8 e 16 mL di terreno di coltura completo caldo). Aggiungere il mezzo goccia a goccia per ridurre il disturbo cellulare agitando leggermente il tubo conico.
- Centrifugare la sospensione cellulare per 5 minuti (300 x g, 20 °C) e rimuovere il surnatante inclinando delicatamente la provetta conica con un movimento fluido.
- Risospendere le cellule in 3 mL di terreno di coltura completo caldo e procedere con la conta cellulare.
- Per il conteggio, miscelare 10 μL di sospensione cellulare con Trypan Blue (diluizione da 1:2 a 1:10 a seconda della densità della sospensione cellulare) per distinguere le cellule vive da quelle morte durante il conteggio. Le cellule morte o danneggiate appariranno blu a causa dell'assorbimento del colorante, mentre le cellule vitali non vengono colorate.
NOTA: Trypan Blue è leggermente citotossico, le cellule colorate non devono essere conservate per più di 5 minuti. - Contare le cellule con un contatore di cellule automatizzato o una camera di conteggio utilizzando 10 μL di soluzione cellulare colorata.
- Quando si utilizza la camera cellulare migliorata da Neubauer, contare tutte le cellule all'interno dei quattro grandi quadrati situati agli angoli e utilizzare la seguente equazione per calcolare la concentrazione della sospensione cellulare.
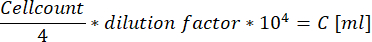
- Quando si utilizza la camera cellulare migliorata da Neubauer, contare tutte le cellule all'interno dei quattro grandi quadrati situati agli angoli e utilizzare la seguente equazione per calcolare la concentrazione della sospensione cellulare.
- Diluire le cellule a una concentrazione finale di 1 x 106 cellule/mL con terreno di coltura completo caldo. Centrifugare solo se il volume richiesto è inferiore a 3 mL e risospendere il pellet cellulare al volume corretto.
- Semina e trattamento cellulare
- Seminare 100 μL di sospensione cellulare per pozzetto in una piastra di coltura cellulare a 96 pozzetti (1 x10 5 cellule/pozzetto).
NOTA: Il numero di celle per la semina è stato ottimizzato per le colture PBMC. Mentre basse concentrazioni cellulari non produrranno una quantità sufficiente di RNA per il sequenziamento, un numero eccessivo di cellule aumenterà il rischio di lisi cellulare non ottimale e di inibizione della reazione di trascrizione inversa. - Preparare diluizioni di farmaci a una concentrazione due volte superiore a quella utilizzata per il trattamento (2x). Scegliere il solvente in base alla solubilità del composto, preferibilmente terreno di coltura completo.
NOTA: PBS o dimetilsolfossido (DMSO) possono essere utilizzati se non è possibile raggiungere una solubilità sufficiente in altro modo. La concentrazione massima di DMSO nel volume di incubazione risultante non deve superare lo 0,5 % per evitare la citotossicità indotta dal solvente. - Aggiungere 100 μL della diluizione del farmaco 2x (concentrazione finale nel pozzetto 1X) e incubare a 37 °C per il tempo definito.
NOTA: Devono essere effettuate indagini complementari al fine di stabilire il tempo di incubazione ottimale del farmaco ed escludere potenziali meccanismi di citotossicità.
- Seminare 100 μL di sospensione cellulare per pozzetto in una piastra di coltura cellulare a 96 pozzetti (1 x10 5 cellule/pozzetto).
- Prelievo e lisi cellulare
- Centrifugare la piastra di coltura cellulare per 10 minuti (300 x g, 20 °C). Rimuovere delicatamente il surnatante con una pompa a vuoto mantenendo la piastra inclinata (30°-45°) mentre si punta la punta verso l'angolo inferiore del pozzetto per rimuovere il minor numero possibile di cellule.
- Lavare le cellule aggiungendo 200 μL di PBS ghiacciato a ciascun pozzetto e centrifugare la piastra per 5 minuti (300 x g, 4 °C). Rimuovere il surnatante con una pompa a vuoto, sempre mantenendo la piastra ad angolo acuto per rimuovere quanto più PBS possibile.
- Preparare il tampone di lisi come descritto nella Tabella 1 per il numero di reazioni (rxn) necessarie, aggiungendo il 10% in eccesso.
- Aggiungere 15 μL di tampone di lisi a ciascun pozzetto e sigillare la piastra con una pellicola sigillante adesiva per proteggere i campioni dalla contaminazione. Agitare la piastra prima della centrifugazione per 1 minuto (1000 x g, 4 °C) e incubare per 5 minuti su ghiaccio.
- Raccogliere 6 μL di soluzione cellulare lisata in una piastra PCR e congelare brevemente il lisato cellulare a -80 °C per garantire una lisi cellulare ottimale. Assicurarsi di evitare più cicli di congelamento delle piastre.
NOTA: PUNTO DI ARRESTO: Se la produzione di cDNA non viene effettuata successivamente, il lisato cellulare può essere conservato a -80 °C per un massimo di diversi mesi senza una notevole diminuzione della qualità dell'RNA.
3. Preparazione della libreria per il sequenziamento
- Reazione della trascrittasi inversa (RT)
NOTA: Quando si lavora con l'RNA, posizionare tutti i campioni su ghiaccio e assicurarsi di utilizzare attrezzature prive di nucleasi (articoli di plastica sterili e monouso) e acqua.- Preparare la miscela di reazione RT come descritto nella Tabella 2 per il numero di reazioni necessarie, aggiungendo il 10% in eccesso. Fate vorticare brevemente la miscela e fatela girare brevemente. Tenere il composto sul ghiaccio fino al momento dell'uso.
- Scongelare il lisato cellulare a temperatura ambiente (RT) e ruotarlo brevemente per raccogliere tutto il lisato cellulare sul fondo della piastra.
- Eseguire la denaturazione dell'mRNA su un termociclatore come da Tabella 3.
- Estrarre la piastra dal termociclatore e ruotarla brevemente verso il basso per raccogliere la potenziale condensa. Aggiungere 6 μL di miscela di reazione RT in ciascun pozzetto per un volume finale di 12 μL. Sigillare la piastra per proteggerla ed evitare l'evaporazione, il vortice e la rotazione verso il basso.
- Posizionare la piastra sul termociclatore e avviare il programma di reazione RT come da Tabella 3.
- Pre-amplificazione
- Scongelare il primer della DNA polimerasi ad alta fedeltà e della PCR in situ (ISPCR) a temperatura ambiente.
- Preparare la miscela di pre-amplificazione come da Tabella 4 per il numero di reazioni necessarie, aggiungendo il 10% di eccedenza. Agitare brevemente la miscela e farla girare.
NOTA: La miscela enzimatica è stabile a temperatura ambiente per ore. - Ruotare la piastra PCR, aggiungere 15 μL della miscela di preamplificazione a ciascun pozzetto e sigillare la piastra.
- Posizionarlo sul termociclatore e avviare la reazione di preamplificazione come da Tabella 5.
- Regolare il numero di cicli in base al contenuto di RNA. Iniziare con un numero più basso e aumentare se la resa di cDNA non è sufficiente.
NOTA: In base alla nostra esperienza, il numero ottimale di cicli dovrebbe essere stabilito per ogni tipo di cellula, le condizioni sperimentali e il trattamento non influenzeranno il numero ottimale di cicli. Pertanto, raccomandiamo di stabilire sperimentalmente il numero ottimale di cicli quando si stabilisce questo protocollo per un nuovo tipo di cellula. In generale, le cellule primarie richiedono un numero maggiore di cicli rispetto alle linee cellulari. PUNTO DI ARRESTO: Il prodotto di preamplificazione può essere conservato a - 20 °C.
- Regolare il numero di cicli in base al contenuto di RNA. Iniziare con un numero più basso e aumentare se la resa di cDNA non è sufficiente.
- Pulizia del cDNA e controllo qualità (QC)
NOTA: La pulizia del campione può essere eseguita in sequenza per ciascuna piastra poiché i campioni sono piuttosto stabili in questa fase. L'aumento del numero di campioni porterà a una maggiore durata del protocollo, ma non limiterà il numero di campioni che possono essere processati contemporaneamente.- Prima di iniziare, portare le perle di purificazione magnetica a temperatura ambiente e vorticare ad alta velocità per 1 minuto per risospendere completamente le perline.
- Aggiungere 0,8 x v/v (20 μL) di microsfere di purificazione magnetica a ciascun pozzetto e incubare a temperatura ambiente per 5 minuti.
- Posizionare la piastra PCR su un rack magnetico per 5 minuti fino a quando le perle non sono completamente separate. Rimuovere con cautela il surnatante.
- Tenendo la piastra sulla griglia magnetica, aggiungere delicatamente 100 μL di etanolo all'80% appena preparato per lavare le perle e incubare per 30 s. Utilizzare una pipetta di piccolo volume per rimuovere il surnatante. Ripetere l'operazione per un'ulteriore fase di lavaggio. Assicurati di rimuovere quanto più etanolo possibile.
- Asciugare all'aria le perline sulla griglia magnetica a temperatura ambiente per un massimo di 5 minuti fino a quando l'etanolo non è completamente evaporato e le perline non sembrano più lucide.
- Rimuovere la piastra dal rack magnetico, risospendere le perle in 20 μL di acqua priva di nucleasi e incubare per 2 minuti.
- Riposizionare la piastra sulla griglia magnetica fino a quando le perline non si sono separate.
- Recuperare l'eluato in una nuova piastra PCR per il controllo qualità e la codifica del cDNA.
- Eseguire un test TapeStation o FragmentAnalyser per valutare la distribuzione dimensionale e la concentrazione della libreria di cDNA (consigliato: saggio TapeStation D5000). Per i dettagli, vedere le istruzioni del produttore. Ci si può aspettare una resa tipica di 20 ng.
- Tagmentazione con kit
NOTA: È possibile utilizzare altri protocolli di tagmentazione se già stabiliti.- Diluire il cDNA a una concentrazione finale di 150 - 300 pg/μL utilizzando acqua priva di nucleasi.
- Preprogrammare il termociclatore come da Tabella 6 per garantire un avvio immediato della reazione di tagmentazione dopo aver aggiunto il cDNA alla miscela enzimatica.
- Preparare la miscela di tagmentazione come da Tabella 7 per il numero di reazioni necessarie, aggiungendo il 10% in eccesso.
- Erogare 3 μL della miscela di tagmentazione per reazione su una nuova piastra PCR e aggiungere 1 μL di cDNA a ciascuna reazione.
- Iniziare la reazione di tagmentazione subito dopo aver aggiunto il cDNA.
- Inattivare la reazione aggiungendo 1 μL di tampone di neutralizzazione (NT; in alternativa, è possibile utilizzare lo 0,2% di sodio dodecil solfato (SDS)).
- PCR di arricchimento
- Preparare la miscela di arricchimento PCR Tabella 7 per il numero di reazioni, aggiungendo il 10% in eccesso. (Consigliato: Nextera UDI impostato per impedire il salto dell'indice su celle di flusso modellate (ad esempio, Illumina NovaSeq 6000)).
- Aggiungere 9 μL della miscela di arricchimento PCR a ciascuna reazione per un volume totale di 14 μL.
- Eseguire il programma di arricchimento PCR come indicato nella Tabella 8.
NOTA: Per la PCR di arricchimento, sono standard 16 cicli. Se la qualità del cDNA è scarsa, è possibile aggiungere ulteriori cicli.
- Pulizia e controllo qualità
- Prima di iniziare, portare le perle di purificazione magnetica a temperatura ambiente e vorticare ad alta velocità per 1 minuto per risospendere completamente le perle.
- Aggiungere 1,0 x v/v (14 μL) di microsfere di purificazione magnetica e incubare a temperatura ambiente per 5 minuti.
- Posizionare il piatto su una griglia magnetica e attendere 5 minuti fino a quando le perline non sono completamente separate. Rimuovere con cautela il surnatante.
- Tenendo la piastra sulla griglia magnetica, aggiungere delicatamente 100 μL di etanolo all'80% appena preparato per lavare le perle e incubare per 30 s. Utilizzare una pipetta di piccolo volume per rimuovere il surnatante. Ripetere l'operazione per un'ulteriore fase di lavaggio. Assicurati di rimuovere quanto più etanolo possibile.
- Asciugare le perline all'aria a temperatura ambiente per 3 minuti o fino a quando non sembrano più lucide.
- Rimuovere la piastra dal rack magnetico, risospendere le perle in 20 μL di acqua priva di nucleasi e incubare per 2 minuti.
- Posizionare nuovamente la piastra sul rack magnetico fino a quando le perle non si separano e recuperare l'eluato in una nuova piastra PCR per il controllo qualità della libreria.
- Eseguire un test TapeStation o Fragment Analyzer per valutare la distribuzione dimensionale e la concentrazione della libreria di cDNA (consigliato: saggio ad alta sensibilità TapeStation D1000). Per i dettagli, vedere le istruzioni del produttore. In media, ci si può aspettare una resa di 10 ng.
NOTA: PUNTO DI ARRESTO: Il prodotto PCR può essere conservato a - 20 °C.
4. Sequenziamento e pre-elaborazione dei dati
- Sequenziamento
NOTA: Le seguenti linee guida saranno applicabili a tutti gli strumenti Illumina per il sequenziamento a lettura breve. Se la strumentazione non è disponibile, il sequenziamento può essere eseguito da una struttura di sequenziamento esterna. Potrebbero essere utilizzati anche altri approcci di sequenziamento. Per semplicità, abbiamo scelto di riportare solo la tecnologia di sequenziamento più utilizzata.
NOTA: I seguenti passaggi relativi all'utilizzo del software descrivono la procedura su un sequencer Illumina NovaSeq6000.- Raggruppare le librerie indicizzate in modo univoco in un rapporto equimolare in base ai risultati ottenuti nel passaggio 3.6.8.
- Misurare la concentrazione del pool finale con un test ad alta sensibilità per calcolare la molarità del campione come segue:
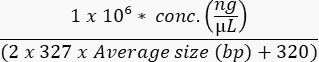
- Caricare la cella di flusso secondo le specifiche dello strumento e l'ottimizzazione sperimentale. Esempi di concentrazioni di carico di strumenti comuni sono riportati nella tabella 9.
- Toccando lo schermo, selezionare Sequenza per avviare l'impostazione dell'esecuzione.
- Seguire le istruzioni sullo schermo e sulla cella di flusso di carico, sulla cartuccia di sequenza per sintesi, sulla cartuccia di clustering, sulla cartuccia tampone e assicurarsi che i contenitori dei rifiuti siano vuoti.
- Una volta che tutti i reagenti sono stati riconosciuti dallo strumento, fare clic su Run Setup. Definire qui il nome dell'esecuzione e la cartella di output in cui memorizzare i dati.
- Definire i dettagli della sequenza come paired-end con entrambe le letture di 51 bp. Vengono inoltre sequenziate due letture dell'indice con 8 bp ciascuna.
- Premere Review e, dopo aver verificato se tutti i dettagli della sequenza sono corretti, premere Start Run.
NOTA: La profondità di sequenziamento consigliata è di 5 x 106 letture/campione, con un minimo di 1 x 106 letture/campione.
- Pre-elaborazione dei dati
- Trasforma i dati di sequenziamento grezzi in formato FASTQ e demultiplexa in base agli indici del campione con lo strumento Bcl2Fastq2. Eseguire il demultiplexing con le impostazioni predefinite. Per istruzioni dettagliate su Bcl2Fastq2, vedere il manuale di riferimento.
NOTA: La conversione e il demultiplexing di FASTQ vengono solitamente eseguiti dalla struttura di sequenziamento se il sequenziamento non viene eseguito internamente. Le strutture di sequenziamento di solito forniscono FASTQ demultiplexato per ulteriori elaborazioni. - Sono disponibili diverse opzioni per l'allineamento dei dati e la quantificazione dell'abbondanza delle letture di sequenziamento (consigliato: pipeline nf-core RNA-seq (https://nf-co.re/rnaseq)). La pipeline offre diverse opzioni, utilizzare l'impostazione predefinita con STAR8 come allineato e Salmon9 per quantificare l'abbondanza della trascrizione.
- NOTA: Per ulteriori analisi bioinformatiche, sono disponibili diversi metodi. Non rientra nell'ambito di questo protocollo coprire tutti i protocolli (consigliato: pipeline DEseq210). Uno script standard basato sul flusso di lavoro DEseq2 da noi sviluppato è disponibile su GitHub (https://github.com/jsschrepping/RNA-DESeq2).
- Trasforma i dati di sequenziamento grezzi in formato FASTQ e demultiplexa in base agli indici del campione con lo strumento Bcl2Fastq2. Eseguire il demultiplexing con le impostazioni predefinite. Per istruzioni dettagliate su Bcl2Fastq2, vedere il manuale di riferimento.
Risultati
Seguendo il protocollo riportato, le PBMC umane sono state seminate, trattate con diversi farmaci immunomodulatori e, dopo diversi tempi di incubazione, raccolte per l'analisi trascrittomica di massa utilizzando il protocollo di sequenziamento (Figura 1).
Le concentrazioni ideali del farmaco e i tempi di incubazione per i composti in esame dovrebbero essere identificati a monte di questo protocollo con l'aiuto di strategie sperimentali complementari e sulla base d...
Discussione
La scoperta e lo sviluppo di farmaci possono trarre grandi benefici dalla visione olistica dei processi cellulari che la trascrittomica di massa può fornire. Tuttavia, questo approccio è spesso limitato dall'alto costo dell'esperimento con il protocollo standard di RNA-seq di massa, che ne impedisce l'applicazione in ambienti accademici e dal suo potenziale di scalabilità industriale.
Le fasi più critiche del protocollo sono lo scongelamento delle cellule e le fasi iniziali della preparazi...
Divulgazioni
Gli autori dichiarano di non avere interessi contrastanti.
Riconoscimenti
J.L.S. è sostenuta dalla Fondazione tedesca per la ricerca (DFG) nell'ambito della strategia di eccellenza tedesca (EXC2151-390873048), nonché nell'ambito dello SCHU 950/8-1; GRK 2168, TP11; CRC SFB 1454 Metaflammation, IRTG GRK 2168, WGGC INST 216/981-1, CCU INST 217/988-1, il progetto di eccellenza finanziato dal BMBF Diet-Body-Brain (DietBB); e il progetto europeo SYSCID con il numero di sovvenzione 733100. M.B. è supportato da DFG (IRTG2168-272482170, SFB1454-432325352). L.B. è sostenuta da DFG (ImmuDiet BO 6228/2-1 - Progetto numero 513977171) e dalla Strategia di Eccellenza della Germania (EXC2151-390873048). Immagini create con BioRender.com.
Materiali
| Name | Company | Catalog Number | Comments |
| 50 mL conical tube | fisher scientific | 10203001 | |
| Adhesive PCR Plate Seals | Thermo Fisher Scientific | AB0558 | |
| Amplicon Tagment Mix (ATM) | Illumina | FC-131-1096 | Nextera XT DNA Library Prep Kit (96 samples) |
| AMPure XP beads | Beckman Coulter | A 63881 | |
| Betaine | Sigma-Aldrich | 61962 | |
| Cell culture grade 96-well plates | Thermo Fisher Scientific | 260860 | |
| Cell culture vacuum pump (VACUSAFE) | Integra Bioscience | 158300 | |
| Deoxynucleotide triphosphates (dNTPs) mix 10 mM each | Fermentas | R0192 | |
| DMSO | Sigma-Aldrich | 276855 | |
| DTT (100 mM) | Invitrogen | 18064-014 | |
| EDTA | Sigma-Aldrich | 798681 | for adherent cells |
| Ethanol | Sigma-Aldrich | 51976 | |
| Fetal Bovine Serum | Thermo Fisher Scientific | 26140079 | |
| Filter tips (10 µL) | Gilson | F171203 | |
| Filter tips (100 µL) | Gilson | F171403 | |
| Filter tips (20 µL) | Gilson | F171303 | |
| Filter tips (200 µL) | Gilson | F171503 | |
| Guanidine Hydrochloride | Sigma-Aldrich | G3272 | |
| ISPCR primer (10 µM) | Biomers.net GmbH | SP10006 | 5′-AAGCAGTGGTATCAACGCAGAG T-3′ |
| KAPA HiFi HotStart ReadyMix (2X) | KAPA Biosystems | KK2601 | |
| Magnesium chloride (MgCl2) | Sigma-Aldrich | M8266 | |
| Magnetic stand 96 | Ambion | AM10027 | |
| Neutralize Tagment (NT) Buffer | Illumina | FC-131-1096 | Nextera XT DNA Library Prep Kit (96 samples), alternatively 0.2 % SDS |
| Nextera-compatible indexing primer | Illumina | ||
| Nuclease-free water | Invitrogen | 10977049 | |
| PBS | Thermo Fisher Scientific | AM9624 | |
| PCR 96-well plates | Thermo Fisher Scientific | AB0600 | |
| PCR plate sealer | Thermo Fisher Scientific | HSF0031 | |
| Penicillin / Streptomycin | Thermo Fisher Scientific | 15070063 | |
| Qubit 4 fluorometer | Invitrogen | 15723679 | |
| Recombinant RNase inhibitor (40 U/ul) | TAKARA | 2313A | |
| RPMI-1640 cell culture medium | Gibco | 61870036 | If not working with PBMCs, adjust to cell type |
| SMART dT30VN primer | Sigma-Aldrich | 5' Bio-AAGCAGTGGTATCAACGCAGAG TACT30VN-3 | |
| Standard lab equipment | various | various | e.g. centrifuge, ice machine, ice bucket, distilled water, water bath |
| SuperScript II Reverse Transcriptase (SSRT II) | Thermo Fisher Scientific | 18064-014 | |
| SuperScript II Reverse Transcriptase (SSRT II) buffer (5x) | Thermo Fisher Scientific | 18064-014 | |
| Tagment DNA Buffer (TD) | Illumina | FC-131-1096 | Nextera XT DNA Library Prep Kit (96 samples) |
| TapeStation system 4200 | Agilent | G2991BA | |
| Thermocycler (S1000) | Bio-Rad | 1852148 | |
| TSO-LNA (100 uM) | Eurogentec | 5' Biotin AAGCAGTGGTATCAACGCAGAG TACAT(G)(G){G | |
| Vortex-Genie 2 Mixer | Sigma-Aldrich | Z258415 |
Riferimenti
- Hughes, J. P., Rees, S., Kalindjian, S. B., Philpott, K. L. Principles of early drug discovery. Br J Pharmacol. 162 (6), 1239-1249 (2011).
- Yang, X., et al. High-throughput transcriptome profiling in drug and biomarker discovery. Front Genet. 11, 19 (2020).
- Bonaguro, L., et al. A guide to systems-level immunomics. Nat Immunol. 23 (10), 1412-1423 (2022).
- Carraro, C., et al. Decoding mechanism of action and sensitivity to drug candidates from integrated transcriptome and chromatin state. ELife. 11, 78012 (2022).
- Picelli, S., et al. Smart-seq2 for sensitive full-length transcriptome profiling in single cells. Nat Methods. 10 (11), 1096-1098 (2013).
- Picelli, S., et al. Full-length RNA-seq from single cells using Smart-seq2. Nat Protoc. 9 (1), 171-181 (2014).
- De Domenico, E., et al. Optimized workflow for single-cell transcriptomics on infectious diseases including COVID-19. STAR Protoc. 1 (3), 100233 (2020).
- Dobin, A., et al. ultrafast universal RNA-seq aligner. Bioinformatics. 29 (1), 15-21 (2013).
- Patro, R., et al. Salmon provides fast and bias-aware quantification of transcript expression. Nat Methods. 14 (4), 417-419 (2017).
- Love, M. I., Huber, W., Anders, S. Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2. Genome Biol. 15 (12), 550 (2014).
- Frankish, A., et al. GENCODE reference annotation for the human and mouse genomes. Nucleic Acids Res. 47, D766-D773 (2019).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon