A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
בדיקת תרופות מבוססת תמלול חסכונית
In This Article
Summary
פרוטוקול זה מתאר זרימת עבודה מתרביות תאים ex vivo או in vitro לעיבוד מקדים של נתוני תמלול לסינון תרופות מבוסס תמלול חסכוני.
Abstract
תמלול מאפשר לקבל תובנות מקיפות על תוכניות סלולריות ותגובותיהן להפרעות. למרות ירידה משמעותית בעלויות הייצור והריצוף של ספריות בעשור האחרון, יישום טכנולוגיות אלה בהיקף הדרוש לסינון תרופות נותר יקר מאוד, וחוסם את הפוטנציאל העצום של שיטות אלה. המחקר שלנו מציג מערכת חסכונית לבדיקת תרופות מבוססת תמלול, המשלבת תרביות הפרעה ממוזערות עם מיני-תמלול בתפזורת. פרוטוקול המיני-צובר הממוטב מספק אותות ביולוגיים אינפורמטיביים בעומק ריצוף חסכוני, ומאפשר סינון נרחב של תרופות ידועות ומולקולות חדשות. בהתאם לזמן הטיפול והדגירה שנבחר, פרוטוקול זה יביא לספריות רצף תוך כיומיים. בשל מספר נקודות עצירה בפרוטוקול זה, הכנת הספרייה, כמו גם הרצף, יכולים להתבצע ללא תלות בזמן. עיבוד בו זמנית מספר גבוה של דגימות אפשרי; מדידה של עד 384 דגימות נבדקה ללא אובדן איכות הנתונים. כמו כן, לא ידועות מגבלות על מספר המצבים ו/או התרופות, למרות שלוקחים בחשבון את השונות בזמני הדגירה האופטימליים של התרופה.
Introduction
פיתוח תרופות חדשות הוא תהליך מורכב וגוזל זמן הכולל זיהוי תרופות פוטנציאליות ומטרותיהן, אופטימיזציה וסינתזה של תרופות מועמדות ובדיקת יעילותן ובטיחותן בניסויים פרה-קליניים וקליניים1. שיטות מסורתיות לסינון תרופות, כלומר, הערכה שיטתית של ספריות של תרכובות מועמדות למטרות טיפוליות, כוללות שימוש במודלים של בעלי חיים או בדיקות מבוססות תאים כדי לבחון את ההשפעות על מטרות או מסלולים ספציפיים. בעוד ששיטות אלה הצליחו לזהות מועמדים לתרופה, לעתים קרובות הן לא סיפקו תובנות מספקות לגבי המנגנונים המולקולריים המורכבים העומדים בבסיס יעילות התרופה וגם רעילות ומנגנונים של תופעות לוואי אפשריות.
הערכת מצבי שעתוק כלל גנומיים מציגה גישה רבת עוצמה להתגברות על המגבלות הנוכחיות בבדיקת תרופות, שכן היא מאפשרת הערכה מקיפה של ביטוי גנים בתגובה לטיפולים תרופתיים2. על ידי מדידת תעתיקי RNA באופן כלל גנומי המתבטא בזמן נתון, שעתוק שואפת לספק מבט הוליסטי על שינויי השעתוק המתרחשים בתגובה לתרופות, כולל שינויים בדפוסי ביטוי גנים, שחבור חלופי וביטוי RNA לא מקודד3. ניתן להשתמש במידע זה כדי לקבוע מטרות לתרופות, לחזות יעילות ורעילות של תרופות ולמטב את מינון התרופות ואת משטרי הטיפול.
אחד היתרונות המרכזיים של שילוב תמלול עם סינון סמים בלתי משוחד הוא הפוטנציאל לזהות מטרות סמים חדשות שלא נלקחו בחשבון בעבר. גישות קונבנציונליות לסינון תרופות מתמקדות לעתים קרובות במולקולות מטרה או מסלולים מבוססים, מעכבים את זיהוי המטרות החדשות ועלולים לגרום לתרופות עם תופעות לוואי בלתי צפויות ויעילות מוגבלת. Transcriptomics יכול להתגבר על מגבלות אלה על ידי מתן תובנות לגבי השינויים המולקולריים המתרחשים בתגובה לטיפול תרופתי, חשיפת מטרות פוטנציאליות או מסלולים שאולי לא נחשבו בעבר2.
בנוסף לזיהוי מטרות תרופות חדשות, ניתן להשתמש בתמלול גם כדי לחזות יעילות ורעילות של תרופות. על ידי ניתוח דפוסי ביטוי הגנים הקשורים לתגובות לתרופות, ניתן לפתח סמנים ביולוגיים שניתן להשתמש בהם כדי לחזות את תגובת המטופל לתרופה מסוימת או למשטר טיפול מסוים. זה יכול גם לעזור לייעל את מינון התרופה ולהפחית את הסיכון לתופעות לוואי שליליות4.
למרות היתרונות הפוטנציאליים שלה, עלות התמלול נותרה מחסום משמעותי ליישומה הנרחב בסינון תרופות. ניתוח תמלול דורש ציוד מיוחד, מומחיות טכנית וניתוח נתונים, מה שיכול להקשות על צוותי מחקר קטנים יותר או ארגונים עם מימון מוגבל להשתמש בתמלול בסינון תרופות. עם זאת, עלות התמלול יורדת בהתמדה, מה שהופך אותה לנגישה יותר לקהילות המחקר. בנוסף, התקדמות הטכנולוגיה ושיטות ניתוח הנתונים הפכו את התמלול ליעיל וחסכוני יותר, והגדילו עוד יותר את נגישותו2.
בפרוטוקול זה, אנו מתארים מערכת רב-ממדית וחקרנית לבדיקת תרופות מבוססת תמלול, המשלבת תרביות הפרעה ממוזערות עם ניתוח תמלול בתפזורתקטנה 5,6. באמצעות פרוטוקול זה, ניתן להפחית את העלות לדגימה ל -1/6 מהעלות הנוכחית של פתרונות מסחריים לריצוף mRNA באורך מלא. הפרוטוקול דורש רק ציוד מעבדה סטנדרטי, כאשר היוצא מן הכלל היחיד הוא השימוש בטכנולוגיות ריצוף בקריאה קצרה, שניתן לבצע במיקור חוץ אם מכשירי ריצוף אינם זמינים בתוך החברה. פרוטוקול המיני-צובר הממוטב מספק אותות ביולוגיים עשירים במידע בעומק ריצוף חסכוני, ומאפשר סריקה נרחבת של תרופות ידועות ומולקולות חדשות.
מטרת הניסוי היא לסנן פעילות תרופתית על PBMCs בהקשרים ביולוגיים שונים. פרוטוקול זה יכול להיות מיושם על כל שאלה ביולוגית שבה מספר תרופות צריכות להיבדק עם קריאה תמלולית, נותן מבט רחב על התעתיק של ההשפעה התאית של הטיפול.
Protocol
פרוטוקול זה עוקב אחר הנחיות ועדות האתיקה המקומיות של אוניברסיטת בון.
1. הכנת חוצצים, פתרונות וציוד
- הכן את הפתרונות ואסוף את החומרים המתוארים בטבלת החומרים.
- חממו את אמבט המים ל-37°C וחממו את מדיום הגידול המלא (RPMI-1640 + 10% סרום עגל עוברי (FCS) + 1% פניצילין/סטרפטומיצין).
- לקצירת תאים, השתמשו במי מלח חוצצי פוספט קר כקרח (PBS).
הערה: שמור על סביבה נקייה בעת עבודה עם תאים כדי לשמור על סטריליות.
2. טיפול בתאים
הערה: פרוטוקול מפורט לשימור בהקפאה של תאי דם חד-גרעיניים היקפיים (PBMC) מדם אנושי ניתן למצואב-7.
- הפשרת תאים וספירה
- הסר את cryovials מחנקן נוזלי והפשיר אותם באמבט מים ב 37 ° C במשך 2 - 3 דקות תוך היפוך עדין.
- מעבירים את התאים המופשרים לתוך צינור חרוטי 50 מ"ל.
- לשטוף את cryovial עם 1 מ"ל חם להשלים מדיום הצמיחה ולהוסיף פתרון זה טיפה לתאים בצינור (1 שלב דילולרחוב ).
- חזור על הדילול ביחס של 1:1 עד לקבלת נפח של 32 מ"ל (סה"כ 5 דילולים עם 1, 2, 4, 8 ו-16 מ"ל בהתאמה של מדיום גידול מלא חם). הוסף את טיפת המדיום כדי להפחית את הפרעות התא תוך כדי התסיסה קלה של הצינור החרוטי.
- צנטריפוגו את מתלה התא למשך 5 דקות (300 x גרם, 20°C) והוציאו את הסופרנאטנט על ידי הטייה עדינה של הצינור החרוטי בתנועה שוטפת אחת.
- להשהות מחדש את התאים ב 3 מ"ל של מדיום צמיחה מלא חם ולהמשיך עם ספירת תאים.
- לצורך ספירה, ערבבו 10 μL של תרחיף תאים עם Trypan Blue (דילול 1:2 עד 1:10 בהתאם לצפיפות תרחיף התא) כדי להבחין בין תאים חיים לתאים מתים בזמן הספירה. תאים מתים או פגומים ייראו כחולים עקב ספיגת הצבע, בעוד תאים חיוניים אינם מוכתמים.
הערה: Trypan Blue הוא מעט ציטוטוקסי, תאים מוכתמים לא צריך להיות מאוחסן במשך יותר מ 5 דקות. - ספור את התאים באמצעות מונה תאים אוטומטי או תא ספירה באמצעות 10 μL של תמיסת תאים מוכתמים.
- בעת שימוש בתא התא המשופר על ידי נויבאואר, ספור את כל התאים בתוך ארבעת הריבועים הגדולים הממוקמים בפינות והשתמש במשוואה הבאה כדי לחשב את ריכוז תרחיף התא.
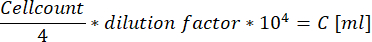
- בעת שימוש בתא התא המשופר על ידי נויבאואר, ספור את כל התאים בתוך ארבעת הריבועים הגדולים הממוקמים בפינות והשתמש במשוואה הבאה כדי לחשב את ריכוז תרחיף התא.
- לדלל את התאים לריכוז סופי של 1 x 106 תאים / מ"ל עם מצע צמיחה מלא חם. צנטריפוגה רק אם הנפח הנדרש נמוך מ -3 מ"ל ולהשעות מחדש את גלולת התא לנפח הנכון.
- זריעת תאים וטיפול בהם
- זרע 100 μL של תרחיף תאים לכל באר בצלחת תרבית תאים 96 באר (1 x 105 תאים / באר).
הערה: מספר התא לזריעה עבר אופטימיזציה לתרביות PBMC. בעוד שריכוזי תאים נמוכים לא יניבו כמות מספקת של RNA לריצוף, מספר מוגזם של תאים יגדיל את הסיכון לליזה תת-אופטימלית של התא ולעיכוב תגובת השעתוק ההפוך. - הכינו דילול תרופות בריכוז גדול פי שניים מזה המשמש לטיפול (פי 2). בחר את הממס על פי מסיסות של התרכובת, רצוי מדיום צמיחה מלא.
הערה: PBS או dimethyl sulfoxide (DMSO) ניתן להשתמש אם לא ניתן להגיע למסיסות מספקת אחרת. הריכוז המרבי של DMSO בנפח הדגירה המתקבל לא יעלה על 0.5% כדי למנוע ציטוטוקסיות הנגרמת על ידי הממס. - מוסיפים 100 μL של דילול התרופה 2x (ריכוז סופי בבאר 1X) ודגרים ב 37 ° C למשך הזמן שהוגדר.
הערה: יש לבצע חקירות משלימות על מנת לקבוע את זמן הדגירה האופטימלי של התרופה ולשלול מנגנונים פוטנציאליים של ציטוטוקסיות.
- זרע 100 μL של תרחיף תאים לכל באר בצלחת תרבית תאים 96 באר (1 x 105 תאים / באר).
- קצירת תאים וליזיס
- צנטריפוגה את צלחת תרבית התא במשך 10 דקות (300 x גרם, 20 ° C). הסירו בעדינות את הסופרנאטנט בעזרת משאבת ואקום תוך שמירה על הצלחת בזווית (30°-45°) תוך כיוון הקצה לפינה הנמוכה של הבאר כדי להסיר כמה שפחות תאים.
- שטפו את התאים על ידי הוספת 200 מיקרוליטר של PBS קר כקרח לכל באר וצנטריפוגו את הצלחת למשך 5 דקות (300 x גרם, 4 מעלות צלזיוס). הסר את supernatant עם משאבת ואקום, שוב, שמירה על הצלחת בזווית חדה כדי להסיר PBS ככל האפשר.
- הכן את מאגר הליזיס כמתואר בטבלה 1 עבור מספר התגובות (rxn) הדרושות, תוך הוספת עודף של 10%.
- הוסף 15 μL של חיץ ליזיס לכל באר ואטם את הצלחת עם סרט איטום דבק כדי להגן על הדגימות מפני זיהום. מערבבים את הצלחת לפני הצנטריפוגה למשך דקה אחת (1000 x גרם, 4 מעלות צלזיוס) ודגרים במשך 5 דקות על קרח.
- לאסוף 6 μL של תמיסת תאים lysed בצלחת PCR ולהקפיא את התא lysate ב -80 ° C כדי להבטיח ליזה התא אופטימלי. הקפידו להימנע ממחזורי הקפאה מרובים של הצלחות.
הערה: נקודת עצירה: אם ייצור cDNA אינו מתבצע לאחר מכן, התא ליזט יכול להיות מאוחסן ב -80 ° C עד מספר חודשים ללא ירידה ניכרת באיכות RNA.
3. הכנת הספרייה לרצף
- תגובת שעתוק לאחור (RT)
הערה: בעת עבודה עם RNA, הניחו את כל הדגימות על קרח והקפידו להשתמש בציוד ללא נוקלאז (כלי פלסטיק סטריליים חד פעמיים) ובמים.- הכינו את תמהיל תגובות RT כמתואר בטבלה 2 למספר התגובות הדרושות, והוסיפו 10% עודף. מערבלים לרגע את התערובת ותוך זמן קצר מסובבים אותה. שומרים את התערובת על קרח עד לשימוש.
- הפשירו את הליזט התא בטמפרטורת החדר (RT) וסובבו אותו לזמן קצר כלפי מטה כדי לאסוף את כל הליזט התא בתחתית הצלחת.
- בצע דנטורציה mRNA על thermocycler לפי טבלה 3.
- הוציאו את הצלחת מהתרמוסייקלר וסובבו אותה תוך זמן קצר כדי לאסוף עיבוי פוטנציאלי. הוסיפו 6 μL של תערובת תגובת RT לכל באר לקבלת נפח סופי של 12 μL. אטמו את הצלחת כדי להגן על הצלחת ולמנוע אידוי, מערבולות וסובבו אותה.
- הניחו את הצלחת על התרמוסייקלר והתחילו את תוכנית תגובת RT לפי טבלה 3.
- טרום הגברה
- הפשירו את ה-DNA פולימראז באיכות גבוהה ואת פריימר ה-PCR (ISPCR) באתרו בטמפרטורת החדר.
- הכינו את תמהיל ההגברה מראש לפי טבלה 4 למספר התגובות הדרושות, והוסיפו 10% עודף. מערבלים לרגע את התערובת ומסובבים אותה.
הערה: תערובת האנזימים יציבה בטמפרטורת החדר למשך שעות. - סובבו את צלחת ה-PCR, הוסיפו 15 מיקרוליטר של תערובת קדם-הגברה לכל באר ואטמו את הצלחת.
- הניחו אותו על התרמוסייקלר והתחילו את תגובת קדם-ההגברה לפי טבלה 5.
- התאם את מספר המחזורים עקב תכולת RNA. התחל עם מספר נמוך יותר והגדל אם תפוקת cDNA אינה מספקת.
הערה: מניסיוננו, יש לקבוע מספר אופטימלי של מחזורים לכל סוג תא, מצב הניסוי והטיפול לא ישפיעו על מספר המחזורים האופטימלי. לכן, אנו ממליצים לקבוע את מספר המחזורים האופטימלי באופן ניסיוני בעת קביעת פרוטוקול זה עבור סוג תא חדש. באופן כללי, תאים ראשוניים ידרשו מספר רב יותר של מחזורים בהשוואה לשורות תאים. נקודת עצירה: ניתן לאחסן את מוצר ההגברה מראש ב- 20 מעלות צלזיוס.
- התאם את מספר המחזורים עקב תכולת RNA. התחל עם מספר נמוך יותר והגדל אם תפוקת cDNA אינה מספקת.
- ניקוי ובקרת איכות cDNA (QC)
הערה: ניקוי הדגימה יכול להתבצע ברצף עבור כל צלחת מכיוון שהדגימות יציבות למדי בשלב זה. הגדלת מספר הדגימות תוביל למשך זמן ארוך יותר של הפרוטוקול אך לא תגביל את מספר הדגימות שניתן לעבד בו זמנית.- לפני שמתחילים, הביאו את חרוזי הטיהור המגנטי לטמפרטורת החדר ומערבלים במהירות גבוהה למשך דקה אחת כדי להשהות מחדש את החרוזים במלואם.
- הוסיפו חרוזי טיהור מגנטיים 0.8x v/v (20 μL) לכל באר ודגרו בטמפרטורת החדר למשך 5 דקות.
- הניחו את לוחית ה-PCR על מדף מגנטי למשך 5 דקות עד להפרדה מלאה בין החרוזים. הסר את supernatant בזהירות.
- שומרים את הצלחת על המדף המגנטי, מוסיפים בעדינות 100 μL של 80% אתנול טרי שהוכן כדי לשטוף את החרוזים ולדגור במשך 30 שניות. השתמש פיפטה נפח קטן כדי להסיר את supernatant. חזרו על הפעולה לשלב שטיפה נוסף. הקפד להסיר אתנול רב ככל האפשר.
- יבשו באוויר את החרוזים על המדף המגנטי בטמפרטורת החדר למשך עד 5 דקות עד שהאתנול יתאדה במלואו והחרוזים כבר לא ייראו מבריקים.
- הסר את הצלחת מהמדף המגנטי, השהה מחדש את החרוזים ב -20 מיקרוליטר מים נטולי נוקלאז ודגור למשך 2 דקות.
- הניחו את הצלחת שוב על המדף המגנטי עד להפרדת החרוזים.
- לשחזר את eluate בצלחת PCR חדשה עבור cDNA QC ותיוג.
- בצע בדיקת TapeStation או FragmentAnalyser כדי להעריך את התפלגות הגודל והריכוז של ספריית cDNA (מומלץ: בדיקת TapeStation D5000). לקבלת פרטים, עיין בהוראות היצרן. יש לצפות ליבול טיפוסי של 20 ננוגרם.
- תיוג עם ערכה
הערה: ניתן להשתמש בפרוטוקולי תיוג אחרים אם כבר הוקמו.- לדלל את cDNA לריכוז סופי של 150 - 300 pg/μL באמצעות מים נטולי נוקלאז.
- תכנת מראש את התרמוסייקלר לפי טבלה 6 כדי להבטיח התחלה מיידית של תגובת התיוג לאחר הוספת cDNA לתערובת האנזימים.
- הכינו את תמהיל התיוג לפי טבלה 7 למספר התגובות הדרושות, והוסיפו 10% חריגה.
- יש להוציא 3 μL של תערובת התיוג לכל תגובה על צלחת PCR חדשה ולהוסיף 1 μL של cDNA לכל תגובה.
- התחל את תגובת התיוג מיד לאחר הוספת cDNA.
- נטרל את התגובה על ידי הוספת 1 μL של מאגר תג נטרול (NT; לחלופין, ניתן להשתמש ב- 0.2% נתרן דודציל סולפט (SDS).
- PCR העשרה
- הכינו את תמהיל ה-PCR להעשרה טבלה 7 למספר התגובות, והוסיפו 10% עודף. (מומלץ: ערכת Nextera UDI למניעת דילוג אינדקס על תאי זרימה בתבנית (למשל, Illumina NovaSeq 6000)).
- הוסף 9 μL של תערובת PCR העשרה לכל תגובה עבור נפח כולל של 14 μL.
- הפעל את תוכנית ה-PCR להעשרה לפי טבלה 8.
הערה: עבור PCR העשרה, 16 מחזורים הם סטנדרטיים. אם איכות cDNA ירודה, ניתן להוסיף מחזורים נוספים.
- ניקוי ובקרת איכות
- לפני שמתחילים, הביאו חרוזי טיהור מגנטיים לטמפרטורת החדר וערברו במהירות גבוהה למשך דקה אחת כדי להשהות מחדש את החרוזים במלואם.
- מוסיפים 1.0x v/v (14 μL) חרוזי טיהור מגנטיים ודגרים בטמפרטורת החדר למשך 5 דקות.
- הניחו את הצלחת על מדף מגנטי והמתינו 5 דקות עד שהחרוזים מופרדים לחלוטין. הסר את supernatant בזהירות.
- שומרים את הצלחת על המדף המגנטי, מוסיפים בעדינות 100 μL של 80% אתנול טרי שהוכן כדי לשטוף את החרוזים ולדגור במשך 30 שניות. השתמש פיפטה נפח קטן כדי להסיר את supernatant. חזרו על הפעולה לשלב שטיפה נוסף. הקפד להסיר אתנול רב ככל האפשר.
- יבשו את החרוזים באוויר בטמפרטורת החדר למשך 3 דקות או עד שהם כבר לא נראים מבריקים.
- הסר את הצלחת מהמדף המגנטי, השהה מחדש את החרוזים ב -20 מיקרוליטר מים נטולי נוקלאז ודגור למשך 2 דקות.
- הניחו את הצלחת שוב על המדף המגנטי עד שהחרוזים ייפרדו ושחזרו את הפליט בצלחת PCR חדשה עבור בקרת איכות בספרייה.
- בצע בדיקת TapeStation או Fragment Analyzer כדי להעריך את התפלגות הגודל והריכוז של ספריית cDNA (מומלץ: בדיקת רגישות גבוהה TapeStation D1000). לקבלת פרטים, עיין בהוראות היצרן. בממוצע, יש לצפות ליבול של 10 ננוגרם.
הערה: נקודת עצירה: ניתן לאחסן את מוצר ה- PCR ב- 20 °C.
4. רצף ועיבוד מראש של נתונים
- רצף
הערה: ההנחיה הבאה תחול על כל מכשירי Illumina לריצוף בקריאה קצרה. אם המכשור אינו זמין, ניתן לבצע את הריצוף על ידי מתקן ריצוף חיצוני. ניתן להשתמש גם בגישות ריצוף אחרות. לשם הפשטות, בחרנו לדווח על טכנולוגיית הריצוף הנפוצה ביותר בלבד.
הערה: השלבים הבאים הקשורים לשימוש בתוכנה מתארים את ההליך ברצף Illumina NovaSeq6000.- מאגר ספריות אינדקס ייחודיות ביחס שווה משקל בהתאם לתוצאות שהתקבלו בשלב 3.6.8.
- מדוד את ריכוז המאגר הסופי עם בדיקת רגישות גבוהה כדי לחשב את טוחנות הדגימה באופן הבא:
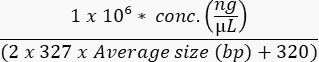
- טען את תא הזרימה בהתאם למפרט המכשיר ולאופטימיזציה ניסיונית. דוגמאות לריכוזי העמסה של מכשירים נפוצים מוצגות בטבלה 9.
- על ידי נגיעה במסך בחר רצף כדי להתחיל את הגדרת ההפעלה.
- בצע את ההוראות שעל המסך וטען תא זרימה, מחסנית רצף אחר סינתזה, מחסנית אשכולות, מחסנית מאגר וודא שמיכלי הפסולת ריקים.
- לאחר שכל הריאגנטים זוהו על ידי המכשיר, לחץ על הפעל התקנה. הגדר כאן את שם ההפעלה ואת תיקיית הפלט לאחסון הנתונים.
- הגדר את פרטי הרצף כזוג עם שתי קריאות של 51 bp. שתי קריאות אינדקס מסודרות גם הן ברצף של 8 bp כל אחת.
- לחץ על סקירה ולאחר בדיקה אם כל פרטי הרצף נכונים לחץ על התחל הפעלה.
הערה: עומק הרצף המומלץ הוא 5 x 106 קריאות לדגימה, עם מינימום 1 x 106 קריאות לדגימה.
- עיבוד מקדים של נתונים
- המר נתוני רצף גולמיים לפורמט FASTQ ו- demultiplex בהתאם לאינדקסים לדוגמה באמצעות הכלי Bcl2Fastq2. בצע demultiplexing עם הגדרות ברירת המחדל. לקבלת הוראות מפורטות על Bcl2Fastq2, עיין במדריך העיון.
הערה: המרת FASTQ וריבוי מבוצעים בדרך כלל על ידי מתקן הריצוף אם הרצף אינו מבוצע בתוך הבית. מתקני ריצוף יספקו בדרך כלל FASTQ מרובה לעיבוד נוסף. - קיימות מספר אפשרויות ליישור נתונים וכימות שפע של קריאות רצף (מומלץ: צינור RNA-seq של ליבת nf (https://nf-co.re/rnaseq)). הצינור מספק מספר אפשרויות, השתמש בהגדרת ברירת המחדל עם STAR8 מיושר וסלמון9 כדי לכמת את שפע התמלול.
- הערה: לניתוח ביואינפורמטיקה נוסף, קיימות מספר שיטות. זה לא במסגרת פרוטוקול זה לכסות את כל (מומלץ: צינור DEseq210). סקריפט סטנדרטי המבוסס על זרימת העבודה DEseq2 שפותחה על ידינו ניתן למצוא ב- GitHub (https://github.com/jsschrepping/RNA-DESeq2).
- המר נתוני רצף גולמיים לפורמט FASTQ ו- demultiplex בהתאם לאינדקסים לדוגמה באמצעות הכלי Bcl2Fastq2. בצע demultiplexing עם הגדרות ברירת המחדל. לקבלת הוראות מפורטות על Bcl2Fastq2, עיין במדריך העיון.
תוצאות
בעקבות הפרוטוקול המדווח, PBMCs אנושיים נזרעו, טופלו בתרופות אימונומודולטוריות שונות, ולאחר זמני דגירה שונים, נקטפו לצורך ניתוח שעתוק בתפזורת באמצעות פרוטוקול הריצוף (איור 1).
יש לזהות ריכוזי תרופות אידיאליים וזמני דגירה של תרכובות בדיקה במעלה הזרם פרוטוקול זה...
Discussion
גילוי תרופות ופיתוח תרופות יכולים להפיק תועלת רבה מהראייה ההוליסטית של תהליכים תאיים שתעתיק בתפזורת יכול לספק. עם זאת, גישה זו מוגבלת לעתים קרובות על ידי העלות הגבוהה של הניסוי עם פרוטוקול RNA-seq סטנדרטי בתפזורת, האוסר על יישומו במסגרות אקדמיות, כמו גם הפוטנציאל שלו למדרגיות תעשייתית.
Disclosures
המחברים מצהירים כי אין אינטרסים מתחרים.
Acknowledgements
J.L.S. נתמכת על ידי קרן המחקר הגרמנית (DFG) תחת אסטרטגיית המצוינות של גרמניה (EXC2151-390873048), כמו גם תחת SCHU 950/8-1; GRK 2168, TP11; CRC SFB 1454 Metaflammation, IRTG GRK 2168, WGGC INST 216/981-1, CCU INST 217/988-1, פרויקט מצוינות במימון BMBF דיאטה-גוף-מוח (DietBB); ופרויקט האיחוד האירופי SYSCID תחת מענק מספר 733100. מ.ב. נתמך על ידי DFG (IRTG2168-272482170, SFB1454-432325352). L.B. נתמכת על ידי DFG (ImmuDiet BO 6228/2-1 - פרויקט מספר 513977171) ואסטרטגיית המצוינות של גרמניה (EXC2151-390873048). תמונות שנוצרו באמצעות BioRender.com.
Materials
| Name | Company | Catalog Number | Comments |
| 50 mL conical tube | fisher scientific | 10203001 | |
| Adhesive PCR Plate Seals | Thermo Fisher Scientific | AB0558 | |
| Amplicon Tagment Mix (ATM) | Illumina | FC-131-1096 | Nextera XT DNA Library Prep Kit (96 samples) |
| AMPure XP beads | Beckman Coulter | A 63881 | |
| Betaine | Sigma-Aldrich | 61962 | |
| Cell culture grade 96-well plates | Thermo Fisher Scientific | 260860 | |
| Cell culture vacuum pump (VACUSAFE) | Integra Bioscience | 158300 | |
| Deoxynucleotide triphosphates (dNTPs) mix 10 mM each | Fermentas | R0192 | |
| DMSO | Sigma-Aldrich | 276855 | |
| DTT (100 mM) | Invitrogen | 18064-014 | |
| EDTA | Sigma-Aldrich | 798681 | for adherent cells |
| Ethanol | Sigma-Aldrich | 51976 | |
| Fetal Bovine Serum | Thermo Fisher Scientific | 26140079 | |
| Filter tips (10 µL) | Gilson | F171203 | |
| Filter tips (100 µL) | Gilson | F171403 | |
| Filter tips (20 µL) | Gilson | F171303 | |
| Filter tips (200 µL) | Gilson | F171503 | |
| Guanidine Hydrochloride | Sigma-Aldrich | G3272 | |
| ISPCR primer (10 µM) | Biomers.net GmbH | SP10006 | 5′-AAGCAGTGGTATCAACGCAGAG T-3′ |
| KAPA HiFi HotStart ReadyMix (2X) | KAPA Biosystems | KK2601 | |
| Magnesium chloride (MgCl2) | Sigma-Aldrich | M8266 | |
| Magnetic stand 96 | Ambion | AM10027 | |
| Neutralize Tagment (NT) Buffer | Illumina | FC-131-1096 | Nextera XT DNA Library Prep Kit (96 samples), alternatively 0.2 % SDS |
| Nextera-compatible indexing primer | Illumina | ||
| Nuclease-free water | Invitrogen | 10977049 | |
| PBS | Thermo Fisher Scientific | AM9624 | |
| PCR 96-well plates | Thermo Fisher Scientific | AB0600 | |
| PCR plate sealer | Thermo Fisher Scientific | HSF0031 | |
| Penicillin / Streptomycin | Thermo Fisher Scientific | 15070063 | |
| Qubit 4 fluorometer | Invitrogen | 15723679 | |
| Recombinant RNase inhibitor (40 U/ul) | TAKARA | 2313A | |
| RPMI-1640 cell culture medium | Gibco | 61870036 | If not working with PBMCs, adjust to cell type |
| SMART dT30VN primer | Sigma-Aldrich | 5' Bio-AAGCAGTGGTATCAACGCAGAG TACT30VN-3 | |
| Standard lab equipment | various | various | e.g. centrifuge, ice machine, ice bucket, distilled water, water bath |
| SuperScript II Reverse Transcriptase (SSRT II) | Thermo Fisher Scientific | 18064-014 | |
| SuperScript II Reverse Transcriptase (SSRT II) buffer (5x) | Thermo Fisher Scientific | 18064-014 | |
| Tagment DNA Buffer (TD) | Illumina | FC-131-1096 | Nextera XT DNA Library Prep Kit (96 samples) |
| TapeStation system 4200 | Agilent | G2991BA | |
| Thermocycler (S1000) | Bio-Rad | 1852148 | |
| TSO-LNA (100 uM) | Eurogentec | 5' Biotin AAGCAGTGGTATCAACGCAGAG TACAT(G)(G){G | |
| Vortex-Genie 2 Mixer | Sigma-Aldrich | Z258415 |
References
- Hughes, J. P., Rees, S., Kalindjian, S. B., Philpott, K. L. Principles of early drug discovery. Br J Pharmacol. 162 (6), 1239-1249 (2011).
- Yang, X., et al. High-throughput transcriptome profiling in drug and biomarker discovery. Front Genet. 11, 19 (2020).
- Bonaguro, L., et al. A guide to systems-level immunomics. Nat Immunol. 23 (10), 1412-1423 (2022).
- Carraro, C., et al. Decoding mechanism of action and sensitivity to drug candidates from integrated transcriptome and chromatin state. ELife. 11, 78012 (2022).
- Picelli, S., et al. Smart-seq2 for sensitive full-length transcriptome profiling in single cells. Nat Methods. 10 (11), 1096-1098 (2013).
- Picelli, S., et al. Full-length RNA-seq from single cells using Smart-seq2. Nat Protoc. 9 (1), 171-181 (2014).
- De Domenico, E., et al. Optimized workflow for single-cell transcriptomics on infectious diseases including COVID-19. STAR Protoc. 1 (3), 100233 (2020).
- Dobin, A., et al. ultrafast universal RNA-seq aligner. Bioinformatics. 29 (1), 15-21 (2013).
- Patro, R., et al. Salmon provides fast and bias-aware quantification of transcript expression. Nat Methods. 14 (4), 417-419 (2017).
- Love, M. I., Huber, W., Anders, S. Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2. Genome Biol. 15 (12), 550 (2014).
- Frankish, A., et al. GENCODE reference annotation for the human and mouse genomes. Nucleic Acids Res. 47, D766-D773 (2019).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved