JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
비용 효율적인 전사체 기반 약물 스크리닝
요약
이 프로토콜은 비용 효율적인 전사체 기반 약물 스크리닝을 위해 체외 또는 체외 세포 배양에서 전사체 데이터 전처리까지의 워크플로우를 설명합니다.
초록
전사체학을 통해 세포 프로그램과 섭동에 대한 반응에 대한 포괄적인 통찰력을 얻을 수 있습니다. 지난 10년 동안 라이브러리 생산 및 염기서열 분석 비용이 크게 감소했음에도 불구하고 약물 스크리닝에 필요한 규모로 이러한 기술을 적용하는 것은 여전히 엄청난 비용이 들기 때문에 이러한 방법의 엄청난 잠재력을 가로막고 있습니다. 우리의 연구는 소형화된 섭동 배양과 미니 벌크 전사체학을 결합한 전사체 기반 약물 스크리닝을 위한 비용 효율적인 시스템을 제시합니다. 최적화된 mini-bulk 프로토콜은 비용 효율적인 염기서열분석 깊이에서 유익한 생물학적 신호를 제공하여 알려진 약물 및 새로운 분자에 대한 광범위한 스크리닝을 가능하게 합니다. 선택한 처리 및 배양 시간에 따라 이 프로토콜은 약 2일 이내에 시퀀싱 라이브러리를 생성합니다. 이 프로토콜 내의 여러 중지 지점으로 인해 라이브러리 준비와 염기서열분석은 시간에 관계없이 수행할 수 있습니다. 많은 수의 샘플을 동시에 처리할 수 있습니다. 최대 384개의 샘플 측정이 데이터 품질 손실 없이 테스트되었습니다. 또한 최적의 약물 배양 시간의 변동성을 고려했음에도 불구하고 조건 및/또는 약물의 수에 대한 알려진 제한은 없습니다.
서문
신약 개발은 잠재적 약물과 표적을 식별하고, 약물 후보를 최적화 및 합성하고, 전임상 및 임상 시험에서 효능과 안전성을 테스트하는 복잡하고 시간이 많이 소요되는 프로세스입니다1. 약물 스크리닝을 위한 전통적인 방법, 즉 치료 목적을 위한 후보 화합물 라이브러리의 체계적인 평가에는 특정 표적 또는 경로에 대한 효과를 테스트하기 위해 동물 모델 또는 세포 기반 분석을 사용하는 것이 포함됩니다. 이러한 방법은 약물 후보를 식별하는 데 성공적이었지만 약물 효능의 기초가 되는 복잡한 분자 메커니즘과 잠재적 부작용의 독성 및 메커니즘에 대한 충분한 통찰력을 제공하지 못하는 경우가 많았습니다.
게놈 전반의 전사 상태를 평가하는 것은 약물 치료에 대한 반응으로 유전자 발현을 포괄적으로 평가할 수 있기 때문에 현재 약물 스크리닝의 한계를 극복할 수 있는 강력한 접근 방식을 제시합니다2. 주어진 시간에 발현되는 게놈 전체 방식으로 RNA 전사체를 측정함으로써 전사체학은 유전자 발현 패턴의 변화, 선택적 스플라이싱 및 비코딩 RNA 발현 3을 포함하여 약물에 대한 반응에서 발생하는 전사 변화에 대한 전체적인 관점을 제공하는 것을 목표로 합니다3. 이 정보는 약물 표적을 결정하고, 약물 효능 및 독성을 예측하고, 약물 투여 및 치료 요법을 최적화하는 데 사용할 수 있습니다.
트랜스크립토믹스와 편견 없는 약물 스크리닝을 결합하는 주요 이점 중 하나는 이전에 고려되지 않았던 새로운 약물 표적을 식별할 수 있다는 것입니다. 기존의 약물 스크리닝 접근법은 확립된 표적 분자 또는 경로에 초점을 맞추는 경우가 많아 새로운 표적의 식별을 방해하고 잠재적으로 예상치 못한 부작용과 제한된 효과를 가진 약물을 초래할 수 있습니다. 전사체학은 약물 치료에 반응하여 발생하는 분자 변화에 대한 통찰력을 제공하고 이전에는 고려되지 않았을 수 있는 잠재적인 표적 또는 경로를 밝혀냄으로써 이러한 한계를 극복할 수 있습니다2.
새로운 약물 표적을 식별하는 것 외에도 전사체학은 약물 효능 및 독성을 예측하는 데에도 사용할 수 있습니다. 약물 반응과 관련된 유전자 발현 패턴을 분석함으로써 특정 약물 또는 치료 요법에 대한 환자의 반응을 예측하는 데 사용할 수 있는 바이오마커를 개발할 수 있습니다. 이는 또한 약물 투여를 최적화하고 부작용의 위험을 줄이는 데 도움이 될 수 있다4.
잠재적인 이점에도 불구하고 전사체학의 비용은 약물 스크리닝에 널리 적용되는 데 중요한 장벽으로 남아 있습니다. 전사체 분석에는 전문 장비, 기술 전문 지식 및 데이터 분석이 필요하므로 소규모 연구팀이나 자금이 제한된 조직에서는 약물 스크리닝에 전사체학을 활용하기가 어려울 수 있습니다. 그러나 전사체학의 비용은 꾸준히 감소하여 연구 커뮤니티에서 더 쉽게 접근할 수 있게 되었습니다. 또한 기술 및 데이터 분석 방법의 발전으로 전사체학이 보다 효율적이고 비용 효율적이며 접근성이 더욱 높아졌습니다2.
이 프로토콜에서는 소형 섭동 배양과 미니 벌크 전사체학 분석을 결합한 전사체 기반 약물 스크리닝을 위한 고차원 탐색 시스템을 설명합니다 5,6. 이 프로토콜을 사용하면 샘플당 비용을 전체 길이 mRNA 염기서열분석을 위한 현재 상용 솔루션비용의 1/6로 줄일 수 있습니다. 이 프로토콜에는 표준 실험실 장비만 필요하며, 유일한 예외는 짧은 판독 염기서열분석 기술을 사용하는 것이며, 염기서열분석 장비를 사내에서 사용할 수 없는 경우 아웃소싱할 수 있습니다. 최적화된 미니 벌크 프로토콜은 비용 효율적인 염기서열분석 깊이에서 정보가 풍부한 생물학적 신호를 제공하여 알려진 약물 및 새로운 분자에 대한 광범위한 스크리닝을 가능하게 합니다.
이 실험의 목적은 다양한 생물학적 맥락에서 PBMC의 약물 활성을 스크리닝하는 것입니다. 이 프로토콜은 전사체 판독으로 여러 약물을 테스트해야 하는 모든 생물학적 질문에 적용할 수 있으며, 치료의 세포 효과에 대한 전사체 전체 보기를 제공합니다.
프로토콜
이 프로토콜은 본 대학교의 지역 윤리 위원회의 지침을 따릅니다.
1. 완충액, 용액 및 장비 준비
- 용액을 준비하고 재료 목차에 설명된 재료를 수집합니다.
- 수조를 37°C로 가열하고 완전 성장 배지(RPMI-1640 + 10% 태아 송아지 혈청(FCS) + 1% 페니실린/스트렙토마이신)을 예열합니다.
- 세포 채취의 경우 얼음처럼 차가운 인산염 완충 식염수(PBS)를 사용합니다.
알림: 무균 상태를 유지하기 위해 세포와 함께 작업하는 동안 깨끗한 환경을 유지하십시오.
2. 세포 취급
참고: 인간 혈액에서 말초 혈액 단핵 세포(PBMC)의 동결 보존을 위한 자세한 프로토콜은7에서 찾을 수 있습니다.
- 세포 해동 및 계수
- 액체 질소에서 극저온을 제거하고 37°C의 수조에서 2 - 3분 동안 부드럽게 뒤집으면서 해동합니다.
- 해동된 세포를 50mL 원뿔형 튜브에 옮깁니다.
- 1mL의 따뜻한 완전 성장 배지로 cryovial을 헹구고 이 용액을 튜브의 세포에 적가합니다(1차 희석 단계).
- 32mL의 부피가 될 때까지 1:1로 희석을 반복합니다(각각 1, 2, 4, 8 및 16mL의 따뜻한 완전 성장 배지로 총 5회 희석). 원뿔형 튜브를 약간 흔들면서 세포 교란을 줄이기 위해 배지를 적하 방향으로 추가합니다.
- 셀 현탁액을 5분(300 x g, 20°C) 동안 원심분리하고 원뿔형 튜브를 한 번의 유창한 동작으로 부드럽게 기울여 상층액을 제거합니다.
- 3mL의 따뜻한 완전 성장 배지에 세포를 재현탁시키고 세포 계수를 진행합니다.
- 계수를 위해 10μL의 세포 현탁액을 Trypan Blue(세포 현탁액의 밀도에 따라 1:2에서 1:10 희석)와 혼합하여 계수하는 동안 살아있는 세포와 죽은 세포를 구별합니다. 죽거나 손상된 세포는 염료의 흡수로 인해 파란색으로 보이지만 중요한 세포는 염색되지 않습니다.
참고: 트리판 블루는 약간 세포 독성이 있으므로 염색된 세포를 5분 이상 보관해서는 안 됩니다. - 10μL의 염색된 세포 용액을 사용하여 자동 세포 카운터 또는 계수 챔버로 세포를 계수합니다.
- Neubauer가 개선한 세포 챔버를 사용할 때 모서리에 있는 4개의 큰 사각형 내에 있는 모든 세포를 계산하고 다음 방정식을 사용하여 세포 현탁액의 농도를 계산합니다.
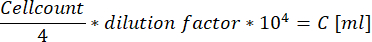
- Neubauer가 개선한 세포 챔버를 사용할 때 모서리에 있는 4개의 큰 사각형 내에 있는 모든 세포를 계산하고 다음 방정식을 사용하여 세포 현탁액의 농도를 계산합니다.
- 따뜻한 완전 성장 배지로 세포를 1 x 106 cells/mL의 최종 농도로 희석합니다. 필요한 부피가 3mL 미만인 경우에만 원심분리하고 세포 펠릿을 올바른 부피로 재현탁시킵니다.
- 세포 파종 및 처리
- 96웰 세포 배양 플레이트(1 x 105 세포/웰)에 웰당 100μL의 세포 현탁액을 시드합니다.
참고: 파종을 위한 세포 수는 PBMC 배양에 최적화되었습니다. 낮은 세포 농도는 염기서열분석을 위한 충분한 양의 RNA를 생성하지 못하지만, 과도한 수의 세포는 최적이 아닌 세포 용해의 위험을 증가시키고 역전사 반응의 억제를 증가시킵니다. - 치료에 사용되는 농도의 2배 농도로 약물 희석액을 준비합니다(2x). 화합물의 용해도에 따라 용매를 선택하고, 바람직하게는 완전한 성장 배지를 선택하십시오.
참고: PBS 또는 디메틸 설폭사이드(DMSO)는 충분한 용해도에 도달할 수 없는 경우 사용할 수 있습니다. 생성된 배양 부피에서 DMSO의 최대 농도는 용매에 의해 유도된 세포 독성을 방지하기 위해 0.5%를 초과해서는 안 됩니다. - 2x 약물 희석액 100μL(웰 1X의 최종 농도)를 추가하고 정의된 시간 동안 37°C에서 배양합니다.
참고: 최적의 약물 배양 시간을 설정하고 세포 독성의 잠재적 메커니즘을 배제하기 위해 보완 조사를 수행해야 합니다.
- 96웰 세포 배양 플레이트(1 x 105 세포/웰)에 웰당 100μL의 세포 현탁액을 시드합니다.
- 세포 수확 및 용해
- 세포 배양 플레이트를 10분(300 x g, 20°C) 동안 원심분리합니다. 플레이트를 비스듬히(30°-45°) 유지하면서 진공 펌프로 상층액을 부드럽게 제거하면서 팁을 웰의 낮은 모서리로 조준하여 가능한 한 적은 세포를 제거합니다.
- 각 웰에 200μL의 얼음처럼 차가운 PBS를 추가하여 세포를 세척하고 플레이트를 5분(300 x g, 4°C) 동안 원심분리합니다. 진공 펌프로 상층액을 다시 제거하고 플레이트를 예리한 각도로 유지하여 가능한 한 많은 PBS를 제거합니다.
- 필요한 반응 수(rxn)에 대해 표 1 에 설명된 대로 용해 완충액을 준비하고, 10% 초과분을 추가합니다.
- 각 웰에 15μL의 용해 완충액을 추가하고 접착 밀봉 필름으로 플레이트를 밀봉하여 샘플을 오염으로부터 보호합니다. 1분(1000 x g, 4°C) 동안 원심분리하기 전에 플레이트를 소용돌이치고 얼음 위에서 5분 동안 배양합니다.
- PCR 플레이트에서 용해된 세포 용액 6μL를 수집하고 세포 용해물을 -80°C에서 잠시 동결하여 최적의 세포 용해를 보장합니다. 플레이트의 여러 동결 주기를 피하십시오.
참고: 중지점: cDNA 생산이 이후에 수행되지 않으면 세포 용해물을 RNA 품질의 상당한 저하 없이 최대 몇 개월 동안 -80°C에서 보관할 수 있습니다.
3. 염기서열분석을 위한 라이브러리 준비
- 역전사 효소(RT) 반응
참고: RNA로 작업할 때 모든 샘플을 얼음 위에 놓고 뉴클레아제가 없는 장비(멸균, 일회용 플라스틱 용기)와 물을 사용해야 합니다.- 필요한 반응 수에 대해 표 2 에 설명된 대로 RT 반응 혼합물을 준비하고, 10% 초과분을 추가합니다. 믹스를 간단히 소용돌이치고 짧게 회전시킵니다. 사용할 때까지 혼합물을 얼음 위에 두십시오.
- 세포 용해물을 실온(RT)에서 해동하고 잠시 회전시켜 플레이트 바닥에 있는 모든 세포 용해물을 수집합니다.
- 표 3에 따라 thermocycler에서 mRNA 변성을 수행합니다.
- 열순환기에서 플레이트를 꺼내고 잠시 회전시켜 잠재적인 응축수를 수집합니다. 6μL의 RT 반응 혼합물을 각 웰에 추가하여 최종 부피를 12μL로 만듭니다. 플레이트를 보호하고 증발, 소용돌이를 방지하기 위해 플레이트를 밀봉하고 스핀다운합니다.
- 플레이트를 열순환기에 놓고 표 3에 따라 RT 반응 프로그램을 시작합니다.
- 사전 증폭
- 고충실도 DNA 중합효소 및 현장 PCR(ISPCR) 프라이머를 실온에서 해동합니다.
- 필요한 반응 수에 대해 표 4 에 따라 사전 증폭 혼합물을 준비하고 10% 초과분을 추가합니다. 믹스를 간단히 소용돌이치고 회전시킵니다.
알림: 효소 혼합물은 실온에서 몇 시간 동안 안정적입니다. - PCR 플레이트를 회전시키고 각 웰에 15μL의 사전 증폭 혼합물을 추가하고 플레이트를 밀봉합니다.
- thermocycler에 그것을 두고 표 5에 의하여 전 증폭 반응을 시작하십시오.
- RNA 함량으로 인한 사이클 수를 조정합니다. 더 낮은 숫자로 시작하여 cDNA 수율이 충분하지 않은 경우 증가시킵니다.
참고: 우리의 경험에 비추어 볼 때, 각 세포 유형에 대해 최적의 주기 수를 설정해야 하며, 실험 조건 및 처리는 최적의 주기 수에 영향을 미치지 않습니다. 따라서 새로운 세포 유형에 대해 이 프로토콜을 설정할 때 실험적으로 최적의 사이클 수를 설정하는 것이 좋습니다. 일반적으로 일차 세포는 세포주에 비해 더 많은 수의 사이클이 필요합니다. 중지점: 사전 증폭 제품은 -20°C에서 보관할 수 있습니다.
- RNA 함량으로 인한 사이클 수를 조정합니다. 더 낮은 숫자로 시작하여 cDNA 수율이 충분하지 않은 경우 증가시킵니다.
- cDNA 세척 및 품질 관리(QC)
참고: 샘플 청소는 샘플이 이 단계에서 다소 안정적이므로 각 플레이트에 대해 순차적으로 수행할 수 있습니다. 샘플 수를 늘리면 프로토콜의 지속 시간이 길어지지만 동시에 처리할 수 있는 샘플 수는 제한되지 않습니다.- 시작하기 전에 자기 정화 비드를 실온으로 가져오고 비드를 완전히 재현탁하기 위해 1분 동안 고속으로 소용돌이칩니다.
- 각 웰에 0.8x v/v(20μL) 자기 정제 비드를 추가하고 실온에서 5분 동안 배양합니다.
- 비드가 완전히 분리될 때까지 PCR 플레이트를 마그네틱 랙에 5분 동안 놓습니다. 상층액을 조심스럽게 제거하십시오.
- 접시를 마그네틱 랙에 놓고 갓 준비한 80% 에탄올 100μL를 부드럽게 추가하여 비드를 씻고 30초 동안 배양합니다. 소량의 피펫을 사용하여 상층액을 제거합니다. 추가 세척 단계를 반복합니다. 가능한 한 많은 에탄올을 제거하십시오.
- 에탄올이 완전히 증발하고 비드가 더 이상 광택이 나지 않을 때까지 실온에서 최대 5분 동안 마그네틱 랙의 비드를 자연 건조합니다.
- 마그네틱 랙에서 플레이트를 제거하고 비드를 20μL의 뉴클레아제가 없는 물에 재현탁시키고 2분 동안 배양합니다.
- 비드가 분리될 때까지 플레이트를 마그네틱 랙에 다시 놓습니다.
- cDNA QC 및 tagmentation을 위해 새로운 PCR 플레이트에서 용리액을 회수합니다.
- TapeStation 또는 FragmentAnalyser 어세이를 수행하여 cDNA 라이브러리의 크기 분포 및 농도를 평가합니다(권장: TapeStation D5000 어세이). 자세한 내용은 제조업체 지침을 참조하십시오. 일반적인 수율은 20ng입니다.
- 키트를 사용한 태그 지정
알림: 이미 설정된 경우 다른 Tagmentation 프로토콜을 사용할 수 있습니다.- 뉴클레아제가 없는 물을 사용하여 cDNA를 최종 농도인 150 - 300pg/μL로 희석합니다.
- 표 6에 의하여 효소 혼합물에 cDNA를 추가한 후에 tagmentation 반응의 즉각 시작을 지키기 위하여 thermocycler를 미리 프로그램하십시오.
- 필요한 반응 수에 대해 표 7 에 따라 태그 혼합물을 준비하고 10% 초과분을 추가합니다.
- 반응당 3μL의 tagmentation 혼합물을 새 PCR 플레이트에 분주하고 각 반응에 1μL의 cDNA를 추가합니다.
- cDNA를 첨가한 직후 태그 부착 반응을 시작합니다.
- 1μL의 중화 태그 완충액(NT; 또는 0.2% 소듐 도데실 설페이트(SDS)을 사용할 수 있음)을 첨가하여 반응을 비활성화합니다.
- 농축 PCR
- 반응 횟수에 대한 농축 PCR 혼합물을 표 7 에 준비하고, 10% 초과분을 첨가한다. (권장: 패턴화된 플로우 셀(예: Illumina NovaSeq 6000)에서 인덱스 호핑을 방지하기 위해 설정된 Nextera UDI).
- 농축 PCR 혼합물 9μL를 각 반응에 첨가하여 총 부피 14μL를 만듭니다.
- 표 8에 따라 농축 PCR 프로그램을 실행합니다.
참고: 농축 PCR의 경우 16 사이클이 표준입니다. cDNA 품질이 좋지 않은 경우 추가 사이클을 추가할 수 있습니다.
- 세척 및 QC
- 시작하기 전에 자기 정화 비드를 실온으로 가져와 1분 동안 고속으로 소용돌이쳐 비드를 완전히 재현탁합니다.
- 1.0x v/v(14μL) 자기 정제 비드를 추가하고 실온에서 5분 동안 배양합니다.
- 접시를 마그네틱 랙에 놓고 비드가 완전히 분리될 때까지 5분 동안 기다립니다. 상층액을 조심스럽게 제거하십시오.
- 접시를 마그네틱 랙에 놓고 갓 준비한 80% 에탄올 100μL를 부드럽게 추가하여 비드를 씻고 30초 동안 배양합니다. 소량의 피펫을 사용하여 상층액을 제거합니다. 추가 세척 단계를 반복합니다. 가능한 한 많은 에탄올을 제거하십시오.
- 비드를 실온에서 3분 동안 또는 더 이상 광택이 나지 않을 때까지 자연 건조시킵니다.
- 마그네틱 랙에서 플레이트를 제거하고 비드를 20μL의 뉴클레아제가 없는 물에 재현탁시키고 2분 동안 배양합니다.
- 비드가 분리될 때까지 플레이트를 마그네틱 랙에 다시 놓고 라이브러리 QC를 위해 새 PCR 플레이트에서 용리액을 회수합니다.
- TapeStation 또는 Fragment Analyzer 어세이를 수행하여 cDNA 라이브러리의 크기 분포 및 농도를 평가합니다(권장: TapeStation D1000 고감도 어세이). 자세한 내용은 제조업체 지침을 참조하십시오. 평균적으로 10ng의 수율이 예상됩니다.
참고: 정지점: PCR 산물은 -20°C에서 보관할 수 있습니다.
4. 염기서열분석 및 데이터 전처리
- 시퀀싱
알림: 다음 지침은 짧은 판독 시퀀싱을 위해 모든 Illumina 기기에 적용됩니다. 계측을 사용할 수 없는 경우 외부 시퀀싱 기능에서 시퀀싱을 수행할 수 있습니다. 다른 염기서열분석 방법도 사용할 수 있습니다. 단순화를 위해 가장 널리 사용되는 염기서열분석 기술에 대해서만 보고하기로 결정했습니다.
알림: 소프트웨어 사용과 관련된 다음 단계는 Illumina NovaSeq6000 시퀀서의 절차를 설명합니다.- 3.6.8단계에서 얻은 결과에 따라 등몰비로 고유하게 인덱싱된 라이브러리를 풀링합니다.
- 고감도 분석으로 최종 풀의 농도를 측정하여 다음과 같이 샘플 몰 농도를 계산합니다.
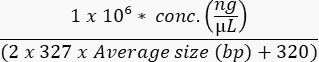
- 기기의 사양과 실험 최적화에 따라 플로우 셀을 로드합니다. 일반적인 기기의 부하 농도에 대한 예는 표 9에 나와 있습니다.
- 화면을 터치하여 시퀀스 를 선택하여 실행 설정을 시작합니다.
- 화면의 지침을 따르고 플로우 셀, 합성 시퀀스 카트리지, 클러스터링 카트리지, 버퍼 카트리지를 로드하고 폐기물 용기가 비어 있는지 확인합니다.
- 기기에서 모든 시약을 인식하면 Run Setup(설정 실행)을 클릭합니다. 여기에서 실행 이름과 데이터를 저장할 출력 폴더를 정의합니다.
- 염기서열분석 세부 정보를 두 판독 모두 51bp의 쌍 끝으로 정의합니다. 두 개의 인덱스 읽기도 각각 8bp로 시퀀싱됩니다.
- Review를 누르고 모든 시퀀싱 세부 정보가 올바른지 확인한 후 Start Run을 누릅니다.
참고: 권장 시퀀싱 깊이는 5 x 106 읽기/샘플, 최소 1 x 106 읽기/샘플.
- 데이터 전처리
- 원시 염기서열분석 데이터를 FASTQ 형식으로 변환하고 Bcl2Fastq2 도구를 사용하여 샘플 인덱스에 따라 역다중화합니다. 기본 설정으로 역다중화를 수행합니다. Bcl2Fastq2에 대한 자세한 지침은 참조 설명서를 참조하십시오.
참고: FASTQ 변환 및 역다중화는 시퀀싱이 사내에서 수행되지 않는 경우 일반적으로 시퀀싱 시설에서 수행됩니다. 염기서열분석 시설은 일반적으로 추가 처리를 위해 역다중화된 FASTQ를 제공합니다. - 염기서열분석 판독의 데이터 정렬 및 풍부도 정량화를 위해 몇 가지 옵션을 사용할 수 있습니다(권장: nf-core RNA-seq 파이프라인(https://nf-co.re/rnaseq)). 파이프라인은 몇 가지 옵션을 제공하며, STAR8 을 정렬하고 Salmon9 를 사용하여 전사체 풍부도를 정량화하는 기본 설정을 사용합니다.
- 참고: 추가 생물정보학 분석을 위해 여러 가지 방법을 사용할 수 있습니다. 모든 것을 다루는 것은 이 프로토콜의 범위에 속하지 않습니다(권장: DEseq2 파이프라인10). 당사에서 개발한 DEseq2 워크플로를 기반으로 하는 표준 스크립트는 GitHub(https://github.com/jsschrepping/RNA-DESeq2)에서 찾을 수 있습니다.
- 원시 염기서열분석 데이터를 FASTQ 형식으로 변환하고 Bcl2Fastq2 도구를 사용하여 샘플 인덱스에 따라 역다중화합니다. 기본 설정으로 역다중화를 수행합니다. Bcl2Fastq2에 대한 자세한 지침은 참조 설명서를 참조하십시오.
결과
보고된 프로토콜에 따라 인간 PBMC를 파종하고, 다양한 면역조절 약물로 처리하고, 서로 다른 배양 시간 후에 염기서열분석 프로토콜을 사용하여 대량 전사체 분석을 위해 수확했습니다(그림 1).
테스트 화합물에 대한 이상적인 약물 농도 및 배양 시간은 보완적인 실험 전략의 도움과 특정 과학적 질문을 기반으로 이 프로토콜의 업스트림에서 식별되어야...
토론
약물 발견 및 약물 개발은 벌크 전사체학이 제공할 수 있는 세포 과정에 대한 전체론적 관점으로부터 큰 이점을 얻을 수 있습니다. 그럼에도 불구하고 이 접근법은 표준 벌크 RNA-seq 프로토콜을 사용한 실험의 높은 비용으로 인해 제한되는 경우가 많으며, 이는 학술 환경에서의 적용과 산업 확장성 가능성을 금지합니다.
프로토콜의 가장 중요한 단계는 세포 해동과 라이브러리...
공개
저자는 상충되는 이해관계가 없음을 선언합니다.
감사의 말
JLS는 독일의 우수성 전략(EXC2151-390873048)과 SCHU 950/8-1에 따라 독일 연구 재단(DFG)의 지원을 받습니다. GRK 2168, TP11; CRC SFB 1454 Metaflammation, IRTG GRK 2168, WGGC INST 216/981-1, CCU INST 217/988-1, BMBF 자금 지원 우수 프로젝트 Diet-Body-Brain (DietBB); 및 보조금 번호 733100에 따른 EU 프로젝트 SYSCID. M.B.는 DFG에서 지원됩니다(IRTG2168-272482170, SFB1454-432325352). L.B.는 DFG(ImmuDiet BO 6228/2-1 - 프로젝트 번호 513977171) 및 Germany's Excellence Strategy(EXC2151-390873048)의 지원을 받습니다. BioRender.com 로 만든 이미지.
자료
| Name | Company | Catalog Number | Comments |
| 50 mL conical tube | fisher scientific | 10203001 | |
| Adhesive PCR Plate Seals | Thermo Fisher Scientific | AB0558 | |
| Amplicon Tagment Mix (ATM) | Illumina | FC-131-1096 | Nextera XT DNA Library Prep Kit (96 samples) |
| AMPure XP beads | Beckman Coulter | A 63881 | |
| Betaine | Sigma-Aldrich | 61962 | |
| Cell culture grade 96-well plates | Thermo Fisher Scientific | 260860 | |
| Cell culture vacuum pump (VACUSAFE) | Integra Bioscience | 158300 | |
| Deoxynucleotide triphosphates (dNTPs) mix 10 mM each | Fermentas | R0192 | |
| DMSO | Sigma-Aldrich | 276855 | |
| DTT (100 mM) | Invitrogen | 18064-014 | |
| EDTA | Sigma-Aldrich | 798681 | for adherent cells |
| Ethanol | Sigma-Aldrich | 51976 | |
| Fetal Bovine Serum | Thermo Fisher Scientific | 26140079 | |
| Filter tips (10 µL) | Gilson | F171203 | |
| Filter tips (100 µL) | Gilson | F171403 | |
| Filter tips (20 µL) | Gilson | F171303 | |
| Filter tips (200 µL) | Gilson | F171503 | |
| Guanidine Hydrochloride | Sigma-Aldrich | G3272 | |
| ISPCR primer (10 µM) | Biomers.net GmbH | SP10006 | 5′-AAGCAGTGGTATCAACGCAGAG T-3′ |
| KAPA HiFi HotStart ReadyMix (2X) | KAPA Biosystems | KK2601 | |
| Magnesium chloride (MgCl2) | Sigma-Aldrich | M8266 | |
| Magnetic stand 96 | Ambion | AM10027 | |
| Neutralize Tagment (NT) Buffer | Illumina | FC-131-1096 | Nextera XT DNA Library Prep Kit (96 samples), alternatively 0.2 % SDS |
| Nextera-compatible indexing primer | Illumina | ||
| Nuclease-free water | Invitrogen | 10977049 | |
| PBS | Thermo Fisher Scientific | AM9624 | |
| PCR 96-well plates | Thermo Fisher Scientific | AB0600 | |
| PCR plate sealer | Thermo Fisher Scientific | HSF0031 | |
| Penicillin / Streptomycin | Thermo Fisher Scientific | 15070063 | |
| Qubit 4 fluorometer | Invitrogen | 15723679 | |
| Recombinant RNase inhibitor (40 U/ul) | TAKARA | 2313A | |
| RPMI-1640 cell culture medium | Gibco | 61870036 | If not working with PBMCs, adjust to cell type |
| SMART dT30VN primer | Sigma-Aldrich | 5' Bio-AAGCAGTGGTATCAACGCAGAG TACT30VN-3 | |
| Standard lab equipment | various | various | e.g. centrifuge, ice machine, ice bucket, distilled water, water bath |
| SuperScript II Reverse Transcriptase (SSRT II) | Thermo Fisher Scientific | 18064-014 | |
| SuperScript II Reverse Transcriptase (SSRT II) buffer (5x) | Thermo Fisher Scientific | 18064-014 | |
| Tagment DNA Buffer (TD) | Illumina | FC-131-1096 | Nextera XT DNA Library Prep Kit (96 samples) |
| TapeStation system 4200 | Agilent | G2991BA | |
| Thermocycler (S1000) | Bio-Rad | 1852148 | |
| TSO-LNA (100 uM) | Eurogentec | 5' Biotin AAGCAGTGGTATCAACGCAGAG TACAT(G)(G){G | |
| Vortex-Genie 2 Mixer | Sigma-Aldrich | Z258415 |
참고문헌
- Hughes, J. P., Rees, S., Kalindjian, S. B., Philpott, K. L. Principles of early drug discovery. Br J Pharmacol. 162 (6), 1239-1249 (2011).
- Yang, X., et al. High-throughput transcriptome profiling in drug and biomarker discovery. Front Genet. 11, 19 (2020).
- Bonaguro, L., et al. A guide to systems-level immunomics. Nat Immunol. 23 (10), 1412-1423 (2022).
- Carraro, C., et al. Decoding mechanism of action and sensitivity to drug candidates from integrated transcriptome and chromatin state. ELife. 11, 78012 (2022).
- Picelli, S., et al. Smart-seq2 for sensitive full-length transcriptome profiling in single cells. Nat Methods. 10 (11), 1096-1098 (2013).
- Picelli, S., et al. Full-length RNA-seq from single cells using Smart-seq2. Nat Protoc. 9 (1), 171-181 (2014).
- De Domenico, E., et al. Optimized workflow for single-cell transcriptomics on infectious diseases including COVID-19. STAR Protoc. 1 (3), 100233 (2020).
- Dobin, A., et al. ultrafast universal RNA-seq aligner. Bioinformatics. 29 (1), 15-21 (2013).
- Patro, R., et al. Salmon provides fast and bias-aware quantification of transcript expression. Nat Methods. 14 (4), 417-419 (2017).
- Love, M. I., Huber, W., Anders, S. Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2. Genome Biol. 15 (12), 550 (2014).
- Frankish, A., et al. GENCODE reference annotation for the human and mouse genomes. Nucleic Acids Res. 47, D766-D773 (2019).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유