Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Dépistage de drogues basé sur la transcriptomique rentable
Dans cet article
Résumé
Ce protocole décrit un flux de travail allant des cultures cellulaires ex vivo ou in vitro au prétraitement des données transcriptomiques pour un criblage rentable de médicaments basés sur le transcriptome.
Résumé
La transcriptomique permet d’obtenir des informations complètes sur les programmes cellulaires et leurs réponses aux perturbations. Malgré une baisse significative des coûts de production et de séquençage des bibliothèques au cours de la dernière décennie, l’application de ces technologies à l’échelle nécessaire au criblage des médicaments reste prohibitive, ce qui entrave l’immense potentiel de ces méthodes. Notre étude présente un système rentable de criblage de médicaments basé sur le transcriptome, combinant des cultures de perturbation miniaturisées avec une transcriptomique mini-en vrac. Le protocole mini-bulk optimisé fournit des signaux biologiques informatifs à une profondeur de séquençage rentable, ce qui permet un criblage approfondi de médicaments connus et de nouvelles molécules. En fonction du traitement choisi et du temps d’incubation, ce protocole permettra d’obtenir des banques de séquençage dans un délai d’environ 2 jours. En raison de plusieurs points d’arrêt au sein de ce protocole, la préparation de la bibliothèque, ainsi que le séquençage, peuvent être effectués indépendamment du temps. Il est possible de traiter simultanément un grand nombre d’échantillons ; La mesure de jusqu’à 384 échantillons a été testée sans perte de qualité des données. Il n’y a pas non plus de limites connues au nombre d’affections et/ou de médicaments, malgré la prise en compte de la variabilité des temps d’incubation optimaux des médicaments.
Introduction
Le développement de nouveaux médicaments est un processus complexe et chronophage qui implique l’identification de médicaments potentiels et de leurs cibles, l’optimisation et la synthèse de candidats-médicaments, et le test de leur efficacité et de leur innocuité dans le cadre d’essais précliniques et cliniques1. Les méthodes traditionnelles de criblage de médicaments, c’est-à-dire l’évaluation systématique de bibliothèques de composés candidats à des fins thérapeutiques, impliquent l’utilisation de modèles animaux ou de tests cellulaires pour tester les effets sur des cibles ou des voies spécifiques. Bien que ces méthodes aient permis d’identifier des médicaments candidats, elles n’ont souvent pas fourni suffisamment d’informations sur les mécanismes moléculaires complexes sous-jacents à l’efficacité des médicaments, ainsi que sur leur toxicité et leurs mécanismes d’effets secondaires potentiels.
L’évaluation des états transcriptionnels à l’échelle du génome présente une approche puissante pour surmonter les limites actuelles du criblage des médicaments, car elle permet des évaluations complètes de l’expression des gènes en réponse aux traitements médicamenteux2. En mesurant les transcrits d’ARN exprimés à l’échelle du génome à un moment donné, la transcriptomique vise à fournir une vue holistique des changements transcriptionnels qui se produisent en réponse aux médicaments, y compris les changements dans les modèles d’expression génique, l’épissage alternatif et l’expression de l’ARN non codant3. Ces informations peuvent être utilisées pour déterminer les cibles des médicaments, prédire l’efficacité et la toxicité des médicaments et optimiser les schémas posologiques et de traitement des médicaments.
L’un des principaux avantages de la combinaison de la transcriptomique et du criblage non biaisé des médicaments est la possibilité d’identifier de nouvelles cibles médicamenteuses qui n’ont pas été envisagées auparavant. Les approches conventionnelles de criblage de médicaments se concentrent souvent sur des molécules ou des voies cibles établies, ce qui entrave l’identification de nouvelles cibles et peut entraîner la mise en place de médicaments ayant des effets secondaires imprévus et une efficacité limitée. La transcriptomique peut surmonter ces limites en fournissant des informations sur les changements moléculaires qui se produisent en réponse au traitement médicamenteux, en découvrant des cibles ou des voies potentielles qui n’auraient peut-être pas été envisagées auparavant2.
En plus de l’identification de nouvelles cibles médicamenteuses, la transcriptomique peut également être utilisée pour prédire l’efficacité et la toxicité des médicaments. En analysant les modèles d’expression génique associés aux réponses aux médicaments, il est possible de développer des biomarqueurs qui peuvent être utilisés pour prédire la réponse d’un patient à un médicament ou à un schéma thérapeutique particulier. Cela peut également aider à optimiser la posologie des médicaments et à réduire le risque d’effets secondaires indésirables4.
Malgré ses avantages potentiels, le coût de la transcriptomique reste un obstacle important à son application généralisée dans le dépistage des drogues. L’analyse transcriptomique nécessite un équipement spécialisé, une expertise technique et une analyse des données, ce qui peut rendre difficile l’utilisation de la transcriptomique pour les petites équipes de recherche ou les organisations disposant d’un financement limité dans le dépistage des médicaments. Cependant, le coût de la transcriptomique n’a cessé de diminuer, ce qui la rend plus accessible aux communautés de recherche. De plus, les progrès de la technologie et des méthodes d’analyse des données ont rendu la transcriptomique plus efficace et plus rentable, ce qui en a encore accru l’accessibilité2.
Dans ce protocole, nous décrivons un système exploratoire de grande dimension pour le criblage de médicaments basé sur le transcriptome, combinant des cultures de perturbation miniaturisées avec une analyse transcriptomique en mini-vrac 5,6. Avec ce protocole, il est possible de réduire le coût par échantillon à 1/6ème du coût actuel des solutions commerciales pour le séquençage de l’ARNm pleine longueur. Le protocole ne nécessite que des équipements de laboratoire standard, la seule exception étant l’utilisation de technologies de séquençage à lecture courte, qui peuvent être externalisées si les instruments de séquençage ne sont pas disponibles en interne. Le protocole mini-bulk optimisé fournit des signaux biologiques riches en informations à une profondeur de séquençage rentable, ce qui permet un criblage approfondi de médicaments connus et de nouvelles molécules.
L’objectif de l’expérience est de dépister l’activité des médicaments sur les PBMC dans différents contextes biologiques. Ce protocole peut être appliqué à n’importe quelle question biologique où plusieurs médicaments doivent être testés avec une lecture transcriptomique, donnant une vue à l’échelle du transcriptome de l’effet cellulaire du traitement.
Protocole
Ce protocole suit les directives des comités d’éthique locaux de l’Université de Bonn.
1. Préparation des tampons, des solutions et de l’équipement
- Préparez les solutions et rassemblez les matériaux décrits dans le tableau des matériaux.
- Chauffer le bain-marie à 37 °C et réchauffer le milieu de croissance complet (RPMI-1640 + 10 % de sérum de veau fœtal (FCS) + 1 % de pénicilline/streptomycine).
- Pour le prélèvement de cellules, utilisez une solution saline tamponnée au phosphate (PBS) glacée.
REMARQUE : Gardez un environnement propre lorsque vous travaillez avec des cellules pour maintenir la stérilité.
2. Manipulation des cellules
NOTE : Un protocole détaillé pour la cryoconservation des cellules mononucléées du sang périphérique (PBMC) à partir de sang humain se trouve aupoint 7.
- Décongélation et comptage des cellules
- Retirez les cryoflacons de l’azote liquide et décongelez-les au bain-marie à 37 °C pendant 2 à 3 min tout en les inversant doucement.
- Transférer les cellules décongelées dans un tube conique de 50 ml.
- Rincez le cryoflacon avec 1 mL de milieu de culture complet chaud et ajoutez cette solution goutte à goutte aux cellules du tube (1ère étape de dilution).
- Répétez la dilution 1 :1 jusqu’à ce que vous atteigniez un volume de 32 mL (5 dilutions au total avec respectivement 1, 2, 4, 8 et 16 mL de milieu de culture complet chaud). Ajouter le milieu goutte à goutte pour réduire la perturbation cellulaire tout en agitant légèrement le tube conique.
- Centrifuger la suspension cellulaire pendant 5 min (300 x g, 20 °C) et retirer le surnageant en inclinant doucement le tube conique d’un seul mouvement fluide.
- Remettre les cellules en suspension dans 3 mL de milieu de culture complet chaud et procéder au comptage des cellules.
- Pour le comptage, mélangez 10 μL de suspension cellulaire avec du bleu de trypan (dilution de 1 :2 à 1 :10 selon la densité de la suspension cellulaire) pour distinguer les cellules vivantes des cellules mortes pendant le comptage. Les cellules mortes ou endommagées apparaîtront bleues en raison de l’absorption du colorant, tandis que les cellules vitales ne sont pas colorées.
REMARQUE : Le bleu de trypan est légèrement cytotoxique, les cellules colorées ne doivent pas être stockées plus de 5 minutes. - Comptez les cellules à l’aide d’un compteur de cellules automatisé ou d’une chambre de comptage en utilisant 10 μL de solution cellulaire colorée.
- Lors de l’utilisation de la chambre cellulaire améliorée par Neubauer, comptez toutes les cellules à l’intérieur des quatre grands carrés situés aux coins et utilisez l’équation suivante pour calculer la concentration de la suspension cellulaire.
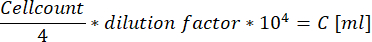
- Lors de l’utilisation de la chambre cellulaire améliorée par Neubauer, comptez toutes les cellules à l’intérieur des quatre grands carrés situés aux coins et utilisez l’équation suivante pour calculer la concentration de la suspension cellulaire.
- Diluer les cellules jusqu’à une concentration finale de 1 x 106 cellules/mL avec un milieu de culture complet chaud. Centrifuger uniquement si le volume requis est inférieur à 3 mL et remettre en suspension la pastille cellulaire au bon volume.
- Ensemencement et traitement cellulaire
- Ensemencer 100 μL de suspension cellulaire par puits dans une plaque de culture cellulaire à 96 puits (1 x 105 cellules/puits).
REMARQUE : Le nombre de cellules pour l’ensemencement a été optimisé pour les cultures PBMC. Bien que de faibles concentrations cellulaires ne produisent pas une quantité suffisante d’ARN pour le séquençage, un nombre excessif de cellules augmentera le risque de lyse cellulaire sous-optimale et d’inhibition de la réaction de transcription inverse. - Préparer des dilutions de médicaments à une concentration deux fois supérieure à celle utilisée pour le traitement (2x). Choisir le solvant en fonction de la solubilité du composé, de préférence milieu de culture complet.
REMARQUE : Le PBS ou le diméthylsulfoxyde (DMSO) peut être utilisé si une solubilité suffisante ne peut être atteinte autrement. La concentration maximale de DMSO dans le volume d’incubation résultant ne doit pas dépasser 0,5 % pour éviter la cytotoxicité induite par le solvant. - Ajouter 100 μL de la dilution 2x du médicament (concentration finale dans le puits 1X) et incuber à 37 °C pendant la durée définie.
NOTA : Des investigations complémentaires doivent être menées afin d’établir le temps d’incubation optimal du médicament et d’exclure les mécanismes potentiels de cytotoxicité.
- Ensemencer 100 μL de suspension cellulaire par puits dans une plaque de culture cellulaire à 96 puits (1 x 105 cellules/puits).
- Prélèvement et lyse cellulaires
- Centrifuger la plaque de culture cellulaire pendant 10 min (300 x g, 20 °C). Retirez délicatement le surnageant à l’aide d’une pompe à vide en maintenant la plaque en biais (30°-45°) tout en dirigeant la pointe vers le coin bas du puits pour éliminer le moins de cellules possible.
- Lavez les cellules en ajoutant 200 μL de PBS glacé dans chaque puits et centrifugez la plaque pendant 5 min (300 x g, 4 °C). Retirez le surnageant à l’aide d’une pompe à vide, encore une fois, en gardant la plaque à un angle aigu pour éliminer autant de PBS que possible.
- Préparez le tampon de lyse comme décrit dans le tableau 1 pour le nombre de réactions (rxn) nécessaires, en ajoutant 10 % de dépassement.
- Ajouter 15 μL de tampon de lyse dans chaque puits et sceller la plaque avec un film d’étanchéité adhésif pour protéger les échantillons contre la contamination. Faire vortex la plaque avant de la centrifuger pendant 1 min (1000 x g, 4 °C) et incuber pendant 5 min sur glace.
- Prélever 6 μL de solution cellulaire lysée dans une plaque PCR et congeler brièvement le lysat cellulaire à -80 °C pour assurer une lyse cellulaire optimale. Assurez-vous d’éviter les multiples cycles de congélation des plaques.
REMARQUE : POINT D’ARRÊT : Si la production d’ADNc n’est pas effectuée par la suite, le lysat cellulaire peut être stocké à -80 °C pendant plusieurs mois sans diminution considérable de la qualité de l’ARN.
3. Préparation de la bibliothèque pour le séquençage
- Réaction de transcriptase inverse (RT)
REMARQUE : Lorsque vous travaillez avec de l’ARN, placez tous les échantillons sur de la glace et assurez-vous d’utiliser du matériel exempt de nucléases (articles en plastique stériles et jetables) et de l’eau.- Préparez le mélange réactionnel RT tel que décrit dans le tableau 2 pour le nombre de réactions nécessaires, en ajoutant 10 % de dépassement. Faites tourner brièvement le mélange et faites-le tourner brièvement vers le bas. Conservez le mélange sur de la glace jusqu’à utilisation.
- Décongelez le lysat cellulaire à température ambiante (RT) et faites-le tourner brièvement pour recueillir tout le lysat cellulaire au fond de la plaque.
- Effectuer la dénaturation de l’ARNm sur un thermocycleur conformément au tableau 3.
- Sortez la plaque du thermocycleur et essorez-la brièvement pour recueillir le condensat potentiel. Ajouter 6 μL de mélange réactionnel RT dans chaque puits pour obtenir un volume final de 12 μL. Scellez la plaque pour la protéger et éviter l’évaporation, le vortex et la faire tourner.
- Placez la plaque sur le thermocycleur et démarrez le programme de réaction RT comme indiqué dans le tableau 3.
- Pré-amplification
- Décongeler l’amorce d’ADN polymérase haute fidélité et de PCR in situ (ISPCR) à température ambiante.
- Préparez le mélange de préamplification selon le tableau 4 pour le nombre de réactions nécessaires, en ajoutant 10 % de dépassement. Faites tourner brièvement le mélange et faites-le tourner.
REMARQUE : Le mélange enzymatique est stable à température ambiante pendant des heures. - Essorez la plaque PCR, ajoutez 15 μL du mélange de préamplification dans chaque puits et scellez la plaque.
- Placez-le sur le thermocycleur et démarrez la réaction de pré-amplification comme indiqué dans le tableau 5.
- Ajustez le nombre de cycles en fonction de la teneur en ARN. Commencez par un nombre plus faible et augmentez si le rendement en ADNc n’est pas suffisant.
REMARQUE : D’après notre expérience, le nombre optimal de cycles doit être établi pour chaque type de cellule, les conditions expérimentales et le traitement n’influenceront pas le nombre optimal de cycles. Nous recommandons donc d’établir expérimentalement le nombre optimal de cycles lors de l’établissement de ce protocole pour un nouveau type de cellule. En général, les cellules primaires nécessiteront un nombre de cycles plus élevé que les lignées cellulaires. POINT D’ARRÊT : Le produit de préamplification peut être stocké à - 20 °C.
- Ajustez le nombre de cycles en fonction de la teneur en ARN. Commencez par un nombre plus faible et augmentez si le rendement en ADNc n’est pas suffisant.
- Nettoyage de l’ADNc et contrôle de la qualité (CQ)
REMARQUE : Le nettoyage des échantillons peut être effectué séquentiellement pour chaque plaque car les échantillons sont plutôt stables à ce stade. L’augmentation du nombre d’échantillons entraînera une durée plus longue du protocole, mais ne limitera pas le nombre d’échantillons pouvant être traités simultanément.- Avant de commencer, amenez les billes de purification magnétique à température ambiante et à vortex à grande vitesse pendant 1 min pour les remettre complètement en suspension.
- Ajouter 0,8 bille de purification magnétique v/v (20 μL) dans chaque puits et incuber à température ambiante pendant 5 min.
- Placez la plaque PCR sur une grille magnétique pendant 5 min jusqu’à ce que les billes soient complètement séparées. Retirez le surnageant avec précaution.
- En gardant la plaque sur la grille magnétique, ajoutez délicatement 100 μL d’éthanol à 80 % fraîchement préparé pour laver les billes et incuber pendant 30 s. Utilisez une pipette de petit volume pour retirer le surnageant. Répétez l’opération pour une étape de lavage supplémentaire. Assurez-vous d’éliminer autant d’éthanol que possible.
- Faites sécher les billes à l’air libre sur la grille magnétique à température ambiante jusqu’à 5 minutes jusqu’à ce que l’éthanol soit complètement évaporé et que les billes ne soient plus brillantes.
- Retirer la plaque de la grille magnétique, remettre les billes en suspension dans 20 μL d’eau sans nucléase et incuber pendant 2 min.
- Placez à nouveau la plaque sur la grille magnétique jusqu’à ce que les perles soient séparées.
- Récupérer l’éluat dans une nouvelle plaque PCR pour le contrôle qualité de l’ADNc et le marquage.
- Effectuez un test TapeStation ou FragmentAnalyser pour évaluer la distribution granulométrique et la concentration de la banque d’ADNc (Recommandé : test TapeStation D5000). Pour plus de détails, consultez les instructions du fabricant. Il faut s’attendre à un rendement typique de 20 ng.
- Tagmentation avec kit
REMARQUE : D’autres protocoles de marquage peuvent être utilisés s’ils sont déjà établis.- Diluer l’ADNc à une concentration finale de 150 à 300 pg/μL en utilisant de l’eau sans nucléase.
- Préprogrammez le thermocycleur conformément au tableau 6 pour assurer un démarrage immédiat de la réaction de marquage après l’ajout de l’ADNc au mélange enzymatique.
- Préparez le mélange de marquage selon le tableau 7 pour le nombre de réactions nécessaires, en ajoutant 10 % de dépassement.
- Verser 3 μL du mélange de marquage par réaction sur une nouvelle plaque PCR et ajouter 1 μL d’ADNc à chaque réaction.
- Démarrez la réaction de marquage immédiatement après l’ajout de l’ADNc.
- Inactiver la réaction en ajoutant 1 μL de tampon de marquage neutralisant (NT ; on peut également utiliser du dodécylsulfate de sodium (SDS) à 0,2 %.
- PCR d’enrichissement
- Préparez le mélange d’enrichissement PCR Tableau 7 pour le nombre de réactions, en ajoutant 10 % de dépassement. (Recommandé : Nextera UDI réglé pour empêcher le saut d’index sur les cellules d’écoulement à motifs (par exemple, Illumina NovaSeq 6000)).
- Ajouter 9 μL du mélange de PCR d’enrichissement à chaque réaction pour un volume total de 14 μL.
- Exécutez le programme de PCR d’enrichissement conformément au tableau 8.
REMARQUE : Pour la PCR d’enrichissement, 16 cycles sont standard. Si la qualité de l’ADNc est médiocre, des cycles supplémentaires peuvent être ajoutés.
- Nettoyage et contrôle qualité
- Avant de commencer, amenez les billes de purification magnétiques à température ambiante et à vortex à grande vitesse pendant 1 min pour remettre complètement les billes en suspension.
- Ajouter 1,0 billes de purification magnétique v/v (14 μL) et incuber à température ambiante pendant 5 min.
- Placez l’assiette sur une grille magnétique et attendez 5 min jusqu’à ce que les perles soient complètement séparées. Retirez le surnageant avec précaution.
- En gardant la plaque sur la grille magnétique, ajoutez délicatement 100 μL d’éthanol à 80 % fraîchement préparé pour laver les billes et incuber pendant 30 s. Utilisez une pipette de petit volume pour retirer le surnageant. Répétez l’opération pour une étape de lavage supplémentaire. Assurez-vous d’éliminer autant d’éthanol que possible.
- Faites sécher les perles à l’air libre à température ambiante pendant 3 minutes ou jusqu’à ce qu’elles ne soient plus brillantes.
- Retirer la plaque de la grille magnétique, remettre les billes en suspension dans 20 μL d’eau sans nucléase et incuber pendant 2 min.
- Placez à nouveau la plaque sur la grille magnétique jusqu’à ce que les billes se séparent et récupérez l’éluat dans une nouvelle plaque PCR pour le contrôle qualité de la bibliothèque.
- Effectuez un test TapeStation ou Fragment Analyzer pour évaluer la distribution granulométrique et la concentration de la banque d’ADNc (recommandé : test haute sensibilité TapeStation D1000). Pour plus de détails, consultez les instructions du fabricant. En moyenne, il faut s’attendre à un rendement de 10 ng.
REMARQUE : POINT D’ARRÊT : Le produit PCR peut être conservé à - 20 °C.
4. Séquençage et prétraitement des données
- Séquençage
REMARQUE : La directive suivante s’appliquera à tous les instruments Illumina pour le séquençage à lecture courte. Si l’instrumentation n’est pas disponible, le séquençage peut être effectué par une installation de séquençage externe. D’autres approches de séquençage pourraient également être utilisées. Pour simplifier, nous avons choisi de ne rapporter que la technologie de séquençage la plus utilisée.
REMARQUE : Les étapes suivantes liées à l’utilisation du logiciel décrivent la procédure sur un séquenceur Illumina NovaSeq6000.- Regrouper des bibliothèques indexées de manière unique dans un rapport équimolaire selon les résultats obtenus à l’étape 3.6.8.
- Mesurer la concentration de la piscine finale à l’aide d’un test à haute sensibilité pour calculer la molarité de l’échantillon comme suit :
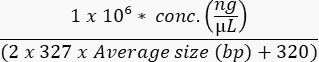
- Charger la cellule d’écoulement selon les spécifications de l’instrument et l’optimisation expérimentale. Le tableau 9 donne des exemples de concentrations de charge d’instruments courants.
- En touchant l’écran, sélectionnez Séquence pour lancer la configuration de l’exécution.
- Suivez les instructions à l’écran et chargez la cellule d’écoulement, la cartouche séquence par synthèse, la cartouche de clustering, la cartouche tampon et assurez-vous que les conteneurs à déchets sont vides.
- Une fois que tous les réactifs ont été reconnus par l’instrument, cliquez sur Run Setup (Exécuter la configuration). Définissez ici le nom de l’exécution et le dossier de sortie pour stocker les données.
- Définissez les détails de séquençage comme appariés avec les deux lectures de 51 pb. Deux lectures d’index sont également séquencées avec 8 pb chacune.
- Appuyez sur Vérifier et après avoir vérifié si tous les détails du séquençage sont corrects, appuyez sur Démarrer l’exécution.
REMARQUE : La profondeur de séquençage recommandée est de 5 x 106 lectures/échantillon, avec un minimum de 1 x 106 lectures/échantillon.
- Pré-traitement des données
- Transformez les données brutes de séquençage au format FASTQ et démultiplexez en fonction des index d’échantillons avec l’outil Bcl2Fastq2. Effectuez le démultiplexage avec les paramètres par défaut. Pour obtenir des instructions détaillées sur Bcl2Fastq2, consultez le manuel de référence.
REMARQUE : La conversion et le démultiplexage FASTQ sont généralement effectués par l’installation de séquençage si le séquençage n’est pas effectué en interne. Les installations de séquençage fournissent généralement un FASTQ démultiplexé pour un traitement ultérieur. - Plusieurs options sont disponibles pour l’alignement des données et la quantification de l’abondance des lectures de séquençage (recommandé : pipeline de séquençage de l’ARN nf-core (https://nf-co.re/rnaseq)). Le pipeline offre plusieurs options, utilisez le paramètre par défaut avec STAR8 comme aligné et Salmon9 pour quantifier l’abondance du transcrit.
- REMARQUE : Pour une analyse bioinformatique plus poussée, plusieurs méthodes sont disponibles. Il n’entre pas dans le cadre de ce protocole de couvrir tous les (Recommandé : DEseq2 pipeline10). Un script standard basé sur le workflow DEseq2 que nous avons développé est disponible sur GitHub (https://github.com/jsschrepping/RNA-DESeq2).
- Transformez les données brutes de séquençage au format FASTQ et démultiplexez en fonction des index d’échantillons avec l’outil Bcl2Fastq2. Effectuez le démultiplexage avec les paramètres par défaut. Pour obtenir des instructions détaillées sur Bcl2Fastq2, consultez le manuel de référence.
Résultats
Suivant le protocole rapporté, les PBMC humains ont été ensemencés, traités avec différents médicaments immunomodulateurs et, après différents temps d’incubation, récoltés pour une analyse transcriptomique en vrac à l’aide du protocole de séquençage (Figure 1).
Les concentrations idéales de médicaments et les temps d’incubation des composés d’essai doivent être identifiés en amont de ce protocole à l’aide de stratégies expérimentale...
Discussion
La découverte et le développement de médicaments peuvent grandement bénéficier de la vision holistique des processus cellulaires que la transcriptomique de masse peut fournir. Néanmoins, cette approche est souvent limitée par le coût élevé de l’expérience avec le protocole standard de séquençage de l’ARN en vrac, ce qui interdit son application dans les milieux académiques ainsi que son potentiel d’évolutivité industrielle.
Les étapes les plus critiques du protocole sont...
Déclarations de divulgation
Les auteurs ne déclarent pas d’intérêts concurrents.
Remerciements
J.L.S. est soutenu par la Fondation allemande pour la recherche (DFG) dans le cadre de la stratégie d’excellence de l’Allemagne (EXC2151-390873048), ainsi que dans le cadre de la SCHU 950/8-1 ; GRK 2168, TP11 ; CRC SFB 1454 Metaflammation, IRTG GRK 2168, WGGC INST 216/981-1, CCU INST 217/988-1, le projet d’excellence Diet-Body-Brain (DietBB) financé par le BMBF ; et le projet européen SYSCID sous le numéro de subvention 733100. M.B. est soutenu par DFG (IRTG2168-272482170, SFB1454-432325352). L.B. est soutenu par la DFG (ImmuDiet BO 6228/2-1 - numéro de projet 513977171) et la stratégie d’excellence de l’Allemagne (EXC2151-390873048). Images créées avec BioRender.com.
matériels
| Name | Company | Catalog Number | Comments |
| 50 mL conical tube | fisher scientific | 10203001 | |
| Adhesive PCR Plate Seals | Thermo Fisher Scientific | AB0558 | |
| Amplicon Tagment Mix (ATM) | Illumina | FC-131-1096 | Nextera XT DNA Library Prep Kit (96 samples) |
| AMPure XP beads | Beckman Coulter | A 63881 | |
| Betaine | Sigma-Aldrich | 61962 | |
| Cell culture grade 96-well plates | Thermo Fisher Scientific | 260860 | |
| Cell culture vacuum pump (VACUSAFE) | Integra Bioscience | 158300 | |
| Deoxynucleotide triphosphates (dNTPs) mix 10 mM each | Fermentas | R0192 | |
| DMSO | Sigma-Aldrich | 276855 | |
| DTT (100 mM) | Invitrogen | 18064-014 | |
| EDTA | Sigma-Aldrich | 798681 | for adherent cells |
| Ethanol | Sigma-Aldrich | 51976 | |
| Fetal Bovine Serum | Thermo Fisher Scientific | 26140079 | |
| Filter tips (10 µL) | Gilson | F171203 | |
| Filter tips (100 µL) | Gilson | F171403 | |
| Filter tips (20 µL) | Gilson | F171303 | |
| Filter tips (200 µL) | Gilson | F171503 | |
| Guanidine Hydrochloride | Sigma-Aldrich | G3272 | |
| ISPCR primer (10 µM) | Biomers.net GmbH | SP10006 | 5′-AAGCAGTGGTATCAACGCAGAG T-3′ |
| KAPA HiFi HotStart ReadyMix (2X) | KAPA Biosystems | KK2601 | |
| Magnesium chloride (MgCl2) | Sigma-Aldrich | M8266 | |
| Magnetic stand 96 | Ambion | AM10027 | |
| Neutralize Tagment (NT) Buffer | Illumina | FC-131-1096 | Nextera XT DNA Library Prep Kit (96 samples), alternatively 0.2 % SDS |
| Nextera-compatible indexing primer | Illumina | ||
| Nuclease-free water | Invitrogen | 10977049 | |
| PBS | Thermo Fisher Scientific | AM9624 | |
| PCR 96-well plates | Thermo Fisher Scientific | AB0600 | |
| PCR plate sealer | Thermo Fisher Scientific | HSF0031 | |
| Penicillin / Streptomycin | Thermo Fisher Scientific | 15070063 | |
| Qubit 4 fluorometer | Invitrogen | 15723679 | |
| Recombinant RNase inhibitor (40 U/ul) | TAKARA | 2313A | |
| RPMI-1640 cell culture medium | Gibco | 61870036 | If not working with PBMCs, adjust to cell type |
| SMART dT30VN primer | Sigma-Aldrich | 5' Bio-AAGCAGTGGTATCAACGCAGAG TACT30VN-3 | |
| Standard lab equipment | various | various | e.g. centrifuge, ice machine, ice bucket, distilled water, water bath |
| SuperScript II Reverse Transcriptase (SSRT II) | Thermo Fisher Scientific | 18064-014 | |
| SuperScript II Reverse Transcriptase (SSRT II) buffer (5x) | Thermo Fisher Scientific | 18064-014 | |
| Tagment DNA Buffer (TD) | Illumina | FC-131-1096 | Nextera XT DNA Library Prep Kit (96 samples) |
| TapeStation system 4200 | Agilent | G2991BA | |
| Thermocycler (S1000) | Bio-Rad | 1852148 | |
| TSO-LNA (100 uM) | Eurogentec | 5' Biotin AAGCAGTGGTATCAACGCAGAG TACAT(G)(G){G | |
| Vortex-Genie 2 Mixer | Sigma-Aldrich | Z258415 |
Références
- Hughes, J. P., Rees, S., Kalindjian, S. B., Philpott, K. L. Principles of early drug discovery. Br J Pharmacol. 162 (6), 1239-1249 (2011).
- Yang, X., et al. High-throughput transcriptome profiling in drug and biomarker discovery. Front Genet. 11, 19 (2020).
- Bonaguro, L., et al. A guide to systems-level immunomics. Nat Immunol. 23 (10), 1412-1423 (2022).
- Carraro, C., et al. Decoding mechanism of action and sensitivity to drug candidates from integrated transcriptome and chromatin state. ELife. 11, 78012 (2022).
- Picelli, S., et al. Smart-seq2 for sensitive full-length transcriptome profiling in single cells. Nat Methods. 10 (11), 1096-1098 (2013).
- Picelli, S., et al. Full-length RNA-seq from single cells using Smart-seq2. Nat Protoc. 9 (1), 171-181 (2014).
- De Domenico, E., et al. Optimized workflow for single-cell transcriptomics on infectious diseases including COVID-19. STAR Protoc. 1 (3), 100233 (2020).
- Dobin, A., et al. ultrafast universal RNA-seq aligner. Bioinformatics. 29 (1), 15-21 (2013).
- Patro, R., et al. Salmon provides fast and bias-aware quantification of transcript expression. Nat Methods. 14 (4), 417-419 (2017).
- Love, M. I., Huber, W., Anders, S. Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2. Genome Biol. 15 (12), 550 (2014).
- Frankish, A., et al. GENCODE reference annotation for the human and mouse genomes. Nucleic Acids Res. 47, D766-D773 (2019).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon