Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Экономичный транскриптомный скрининг лекарственных препаратов
В этой статье
Резюме
Этот протокол описывает рабочий процесс от культивирования клеток ex vivo или in vitro до предварительной обработки транскриптомных данных для экономически эффективного скрининга лекарств на основе транскриптома.
Аннотация
Транскриптомика позволяет получить всестороннее представление о клеточных программах и их реакциях на возмущения. Несмотря на значительное снижение затрат на библиотечное производство и секвенирование в последнее десятилетие, применение этих технологий в масштабах, необходимых для скрининга лекарств, остается непомерно дорогим, что препятствует огромному потенциалу этих методов. В нашем исследовании представлена экономически эффективная система скрининга лекарственных средств на основе транскриптома, сочетающая миниатюрные культуры возмущений с мини-объемной транскриптомикой. Оптимизированный протокол mini-bulk обеспечивает информативные биологические сигналы на экономически эффективной глубине секвенирования, что позволяет проводить обширный скрининг известных лекарств и новых молекул. В зависимости от выбранного лечения и инкубационного времени, этот протокол приведет к секвенированию библиотек в течение примерно 2 дней. Благодаря наличию нескольких остановочных точек в рамках этого протокола, подготовка библиотеки, а также секвенирование могут выполняться независимо от времени. Возможна одновременная обработка большого количества образцов; Измерение до 384 образцов было проведено без потери качества данных. Также не существует известных ограничений по количеству состояний и/или препаратов, несмотря на вариабельность оптимальных сроков инкубации лекарств.
Введение
Разработка новых лекарственных средств представляет собой сложный и трудоемкий процесс, который включает в себя идентификацию потенциальных лекарственных средств и их мишеней, оптимизацию и синтез лекарственных препаратов-кандидатов, а также проверку их эффективности и безопасности в доклинических иклинических испытаниях1. Традиционные методы скрининга лекарственных средств, т.е. систематическая оценка библиотек соединений-кандидатов для терапевтических целей, предполагают использование животных моделей или клеточных анализов для проверки воздействия на конкретные мишени или пути. Несмотря на то, что эти методы были успешны в выявлении лекарств-кандидатов, они часто не давали достаточного представления о сложных молекулярных механизмах, лежащих в основе эффективности лекарств, а также о токсичности и механизмах потенциальных побочных эффектов.
Оценка полногеномных транскрипционных состояний представляет собой мощный подход к преодолению существующих ограничений в скрининге лекарственных препаратов, поскольку она позволяет проводить всестороннюю оценку экспрессии генов в ответ на медикаментозное лечение. Измеряя транскриптомы РНК в масштабах всего генома, экспрессируемые в определенный момент времени, транскриптомика стремится обеспечить целостное представление о транскрипционных изменениях, которые происходят в ответ на лекарственные препараты, включая изменения в паттернах экспрессии генов, альтернативный сплайсинги экспрессию некодирующей РНК. Эта информация может быть использована для определения мишеней для лекарственных препаратов, прогнозирования эффективности и токсичности лекарственных средств, а также оптимизации дозирования и схем лечения.
Одним из ключевых преимуществ сочетания транскриптомики с объективным скринингом лекарственных препаратов является возможность выявления новых мишеней для лекарственных препаратов, которые ранее не рассматривались. Традиционные подходы к скринингу лекарственных средств часто фокусируются на установленных молекулах-мишенях или путях, что затрудняет идентификацию новых мишеней и потенциально приводит к появлению лекарств с непредвиденными побочными эффектами и ограниченной эффективностью. Транскриптомика может преодолеть эти ограничения, предоставляя представление о молекулярных изменениях, которые происходят в ответ на медикаментозное лечение, раскрывая потенциальные мишени или пути, которые, возможно, ранеене рассматривались.
В дополнение к идентификации новых мишеней для лекарств, транскриптомика также может быть использована для прогнозирования эффективности и токсичности лекарств. Анализируя паттерны экспрессии генов, связанные с реакцией на лекарственные препараты, можно разработать биомаркеры, которые можно использовать для прогнозирования реакции пациента на конкретное лекарство или схему лечения. Это также может помочь оптимизировать дозировку лекарств и снизить риск неблагоприятных побочных эффектов4.
Несмотря на потенциальные преимущества, стоимость транскриптомики остается существенным препятствием для ее широкого применения в скрининге лекарственных препаратов. Транскриптомный анализ требует специализированного оборудования, технических знаний и анализа данных, что может затруднить использование транскриптомики в скрининге лекарств для небольших исследовательских групп или организаций с ограниченным финансированием. Тем не менее, стоимость транскриптомики неуклонно снижается, что делает ее более доступной для исследовательских сообществ. Кроме того, достижения в области технологий и методов анализа данных сделали транскриптомику более эффективной и экономичной, что еще больше повысило ее доступность2.
В этом протоколе мы описываем многомерную и исследовательскую систему для скрининга лекарств на основе транскриптома, сочетающую миниатюрные культуры возмущений с мини-объемным транскриптомным анализом 5,6. С помощью этого протокола можно снизить стоимость одного образца до 1/6от текущей стоимости коммерческих растворов для полноразмерного секвенирования мРНК. Протокол требует только стандартного лабораторного оборудования, единственным исключением является использование технологий секвенирования с коротким считыванием, которые могут быть переданы на аутсорсинг, если инструменты для секвенирования отсутствуют в компании. Оптимизированный протокол mini-bulk обеспечивает информационно насыщенные биологические сигналы на экономически эффективной глубине секвенирования, что позволяет проводить обширный скрининг известных лекарств и новых молекул.
Целью эксперимента является скрининг активности лекарственных средств на КПМК в различных биологических контекстах. Этот протокол может быть применен к любому биологическому вопросу, где несколько препаратов должны быть протестированы с транскриптомным считыванием, что дает общее представление о клеточном эффекте лечения.
протокол
Этот протокол соответствует рекомендациям местных комитетов по этике Боннского университета.
1. Приготовление буферов, растворов и оборудования
- Приготовьте растворы и соберите материалы, описанные в Таблице материалов.
- Водяную баню нагреть до 37 °C и прогреть полную питательную среду (RPMI-1640 + 10% фетальной телячьей сыворотки (FCS) + 1% пенициллин/стрептомицин).
- Для сбора клеток используйте ледяной фосфатно-солевой буфер (PBS).
ПРИМЕЧАНИЕ: Поддерживайте чистоту окружающей среды во время работы с клетками, чтобы поддерживать стерильность.
2. Работа с ячейками
ПРИМЕЧАНИЕ: Подробный протокол криоконсервации мононуклеарных клеток периферической крови (МПКМ) из крови человека можно найти в7.
- Размораживание и подсчет клеток
- Извлеките криовиалы из жидкого азота и разморозьте их на водяной бане при температуре 37 °C в течение 2-3 минут, осторожно переворачивая.
- Переложите размороженные клетки в коническую пробирку объемом 50 мл.
- Промойте криовиал 1 мл теплой питательной средой и добавьте этот раствор по каплям в клетки в пробирке (1-я ступень разведения).
- Повторите разведение 1:1 до достижения объема 32 мл (всего 5 разведений с соответственно 1, 2, 4, 8 и 16 мл теплой питательной среды). Добавьте среду по каплям, чтобы уменьшить повреждение клеток, слегка перемешивая коническую трубку.
- Центрифугируйте клеточную суспензию в течение 5 мин (300 x g, 20 °C) и удалите надосадочную жидкость, осторожно опрокинув коническую пробирку одним плавным движением.
- Ресуспендируйте клетки в 3 мл теплой питательной среды и приступайте к подсчету клеток.
- Для подсчета смешайте 10 мкл клеточной суспензии с трипановым синим (разбавление от 1:2 до 1:10 в зависимости от плотности клеточной суспензии), чтобы отличить живые клетки от мертвых во время подсчета. Мертвые или поврежденные клетки будут казаться синими из-за поглощения красителя, в то время как жизненно важные клетки не окрашиваются.
ПРИМЕЧАНИЕ: Трипан синий слегка цитотоксичен, окрашенные клетки не следует хранить более 5 минут. - Подсчитайте количество клеток с помощью автоматического счетчика клеток или счетной камеры, используя 10 мкл окрашенного клеточного раствора.
- При использовании улучшенной клеточной камеры Нойбауэра подсчитайте все клетки в пределах четырех больших квадратов, расположенных по углам, и используйте следующее уравнение для вычисления концентрации клеточной суспензии.
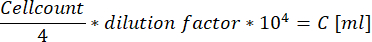
- При использовании улучшенной клеточной камеры Нойбауэра подсчитайте все клетки в пределах четырех больших квадратов, расположенных по углам, и используйте следующее уравнение для вычисления концентрации клеточной суспензии.
- Разбавьте клетки до конечной концентрации 1 x 106 клеток/мл теплой питательной средой. Центрифугу только в том случае, если требуемый объем меньше 3 мл, и ресуспендируйте гранулу ячейки до нужного объема.
- Посев и обработка клеток
- Высевают 100 мкл клеточной суспензии на лунку в 96-луночный планшет для клеточной культуры (1 x 10,5 клеток/лунку).
ПРИМЕЧАНИЕ: Количество клеток для посева было оптимизировано для культур PBMC. В то время как низкая концентрация клеток не даст достаточного количества РНК для секвенирования, чрезмерное количество клеток увеличит риск субоптимального лизиса клеток и ингибирования реакции обратной транскрипции. - Готовят разведения препарата в концентрации, в два раза превышающей ту, которая используется для лечения (2 раза). Выбирайте растворитель в соответствии с растворимостью соединения, предпочтительно полную питательную среду.
ПРИМЕЧАНИЕ: PBS или диметилсульфоксид (ДМСО) можно использовать, если удовлетворительная растворимость не может быть достигнута иным способом. Максимальная концентрация ДМСО в полученном инкубационном объеме не должна превышать 0,5 % для предотвращения цитотоксичности, индуцированной растворителем. - Добавьте 100 мкл 2-кратного разведения препарата (конечная концентрация в лунке 1X) и инкубируйте при 37 °C в течение определенного времени.
ПРИМЕЧАНИЕ: Необходимо провести дополнительные исследования для установления оптимального инкубационного периода препарата и исключения потенциальных механизмов цитотоксичности.
- Высевают 100 мкл клеточной суспензии на лунку в 96-луночный планшет для клеточной культуры (1 x 10,5 клеток/лунку).
- Забор и лизис клеток
- Центрифугируйте планшет для клеточных культур в течение 10 мин (300 x g, 20 °C). Осторожно удалите надосадочную жидкость с помощью вакуумного насоса, удерживая пластину под углом (30°-45°), направляя наконечник в нижний угол лунки, чтобы удалить как можно меньше клеток.
- Промойте ячейки, добавив 200 мкл ледяного PBS в каждую лунку, и центрифугируйте планшет в течение 5 минут (300 x g, 4 °C). Удалите надосадочную жидкость с помощью вакуумного насоса, опять же, держа пластину под острым углом, чтобы удалить как можно больше PBS.
- Подготовьте лизисный буфер, как описано в таблице 1 , для необходимого количества реакций (rxn), добавив 10% избытка.
- Добавьте 15 мкл лизисного буфера в каждую лунку и запечатайте планшет клейкой герметизирующей пленкой, чтобы защитить образцы от загрязнения. Перед центрифугированием вверните планшет в течение 1 минуты (1000 x g, 4 °C) и инкубируйте в течение 5 минут на льду.
- Соберите 6 мкл раствора лизированных клеток в ПЦР-планшет и коротко заморозьте клеточный лизат при -80 °C, чтобы обеспечить оптимальный лизис клеток. Обязательно избегайте многократных циклов замораживания пластин.
ПРИМЕЧАНИЕ: ТОЧКА ОСТАНОВКИ: Если производство кДНК не будет осуществлено впоследствии, клеточный лизат может храниться при -80 °C в течение нескольких месяцев без значительного снижения качества РНК.
3. Подготовка библиотеки к секвенированию
- Реакция обратной транскриптазы (ОТ)
ПРИМЕЧАНИЕ: При работе с РНК поместите все образцы на лед и обязательно используйте безнуклеазное оборудование (стерильную, одноразовую пластиковую посуду) и воду.- Приготовьте реакционную смесь RT, как описано в таблице 2 , для необходимого количества реакций, добавив 10% избытка. Ненадолго взболтайте смесь и быстро раскрутите ее. Держите смесь на льду до использования.
- Разморозьте клеточный лизат при комнатной температуре (RT) и кратковременно открутите его, чтобы собрать весь клеточный лизат на дне пластины.
- Выполните денатурацию мРНК на амплификаторе в соответствии с таблицей 3.
- Выньте пластину из амплификатора и коротко раскрутите ее, чтобы собрать потенциальный конденсат. Добавьте 6 мкл реакционной смеси RT в каждую лунку для получения окончательного объема 12 мкл. Запечатайте пластину, чтобы защитить планшет и избежать испарения, завихрения и вращения.
- Поместите пластину на амплификатор и запустите программу реакции RT в соответствии с таблицей 3.
- Предварительное усиление
- Разморозьте высокоточную ДНК-полимеразу и праймер для ПЦР in situ (ISPCR) при комнатной температуре.
- Приготовьте предварительную амплификационную смесь в соответствии с таблицей 4 для необходимого количества реакций, добавив 10% избытка. Ненадолго взболтайте смесь и раскрутите ее.
ПРИМЕЧАНИЕ: Смесь ферментов стабильна при комнатной температуре в течение нескольких часов. - Раскрутите ПЦР-планшет, добавьте 15 мкл смеси для предварительной амплификации в каждую лунку и запечатайте планшет.
- Поместите его в амплификатор и запустите реакцию предварительного усиления в соответствии с таблицей 5.
- Отрегулируйте количество циклов в зависимости от содержания РНК. Начните с меньшего числа и увеличивайте, если выход кДНК недостаточен.
ПРИМЕЧАНИЕ: По нашему опыту, оптимальное количество циклов должно быть установлено для каждого типа клеток, условия эксперимента и лечение не будут влиять на оптимальное количество циклов. Поэтому мы рекомендуем установить оптимальное количество циклов экспериментально при установлении этого протокола для нового типа клеток. Как правило, первичные клетки требуют большего количества циклов по сравнению с клеточными линиями. ТОЧКА ОСТАНОВКИ: Продукт предварительного усиления можно хранить при температуре -20 °C.
- Отрегулируйте количество циклов в зависимости от содержания РНК. Начните с меньшего числа и увеличивайте, если выход кДНК недостаточен.
- Очистка кДНК и контроль качества (QC)
ПРИМЕЧАНИЕ: Очистка образца может быть выполнена последовательно для каждой пластины, так как образцы на этом этапе довольно стабильны. Увеличение количества образцов приведет к увеличению продолжительности протокола, но не ограничит количество образцов, которые могут быть обработаны одновременно.- Перед началом работы доведите шарики магнитной очистки до комнатной температуры и встряхните на высокой скорости в течение 1 минуты, чтобы полностью ресуспендировать шарики.
- Добавьте 0,8x v/v (20 мкл) магнитных гранул очистки в каждую лунку и инкубируйте при комнатной температуре в течение 5 минут.
- Поместите ПЦР-планшет на магнитную стойку на 5 минут до полного отделения шариков. Осторожно удалите надосадочную жидкость.
- Держа планшет на магнитной стойке, аккуратно добавьте 100 мкл свежеприготовленного 80% этанола, чтобы промыть шарики и инкубировать в течение 30 с. Используйте пипетку небольшого объема, чтобы удалить надосадочную жидкость. Повторите то же самое для дополнительного этапа стирки. Обязательно удалите как можно больше этанола.
- Высушите шарики на магнитной стойке при комнатной температуре до 5 минут, пока этанол полностью не испарится и шарики не перестанут выглядеть блестящими.
- Снимите планшет с магнитного штатива, повторно суспендируйте шарики в 20 мкл воды, не содержащей нуклеаз, и инкубируйте в течение 2 минут.
- Снова поместите пластину на магнитную стойку, пока шарики не разделятся.
- Восстановите элюат в новой ПЦР-пробирке для контроля качества кДНК и мечения.
- Выполните анализ TapeStation или FragmentAnalyser для оценки распределения по размерам и концентрации библиотеки кДНК (рекомендуется: анализ TapeStation D5000). Подробные сведения см. в инструкциях производителя. Типичный урожай составляет 20 нг.
- Маркировка с помощью комплекта
ПРИМЕЧАНИЕ: Другие протоколы Tagmentation могут быть использованы, если они уже установлены.- Разбавляют кДНК до конечной концентрации 150–300 пг/мкл, используя воду, не содержащую нуклеаз.
- Предварительно запрограммируйте амплификатор в соответствии с таблицей 6 , чтобы обеспечить немедленный запуск реакции мечения после добавления кДНК в смесь ферментов.
- Приготовьте смесь для маркировки в соответствии с таблицей 7 для необходимого количества реакций, добавив 10% избытка.
- Распределите 3 мкл смеси для мечения на реакцию на новую ПЦР-планшет и добавьте 1 мкл кДНК в каждую реакцию.
- Реакцию мечения запускают сразу после добавления кДНК.
- Инактивируйте реакцию, добавив 1 мкл нейтрализующего буфера (NT; в качестве альтернативы можно использовать 0,2% додецилсульфат натрия (SDS)).
- ПЦР с обогащением
- Приготовьте обогащение ПЦР-смесью таблицы 7 по числу реакций, добавив 10% избытка. (Рекомендуется: Nextera UDI настроен для предотвращения скачкообразных значений индексов на шаблонных проточных ячейках (например, Illumina NovaSeq 6000)).
- Добавьте 9 мкл обогащенной ПЦР-смеси в каждую реакцию для получения общего объема 14 мкл.
- Запустите программу обогащения ПЦР в соответствии с таблицей 8.
ПРИМЕЧАНИЕ: Для ПЦР с обогащением стандартными являются 16 циклов. Если качество кДНК низкое, могут быть добавлены дополнительные циклы.
- Очистка и контроль качества
- Перед началом работы доведите шарики магнитной очистки до комнатной температуры и встряхните на высокой скорости в течение 1 минуты, чтобы полностью ресуспендировать шарики.
- Добавьте 1,0x v/v (14 мкл) шариков магнитной очистки и инкубируйте при комнатной температуре в течение 5 минут.
- Поместите пластину на магнитную стойку и подождите 5 минут, пока бусины полностью не отделятся. Осторожно удалите надосадочную жидкость.
- Держа планшет на магнитной стойке, аккуратно добавьте 100 мкл свежеприготовленного 80% этанола, чтобы промыть шарики и инкубировать в течение 30 с. Используйте пипетку небольшого объема, чтобы удалить надосадочную жидкость. Повторите то же самое для дополнительного этапа стирки. Обязательно удалите как можно больше этанола.
- Высушите бусины на воздухе при комнатной температуре в течение 3 минут или пока они не перестанут выглядеть блестящими.
- Снимите планшет с магнитного штатива, повторно суспендируйте шарики в 20 мкл воды, не содержащей нуклеаз, и инкубируйте в течение 2 минут.
- Снова поместите планшет на магнитную стойку, пока шарики не разделятся, и извлеките элюат в новую ПЦР-планшет для библиотечного контроля качества.
- Выполните анализ с помощью TapeStation или Fragment Analyzer для оценки распределения по размерам и концентрации библиотеки кДНК (рекомендуется: высокочувствительный анализ TapeStation D1000). Подробные сведения см. в инструкциях производителя. В среднем следует ожидать урожайность 10 нг.
ПРИМЕЧАНИЕ: МЕСТО ОСТАНОВКИ: Продукт ПЦР можно хранить при температуре -20 °C.
4. Секвенирование и предварительная обработка данных
- Секвенирование
ПРИМЕЧАНИЕ: Следующие рекомендации будут применимы ко всем приборам Illumina для секвенирования с коротким считыванием. Если инструментарий недоступен, виртуализация может быть выполнена с помощью внешнего средства виртуализации. Можно использовать и другие подходы к секвенированию. Для простоты мы решили рассказать только о наиболее широко используемой технологии секвенирования.
ПРИМЕЧАНИЕ: Следующие шаги, связанные с использованием программного обеспечения, описывают процедуру на секвенсоре Illumina NovaSeq6000.- Объедините уникально индексированные библиотеки в эквимолярном соотношении в соответствии с результатами, полученными на шаге 3.6.8.
- Измерьте концентрацию конечного бассейна с помощью высокочувствительного анализа, чтобы рассчитать молярность образца следующим образом:
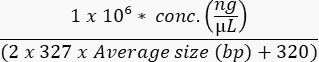
- Загрузите проточную ячейку в соответствии со спецификацией прибора и экспериментальной оптимизацией. Примеры нагрузочных концентраций распространенных приборов приведены в таблице 9.
- Коснувшись экрана, выберите Последовательность , чтобы начать настройку запуска.
- Следуйте инструкциям на экране и загрузочной ячейке, картридже последовательности синтеза, кластерном картридже, буферном картридже и убедитесь, что контейнеры для отходов пусты.
- После того, как все реагенты будут распознаны прибором, нажмите кнопку Run Setup. Здесь определите имя запуска и выходную папку для хранения данных.
- Определите детали последовательности как парные с обоими чтениями 51.н. Два считывания индекса также секвенируются по 8.н. каждое.
- Нажмите «Обзор» и, проверив правильность всех сведений о последовательности, нажмите «Начать выполнение».
ПРИМЕЧАНИЕ: Рекомендуемая глубина секвенирования составляет 5 x 106 прочтений на выборку, при этом минимальная глубина секвенирования составляет 1 x 106 прочтений на выборку.
- Предварительная обработка данных
- Преобразуйте необработанные данные секвенирования в формат FASTQ и демультиплексируйте в соответствии с индексами выборки с помощью инструмента Bcl2Fastq2. Выполните демультиплексирование с настройками по умолчанию. Подробные инструкции по Bcl2Fastq2 см. в справочном руководстве.
ПРИМЕЧАНИЕ: Преобразование и демультиплексирование FASTQ обычно выполняются на предприятии секвенирования, если секвенирование не выполняется собственными силами. Оборудование для секвенирования, как правило, предоставляет демультиплексированный FASTQ для дальнейшей обработки. - Существует несколько вариантов выравнивания данных и количественной оценки количества считываний секвенирования (рекомендуется: nf-core RNA-seq pipeline (https://nf-co.re/rnaseq)). Конвейер предоставляет несколько вариантов: используйте настройку по умолчанию со звездой8 в качестве выравнивания и Salmon9 для количественной оценки распространенности транскрипции.
- ПРИМЕЧАНИЕ: Для дальнейшего биоинформатического анализа доступно несколько методов. В рамки данного протокола не входит охват всех (рекомендуется: конвейер DEseq210). Стандартный скрипт на основе разработанного нами расчетного процесса DEseq2 можно найти на GitHub (https://github.com/jsschrepping/RNA-DESeq2).
- Преобразуйте необработанные данные секвенирования в формат FASTQ и демультиплексируйте в соответствии с индексами выборки с помощью инструмента Bcl2Fastq2. Выполните демультиплексирование с настройками по умолчанию. Подробные инструкции по Bcl2Fastq2 см. в справочном руководстве.
Результаты
В соответствии с описанным протоколом ПБМК человека высевали, обрабатывали различными иммуномодулирующими препаратами и после разного инкубационного периода собирали для массового транскриптомного анализа с использованием протокола секвенирования (рис. 1).
Обсуждение
Открытие и разработка лекарств могут значительно выиграть от целостного взгляда на клеточные процессы, который может обеспечить массовая транскриптомика. Тем не менее, этот подход часто ограничен высокой стоимостью эксперимента со стандартным протоколом массового секвенирования РН...
Раскрытие информации
Авторы заявляют об отсутствии конкурирующих интересов.
Благодарности
J.L.S. поддерживается Немецким научно-исследовательским обществом (DFG) в рамках Стратегии совершенствования Германии (EXC2151-390873048), а также в соответствии с SCHU 950/8-1; ГРК 2168, ТП11; CRC SFB 1454 Metaflammation, IRTG GRK 2168, WGGC INST 216/981-1, CCU INST 217/988-1, финансируемый BMBF проект передового опыта «Диета-Тело-Мозг» (DietBB); и проект ЕС SYSCID по гранту No 733100. М.Б. поддерживается DFG (IRTG2168-272482170, SFB1454-432325352). L.B. поддерживается DFG (ImmuDiet BO 6228/2-1 - номер проекта 513977171) и Стратегией превосходства Германии (EXC2151-390873048). Изображения, созданные с помощью BioRender.com.
Материалы
| Name | Company | Catalog Number | Comments |
| 50 mL conical tube | fisher scientific | 10203001 | |
| Adhesive PCR Plate Seals | Thermo Fisher Scientific | AB0558 | |
| Amplicon Tagment Mix (ATM) | Illumina | FC-131-1096 | Nextera XT DNA Library Prep Kit (96 samples) |
| AMPure XP beads | Beckman Coulter | A 63881 | |
| Betaine | Sigma-Aldrich | 61962 | |
| Cell culture grade 96-well plates | Thermo Fisher Scientific | 260860 | |
| Cell culture vacuum pump (VACUSAFE) | Integra Bioscience | 158300 | |
| Deoxynucleotide triphosphates (dNTPs) mix 10 mM each | Fermentas | R0192 | |
| DMSO | Sigma-Aldrich | 276855 | |
| DTT (100 mM) | Invitrogen | 18064-014 | |
| EDTA | Sigma-Aldrich | 798681 | for adherent cells |
| Ethanol | Sigma-Aldrich | 51976 | |
| Fetal Bovine Serum | Thermo Fisher Scientific | 26140079 | |
| Filter tips (10 µL) | Gilson | F171203 | |
| Filter tips (100 µL) | Gilson | F171403 | |
| Filter tips (20 µL) | Gilson | F171303 | |
| Filter tips (200 µL) | Gilson | F171503 | |
| Guanidine Hydrochloride | Sigma-Aldrich | G3272 | |
| ISPCR primer (10 µM) | Biomers.net GmbH | SP10006 | 5′-AAGCAGTGGTATCAACGCAGAG T-3′ |
| KAPA HiFi HotStart ReadyMix (2X) | KAPA Biosystems | KK2601 | |
| Magnesium chloride (MgCl2) | Sigma-Aldrich | M8266 | |
| Magnetic stand 96 | Ambion | AM10027 | |
| Neutralize Tagment (NT) Buffer | Illumina | FC-131-1096 | Nextera XT DNA Library Prep Kit (96 samples), alternatively 0.2 % SDS |
| Nextera-compatible indexing primer | Illumina | ||
| Nuclease-free water | Invitrogen | 10977049 | |
| PBS | Thermo Fisher Scientific | AM9624 | |
| PCR 96-well plates | Thermo Fisher Scientific | AB0600 | |
| PCR plate sealer | Thermo Fisher Scientific | HSF0031 | |
| Penicillin / Streptomycin | Thermo Fisher Scientific | 15070063 | |
| Qubit 4 fluorometer | Invitrogen | 15723679 | |
| Recombinant RNase inhibitor (40 U/ul) | TAKARA | 2313A | |
| RPMI-1640 cell culture medium | Gibco | 61870036 | If not working with PBMCs, adjust to cell type |
| SMART dT30VN primer | Sigma-Aldrich | 5' Bio-AAGCAGTGGTATCAACGCAGAG TACT30VN-3 | |
| Standard lab equipment | various | various | e.g. centrifuge, ice machine, ice bucket, distilled water, water bath |
| SuperScript II Reverse Transcriptase (SSRT II) | Thermo Fisher Scientific | 18064-014 | |
| SuperScript II Reverse Transcriptase (SSRT II) buffer (5x) | Thermo Fisher Scientific | 18064-014 | |
| Tagment DNA Buffer (TD) | Illumina | FC-131-1096 | Nextera XT DNA Library Prep Kit (96 samples) |
| TapeStation system 4200 | Agilent | G2991BA | |
| Thermocycler (S1000) | Bio-Rad | 1852148 | |
| TSO-LNA (100 uM) | Eurogentec | 5' Biotin AAGCAGTGGTATCAACGCAGAG TACAT(G)(G){G | |
| Vortex-Genie 2 Mixer | Sigma-Aldrich | Z258415 |
Ссылки
- Hughes, J. P., Rees, S., Kalindjian, S. B., Philpott, K. L. Principles of early drug discovery. Br J Pharmacol. 162 (6), 1239-1249 (2011).
- Yang, X., et al. High-throughput transcriptome profiling in drug and biomarker discovery. Front Genet. 11, 19 (2020).
- Bonaguro, L., et al. A guide to systems-level immunomics. Nat Immunol. 23 (10), 1412-1423 (2022).
- Carraro, C., et al. Decoding mechanism of action and sensitivity to drug candidates from integrated transcriptome and chromatin state. ELife. 11, 78012 (2022).
- Picelli, S., et al. Smart-seq2 for sensitive full-length transcriptome profiling in single cells. Nat Methods. 10 (11), 1096-1098 (2013).
- Picelli, S., et al. Full-length RNA-seq from single cells using Smart-seq2. Nat Protoc. 9 (1), 171-181 (2014).
- De Domenico, E., et al. Optimized workflow for single-cell transcriptomics on infectious diseases including COVID-19. STAR Protoc. 1 (3), 100233 (2020).
- Dobin, A., et al. ultrafast universal RNA-seq aligner. Bioinformatics. 29 (1), 15-21 (2013).
- Patro, R., et al. Salmon provides fast and bias-aware quantification of transcript expression. Nat Methods. 14 (4), 417-419 (2017).
- Love, M. I., Huber, W., Anders, S. Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2. Genome Biol. 15 (12), 550 (2014).
- Frankish, A., et al. GENCODE reference annotation for the human and mouse genomes. Nucleic Acids Res. 47, D766-D773 (2019).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены