È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Studio comparativo di matrici di membrana basale per il mantenimento di cellule staminali umane e la generazione di organoidi intestinali
In questo articolo
Riepilogo
Gli organoidi sono diventati strumenti preziosi per la modellazione delle malattie. La matrice extracellulare (ECM) guida il destino cellulare durante la generazione degli organoidi e l'utilizzo di un sistema che assomiglia al tessuto nativo può migliorare l'accuratezza del modello. Questo studio confronta la generazione di organoidi intestinali umani derivati da cellule staminali pluripotenti indotte in ECM di origine animale e idrogel privi di xeno.
Abstract
La matrice extracellulare (ECM) svolge un ruolo fondamentale nel comportamento e nello sviluppo cellulare. Gli organoidi generati da cellule staminali pluripotenti indotte umane (hiPSC) sono sotto i riflettori di molte aree di ricerca. Tuttavia, la mancanza di segnali fisiologici nei materiali classici per colture cellulari ostacola un'efficiente differenziazione delle iPSC. L'integrazione della MEC disponibile in commercio nella coltura di cellule staminali fornisce spunti fisici e chimici utili per il mantenimento delle cellule. I prodotti di membrana basale di origine animale disponibili in commercio sono composti da proteine ECM e fattori di crescita che supportano il mantenimento cellulare. Poiché la ECM possiede proprietà tessuto-specifiche in grado di modulare il destino cellulare, le matrici prive di xeno vengono utilizzate per accelerare la traduzione verso gli studi clinici. Sebbene le matrici disponibili in commercio siano ampiamente utilizzate nel lavoro su hiPSC e organoidi, l'equivalenza di queste matrici non è stata ancora valutata. Qui, è stato condotto uno studio comparativo del mantenimento dell'hiPSC e della generazione di organoidi intestinali umani (hIO) in quattro diverse matrici: Matrigel (Matrix 1-AB), Geltrex (Matrix 2-AB), Cultrex (Matrix 3-AB) e VitroGel (Matrix 4-XF). Sebbene le colonie mancassero di una forma perfettamente rotonda, c'era una differenziazione spontanea minima, con oltre l'85% delle cellule che esprimevano il marcatore delle cellule staminali SSEA-4. La matrice 4-XF ha portato alla formazione di grumi rotondi 3D. Inoltre, l'aumento della concentrazione di integratori e fattori di crescita nei terreni utilizzati per produrre la soluzione di idrogel Matrix 4-XF ha migliorato l'espressione di SSEA-4 da parte di hiPSC di 1,3 volte. La differenziazione della hiPSC mantenuta con Matrix 2-AB ha portato a un minor numero di rilasci di sferoidi durante lo stadio medio/posteriore dell'intestino rispetto alle altre membrane basali di origine animale. Rispetto ad altre, la matrice di organoidi xeno-free (Matrix 4-O3) porta a hIO più grandi e più mature, suggerendo che le proprietà fisiche degli idrogel xeno-free possono essere sfruttate per ottimizzare la generazione di organoidi. Nel complesso, i risultati suggeriscono che le variazioni nella composizione di diverse matrici influenzano le fasi di differenziazione dell'IO. Questo studio aumenta la consapevolezza sulle differenze nelle matrici disponibili in commercio e fornisce una guida per l'ottimizzazione delle matrici durante il lavoro iPSC e IO.
Introduzione
La matrice extracellulare (ECM) è un componente dinamico e multifunzionale dei tessuti che svolge un ruolo centrale nella regolazione del comportamento e dello sviluppo cellulare. Essendo una rete complessa, fornisce supporto strutturale, ligandi adesivi cellulari1 e immagazzinamento di fattori di crescita e citochine che regolano la segnalazione cellulare. Ad esempio, durante la guarigione delle ferite, la MEC funge da impalcatura per le cellule in migrazione e da serbatoio di fattori di crescita coinvolti nella riparazione dei tessuti2. Allo stesso modo, la disregolazione nella MEC può portare ad un aumento della gravità di varie malattie come la fibrosi e il cancro 3,4. Durante lo sviluppo embrionale, la MEC guida la morfogenesi dei tessuti. Ad esempio, nello sviluppo del cuore, i componenti della MEC svolgono un ruolo nella creazione della corretta architettura e funzione del tessuto cardiaco5. Oltre un decennio di ricerca ha dimostrato che la rigidità del microambienteda sola 6,7 può controllare la specifica del lignaggio delle cellule staminali. Pertanto, non sorprende che durante la differenziazione cellulare in vitro, la ECM influenzi il destino delle cellule staminali fornendo segnali per la differenziazione.
Gli organoidi possono essere generati da cellule staminali pluripotenti indotte (iPSC). Per generare con successo gli organoidi è necessario partire da una linea iPSC correttamente caratterizzata. Tuttavia, la mancanza di segnali fisiologici nei materiali classici per colture cellulari ostacola l'efficiente differenziazione delle iPSC e la generazione di organoidi. Inoltre, recenti ricerche hanno sottolineato l'importanza della composizione della matrice extracellulare (ECM), delle interazioni tra le cellule e la ECM8, nonché dei segnali meccanici e geometrici 9,10,11 nel contesto dell'espansione e del differenziamento degli organoidi12. L'avanzamento della tecnologia degli organoidi attraverso il miglioramento della riproducibilità comporterà l'incorporazione di segnali fisici e chimici specifici del tessuto.
Gli organoidi mirano a ricapitolare il tessuto nativo all'interno di un microambiente fisiologicamente simile. La scelta di un sistema ECM che imiti fedelmente l'ECM del tessuto nativo è fondamentale per ottenere una rilevanza fisiologica per quanto riguarda il comportamento, la funzione e la risposta delle cellule agli stimoli13. La scelta dei componenti della ECM può influenzare la differenziazione delle cellule staminali in tipi cellulari specifici all'interno dell'organoide. Diverse proteine della ECM e le loro combinazioni possono fornire indizi che guidano il destino cellulare14. Ad esempio, gli studi hanno dimostrato che l'utilizzo di componenti specifici della MEC può promuovere la differenziazione delle cellule staminali intestinali in tipi di cellule intestinali mature, dando vita a organoidi intestinali fisiologicamente rilevanti15. Sebbene gli organoidi siano uno strumento prezioso durante la modellazione delle malattie e i test farmacologici, la selezione di un sistema ECM appropriato è fondamentale per questa applicazione. Un sistema ECM appropriato può migliorare l'accuratezza della modellazione della malattia creando un microambiente che assomiglia al tessuto interessato16. Inoltre, la MEC tessuto-specifica può aiutare a generare organoidi che ricapitolano meglio i fenotipi associati alla malattia e le risposte ai farmaci17. L'ottimizzazione del sistema ECM utilizzato nella differenziazione degli organoidi è fondamentale per ottenere i risultati di differenziazione desiderati.
I sistemi di membrane basali disponibili in commercio derivati da fonti animali di ECM (ad esempio, Matrigel, Cultrex) e idrogel privo di xeno (ad esempio, VitroGel) sono ampiamente utilizzati nella ricerca su iPSC e organoidi. Le aziende che li commercializzano e i ricercatori che li utilizzano hanno elaborato molte istruzioni per i loro prodotti e applicazioni specifici nel corso degli anni. Molte di queste istruzioni sono servite come guida per la generazione di questo protocollo. Inoltre, i benefici e le battute d'arresto associati alle loro proprietà intrinseche sono stati notati individualmente da molti 18,19,20,21. Tuttavia, non esiste un flusso di lavoro sistematico che guidi la selezione dei sistemi ottimali per il lavoro con iPSC e organoidi. In questo caso, viene fornito un flusso di lavoro per valutare sistematicamente l'equivalenza dei sistemi ECM provenienti da varie fonti per il lavoro con iPSC e organoidi. Questo è uno studio comparativo del mantenimento di due diverse linee di iPSC umane (hiPSC) e della generazione di organoidi intestinali umani (hIO) in quattro diverse matrici: Matrigel (Matrix 1-AB), Geltrex (Matrix 2-AB), Cultrex (Matrix 3-AB) e VitroGel (Matrix 4-XF). Per la coltura di organoidi, sono state utilizzate quattro versioni di VitroGel a matrice priva di xeno precedentemente ottimizzate per la coltura di organoidi: ORGANOID 1 (Matrix 4-O1), ORGANOID 2 (Matrix 4-O2), ORGANOID 3 (Matrix 4-O3), ORGANOID 4 (Matrix 4-O4). Inoltre, sono state utilizzate matrici di origine animale ottimizzate per organoidi: Matrigel High Concentration (Matrix 1-ABO) e Cultrex Type 2 (Matrix 3-ABO). Sono stati utilizzati terreni di coltura di cellule staminali disponibili in commercio (mTeSR Plus) e kit di differenziazione degli organoidi (STEMdiff intestinal organoid kit). Questo protocollo combina le istruzioni individuali dei produttori dei prodotti con le esperienze di laboratorio per guidare il lettore verso un'ottimizzazione di successo dell'ECM per il loro specifico lavoro iPSC e organoide. Nel complesso, questo protocollo e i risultati rappresentativi sottolineano l'importanza di selezionare il microambiente ottimale per il lavoro sulle cellule staminali e la differenziazione degli organoidi.
Access restricted. Please log in or start a trial to view this content.
Protocollo
1. Manutenzione hiPSC
ATTENZIONE: Tutto il lavoro viene svolto in una cabina di biosicurezza (BSC) seguendo tecniche asettiche standard. Deve seguire gli standard di sicurezza OSHA per i laboratori, incluso l'uso corretto di dispositivi di protezione individuale come camici da laboratorio, guanti e occhiali.
- Preparazione di matrici, aliquote e terreni di coltura cellulare
- Per membrane basali di derivazione animale (BMs; Matrice 1-AB, Matrice 2-AB, Matrice 3-AB), preparare aliquote dei volumi di lavoro seguendo le raccomandazioni del produttore riassunte nella Tabella 1 e conservarle a -20 °C o -80 °C per la conservazione a lungo termine. Evitare la formazione di bolle. Se si formano delle bolle, prima di conservarle, centrifugare i campioni a 4 °C, a <200 x g, per ~1-2 minuti per forzare la bolla a venire in superficie.
NOTA: La creazione di aliquote monouso aiuta a evitare ripetuti cicli di congelamento-scongelamento che interrompono l'architettura ECM. Poiché la concentrazione di BM varia in base al numero di lotto, assicurarsi di seguire le raccomandazioni del produttore per preparare aliquote monouso e produrre soluzioni di rivestimento. I BM ad alta concentrazione sono viscosi e difficili da pipettare; utilizzare punte fredde precedentemente conservate a -20 °C. - Preparare il terreno di coltura con cellule staminali seguendo le raccomandazioni del produttore. Per preparare il terreno completo specifico disponibile in commercio utilizzato in questo protocollo, aggiungere 100 ml di cocktail di integratori 5x a 400 ml di terreno basale. Quindi, miscelare accuratamente il terreno e aliquotare in volumi da 40 mL, conservati a 20 °C. Per l'uso, scongelare ogni aliquota di terreno completo, utilizzare immediatamente il mezzo completo aliquotato o conservare a 2 - 8 °C per un massimo di 2 settimane. Non ricongelarlo.
- Preparare aliquote del terreno completo di cellule staminali contenente 3 volte la concentrazione del cocktail di integratori per preparare l'idrogel Matrix 4-XE, cioè diluire l'integratore da 5 a 3 volte.
- Per membrane basali di derivazione animale (BMs; Matrice 1-AB, Matrice 2-AB, Matrice 3-AB), preparare aliquote dei volumi di lavoro seguendo le raccomandazioni del produttore riassunte nella Tabella 1 e conservarle a -20 °C o -80 °C per la conservazione a lungo termine. Evitare la formazione di bolle. Se si formano delle bolle, prima di conservarle, centrifugare i campioni a 4 °C, a <200 x g, per ~1-2 minuti per forzare la bolla a venire in superficie.
- Rivestimento di plastica per colture tissutali con BM di origine animale (Matrix 1-3AB)
- Preparare quanto segue prima di iniziare: tenere sempre le matrici nel ghiaccio durante lo scongelamento e la manipolazione per evitare che si gelifichino. Utilizzare terreni freddi per preparare le matrici diluite. Preparare un numero sufficiente di pozzetti di replicazione per condizione per analizzare le cellule prima di iniziare e in ogni fase del processo di differenziazione di hiPSC e generazione di organoidi hIO.
NOTA: In questo caso, per questo studio sono state utilizzate piastre a 24 pozzetti. Fare riferimento alla Tabella 2 per i volumi di rivestimento consigliati per altre dimensioni di lastre. - Per ogni tipo di matrice, preparare 25 mL di ADE a freddo DMEM/F-12 contenente 15 mM di HEPES in una provetta conica da 50 mL. Tienili sul ghiaccio.
- Aggiungere le matrici scongelate al rispettivo avanzamento a freddo DMEM/F-12 e mescolare bene. Mantenere il supporto freddo durante il processo di miscelazione. Controllare visivamente la miscelazione omogenea assicurandosi che non ci siano grumi.
- Utilizzare immediatamente le soluzioni di matrici diluite per rivestire ogni pozzetto selezionato per l'uso (250 μL/pozzetto se si utilizzano piastre da 24 pozzetti).
- Inclinare delicatamente la coltura per distribuire uniformemente la soluzione di rivestimento sulla superficie. Incubare a temperatura ambiente (15 - 25 °C) per almeno 1 ora prima dell'uso.
NOTA: Se non viene utilizzata immediatamente, la lastra può essere conservata a 2 - 8 °C per un massimo di 1 settimana dopo il rivestimento, ma deve essere sigillata con una pellicola trasparente per evitare l'evaporazione. Quando si utilizzano le piastre conservate, lasciarle raggiungere la temperatura ambiente (15 - 25 °C) per 30 minuti prima di procedere al passaggio successivo. - Rimuovere delicatamente la soluzione in eccesso utilizzando una pipetta sierologica o mediante aspirazione. Assicurarsi che la superficie rivestita non sia graffiata.
- Aggiungere immediatamente un terreno di coltura di cellule staminali completo caldo (50% del volume totale necessario per un pozzetto specifico, ad es. 250 μL/pozzetto se si utilizza una piastra a 24 pozzetti).
- Preparare quanto segue prima di iniziare: tenere sempre le matrici nel ghiaccio durante lo scongelamento e la manipolazione per evitare che si gelifichino. Utilizzare terreni freddi per preparare le matrici diluite. Preparare un numero sufficiente di pozzetti di replicazione per condizione per analizzare le cellule prima di iniziare e in ogni fase del processo di differenziazione di hiPSC e generazione di organoidi hIO.
- Rivestimento di plastica per colture tissutali con idrogel Matrix 4-XE
- Prima di iniziare, preparare quanto segue: estrarre l'idrogel Matrix 4-XE dal frigorifero e lasciarlo riscaldare a temperatura ambiente (25 °C). Preparare un numero sufficiente di pozzetti di replicazione per condizione per analizzare le cellule prima di iniziare e in ogni fase del processo di differenziazione di hiPSC e generazione di organoidi hIO.
NOTA: In questo caso, per questo studio sono state utilizzate piastre a 24 pozzetti. Fare riferimento alla Tabella 3 per i volumi di rivestimento consigliati per altre dimensioni di lastre. - Preparare un terreno di coltura completo di cellule staminali contenente una concentrazione di fattore di crescita 3 volte superiore rispetto alla formulazione standard per la coltura di cellule staminali (terreno di coltura con cellule staminali 3 volte).
- Miscelare l'idrogel Matrix 4-XE e il terreno di coltura con cellule staminali 3x a un rapporto di miscelazione 2:1 v/v e pipettare delicatamente su e giù 5x-10x per miscelare accuratamente.
- Trasferire la miscela di idrogel in una piastra a fontana e inclinare con cautela la coltura per distribuire uniformemente la miscela sulla superficie. Utilizzare 250 μL/pozzetto se si utilizza una piastra a 24 pozzetti. Vedere la Tabella 3 per i volumi consigliati.
- Attendere 10-15 minuti a temperatura ambiente per la formazione di un gel morbido. Durante il processo di formazione dell'idrogel, non interrompere l'idrogel inclinando o scuotendo la piastra.
- Prima di iniziare, preparare quanto segue: estrarre l'idrogel Matrix 4-XE dal frigorifero e lasciarlo riscaldare a temperatura ambiente (25 °C). Preparare un numero sufficiente di pozzetti di replicazione per condizione per analizzare le cellule prima di iniziare e in ogni fase del processo di differenziazione di hiPSC e generazione di organoidi hIO.
- passaggio e semina di grumi senza enzimi hiPSC
- Preparare quanto segue prima di iniziare: almeno 1 ora prima del passaggio, rivestire la plastica desiderata con matrici. Aliquotare una quantità sufficiente di terreno di coltura di cellule staminali complete e riscaldarlo a temperatura ambiente (15 - 25 °C). Evitare più cicli di riscaldamento per l'intero fluido.
NOTA: I passaggi seguenti descrivono il passaggio di una coltura confluente già stabilita e >90% di iPSC su una piastra a 6 pozzetti e la loro semina in piastre a 24 pozzetti per la differenziazione di organoidi intestinali. - Sciacquare le celle con 1 mL di D-PBS (senza Ca++ e Mg++) e aspirare. Aggiungere 1 mL di reagente per la selezione di cellule staminali pluripotenti umane prive di enzimi, agitare accuratamente per distribuire uniformemente e aspirare entro 1 minuto. Le colonie devono solo essere esposte a un sottile film di liquido.
- Incubare a 37 °C fino a quando le colonie iniziano a sembrare meno compatte, il che richiederà ~ 3-8 minuti.
NOTA: Il tempo di incubazione ottimale può variare a seconda della linea cellulare utilizzata. Il tempo di incubazione ottimale deve essere determinato quando si passa per la prima volta ciascuna linea cellulare con il reagente per la selezione di cellule staminali pluripotenti umane prive di enzimi. - Aggiungere 1 mL di terreno di coltura completo per cellule staminali. Staccare le colonie picchiettando con decisione sul lato della piastra. Assicurati di tenere il piatto con l'altra mano.
- Con una pipetta da 1 mL o più grande, trasferire la sospensione di grumi cellulari in una provetta conica da 15 mL. Valutare la dimensione dei grumi utilizzando un microscopio a campo chiaro e assicurarsi che la dimensione del grumo di cellule sia compresa tra 50 e 200 μm; Se sono più grandi, scuotere delicatamente il tubo conico per romperli.
- Disegnare una griglia x-y sul fondo di ciascun pozzetto di una piastra a fondo piatto da 96 pozzetti che verrà utilizzata per il conteggio dei grumi e aggiungere 50 μL di D-PBS a ciascuno dei pozzetti. Si consiglia di fare la media dei grumi contati da 3 pozzetti.
- Assicurarsi che i grumi di cellule siano distribuiti uniformemente agitando delicatamente la provetta, quindi trasferire 5 μl di sospensione a grumi in ciascuno dei pozzetti della piastra a 96 pozzetti a fondo piatto.
NOTA: Non è consigliabile utilizzare un contatore automatico di celle per contare i grumi. Si consiglia un conteggio manuale. - Contare il numero totale di grumi in ogni pozzetto che hanno un diametro compreso tra 50 e 200 μm. Se la maggior parte dei grumi di cellule ha un diametro > 200 μm (vedi Figura 1), ripetere i passaggi 1.4.5 - 1.4.7.
- Calcolare il volume (in μL) della sospensione a grumi necessario per seminare 6.000 grumi come segue:
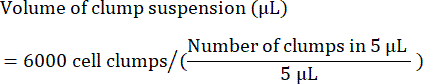
NOTA: La densità di semina ottimale deve essere ottimizzata con ogni linea cellulare. Per determinare la densità di semina ottimale, si consiglia di eseguire una semina iniziale in un intervallo di densità di ciuffi (ad esempio, 4.000, 5.000 e 6.000 grumi per pozzetto) e di controllare 24-48 ore dopo la semina se viene raggiunta la confluenza dell'85%-90%. - Come controllo di qualità, separare un campione di cellule per l'esame di citometria a flusso dei marcatori delle cellule staminali. Fare riferimento alla Tabella 4 per un elenco di marcatori comuni.
- Separare il volume richiesto di sospensione di ciuffi di iPSC da seminare sui pozzetti rivestiti con ECM diversa in provette separate da 15 mL.
- Centrifugare la provetta contenente la soluzione di grumi di iPSC a 200 x g per 5 minuti per rimuovere i terreni contenenti il reagente per la selezione di cellule staminali pluripotenti umane prive di enzimi. In attesa della centrifugazione, aggiungere il 50% del volume desiderato per pozzetto a ciascun pozzetto, se non è già stato fatto.
- Preparare quanto segue prima di iniziare: almeno 1 ora prima del passaggio, rivestire la plastica desiderata con matrici. Aliquotare una quantità sufficiente di terreno di coltura di cellule staminali complete e riscaldarlo a temperatura ambiente (15 - 25 °C). Evitare più cicli di riscaldamento per l'intero fluido.
- Coltura di iPSC in piastre contenenti matrici di origine animale
- Risospendere delicatamente i grumi di iPSC nel volume calcolato al 50% del terreno di coltura di cellule staminali complete per ottenere la densità desiderata e placcare la miscela di aggregati cellulari su pozzetti rivestiti contenenti il 50% del terreno di coltura di cellule staminali complete calcolato.
NOTA: L'aggiunta dell'inibitore ROCK-1 potrebbe essere necessaria per alcune linee cellulari o quando i grumi sono di 50 μm o più piccoli. Si consiglia di aggiungere <10 μm per evitare la gastrulazione precoce. - Aggiungere con cautela il terreno con le celle sopra le matrici, 250 μL/pozzetto se si utilizza una piastra a 24 pozzetti. Per il volume di terreno cellulare consigliato per piastre a pozzetti di altre dimensioni, consultare la Tabella 2.
- Inclinare la piastra con diversi brevi movimenti avanti e indietro e da un lato all'altro per distribuire uniformemente i grumi di cellule.
ATTENZIONE: La distribuzione non uniforme degli aggregati provoca un aumento della differenziazione spontanea delle iPSC umane. Lo spargimento circolare della sospensione dei grumi provoca l'agglomerazione dei grumi sul bordo dei pozzetti e una minore densità al centro. - Incubare la piastra a 37 °C ed eseguire i cambi del terreno utilizzando un terreno completo di cellule staminali ogni giorno o a giorni alterni. Quando si eseguono cambi di terreno, valutare visivamente le colture per monitorare la crescita e determinare se le cellule richiedono un tempo di passaggio o sono pronte per la differenziazione. Per saltare due giorni consecutivi di poppata, aggiungi 2 volte il volume del terreno necessario per un singolo giorno.
- Risospendere delicatamente i grumi di iPSC nel volume calcolato al 50% del terreno di coltura di cellule staminali complete per ottenere la densità desiderata e placcare la miscela di aggregati cellulari su pozzetti rivestiti contenenti il 50% del terreno di coltura di cellule staminali complete calcolato.
- Coltura di iPSC in piastre contenenti Matrix 4-XE
- Risospendere delicatamente i grumi di iPSC nel volume totale calcolato di 3 volte il terreno di coltura delle cellule staminali completo per ottenere la densità desiderata.
- Aggiungere con cautela il terreno con le celle sopra l'idrogel 250 μL/pozzetto se si utilizza una piastra a 24 pozzetti. Per il volume di terreno cellulare consigliato per piastre a pozzetti di altre dimensioni, consultare la Tabella 3.
ATTENZIONE: L'idrogel si gonfierà e occuperà un volume maggiore di quello appena fatto. Le colonie di iPSC saranno parzialmente incorporate nell'idrogel nella parte inferiore della piastra, quindi si consiglia di cambiare il 50%-80% del terreno superiore senza disturbare l'idrogel. - Muovi la piastra con diversi movimenti rapidi, brevi, avanti e indietro e da un lato all'altro per distribuire uniformemente i grumi di cellule.
- Posizionare la piastra in un'incubatrice a 37 °C con il 5% di CO2 e il 95% di umidità. Cambiare il terreno cellulare utilizzando un terreno di coltura completo per cellule staminali. Eseguire cambi medi ogni giorno o a giorni alterni; Per quest'ultimo, aggiungere 2 volte il volume del mezzo.
- Ispeziona visivamente le colture per seguirne la crescita man mano che raggiungono lo stadio adatto alla differenziazione.
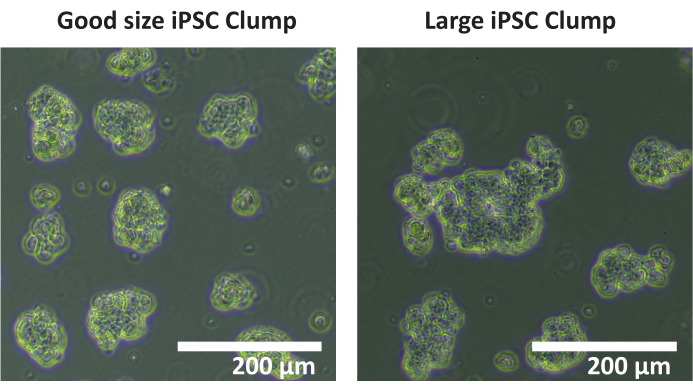
Figura 1: Dimensione ottimale del grumo. Immagini di grumi della linea cellulare iPSC SCTi003A che raffigurano un esempio di dimensione ottimale del grumo. Barra della scala = 200 μm. Fare clic qui per visualizzare una versione più grande di questa figura.
2. Differenziamento di hiPSC e generazione di organoidi intestinali
ATTENZIONE: Tutto il lavoro viene svolto in una cabina di biosicurezza (BSC) seguendo tecniche asettiche standard. Deve seguire gli standard di sicurezza OSHA per i laboratori, incluso l'uso corretto di dispositivi di protezione individuale come camici da laboratorio, guanti e occhiali.
- Prima di iniziare, preparare le aliquote del mezzo di differenziazione necessarie per ogni fase. Dopo aver scongelato le aliquote, utilizzarle immediatamente. Non ricongelare. Seguire la Tabella 5 per una guida generale di ogni tipo di terreno di fase e dei volumi necessari per una piastra a 24 pozzetti.
NOTA: Il tempo ottimale in ogni fase di differenziazione può variare a seconda della linea cellulare. Si raccomanda di controllare l'espressione di ciascun marcatore cellulare stadio per migliorare l'efficacia della differenziazione. La valutazione periodica dell'espressione genica sulle colture di iPSCS non è stata inclusa in questa ricerca poiché abbiamo sfruttato lotti qualificati dal fornitore a bassi passaggi, ma è raccomandata come fase di controllo qualità per linee iPSC nuove/non ancora qualificate durante le successive fasi di differenziazione. - Fase 1: Endoderma definitivo (DE)
- Per valutare la disponibilità ad avviare la differenziazione, verificare i seguenti criteri.
- Utilizzando un microscopio, valutare la confluenza delle colonie di cellule staminali e la quantità di differenziazione spontanea nella coltura. Non fare affidamento solo sulla valutazione morfologica. La confluenza ottimale dovrebbe essere compresa tra l'85% e il 90% (superiore a ~105 cellule/cm2) e la differenziazione spontanea minima osservata dovrebbe essere del <5%.
- Raccogliere le cellule da un pozzetto sacrificale per la caratterizzazione in citometria a flusso dei marcatori hiPSC prima di procedere con la fase successiva. Per la caratterizzazione rappresentativa della citometria a flusso è stata utilizzata una strategia di dispersione diretta e laterale. Se le cellule soddisfano i criteri ottimali, iniziare la differenziazione. Il criterio di passaggio cellulare è >85% di espressione di idealmente 3 marcatori (Tabella 4), specialmente nel caso di linee iPSC nuove/non ancora qualificate in laboratorio.
NOTA: Le colture di iPSC coltivate su matrici diverse subiscono lo stesso processo generale con solo la crescita di iPSC su Matrix 4-XE, che richiede modifiche più delicate dei terreni per non disturbare le cellule semi-incorporate. Si prevede una significativa morte cellulare durante l'induzione dell'endoderma definitivo, poiché le cellule sono particolarmente sensibili in questa fase. Prestare attenzione durante i cambi di terreno, con l'obiettivo di ridurre al minimo la durata di esposizione delle cellule al di fuori dell'ambiente di incubazione a 37 °C.
- Iniziare la differenziazione delle colonie cellulari come descritto di seguito.
- Giorno 0: Scaldare un'aliquota di DE medium a 37 °C. Per evitare la denaturazione dei terreni a causa dei cicli di riscaldamento-raffreddamento, assicurarsi di riscaldare solo il volume richiesto per il giorno 0 (0,7 ml/pozzetto).
- Aspirare il fluido dalle hiPSC. Aggiungere con cautela 0,7 ml di DE Medium per pozzetto lungo il lato del pozzetto. Evitare il pipettaggio aggressivo che potrebbe staccare o danneggiare le colonie. Incubare la piastra a 37 °C con il 5% di CO2 e il 95% di umidità per 24 ore.
- Giorno 1: Riscaldare un'aliquota di terreno DE contenente solo il volume necessario per il giorno 1 (0,5 mL/pozzetto) a 37 °C. Aspirare il terreno DE dalle cellule e aggiungere con cautela 0,5 mL di terreno DE per pozzetto lungo il lato del pozzetto. Incubare a 37 °C con il 5% di CO2 e il 95% di umidità per 24 ore.
- Giorno 3: Le cellule sono pronte per essere analizzate per la formazione definitiva dell'endoderma. Fare riferimento alla Tabella 4 per i marcatori consigliati. Prima di procedere con la fase successiva, sacrificare un pozzetto per eseguire una caratterizzazione in citometria a flusso di specifici marcatori associati alla DE. Per la caratterizzazione rappresentativa della citometria a flusso è stata utilizzata una strategia di dispersione diretta e laterale. Si raccomanda di controllare l'espressione di più di 1 marcatore, soprattutto nel caso di linee iPSC nuove/non ancora qualificate in laboratorio.
- Per valutare la disponibilità ad avviare la differenziazione, verificare i seguenti criteri.
- Fase 2: Intestino medio/intestino posteriore (MH)
- Giorno 3: Riscaldare solo il volume di terreno MH necessario per il giorno 3 (0,5 mL/pozzetto) a 15 - 25 °C. Aspirare il terreno DE dalle cellule e sostituirlo con 0,5 mL di terreno MH. Incubare a 37 °C con il 5% di CO2 e il 95% di umidità per 24 ore.
- Giorni 4 - 9: Prima che il mezzo cambi ogni giorno, utilizzare il microscopio con ingrandimento 2x o 4x per valutare la formazione di sferoidi controllando la presenza di strutture 3D visibili (potrebbe accadere dal giorno 4); sferoidi fluttuanti (potrebbero verificarsi dal giorno 5).
- Una volta che gli sferoidi iniziano a staccarsi, utilizzare un pipettatore da 1 ml per trasferire delicatamente il terreno dalle cellule a una provetta conica sterile da 15 ml per valutare il numero di sferoidi staccati dalle cellule.
NOTA: Evitare qualsiasi forza di taglio che potrebbe staccare le strutture 3D dal monostrato; Una volta pronti, gli sferoidi si staccheranno da soli. - Se si staccano meno di 50 sferoidi, centrifugare a 200 x g per 5 minuti, rimuovere il vecchio terreno e risospendere gli sferoidi su 0,5 mL/pozzetto di terreno e trasferirli nel rispettivo pozzetto fino a quando non sono maturati abbastanza sferoidi.
NOTA: La durata dell'esposizione al mezzo dell'intestino medio/intestino posteriore gioca un ruolo cruciale nel determinare l'identità regionale degli organoidi dell'intestino tenue in via di sviluppo, come il duodeno (esposizione più breve) o l'ileo (esposizione più lunga). Per ottenere colture più uniformi con la stessa identità, mirare a raccogliere sferoidi esposti contemporaneamente al mezzo mediointestino/intestino posteriore. - Ripetere il passaggio 2.3.3 fino a quando non si staccano abbastanza sferoidi per incorporare e avviare la coltura di organoidi intestinali umani (hIO).
- Prima di procedere con la fase successiva, separare un campione di sferoidi ed eseguire una caratterizzazione in citometria a flusso di marcatori specifici associati a Midgut/Hindgut (MH). Per la caratterizzazione rappresentativa della citometria a flusso è stata utilizzata una strategia di dispersione diretta e laterale. Fare riferimento alla Tabella 4 per un elenco dei marcatori di uso comune. Si raccomanda di controllare l'espressione di più di 1 marcatore, soprattutto nel caso di linee iPSC nuove/non ancora qualificate in laboratorio.
- Fase 3: Incorporazione di sferoidi
- Preparare quanto segue prima di iniziare la procedura.
- Scongelare le aliquote di Matrix 1-ABO e Matrix 3-ABO sul ghiaccio. Considerare il numero di sferoidi raccolti per determinare la quantità totale di matrice necessaria se saranno necessari 30-40 μL di matrice per cupola.
- Preparare 25 mL di Advance DMEM/F-12 freddo contenente 15 mM HEPES. Le ECM ad alta concentrazione sono viscose e difficili da pipettare. Posizionare una scatola di puntali per pipette sterili da 100 μl a -20 °C per preparare puntali freddi che possono aiutare il processo. Preparare aliquote di terreno di crescita degli organoidi intestinali (OGM), che richiede 4 somministrazioni (0,5 ml/pozzetto per alimentazione). Vedere la Tabella 5 per la preparazione.
- Scongelare un'aliquota dell'integratore OGM con ghiaccio. Dopo aver scongelato le aliquote, utilizzarle immediatamente. Non ricongelare. Conservare l'OGM completo a 2-8 °C per un massimo di 2 settimane.
- Preparare OGM contenente 3x l'integratore rispetto alla formulazione standard per (3x OGM). Togliete i Matrix 4-XFO1 thought Matrix 4-XFO4 dal frigorifero e lasciateli scaldare a temperatura ambiente (25 °C).
- Per il sistema di derivazione animale, eseguire i passaggi descritti di seguito.
- Posizionare una piastra sterile per colture tissutali a 24 pozzetti nell'incubatore per riscaldarla a 37 °C mentre si preparano gli sferoidi e le matrici. Lasciare che gli sferoidi raccolti si depositino sul fondo di una provetta conica da 15 mL. Aspirare con cura ed eliminare il surnatante.
NOTA: Per effettuare un confronto più accurato dell'effetto delle matrici, assicurarsi di mantenere gli sferoidi raccolti dalla hiPSC differenziati su ciascuna matrice in provette separate. - Aggiungere 1 mL di DMEM/F-12 con 15 mM HEPES agli sferoidi. Centrifugare a 300 x g per 5 minuti a temperatura ambiente (15 - 25 °C).
- Rimuovere con cura la maggior quantità possibile di surnatante. Si consiglia di iniziare la rimozione con un pipettatore da 1 ml e passare a pipette da 100 μl e persino da 10 μl per rimuovere il più possibile senza disturbare il pellet di sferoidi.
NOTA: Maggiore è il volume del supporto rimasto, più diluite saranno le matrici. Le matrici diluite possono portare a problemi di gelificazione o a cupole più morbide. Cupole più morbide portano a maggiori possibilità di collasso con disturbi minimi. - Utilizzando un pipettatore con puntale per pipetta freddo da 100 μL, aggiungere 40 μL/50 sferoidi di Matrix 1-ABO o Matrix 3-ABO freddi (2 - 8 °C) alla rispettiva provetta. Distribuire delicatamente gli sferoidi nella matrice pipettandoli su e giù ~5x. Non svuotare completamente il puntale della pipetta, che potrebbe introdurre bolle.
- Estrarre la piastra dall'incubatrice e, utilizzando una punta per pipetta fredda, trasferire delicatamente gli sferoidi incorporati al centro della piastra seguendo i passaggi della Figura 2. Evitare di erogare Matrix 1-ABO e Matrix 3-ABO troppo rapidamente nel piatto di coltura, poiché ciò appiattirebbe la cupola.
- Trasferire con cautela la piastra in un incubatore a 37 °C e incubare per 30 minuti per garantire la gelificazione della cupola. Prestare particolare attenzione a qualsiasi scuotimento o movimento brusco durante il trasporto e nell'incubatrice.
- Durante l'incubazione della cupola per la gelificazione, riscaldare a 37 °C un volume sufficiente di OGM intestinale (0,5 mL/pozzetto) per il numero di pozzetti da utilizzare.
NOTA: Riscaldare solo il volume necessario. Evitare i cicli di riscaldamento-raffreddamento. Assicurarsi che il supporto sia caldo prima di aggiungerlo alla cupola; I fluidi freddi possono portare al collasso della cupola. - Dopo 30 minuti, aggiungere con cura 0,5 ml di OGM intestinale sul lato del pozzetto per non disturbare la cupola. Incubare a 37 °C con il 5% di CO2 e il 95% di umidità. Eseguire un cambio full-medium ogni 3 - 4 giorni rimuovendo il terreno e quindi aggiungere un terreno fresco.
- Posizionare una piastra sterile per colture tissutali a 24 pozzetti nell'incubatore per riscaldarla a 37 °C mentre si preparano gli sferoidi e le matrici. Lasciare che gli sferoidi raccolti si depositino sul fondo di una provetta conica da 15 mL. Aspirare con cura ed eliminare il surnatante.
- Per i sistemi di organoidi Matrix 4-XF, seguire i passaggi descritti.
NOTA: Il sistema di organoidi Matrix 4-XF include quattro diverse opzioni (O1-O4); La formulazione di ciascun tipo varia in ligandi biofunzionali, rigidità e degradabilità. Di conseguenza, si consiglia di eseguire esperimenti iniziali per determinare il tipo ottimale per l'applicazione specifica. Questo protocollo descrive l'utilizzo delle quattro opzioni per trovare una formulazione ottimale per gli organoidi intestinali derivati da hiPSC. Inoltre, sebbene esistano più protocolli di coltura che possono essere utilizzati (ad esempio, incapsulamento 3D, incapsulamento a cupola), questo protocollo prevede l'utilizzo del metodo a cupola in modo tale che sia possibile confrontarlo direttamente con i metodi comuni utilizzati quando si lavora con sistemi di membrane basali di origine animale.- Aggiungere 1 mL/pozzetto di avanzamento freddo DMEM/F-12 contenente 16 mM HEPES agli sferoidi generati sul sistema Matrix 4-XFO4. Centrifugare a 300 x g per 5 minuti a temperatura ambiente (15 - 25 °C).
NOTA: Per trovare la formulazione ottimale per l'applicazione, assicurarsi di distribuire gli sferoidi in 4 provette separate prima della centrifugazione. Gli sferoidi generati su matrici di origine animale possono anche essere maturati in organoidi intestinali utilizzando questo sistema. - Utilizzando un pipettatore da 1 ml, rimuovere il surnatante senza disturbare gli sferoidi. Aggiungere 50 μL di 3x OGM al pellet di sferoidi (~100 sferoidi). Aggiungere 100 μl della matrice 4-XFO selezionata alla sospensione sferoidale da 50 μl e mescolare delicatamente 5x-10x volte. Mantenere il Matrix 4-XFO a 3x OGM a un rapporto di miscelazione 2:1 v/v per avere una concentrazione finale di 1x.
- Aggiungere 40 μl della miscela idrogel-sferoide al centro di una piastra di coltura tissutale a 24 pozzetti. Trasferire con cura le piastre in un'incubatrice a 37 °C e incubare per 30 minuti.
NOTA: Matrix 4-XFO non richiede un'incubazione a 37 °C per la gelificazione; tuttavia, si raccomanda di mantenere gli organoidi esposti a condizioni simili rispetto ai sistemi Matrix 1-ABO e Matrix 3-ABO che richiedono 37 °C per una migliore gelificazione delle cupole. - Dopo 30 minuti, aggiungere con cura 0,5 ml di OGM intestinale sul lato del pozzetto per non disturbare la cupola. Incubare a 37 °C con il 5% di CO2 e il 95% di umidità. Eseguire un cambio full-medium ogni 3 - 4 giorni rimuovendo il terreno e quindi aggiungendo un terreno fresco.
- Aggiungere 1 mL/pozzetto di avanzamento freddo DMEM/F-12 contenente 16 mM HEPES agli sferoidi generati sul sistema Matrix 4-XFO4. Centrifugare a 300 x g per 5 minuti a temperatura ambiente (15 - 25 °C).
- Preparare quanto segue prima di iniziare la procedura.
- Passaggio e maturazione hIO
- Preparare le stesse sostanze chimiche, soluzioni e reagenti come fatto nel passaggio 2.4.1.
- Aggiungere 1-2 ml di soluzione di risciacquo antiaderente a una provetta conica da 15 ml (1 per condizione) e agitare per rivestire la provetta.
- Rimuovere la soluzione antiaderente e sciacquare le provette con 5 mL di D-PBS (senza Ca++ e Mg++). Tappare tutte le provette rivestite e mantenerle a temperatura ambiente (15 - 25 °C) fino al momento del bisogno.
- Aspirare il fluido dalle cupole. Utilizzando una pipetta da 1 mL, aggiungere 1 mL di DMEM/F-12 freddo direttamente alla cupola. L'obiettivo è quello di staccare le cupole dalla piastra.
- Aggiungere un ulteriore 1 mL di DMEM/F-12 freddo al pozzetto e pipettare su e giù per raccogliere gli organoidi rimanenti. Trasferire nelle provette coniche rivestite da 15 mL.
NOTA: Verificare il successo della raccolta degli organoidi esaminando visivamente il pozzetto al microscopio. Se si osservano organoidi residui, ripetere il passaggio 2.5.5. - Con un pipettatore da 1 mL, eseguire il pipettaggio su e giù della sospensione per disintegrare gli organoidi fino a ottenere una sospensione uniforme del frammento con la dimensione dell'organoide desiderata (ad esempio, 100 - 500 μm).
NOTA: Utilizzare un pipettatore da 200 μl per verificare che gli organoidi siano conformi alle dimensioni consigliate. L'utilizzo del pipettatore da 200 μl facilita la frammentazione aggiuntiva secondo necessità, assicurando che i frammenti possano passare agevolmente attraverso un puntale della pipetta da 200 μl.
ATTENZIONE: Evitare di rompere i frammenti in singole cellule mediante pipettaggio brusco o prolungato. - Stabilire la densità di organoidi desiderata contando i frammenti o utilizzando il rapporto di divisione. Separare un'aliquota ed eseguire il conteggio utilizzando la stessa procedura dei passaggi 1.4.6-1.4.9 per il conteggio dei grumi.
NOTA: La densità ottimale degli organoidi deve essere ottimizzata per riga; In generale, si raccomanda una densità di 40 - 80 organoidi intestinali per cupola. - Assicurarsi che la provetta sia posta sul ghiaccio durante il processo di conteggio degli organoidi. Dopo circa 5 minuti, i frammenti di organoidi si saranno depositati sul fondo del tubo a causa della gravità.
NOTA: Maggiore è il volume della soluzione dell'organoide, più tempo ci vorrà per depositarsi sul fondo. - Eliminare delicatamente quanto surnatante e lo strato torbido formatosi sopra gli organoidi. Nei primi passaggi, quando gli organoidi stanno maturando, questa fase torbida comprende una matrice e singole cellule.
- Aggiungere 2 mL di DMEM/F-12 freddo pipettandolo direttamente sul pellet. Centrifugare a 200 x g per 5 minuti a temperatura ambiente (15 - 25 °C).
- Per il sistema di origine animale, rimuovere e scartare con cura il surnatante seguendo gli stessi passaggi descritti nei passaggi 2.4.2.4-2.4.2.9.
- Per il sistema Matrix 4-XFO, rimuovere ed eliminare con cautela il surnatante seguendo gli stessi passaggi descritti nei passaggi 2.4.3.2-2.4.3.4.
NOTA: Quando si lavora con il sistema Matrix 4-XFO, si consiglia di utilizzare una soluzione di recupero di organoidi idrogel priva di xeno per una rimozione ottimale dei residui di idrogel. Questa soluzione di recupero è particolarmente consigliata quando si passa da sistemi di idrogel di origine animale a sistemi di idrogel privi di xeno per garantire l'eliminazione di qualsiasi materiale xenogenico.
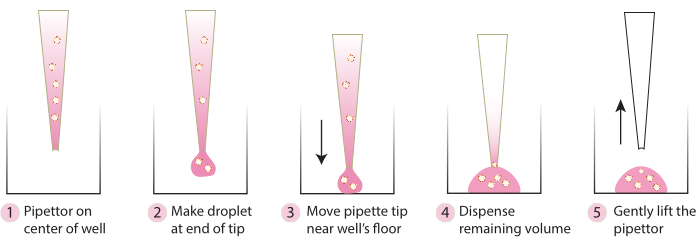
Figura 2. Schema della tecnica consigliata per la formazione a cupola. Lo schema descrive il processo passo dopo passo raccomandato per la formazione di una cupola di successo per tutti i sistemi. Clicca qui per visualizzare una versione più grande di questa figura.
3. Caratterizzazione delle dimensioni IO
NOTA: La dimensione degli organoidi è stata caratterizzata da immagini in campo chiaro scattate a 4x e 10x. L'analisi dell'elaborazione delle immagini è stata automatizzata utilizzando MATLAB. Le fasi generali del processo sono descritte di seguito e un esempio del codice è incluso nel file supplementare 1.
- Definisce la directory contenente le immagini ed elenca tutti i file immagine nella directory. Inizializzare la tabella per archiviare i risultati. Leggi le immagini in campo chiaro in un file.
- Definisci la conversione da pixel a μm e imposta la scala. Se l'immagine non contiene una barra di scala, richiedere all'utente il fattore di conversione da pixel a μm.
- Converti l'immagine in scala di grigi. Applicate un filtro gaussiano per ridurre il rumore nell'immagine. Soglia l'immagine filtrata per separare gli organoidi dallo sfondo.
- Riempi piccoli fori e rimuovi piccoli oggetti per ripulire l'immagine binaria. Esegui l'analisi dei componenti connessi per identificare i singoli organoidi e calcolarne le proprietà come area, baricentro, lunghezza dell'asse maggiore e lunghezza dell'asse minore.
- Calcola la dimensione dell'organoide in base alle lunghezze dell'asse maggiore e minore. Visualizzare gli organoidi segmentati sull'immagine originale ed etichettarli con le loro dimensioni. Stampa le dimensioni degli organoidi e memorizza i risultati in un file .cvs.
NOTA: Le prime due immagini sono state analizzate manualmente per determinare i parametri ottimali per la deviazione standard del filtro gaussiano e l'area fino alla soglia. Il codice nel File Supplementare 1 fornisce un esempio del framework di base per l'analisi delle dimensioni degli organoidi da immagini in campo chiaro; Tuttavia, è necessario un ulteriore perfezionamento per soddisfare i requisiti specifici per ogni tipo di immagine e qualità. Lo stesso processo può essere eseguito utilizzando software open source come FIJI di Image J. - Segui i passaggi seguenti per utilizzare il software FIJI.
- Definisci la conversione da pixel a μm e imposta la scala. Se l'immagine non contiene una barra di scala, richiedere all'utente il fattore di conversione da pixel a μm facendo clic su Analizza > Imposta scala.
- Converti l'immagine in scala di grigi facendo clic su Tipo di immagine > > 8 bit. Applicare un filtro gaussiano per ridurre il rumore nell'immagine facendo clic su Elabora > Filtro > Blurb gaussiano > Sigma (raggio) utilizzato: 2.
- Soglia l'immagine filtrata per separare gli organoidi dallo sfondo facendo clic su Immagine > Soglia > MaxEntropia > Applica.
- Riempi piccoli fori e rimuovi piccoli oggetti per ripulire l'immagine binaria facendo clic su Elabora > rumore > Rimuovi valori anomali > 20 pixel.
- Esegui l'analisi dei componenti collegati per identificare i singoli organoidi e calcolarne le proprietà, come l'area, il baricentro, la lunghezza dell'asse maggiore e la lunghezza dell'asse minore, facendo clic su Analizza > Imposta misurazione > Assicurati che l'area, il perimetro e il diametro siano inclusi > Analizza > analizza particelle > Mostra contorni.
- Analisi statistica
- Valutare la normalità della distribuzione dei dati attraverso il test Saphiro-Wilk utilizzando il software JMP (SAS). Per esaminare le differenze statistiche tra i gruppi, eseguire un'ANOVA bidirezionale e condurre test post-hoc utilizzando il metodo Wilcoxon non parametrico nel software JMP (SAS). La significatività è stata stabilita a un livello alfa di p ≤ 0,05.
Access restricted. Please log in or start a trial to view this content.
Risultati
Seguendo questo protocollo, le membrane basali disponibili in commercio e un sistema di idrogel privo di xeno sono stati utilizzati con successo per coltivare cellule hiPSC e differenziarle in hIO. L'obiettivo principale di questi esperimenti era quello di valutare sistematicamente l'equivalenza di matrici provenienti da varie fonti per il lavoro su hiPSC e hIO. La prima sezione di questo protocollo si è concentrata sul mantenimento e la caratterizzazione di una coltura sana di iPSC che produca un'efficiente generazione...
Access restricted. Please log in or start a trial to view this content.
Discussione
La selezione del microambiente ottimale per il lavoro con cellule staminali e organoidi è un primo passo fondamentale quando si utilizzano queste piattaforme per un'ampia gamma di applicazioni. I nostri risultati rappresentativi mostrano che la matrice 4-XFO3, in combinazione con una maggiore concentrazione di fattori di crescita, porta a organoidi più grandi, suggerendo che le proprietà fisiche degli idrogel privi di xeno possono essere sfruttate per ottimizzare la generazione di organoidi utilizzando questi sistemi....
Access restricted. Please log in or start a trial to view this content.
Divulgazioni
Il Dr. John Huang è fondatore e CEO di TheWell Bioscience.
Riconoscimenti
Gli autori riconoscono la formazione precedente e le raccomandazioni generali riguardanti l'inizio del lavoro su hiPSC e organoidi da parte dei dottori Christina Pacak, Silveli Susuki-Hatano e Russell D'Souza. Ringraziano la dottoressa Chelsey Simmons per la sua guida nell'utilizzo dei sistemi di idrogel per il lavoro di coltura cellulare in vitro . Inoltre, gli autori desiderano ringraziare i dottori Christine Rodriguez e Thomas Allison di STEMCELL Technologies per la loro guida sulla coltura hiPSC. Gli autori ringraziano anche TheWell Bioscience per aver coperto i costi di pubblicazione.
Access restricted. Please log in or start a trial to view this content.
Materiali
| Name | Company | Catalog Number | Comments |
| 24-Well Plate (Culture treated, sterile) | Falcon | 353504 | |
| 37 °C water bath | VWR | ||
| 96-well plate | Fisher Scientific | FB012931 | |
| Advanced DMEM/F12 | Life Technologies | 12634 | |
| Anti-adherence Rinsing Solutio | STEMCELL Technologies | 7010 | |
| Biological safety cabinet (BSC) | Labconco | Logic | |
| Brightfield Microscope | Echo Rebel | REB-01-E2 | |
| BXS0116 | ATCC | ACS-1030 | |
| Centrifuge with temperature control (4 °C capabilities) | ThermoScientific | 75002441 | |
| Conical tubes, 15 mL, sterile | Thermo Fisher Scientific | 339650 | |
| Conical tubes, 50 mL, sterile | Thermo Fisher Scientific | 339652 | |
| Cultrex RGF BME, Type 2 | Bio-techne | 3533-005-02 | |
| Cultrex Stem Cell Qualified RGF BME | Bio-techne | 3434-010-02 | |
| D-PBS (Without Ca++ and Mg++) | Thermo Fisher Scientific | 14190144 | |
| GeltrexLDEV-Free, hESC-Qualified Reduce Growth Factor | Gibco | A14133-02 | |
| GlutaMAX Supplement | Thermo Fischer Scientific | 35050-061 | |
| Guava Muse Cell Analyzer or another flow cytometry equipment (optional) | Luminex | 0500-3115 | |
| HEPES buffer solution | Thermo Fischer Scientific | 15630-056 | |
| Heralcell Vios Cell culture incubator (37 °C, 5% CO2) | Thermo Scientific | 51033775 | |
| JMP Software | SAS Institute | JMP 16 | |
| MATLAB | MathWorks, Inc | R2022b | |
| Matrigel Growth Factor Reduced (GFR) Basement Membrane Matrix LDEV free | Corning | 356231 | |
| Matrigel Matrix High Concentration (HC), Growth Factor Reduced (GFR) LDEV-free | Corning | 354263 | |
| mTeSR Plus Medium | STEMCELL Technologies | 100-0276 | |
| Nunclon Delta surface treated 24-well plate | Thermo Scientific | 144530 | |
| PE Mouse Anti-human CD326 (EpCAM) | BD Pharmingen | 566841 | |
| PE Mouse Anti-human CDX2 | BD Pharmingen | 563428 | |
| PE Mouse Anti-human FOXA2 | BD Pharmingen | 561589 | |
| PerCP-Cy 5.5 Mouse Anti-human SSEA4 | BD Pharmingen | 561565 | |
| ReLeSR | STEMCELL | 5872 | |
| SCTi003-A | STEMCELL Technologies | 200-0510 | |
| Serological pipettes (10 mL) | Fisher Scientific | 13-678-11E | |
| Serological pipettes (5 mL) | Fisher Scientific | 13-678-11D | |
| STEMdiff Intestinal Organoid Growth Medium | STEMCELL Technologies | 5145 | |
| STEMdiff Intestinal Organoid Kit | STEMCELL Technologies | 5140 | |
| Vitrogel Hydrogel Matrix | TheWell Bioscience | VHM01 | |
| VitroGel ORGANOID Discovery Kit | TheWell Bioscience | VHM04-K |
Riferimenti
- Hynes, R. O. Integrins: Bidirectional, allosteric signaling machines. Cell. 110 (6), 673-687 (2002).
- Frantz, C., Stewart, K. M., Weaver, V. M. The extracellular matrix at a glance. J Cell Sci. 123, Pt 24 4195-4200 (2010).
- Hinz, B., Gabbiani, G. Fibrosis: Recent advances in myofibroblast biology and new therapeutic perspectives. F1000 Biol Rep. 2, 78(2010).
- Pickup, M. W., Mouw, J. K., Weaver, V. M. The extracellular matrix modulates the hallmarks of cancer. EMBO Rep. 15 (12), 1243-1253 (2014).
- Rozario, T., DeSimone, D. W. The extracellular matrix in development and morphogenesis: A dynamic view. Dev Biol. 341 (1), 126-140 (2010).
- Even-Ram, S., Artym, V., Yamada, K. M. Matrix control of stem cell fate. Cell. 126 (4), 645-647 (2006).
- Engler, A. J., Sen, S., Sweeney, H. L., Discher, D. E. Matrix elasticity directs stem cell lineage specification. Cell. 126 (4), 677-689 (2006).
- Tran, O. N., et al. Organ-specific extracellular matrix directs trans-differentiation of mesenchymal stem cells and formation of salivary gland-like organoids in vivo. Stem Cell Res Ther. 13 (1), 306(2022).
- Nikolaev, M., et al. Homeostatic mini-intestines through scaffold-guided organoid morphogenesis. Nature. 585 (7826), 574-578 (2020).
- Gjorevski, N., et al. Designer matrices for intestinal stem cell and organoid culture. Nature. 539 (7630), 560-564 (2016).
- Gjorevski, N., et al. Tissue geometry drives deterministic organoid patterning. Science. 375 (6576), (2022).
- Heo, J. H., Kang, D., Seo, S. J., Jin, Y. Engineering the extracellular matrix for organoid culture. Int J Stem Cells. 15 (1), 60-69 (2022).
- Shamir, E. R., Ewald, A. J. Three-dimensional organotypic culture: experimental models of mammalian biology and disease. Nat Rev Mol Cell Biol. 15 (10), 647-664 (2014).
- Clevers, H. Modeling development and disease with organoids. Cell. 165 (7), 1586-1597 (2016).
- Jung, P., et al. Isolation and in vitro expansion of human colonic stem cells. Nat Med. 17 (10), 1225-1227 (2011).
- Lancaster, M. A., Knoblich, J. A. Organogenesis in a dish: Modeling development and disease using organoid technologies. Science. 345 (6194), 1247125(2014).
- Huch, M., et al. In vitro expansion of single Lgr5+ liver stem cells induced by Wnt-driven regeneration. Nature. 494 (7436), 247-250 (2013).
- Greenlee, A. R., Kronenwetter-Koepel, T. A., Kaiser, S. J., Liu, K. Comparison of Matrigel and gelatin substrata for feeder-free culture of undifferentiated mouse embryonic stem cells for toxicity testing. Toxicol In Vitro. 19 (3), 389-397 (2005).
- Geltrex LDEV-Free, HESC-Qualified, Reduced Growth Factor Basement Membrane Matrix User Guide (Pub.No. MAN0007336 3.0. Fisher Scientific. , Available from: https://www.thermofisher.com/document-connect/document-connect.html?url=https://assets.thermofisher.cn/TFS-Assets%2FLSG%2Fmanuals%2FGeltrex_LDEV_Free_hESC_qualified_PI.pdf (2024).
- biotechne R&D Systems. Cultrex Stem Cell Qualified Reduced Growth Factor. biotechne R&D Systems. , (2024).
- VitroGel Organoid Protocol. TheWell Bioscience. , Available from: https://www.thewellbio.com/video-protocols (2024).
- Spence, J. R., et al. Directed differentiation of human pluripotent stem cells into intestinal tissue in vitro. Nature. 470 (7332), 105-110 (2011).
- Henderson, J. K., et al. Preimplantation human embryos and embryonic stem cells show comparable expression of stage-specific embryonic antigens. Stem Cells. 20 (4), 329-337 (2002).
- Haruna, N. F., Huang, J. Investigating the dynamic biophysical properties of a tunable hydrogel for 3D cell culture. J Cytol Tissue Biol. 7, 30(2020).
- Cherne, M. D., et al. A synthetic hydrogel, VitroGel ORGANOID-3, improves immune cell-epithelial interactions in a tissue chip co-culture model of human gastric organoids and dendritic cells. Front Pharmacol. 12, 707891(2021).
- Stewart, D. C., et al. Quantitative assessment of intestinal stiffness and associations with fibrosis in human inflammatory bowel disease. PLoS One. 13, e0200377(2018).
- Hernandez-Gordillo, V., et al. Fully synthetic matrices for in vitro culture of primary human intestinal enteroids and endometrial organoids. Biomaterials. 254, 120125(2020).
- Broguiere, N., et al. Growth of epithelial organoids in a defined hydrogel. Adv Mater. 30, 1801621(2018).
- Barthes, J., et al. Cell microenvironment engineering and monitoring for tissue engineering and regenerative medicine: The recent advances. BioMed Res Int. 2014, 921905(2014).
- Engler, A. J., Sen, S., Sweeney, H. L., Discher, D. E. Matrix elasticity directs stem cell lineage specification. Cell. 126 (4), 677-689 (2006).
- Aisenbrey, E. A., Murphy, W. L. Synthetic alternatives to Matrigel. Nat Rev Mater. 5 (7), 539-551 (2020).
Access restricted. Please log in or start a trial to view this content.
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon