É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Estudo Comparativo de Matrizes de Membrana Basal para Manutenção de Células-Tronco Humanas e Geração de Organoides Intestinais
Neste Artigo
Resumo
Os organoides tornaram-se ferramentas valiosas para a modelagem de doenças. A matriz extracelular (MEC) orienta o destino da célula durante a geração de organoides, e o uso de um sistema que se assemelha ao tecido nativo pode melhorar a precisão do modelo. Este estudo compara a geração de organoides intestinais humanos derivados de células-tronco pluripotentes induzidas em ECM derivada de animais e hidrogéis livres de xeno.
Resumo
A matriz extracelular (MEC) desempenha um papel crítico no comportamento e desenvolvimento celular. Organoides gerados a partir de células-tronco pluripotentes induzidas por humanos (hiPSCs) estão no centro das atenções de muitas áreas de pesquisa. No entanto, a falta de pistas fisiológicas em materiais clássicos de cultura de células dificulta a diferenciação eficiente de iPSC. A incorporação da ECM disponível comercialmente na cultura de células-tronco fornece pistas físicas e químicas benéficas para a manutenção celular. Os produtos de membrana basal comercialmente disponíveis de origem animal são compostos de proteínas ECM e fatores de crescimento que suportam a manutenção celular. Como a ECM possui propriedades específicas do tecido que podem modular o destino celular, matrizes livres de xeno são usadas para transmitir a tradução para estudos clínicos. Embora as matrizes comercialmente disponíveis sejam amplamente utilizadas no trabalho com hiPSC e organoides, a equivalência dessas matrizes ainda não foi avaliada. Aqui, foi realizado um estudo comparativo da manutenção de hiPSC e geração de organoides intestinais humanos (hIO) em quatro matrizes diferentes: Matrigel (Matrix 1-AB), Geltrex (Matrix 2-AB), Cultrex (Matrix 3-AB) e VitroGel (Matrix 4-XF). Embora as colônias não tivessem uma forma perfeitamente redonda, houve uma diferenciação espontânea mínima, com mais de 85% das células expressando o marcador de células-tronco SSEA-4. A Matrix 4-XF levou à formação de aglomerados redondos 3D. Além disso, o aumento da concentração de suplemento e fatores de crescimento no meio usado para fazer a solução de hidrogel Matrix 4-XF melhorou a expressão de hiPSC de SSEA-4 em 1,3 vezes. A diferenciação do hiPSC mantido pela Matrix 2-AB levou a menos liberações de esferóides durante o estágio médio / intestino grosso em comparação com as outras membranas basais derivadas de animais. Em comparação com outras, a matriz organoide livre de xeno (Matriz 4-O3) leva a uma IO maior e mais madura, sugerindo que as propriedades físicas dos hidrogéis livres de xeno podem ser aproveitadas para otimizar a geração de organoides. Em conjunto, os resultados sugerem que variações na composição de diferentes matrizes afetam os estágios de diferenciação de IO. Este estudo aumenta a conscientização sobre as diferenças nas matrizes disponíveis comercialmente e fornece um guia para a otimização da matriz durante o trabalho de iPSC e IO.
Introdução
A matriz extracelular (MEC) é um componente dinâmico e multifuncional dos tecidos que desempenha um papel central na regulação do comportamento e desenvolvimento celular. Como uma rede complexa, fornece suporte estrutural, ligantes adesivos celulares1 e armazenamento de fatores de crescimento e citocinas que regulam a sinalização celular. Por exemplo, durante a cicatrização de feridas, a MEC serve como um andaime para células migratórias e como um reservatório de fatores de crescimento envolvidos no reparo tecidual2. Da mesma forma, a desregulação na MEC pode levar a um aumento na gravidade de várias doenças, como fibrose e câncer 3,4. Durante o desenvolvimento embrionário, a MEC orienta a morfogênese do tecido. Por exemplo, no desenvolvimento do coração, os componentes da ECM desempenham um papel na criação da arquitetura e função corretas do tecido cardíaco5. Mais de uma década de pesquisa mostrou que a rigidez do microambiente por si só 6,7 pode controlar a especificação da linhagem de células-tronco. Portanto, não é surpreendente que, durante a diferenciação celular in vitro, a ECM influencie o destino das células-tronco, fornecendo sinais para diferenciação.
Os organoides podem ser gerados a partir de células-tronco pluripotentes induzidas (iPSCs). Começar com uma linha iPSC devidamente caracterizada é necessário para gerar organoides com sucesso. No entanto, a falta de pistas fisiológicas em materiais clássicos de cultura de células dificulta a diferenciação eficiente de iPSC e a geração de organoides. Além disso, pesquisas recentes enfatizaram a importância da composição da matriz extracelular (MEC), interações entre as células e a MEC8, bem como pistas mecânicas e geométricas 9,10,11 no contexto da expansão e diferenciação de organoides12. O avanço da tecnologia organoide, melhorando a reprodutibilidade, envolverá a incorporação de pistas físicas e químicas específicas do tecido.
Os organoides visam recapitular o tecido nativo dentro de um microambiente fisiologicamente semelhante. A escolha de um sistema de ECM que imite de perto a ECM do tecido nativo é crucial para alcançar relevância fisiológica em relação ao comportamento, função e resposta celular a estímulos13. A escolha dos componentes da ECM pode influenciar a diferenciação de células-tronco em tipos específicos de células dentro do organoide. Diferentes proteínas ECM e suas combinações podem fornecer pistas que orientam o destino celular14. Por exemplo, estudos mostraram que o uso de componentes específicos da ECM pode promover a diferenciação de células-tronco intestinais em tipos de células intestinais maduras, resultando em organoides intestinais fisiologicamente relevantes15. Embora os organoides sejam uma ferramenta valiosa durante a modelagem de doenças e testes de drogas, a seleção de um sistema de ECM apropriado é fundamental para esta aplicação. Um sistema de ECM apropriado pode aumentar a precisão da modelagem da doença, criando um microambiente que se assemelha ao tecido afetado16. Além disso, a ECM específica do tecido pode ajudar a gerar organoides que recapitulam melhor os fenótipos associados à doença e as respostas aos medicamentos17. A otimização do sistema ECM usado na diferenciação de organoides é fundamental para alcançar os resultados de diferenciação desejados.
Sistemas de membrana basal disponíveis comercialmente derivados de fontes animais de ECM (por exemplo, Matrigel, Cultrex) e hidrogel livre de xeno (por exemplo, VitroGel) são amplamente utilizados em pesquisas de iPSC e organoides. As empresas que os comercializam e os pesquisadores que os utilizam estabeleceram muitas instruções para seus produtos e aplicações específicas ao longo dos anos. Muitas dessas instruções serviram de guia para a geração desse protocolo. Além disso, os benefícios e contratempos associados às suas propriedades intrínsecas têm sido observados individualmente por muitos 18,19,20,21. No entanto, não existe um fluxo de trabalho sistemático para orientar a seleção de sistemas ideais para o trabalho com iPSC e organoides. Aqui, é fornecido um fluxo de trabalho para avaliar sistematicamente a equivalência de sistemas ECM de várias fontes para iPSC e trabalho com organoides. Este é um estudo comparativo da manutenção de duas diferentes linhagens de iPSC humanas (hiPSC) e geração de organoides intestinais humanos (hIO) em quatro matrizes diferentes: Matrigel (Matriz 1-AB), Geltrex (Matriz 2-AB), Cultrex (Matriz 3-AB) e VitroGel (Matriz 4-XF). Para a cultura de organoides, foram utilizadas quatro versões da matriz VitroGel sem xeno, previamente otimizadas para cultura de organoides: ORGANOIDE 1 (Matriz 4-O1), ORGANOIDE 2 (Matriz 4-O2), ORGANOIDE 3 (Matriz 4-O3), ORGANOIDE 4 (Matriz 4-O4). Além disso, foram utilizadas matrizes de origem animal otimizadas para organoides: Matrigel High Concentration (Matrix 1-ABO) e Cultrex Type 2 (Matrix 3-ABO). Foram utilizados meios de cultura de células-tronco disponíveis comercialmente (mTeSR Plus) e kit de diferenciação de organoides (kit organoide intestinal STEMdiff). Este protocolo combina as instruções individuais dos fabricantes dos produtos com experiências de laboratório para orientar o leitor em direção a uma otimização bem-sucedida do ECM para seu trabalho específico de iPSC e organoide. Em conjunto, este protocolo e os resultados representativos enfatizam a importância de selecionar o microambiente ideal para o trabalho com células-tronco e diferenciação de organoides.
Access restricted. Please log in or start a trial to view this content.
Protocolo
1. manutenção hiPSC
CUIDADO: Todo o trabalho é feito em um Gabinete de Biossegurança (BSC) seguindo técnicas assépticas padrão. Deve seguir os padrões de segurança da OSHA para laboratórios, incluindo o uso adequado de equipamentos de proteção individual, como jalecos, luvas e óculos de proteção.
- Preparação de matrizes, alíquotas e meios de cultura celular
- Para membranas basais (BMs) derivadas de animais disponíveis comercialmente; Matriz 1-AB, Matriz 2-AB, Matriz 3-AB), preparar alíquotas de volumes de trabalho de acordo com a recomendação do fabricante resumida na Tabela 1 e armazená-las a -20 °C ou -80 °C para armazenamento de longo prazo. Evite a formação de bolhas. Se algumas bolhas forem formadas, antes de armazenar, centrifugue as amostras a 4 °C, a <200 x g, por ~ 1-2 min para forçar a bolha a vir à superfície.
NOTA: Criar alíquotas de uso único ajuda a evitar ciclos repetidos de congelamento e descongelamento que interrompem a arquitetura do ECM. Como a concentração de BM varia de acordo com o número do lote, certifique-se de seguir a recomendação do fabricante para preparar alíquotas de uso único e fazer soluções de revestimento. Os BMs altamente concentrados são viscosos e difíceis de pipetar; use pontas frias previamente armazenadas a -20 °C. - Prepare o meio de cultura de células-tronco seguindo a recomendação do fabricante. Para preparar o meio completo específico disponível comercialmente usado neste protocolo, adicione 100 mL de coquetel de suplemento 5x a 400 mL de meio basal. Em seguida, misture bem este meio e alíquota em volumes de 40 ml, conservados a 20 °C. Para uso, descongele cada alíquota de mídia completa, use a alíquota completa imediatamente ou armazene a 2 - 8 ° C por até 2 semanas. Não o congele novamente.
- Prepare alíquotas do meio completo de células-tronco contendo 3x a concentração do coquetel de suplemento para preparar o Matrix 4-XE Hydrogel, ou seja, dilua 5x suplemento para 3x.
- Para membranas basais (BMs) derivadas de animais disponíveis comercialmente; Matriz 1-AB, Matriz 2-AB, Matriz 3-AB), preparar alíquotas de volumes de trabalho de acordo com a recomendação do fabricante resumida na Tabela 1 e armazená-las a -20 °C ou -80 °C para armazenamento de longo prazo. Evite a formação de bolhas. Se algumas bolhas forem formadas, antes de armazenar, centrifugue as amostras a 4 °C, a <200 x g, por ~ 1-2 min para forçar a bolha a vir à superfície.
- Revestimento de plástico de cultura de tecidos com BMs de origem animal (Matriz 1-3AB)
- Prepare o seguinte antes de começar: sempre mantenha as matrizes no gelo ao descongelar e manusear para evitar que gelificem. Use meios frios para preparar as matrizes diluídas. Prepare poços de replicação suficientes por condição para testar as células antes de iniciar e em todos os estágios do processo de diferenciação hiPSC e geração de organoides hIO.
NOTA: Aqui, placas de 24 poços foram usadas para este estudo. Consulte a Tabela 2 para obter os volumes de revestimento recomendados para outros tamanhos de placa. - Para cada tipo de matriz, prepare 25 mL de DMEM/F-12 de avanço a frio contendo 15 mM de HEPES em um tubo cônico de 50 mL. Mantenha-os no gelo.
- Adicione as matrizes descongeladas ao seu respectivo avanço a frio DMEM / F-12 e misture bem. Mantenha a mídia fria durante o processo de mistura. Verifique visualmente se há mistura homogênea, garantindo que não haja grumos.
- Use imediatamente as soluções de matrizes diluídas para revestir cada poço selecionado para uso (250 μL / poço se estiver usando placas de 24 poços).
- Incline suavemente a cultura para espalhar a solução de revestimento uniformemente pela superfície. Incubar à temperatura ambiente (15 - 25 °C) durante pelo menos 1 h antes de utilizar.
NOTA: Se não for usada imediatamente, a placa pode ser armazenada a 2 - 8 °C por até 1 semana após o revestimento, mas deve ser selada com um filme transparente para evitar a evaporação. Ao usar placas armazenadas, deixe-as atingir a temperatura ambiente (15 - 25 °C) por 30 min antes de prosseguir para a próxima etapa. - Remova suavemente o excesso de solução usando uma pipeta sorológica ou por aspiração. Certifique-se de que a superfície revestida não esteja arranhada.
- Adicione meio de células-tronco completo quente imediatamente (50% do volume total necessário para um poço específico, por exemplo, 250 μL / poço se estiver usando uma placa de 24 poços).
- Prepare o seguinte antes de começar: sempre mantenha as matrizes no gelo ao descongelar e manusear para evitar que gelificem. Use meios frios para preparar as matrizes diluídas. Prepare poços de replicação suficientes por condição para testar as células antes de iniciar e em todos os estágios do processo de diferenciação hiPSC e geração de organoides hIO.
- Revestimento de plástico de cultura de tecidos com hidrogel Matrix 4-XE
- Prepare o seguinte antes de começar: retire o hidrogel Matrix 4-XE da geladeira e deixe-o aquecer até a temperatura ambiente (25 °C). Prepare poços de replicação suficientes por condição para testar as células antes de iniciar e em todos os estágios do processo de diferenciação hiPSC e geração de organoides hIO.
NOTA: Aqui, placas de 24 poços foram usadas para este estudo. Consulte a Tabela 3 para obter os volumes de revestimento recomendados para outros tamanhos de placa. - Prepare um meio de cultura de células-tronco completo contendo 3x concentração de fator de crescimento em comparação com a formulação padrão para cultura de células-tronco (3x meio de células-tronco).
- Misture o hidrogel Matrix 4-XE e o meio de células-tronco 3x em uma proporção de mistura de 2: 1 v / v e pipete suavemente para cima e para baixo 5x-10x para misturar completamente.
- Transfira a mistura de hidrogel para uma placa de poço e incline cuidadosamente a cultura para espalhar a mistura uniformemente pela superfície. Use 250 μL/poço se estiver usando uma placa de 24 poços. Consulte a Tabela 3 para obter os volumes recomendados.
- Aguarde 10-15 min em temperatura ambiente para a formação de gel mole. Durante o processo de formação do hidrogel, não interrompa o hidrogel inclinando ou sacudindo a placa.
- Prepare o seguinte antes de começar: retire o hidrogel Matrix 4-XE da geladeira e deixe-o aquecer até a temperatura ambiente (25 °C). Prepare poços de replicação suficientes por condição para testar as células antes de iniciar e em todos os estágios do processo de diferenciação hiPSC e geração de organoides hIO.
- Passagem e semeadura de aglomerado livre de enzimas hiPSC
- Prepare o seguinte antes de começar: pelo menos 1 h antes de passar, cubra os plásticos desejados com matrizes. Alíquota de meio completo de células-tronco suficiente e quente à temperatura ambiente (15 - 25 °C). Evite vários ciclos de aquecimento para a mídia completa.
NOTA: As etapas abaixo descrevem a passagem de uma cultura já estabelecida e >90% confluente de iPSCs em uma placa de 6 poços e a semeadura em placas de 24 poços para diferenciação de organoides intestinais. - Enxágue as células com 1 mL de D-PBS (sem Ca++ e Mg++) e aspire. Adicione 1 mL de reagente de seleção de células-tronco pluripotentes humanas sem enzimas, agite cuidadosamente para espalhar uniformemente e aspire em 1 minuto. As colônias só precisam ser expostas a uma fina película de líquido.
- Incube a 37 °C até que as colônias comecem a parecer menos compactadas, o que levará ~ 3-8 min.
NOTA: O tempo ideal de incubação pode variar dependendo da linhagem celular usada. O tempo ideal de incubação deve ser determinado ao passar pela primeira vez cada linha celular com o reagente de seleção de células-tronco pluripotentes humanas isentas de enzimas. - Adicione 1 mL de meio completo de células-tronco. Separe as colônias batendo cuidadosamente firmemente na lateral da placa. Certifique-se de segurar o prato com a outra mão.
- Com uma pipeta de 1 mL ou maior, transfira a suspensão de aglomerados de células para um tubo cônico de 15 mL. Avalie o tamanho dos aglomerados usando um microscópio de campo claro e certifique-se de que o tamanho do aglomerado de células esteja entre 50-200 μm; Se forem maiores, agite suavemente o tubo cônico para quebrá-los.
- Desenhe uma grade xy no fundo de cada poço de uma placa de fundo plano de 96 poços que será usada para contagem de touceiras e adicione 50 μL de D-PBS a cada um dos poços. Recomenda-se calcular a média dos aglomerados contados em 3 poços.
- Certifique-se de que os aglomerados de células sejam distribuídos uniformemente agitando suavemente o tubo e, em seguida, transfira 5 μL de suspensão de aglomerado para cada um dos poços da placa de fundo plano de 96 poços.
NOTA: Não é recomendado usar um contador automático de células para contar aglomerados. Recomenda-se uma contagem manual. - Conte o número total de aglomerados em cada poço com 50 a 200 μm de diâmetro. Se a maioria dos aglomerados de células tiver > de 200 μm de diâmetro (ver figura 1), repita os passos 1.4.5 - 1.4.7.
- Calcular o volume (em μL) da suspensão de touceira necessário para semear 6.000 touceiras do seguinte modo:
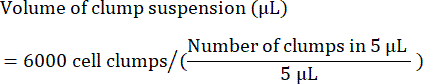
NOTA: A densidade de semeadura ideal deve ser otimizada com cada linhagem celular. Para determinar a densidade de semeadura ideal, recomenda-se realizar uma semeadura inicial em uma variedade de densidades de touceira (por exemplo, 4.000, 5.000 e 6.000 touceiras por poço) e verificar 24-48 h após a semeadura se a confluência de 85% a 90% é atingida. - Como controle de qualidade, separe uma amostra de células para exame de citometria de fluxo de marcadores de células-tronco. Consulte a Tabela 4 para obter uma lista de marcadores comuns.
- Separe o volume necessário de suspensão de aglomerado de iPSC para semear nos poços revestidos com ECM diferente em tubos separados de 15 mL.
- Centrifugue o tubo que contém a solução aglomerada de iPSC a 200 x g durante 5 minutos para remover o meio que contém o reagente de selecção de células estaminais pluripotentes humanas isentas de enzimas. Enquanto aguarda a centrifugação, adicione 50% do volume desejado por poço a cada poço, caso ainda não tenha sido feito.
- Prepare o seguinte antes de começar: pelo menos 1 h antes de passar, cubra os plásticos desejados com matrizes. Alíquota de meio completo de células-tronco suficiente e quente à temperatura ambiente (15 - 25 °C). Evite vários ciclos de aquecimento para a mídia completa.
- Cultura de iPSC em placas contendo matrizes de origem animal
- Ressuspenda suavemente os aglomerados de iPSCs no volume calculado de 50% do meio completo de células-tronco para obter a densidade desejada e coloque a mistura de agregados celulares em poços revestidos contendo 50% do meio completo de células-tronco calculado.
NOTA: A adição do inibidor de ROCK-1 pode ser necessária para algumas linhagens celulares ou quando os aglomerados são de 50 μm ou menores. Recomenda-se adicionar <10 μm para evitar gastrulação precoce. - Adicione cuidadosamente o meio com células no topo das matrizes, 250 μL / poço se estiver usando uma placa de 24 poços. Para o volume recomendado de meio celular para placas de poços de outros tamanhos, consulte a Tabela 2.
- Incline a placa em vários movimentos curtos para frente e para trás e de um lado para o outro para distribuir os aglomerados de células uniformemente.
CUIDADO: A distribuição desigual de agregados resulta em maior diferenciação espontânea de iPSCs humanas. A distribuição circular da suspensão dos aglomerados resulta em aglomeração de aglomerados na borda dos poços e menor densidade no centro. - Incubar a placa a 37 °C e realizar mudanças de meio usando meios completos de células-tronco diariamente ou em dias alternados. Ao realizar mudanças no meio, avalie visualmente as culturas para monitorar o crescimento e determinar se as células precisam de tempo de passagem ou estão prontas para diferenciação. Para pular dois dias consecutivos de alimentação, adicione 2x o volume do meio necessário para um único dia.
- Ressuspenda suavemente os aglomerados de iPSCs no volume calculado de 50% do meio completo de células-tronco para obter a densidade desejada e coloque a mistura de agregados celulares em poços revestidos contendo 50% do meio completo de células-tronco calculado.
- Cultura de iPSC em placas contendo Matrix 4-XE
- Ressuspenda suavemente os aglomerados de iPSCs no volume total calculado de 3x o meio completo de células-tronco para obter a densidade desejada.
- Adicione cuidadosamente o meio com células em cima do hidrogel 250 μL / poço se estiver usando uma placa de 24 poços. Para o volume recomendado de meio celular para placas de poços de outros tamanhos, consulte a Tabela 3.
CUIDADO: O hidrogel inchará e ocupará um volume maior do que o recém-produzido. As colônias de iPSC serão parcialmente incorporadas no hidrogel na parte inferior da placa, por isso é recomendável trocar 50% -80% do meio superior sem perturbar o hidrogel. - Mova a placa em vários movimentos rápidos, curtos, para frente e para trás e de um lado para o outro para distribuir os aglomerados de células uniformemente.
- Coloque a placa em uma incubadora a 37 °C com 5% de CO2 e 95% de umidade. Mude o meio celular usando meios completos de células-tronco. Realizar mudanças médias diariamente ou em dias alternados; para o último, adicione 2x o volume do meio.
- Inspecione visualmente as culturas para acompanhar seu crescimento à medida que atingem o estágio adequado para diferenciação.
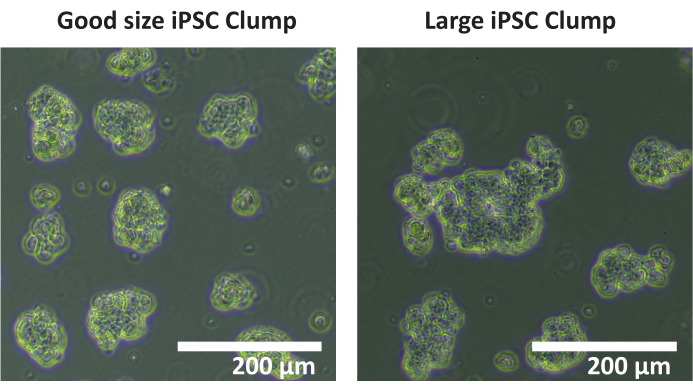
Figura 1: Tamanho ideal da touceira. Imagens de aglomerados de linhagem celular iPSC SCTi003A representando um exemplo de tamanho ideal de aglomerado. Barra de escala = 200 μm. Clique aqui para ver uma versão ampliada desta figura.
2. diferenciação hiPSC e geração de organoides intestinais
CUIDADO: Todo o trabalho é feito em um Gabinete de Biossegurança (BSC) seguindo técnicas assépticas padrão. Deve seguir os padrões de segurança da OSHA para laboratórios, incluindo o uso adequado de equipamentos de proteção individual, como jalecos, luvas e óculos de proteção.
- Preparar o seguinte antes de começar: preparar alíquotas do meio de diferenciação necessário para cada fase. Depois de descongelar as alíquotas, use-as imediatamente. Não volte a congelar. Siga a Tabela 5 para obter um guia geral de cada tipo de mídia de platina e volumes necessários para uma placa de 24 poços.
NOTA: O tempo ideal em cada estágio de diferenciação pode variar de acordo com a linha celular. Recomenda-se verificar a expressão de cada marcador celular de estágio para melhorar a eficácia da diferenciação. A avaliação periódica da expressão gênica nas culturas de iPSCS não foi incluída nesta pesquisa, uma vez que aproveitamos lotes qualificados pelo fornecedor em passagens baixas, mas é recomendada como uma etapa de controle de qualidade para linhas de iPSC novas/ainda não qualificadas durante as etapas de diferenciação subsequentes. - Estágio 1: Endoderme definitivo (DE)
- Para avaliar a prontidão para iniciar a diferenciação, verifique os seguintes critérios.
- Usando um microscópio, avalie a confluência das colônias de células-tronco e a quantidade de diferenciação espontânea na cultura. Não confie apenas na avaliação morfológica. A confluência ideal deve estar entre 85% -90% (acima de ~ 105 células / cm2), e a diferenciação espontânea mínima observada deve ser de <5% de diferenciação.
- Colete células de um poço de sacrifício para caracterização por citometria de fluxo de marcadores hiPSC antes de prosseguir com a próxima etapa. Uma estratégia de bloqueio de dispersão direta e lateral foi usada para a caracterização representativa da citometria de fluxo. Se as células atenderem aos critérios ideais, inicie a diferenciação. O critério de passagem de células é >85% de expressão de idealmente 3 marcadores (Tabela 4), especialmente no caso de linhagens iPSC novas/ainda não qualificadas no laboratório.
NOTA: As culturas de iPSC cultivadas em diferentes matrizes passam pelo mesmo processo geral com apenas o crescimento de iPSC na Matriz 4-XE, exigindo mudanças de meio mais suaves para não perturbar as células semi-incorporadas. Espera-se morte celular significativa durante a indução do endoderma definitivo, pois as células são particularmente sensíveis nesta fase. Tenha cuidado durante as mudanças de meio, com o objetivo de minimizar o tempo de exposição das células fora do ambiente de incubação a 37 °C, tanto quanto possível.
- Inicie a diferenciação das colônias de células conforme descrito abaixo.
- Dia 0: Aqueça uma alíquota de DE médio a 37 °C. Para evitar a desnaturação do meio dos ciclos de aquecimento-resfriamento, certifique-se de aquecer apenas o volume necessário para o Dia 0 (0.7 mL/poço).
- Aspire a mídia de hiPSCs. Adicione cuidadosamente 0,7 mL de meio DE por poço na lateral do poço. Evite pipetagem áspera que possa se soltar ou danificar as colônias. Incubar a placa a 37 °C com 5% de CO2 e 95% de humidade durante 24 h.
- Dia 1: Aqueça uma alíquota de meio DE contendo apenas o volume necessário para o Dia 1 (0,5 mL / poço) a 37 ° C. Aspire o meio DE das células e adicione cuidadosamente 0,5 mL de meio DE por poço na lateral do poço. Incubar a 37 °C com 5% de CO2 e 95% de umidade por 24 h.
- Dia 3: As células estão prontas para serem analisadas para a formação definitiva do endoderme. Consulte a Tabela 4 para obter os marcadores recomendados. Antes de prosseguir com a próxima fase, sacrifique um poço para realizar uma caracterização por citometria de fluxo de marcadores específicos associados à DE. Uma estratégia de bloqueio de dispersão direta e lateral foi usada para a caracterização representativa da citometria de fluxo. Recomenda-se que a expressão de mais de 1 marcador seja verificada, especialmente no caso de linhas iPSC novas/ainda não qualificadas no laboratório.
- Para avaliar a prontidão para iniciar a diferenciação, verifique os seguintes critérios.
- Estágio 2: Intestino médio/intestino posterior (MH)
- Dia 3: Aqueça apenas o volume de meio MH necessário para o dia 3 (0,5 mL / poço) a 15 - 25 ° C. Aspire o meio DE das células e substitua por 0,5 mL de meio MH. Incubar a 37 °C com 5% de CO2 e 95% de umidade por 24 h.
- Dias 4 a 9: Antes das mudanças diárias de meio, use o microscópio com ampliação de 2x ou 4x para avaliar a formação de esferoides, verificando se há estruturas 3D visíveis (pode acontecer a partir do dia 4); esferoides flutuantes (pode ocorrer a partir do dia 5).
- Assim que os esferoides começarem a se desprender, use um pipetador de 1 mL para transferir suavemente o meio das células para um tubo cônico estéril de 15 mL para avaliar o número de esferoides destacados das células.
NOTA: Evite qualquer força de cisalhamento que possa separar estruturas 3D da monocamada; Uma vez prontos, os esferóides se desprenderão por conta própria. - Se menos de 50 esferóides forem destacados, centrifugue a 200 x g por 5 min, remova o meio antigo e ressuspenda os esferóides em 0,5 mL / poço de meio e transfira para o respectivo poço até que esferoides suficientes amadureçam.
NOTA: A duração da exposição ao meio do intestino médio/intestino grosso desempenha um papel crucial na determinação da identidade regional dos organoides do intestino delgado em desenvolvimento, como duodeno (exposição mais curta) ou íleo (exposição mais longa). Para obter culturas mais uniformes com a mesma identidade, procure coletar esferoides expostos ao meio intestino médio / intestino posterior simultaneamente. - Repita a etapa 2.3.3 até que esferoides suficientes sejam destacados para incorporar e iniciar a cultura de organoides intestinais humanos (hIO).
- Antes de prosseguir com a próxima fase, separe uma amostra de esferoides e realize uma caracterização por citometria de fluxo de marcadores específicos associados ao intestino médio/intestino grosso (MH). Uma estratégia de bloqueio de dispersão direta e lateral foi usada para a caracterização representativa da citometria de fluxo. Consulte a Tabela 4 para obter uma lista de marcadores comumente usados. Recomenda-se que a expressão de mais de 1 marcador seja verificada, especialmente no caso de linhas iPSC novas/ainda não qualificadas no laboratório.
- Estágio 3: Incorporação de esferoides
- Prepare o seguinte antes de iniciar o procedimento.
- Descongele alíquotas de Matrix 1-ABO e Matrix 3-ABO no gelo. Considere o número de esferóides coletados para determinar a quantidade total de matriz necessária se 30-40 μL de matriz forem necessários por cúpula.
- Prepare 25 mL de Advance DMEM/F-12 frio contendo 15 mM HEPES. As ECMs altamente concentradas são viscosas e difíceis de pipetar. Coloque uma caixa de ponteiras de pipeta estéreis de 100 μL a -20 °C para preparar ponteiras frias que podem ajudar no processo. Prepare alíquotas de Meio de Crescimento Organoide Intestinal (OGM), que requer 4 alimentos (0,5 mL / poço por alimento). Veja a Tabela 5 para preparação.
- Descongele uma alíquota do suplemento OGM no gelo. Depois de descongelar as alíquotas, use-as imediatamente. Não volte a congelar. Armazene o OGM completo a 2-8 °C por até 2 semanas.
- Prepare OGM contendo 3x do suplemento em comparação com a formulação padrão para (3x OGM). Retire o Matrix 4-XFO1 thought Matrix 4-XFO4 da geladeira e deixe-o aquecer até a temperatura ambiente (25 °C).
- Para o sistema derivado de animais, execute as etapas descritas abaixo.
- Coloque uma placa de cultura de tecidos estéril de 24 poços na incubadora para aquecer a 37 ° C enquanto prepara os esferóides e matrizes. Deixe os esferóides coletados assentarem no fundo de um tubo cônico de 15 mL. Aspire e descarte cuidadosamente o sobrenadante.
NOTA: Para fazer uma comparação mais precisa do efeito das matrizes, certifique-se de manter os esferóides coletados do hiPSC diferenciados em cada matriz em tubos separados. - Adicione 1 mL de DMEM/F-12 com HEPES 15 mM aos esferoides. Centrifugue a 300 x g por 5 min em temperatura ambiente (15 - 25 °C).
- Remova cuidadosamente o máximo possível do sobrenadante. Recomenda-se começar a remover com um pipetador de 1 mL e mudar para pipetas de 100 μL e até 10 μL para remover o máximo possível sem perturbar o pellet de esferóides.
NOTA: Quanto maior o volume de mídia restante, mais diluídas serão as matrizes. Matrizes diluídas podem levar a problemas de gelificação ou cúpulas mais macias. Cúpulas mais macias levam a maiores chances de colapso com distúrbios mínimos. - Usando um pipetador com uma ponta de pipeta fria de 100 μL, adicione 40 μL / 50 esferóides de frio (2 - 8 ° C) Matrix 1-ABO ou Matrix 3-ABO ao respectivo tubo. Distribua suavemente os esferóides na matriz pipetando para cima e para baixo ~5x. Não esvazie completamente a ponta da pipeta, pois pode introduzir bolhas.
- Retire a placa da incubadora e, usando uma ponta de pipeta fria, transfira suavemente os esferoides embutidos para o centro da placa seguindo as etapas da Figura 2. Evite dispensar Matrix 1-ABO e Matrix 3-ABO muito rapidamente no prato de cultura, pois isso achatará a cúpula.
- Transfira cuidadosamente a placa para uma incubadora a 37 °C e incube por 30 min para garantir a gelificação da cúpula. Tenha especial cuidado com qualquer agitação ou movimento brusco durante o transporte e na incubadora.
- Enquanto incuba a cúpula para gelificação, aqueça a 37 ° C um volume suficiente de OGM intestinal (0,5 mL / poço) para o número de poços a serem usados.
NOTA: Aqueça apenas o volume necessário. Evite ciclos de aquecimento-resfriamento. Certifique-se de que a mídia esteja quente antes de adicioná-la à cúpula; O meio frio pode levar ao colapso da cúpula. - Após 30 min, adicione cuidadosamente 0,5 mL de OGM intestinal na lateral do poço para não perturbar a cúpula. Incubar a 37 °C com 5% de CO2 e 95% de humidade. Execute uma troca completa do meio a cada 3 a 4 dias, removendo o meio e, em seguida, adicione meio novo.
- Coloque uma placa de cultura de tecidos estéril de 24 poços na incubadora para aquecer a 37 ° C enquanto prepara os esferóides e matrizes. Deixe os esferóides coletados assentarem no fundo de um tubo cônico de 15 mL. Aspire e descarte cuidadosamente o sobrenadante.
- Para os sistemas organoides Matrix 4-XF, siga as etapas descritas.
NOTA: O sistema organoide Matrix 4-XF inclui quatro opções diferentes (O1-O4); A formulação de cada tipo varia em ligantes biofuncionais, rigidez e degradabilidade. Consequentemente, recomenda-se a realização de experimentos iniciais para determinar o tipo ideal para a aplicação específica. Este protocolo descreve o uso das quatro opções para encontrar uma formulação ideal para organoides intestinais derivados de hiPSC. Além disso, embora existam vários protocolos de cultura que podem ser usados (por exemplo, encapsulamento 3D, encapsulamento de cúpula), este protocolo envolve o uso do método de cúpula de forma que seja possível comparar diretamente com os métodos comuns usados ao trabalhar com sistemas de membrana basal derivados de animais.- Adicione 1 mL / poço de DMEM / F-12 avançado a frio contendo 16 mM HEPES aos esferóides gerados no sistema Matrix 4-XFO4. Centrifugue a 300 x g por 5 min em temperatura ambiente (15 - 25 °C).
NOTA: Para encontrar a formulação ideal para a aplicação, certifique-se de distribuir os esferoides em 4 tubos separados antes da centrifugação. Os esferoides gerados em matrizes derivadas de animais também podem ser amadurecidos em organoides intestinais usando este sistema. - Usando um pipetador de 1 mL, remova o sobrenadante sem perturbar os esferoides. Adicione 50 μL de 3x OGM ao pellet de esferoides (~ 100 esferoides). Adicione 100 μL do Matrix 4-XFO selecionado à suspensão esferóide de 50 μL e misture suavemente 5x-10x vezes. Mantenha o Matrix 4-XFO a 3x OGM em uma proporção de mistura de 2:1 v/v para obter uma concentração final de 1x.
- Adicione 40 μL da mistura hidrogel-esferóide ao centro de uma placa de cultura de tecidos de 24 poços. Transfira cuidadosamente as placas para uma incubadora a 37 °C e incube por 30 min.
NOTA: A matriz 4-XFO não requer incubação de 37 °C para gelificação; no entanto, recomenda-se manter os organoides expostos a condições semelhantes quando comparados aos sistemas Matrix 1-ABO e Matrix 3-ABO que requerem 37 °C para melhor gelificação das cúpulas. - Após 30 min, adicione cuidadosamente 0,5 mL de OGM intestinal na lateral do poço para não perturbar a cúpula. Incubar a 37 °C com 5% de CO2 e 95% de humidade. Execute uma troca de meio completo a cada 3 a 4 dias, removendo o meio e, em seguida, adicionando meio novo.
- Adicione 1 mL / poço de DMEM / F-12 avançado a frio contendo 16 mM HEPES aos esferóides gerados no sistema Matrix 4-XFO4. Centrifugue a 300 x g por 5 min em temperatura ambiente (15 - 25 °C).
- Prepare o seguinte antes de iniciar o procedimento.
- Passagem e maturação de hIO
- Prepare os mesmos produtos químicos, soluções e reagentes feitos na etapa 2.4.1.
- Adicione 1-2 mL de solução de enxágue antiaderente a um tubo cônico de 15 mL (1 por condição) e agite para revestir o tubo.
- Remova a solução antiaderente e enxágue os tubos com 5 mL de D-PBS (sem Ca++ e Mg++). Tampe todos os tubos revestidos e mantenha-os à temperatura ambiente (15 - 25 °C) até que seja necessário.
- Aspire o meio das cúpulas. Usando uma pipeta de 1 mL, adicione 1 mL de DMEM/F-12 frio diretamente à cúpula. O objetivo é separar as cúpulas da placa.
- Adicione mais 1 mL de DMEM/F-12 frio ao poço e pipete para cima e para baixo para colher quaisquer organoides restantes. Transfira para os tubos cônicos revestidos de 15 mL.
NOTA: Verifique a colheita bem-sucedida de organoides examinando visualmente o poço ao microscópio. Se forem observados organoides residuais, repita a etapa 2.5.5. - Com um pipetador de 1 mL, execute a pipetagem para cima e para baixo da suspensão para desintegrar os organoides até que uma suspensão de fragmento uniforme seja alcançada com o tamanho de organoide desejado (por exemplo, 100 - 500 μm).
NOTA: Utilize um pipetador de 200 μL para verificar se os organoides estão em conformidade com o tamanho recomendado. O emprego da pipeta de 200 μL facilita a fragmentação adicional conforme necessário, garantindo que os fragmentos possam passar suavemente por uma ponta de pipeta de 200 μL.
CUIDADO: Evite quebrar fragmentos em células únicas por pipetagem áspera ou prolongada. - Estabeleça a densidade organoide desejada contando fragmentos ou empregando a taxa de divisão. Separe uma alíquota e efectue a contagem utilizando o mesmo procedimento que nos passos 1.4.6 a 1.4.9 para a contagem de touceiras.
NOTA: A densidade organoide ideal deve ser otimizada por linha; Geralmente, recomenda-se uma densidade de 40 a 80 organoides intestinais por cúpula. - Certifique-se de que o tubo seja colocado no gelo durante o processo de contagem de organoides. Após aproximadamente 5 min, fragmentos organoides terão se depositado no fundo do tubo devido à gravidade.
NOTA: Quanto maior o volume da solução organoide, mais tempo levará para se estabelecer no fundo. - Elimine suavemente o máximo de sobrenadante e a camada turva formada sobre os organoides. Nas primeiras passagens, quando os organoides estão amadurecendo, essa fase turva abrange uma matriz e células individuais.
- Adicione 2 mL de DMEM/F-12 frio pipetando diretamente no pellet. Centrifugue a 200 x g por 5 min em temperatura ambiente (15 - 25 °C).
- No caso do sistema derivado de animais, retirar e eliminar cuidadosamente o sobrenadante seguindo os mesmos passos descritos nos passos 2.4.2.4-2.4.2.9.
- Para o sistema Matrix 4-XFO, remova e descarte cuidadosamente o sobrenadante seguindo as mesmas etapas descritas nas etapas 2.4.3.2-2.4.3.4.
NOTA: Ao trabalhar com o sistema Matrix 4-XFO, recomenda-se o uso de uma solução de recuperação de organoide de hidrogel sem xeno para a remoção ideal do resíduo de hidrogel. Esta solução de recuperação é especialmente recomendada ao mudar de sistemas de hidrogel de origem animal para sistemas de hidrogel sem xeno para garantir a eliminação de qualquer material xenogênico.
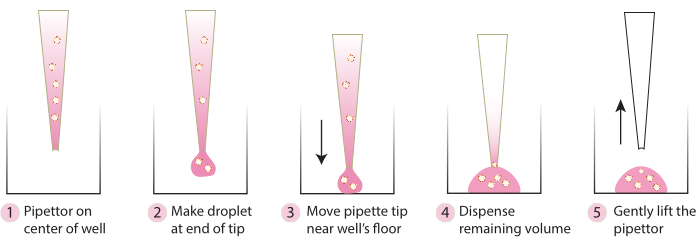
Figura 2. Esquema da técnica recomendada para a formação de cúpulas. O esquema descreve o processo passo a passo recomendado para a formação bem-sucedida de cúpulas para todos os sistemas. Clique aqui para ver uma versão maior desta figura.
3. Caracterização do tamanho de E/S
NOTA: O tamanho dos organoides foi caracterizado por imagens de campo claro tiradas em 4x e 10x. A análise do processamento das imagens foi automatizada usando o MATLAB. As etapas gerais do processo são descritas abaixo e uma amostra do código está incluída no Arquivo Suplementar 1.
- Defina o diretório que contém as imagens e liste todos os arquivos de imagem no diretório. Inicialize a tabela para armazenar os resultados. Leia as imagens de campo claro em um arquivo.
- Defina a conversão de pixel para μm e defina a escala. Se a imagem não contiver uma barra de escala, solicite ao usuário o fator de conversão de pixel para μm.
- Converta a imagem em tons de cinza. Aplique um filtro gaussiano para reduzir o ruído na imagem. Limite a imagem filtrada para separar organoides do plano de fundo.
- Preencha pequenos buracos e remova pequenos objetos para limpar a imagem binária. Realize a análise de componentes conectados para identificar organoides individuais e calcular suas propriedades, como área, centróide, comprimento do eixo principal e comprimento do eixo menor.
- Calcule o tamanho do organoide com base nos comprimentos dos eixos maior e menor. Exiba os organoides segmentados na imagem original e rotule-os com seu tamanho. Imprima os tamanhos dos organoides e armazene os resultados no arquivo .cvs.
NOTA: As primeiras imagens foram analisadas manualmente para determinar os parâmetros ideais para o desvio padrão do filtro gaussiano e a área até o limite. O código no Arquivo Suplementar 1 fornece um exemplo da estrutura básica para análise de tamanho de organoide a partir de imagens de campo claro; no entanto, é necessário um refinamento adicional para atender aos requisitos específicos de cada tipo e qualidade de imagem. O mesmo processo pode ser feito usando software de código aberto como FIJI da Image J. - Siga as etapas abaixo para usar o software FIJI.
- Defina a conversão de pixel para μm e defina a escala. Se a imagem não contiver uma barra de escala, solicite ao usuário o fator de conversão de pixel para μm clicando em Analisar > Definir escala.
- Converta a imagem em tons de cinza clicando em Imagem > Digite > 8 bits. Aplique um filtro gaussiano para reduzir o ruído na imagem clicando em Processar > Filtro > sinopse gaussiana > Sigma (raio) usada: 2.
- Limite a imagem filtrada para separar organoides do plano de fundo clicando em Imagem > Limite > MaxEntropy > Aplicar.
- Preencha pequenos orifícios e remova pequenos objetos para limpar a imagem binária clicando em Processar > Ruído > Remover Valores Atípicos > 20 pixels.
- Execute a análise de componentes conectados para identificar organoides individuais e calcular suas propriedades, como área, centroide, comprimento do eixo principal e comprimento do eixo menor, clicando em Analisar > Definir medição > Certifique-se de que a Área, o Perímetro e o Diâmetro estejam incluídos > Analisar > Analisar Partículas > Mostrar Contornos.
- Análise estatística
- Avaliar a normalidade dos dados por meio do teste de Saphiro-Wilk usando o software JMP (SAS). Para examinar as diferenças estatísticas entre os grupos, realizar uma ANOVA bidirecional e realizar testes post-hoc usando o método não paramétrico de Wilcoxon no software JMP(SAS). A significância foi estabelecida em um nível alfa de p ≤ 0,05.
Access restricted. Please log in or start a trial to view this content.
Resultados
Seguindo este protocolo, membranas basais disponíveis comercialmente e um sistema de hidrogel livre de xeno foram utilizados com sucesso para cultivar células hiPSC e diferenciá-las em hIO. O principal objetivo desses experimentos foi avaliar sistematicamente a equivalência de matrizes de várias fontes para o trabalho de hiPSC e hIO. A primeira seção deste protocolo enfocou a manutenção e caracterização de uma cultura de iPSC saudável que produz uma geração eficiente de organoides intestinais. O processo de...
Access restricted. Please log in or start a trial to view this content.
Discussão
Selecionar o microambiente ideal para o trabalho com células-tronco e organoides é um passo inicial fundamental ao usar essas plataformas para uma ampla gama de aplicações. Nossos resultados representativos mostram que a Matriz 4-XFO3, em combinação com uma maior concentração de fatores de crescimento, leva a organoides maiores, sugerindo que as propriedades físicas dos hidrogéis livres de xeno podem ser aproveitadas para otimizar a geração de organoides usando esses sistemas. Foi demonstrado anteriormente qu...
Access restricted. Please log in or start a trial to view this content.
Divulgações
O Dr. John Huang é fundador e CEO da TheWell Bioscience.
Agradecimentos
Os autores reconhecem o treinamento anterior e as recomendações gerais sobre o início do trabalho com hiPSC e organoides dos Drs. Christina Pacak, Silveli Susuki-Hatano e Russell D'Souza. Eles agradecem à Dra. Chelsey Simmons por sua orientação no uso de sistemas de hidrogel para trabalhos de cultura de células in vitro . Além disso, os autores gostariam de agradecer aos Drs. Christine Rodriguez e Thomas Allison, da STEMCELL Technologies, por sua orientação sobre a cultura hiPSC. Os autores também agradecem à TheWell Bioscience por cobrir os custos de publicação.
Access restricted. Please log in or start a trial to view this content.
Materiais
| Name | Company | Catalog Number | Comments |
| 24-Well Plate (Culture treated, sterile) | Falcon | 353504 | |
| 37 °C water bath | VWR | ||
| 96-well plate | Fisher Scientific | FB012931 | |
| Advanced DMEM/F12 | Life Technologies | 12634 | |
| Anti-adherence Rinsing Solutio | STEMCELL Technologies | 7010 | |
| Biological safety cabinet (BSC) | Labconco | Logic | |
| Brightfield Microscope | Echo Rebel | REB-01-E2 | |
| BXS0116 | ATCC | ACS-1030 | |
| Centrifuge with temperature control (4 °C capabilities) | ThermoScientific | 75002441 | |
| Conical tubes, 15 mL, sterile | Thermo Fisher Scientific | 339650 | |
| Conical tubes, 50 mL, sterile | Thermo Fisher Scientific | 339652 | |
| Cultrex RGF BME, Type 2 | Bio-techne | 3533-005-02 | |
| Cultrex Stem Cell Qualified RGF BME | Bio-techne | 3434-010-02 | |
| D-PBS (Without Ca++ and Mg++) | Thermo Fisher Scientific | 14190144 | |
| GeltrexLDEV-Free, hESC-Qualified Reduce Growth Factor | Gibco | A14133-02 | |
| GlutaMAX Supplement | Thermo Fischer Scientific | 35050-061 | |
| Guava Muse Cell Analyzer or another flow cytometry equipment (optional) | Luminex | 0500-3115 | |
| HEPES buffer solution | Thermo Fischer Scientific | 15630-056 | |
| Heralcell Vios Cell culture incubator (37 °C, 5% CO2) | Thermo Scientific | 51033775 | |
| JMP Software | SAS Institute | JMP 16 | |
| MATLAB | MathWorks, Inc | R2022b | |
| Matrigel Growth Factor Reduced (GFR) Basement Membrane Matrix LDEV free | Corning | 356231 | |
| Matrigel Matrix High Concentration (HC), Growth Factor Reduced (GFR) LDEV-free | Corning | 354263 | |
| mTeSR Plus Medium | STEMCELL Technologies | 100-0276 | |
| Nunclon Delta surface treated 24-well plate | Thermo Scientific | 144530 | |
| PE Mouse Anti-human CD326 (EpCAM) | BD Pharmingen | 566841 | |
| PE Mouse Anti-human CDX2 | BD Pharmingen | 563428 | |
| PE Mouse Anti-human FOXA2 | BD Pharmingen | 561589 | |
| PerCP-Cy 5.5 Mouse Anti-human SSEA4 | BD Pharmingen | 561565 | |
| ReLeSR | STEMCELL | 5872 | |
| SCTi003-A | STEMCELL Technologies | 200-0510 | |
| Serological pipettes (10 mL) | Fisher Scientific | 13-678-11E | |
| Serological pipettes (5 mL) | Fisher Scientific | 13-678-11D | |
| STEMdiff Intestinal Organoid Growth Medium | STEMCELL Technologies | 5145 | |
| STEMdiff Intestinal Organoid Kit | STEMCELL Technologies | 5140 | |
| Vitrogel Hydrogel Matrix | TheWell Bioscience | VHM01 | |
| VitroGel ORGANOID Discovery Kit | TheWell Bioscience | VHM04-K |
Referências
- Hynes, R. O. Integrins: Bidirectional, allosteric signaling machines. Cell. 110 (6), 673-687 (2002).
- Frantz, C., Stewart, K. M., Weaver, V. M. The extracellular matrix at a glance. J Cell Sci. 123, Pt 24 4195-4200 (2010).
- Hinz, B., Gabbiani, G. Fibrosis: Recent advances in myofibroblast biology and new therapeutic perspectives. F1000 Biol Rep. 2, 78(2010).
- Pickup, M. W., Mouw, J. K., Weaver, V. M. The extracellular matrix modulates the hallmarks of cancer. EMBO Rep. 15 (12), 1243-1253 (2014).
- Rozario, T., DeSimone, D. W. The extracellular matrix in development and morphogenesis: A dynamic view. Dev Biol. 341 (1), 126-140 (2010).
- Even-Ram, S., Artym, V., Yamada, K. M. Matrix control of stem cell fate. Cell. 126 (4), 645-647 (2006).
- Engler, A. J., Sen, S., Sweeney, H. L., Discher, D. E. Matrix elasticity directs stem cell lineage specification. Cell. 126 (4), 677-689 (2006).
- Tran, O. N., et al. Organ-specific extracellular matrix directs trans-differentiation of mesenchymal stem cells and formation of salivary gland-like organoids in vivo. Stem Cell Res Ther. 13 (1), 306(2022).
- Nikolaev, M., et al. Homeostatic mini-intestines through scaffold-guided organoid morphogenesis. Nature. 585 (7826), 574-578 (2020).
- Gjorevski, N., et al. Designer matrices for intestinal stem cell and organoid culture. Nature. 539 (7630), 560-564 (2016).
- Gjorevski, N., et al. Tissue geometry drives deterministic organoid patterning. Science. 375 (6576), (2022).
- Heo, J. H., Kang, D., Seo, S. J., Jin, Y. Engineering the extracellular matrix for organoid culture. Int J Stem Cells. 15 (1), 60-69 (2022).
- Shamir, E. R., Ewald, A. J. Three-dimensional organotypic culture: experimental models of mammalian biology and disease. Nat Rev Mol Cell Biol. 15 (10), 647-664 (2014).
- Clevers, H. Modeling development and disease with organoids. Cell. 165 (7), 1586-1597 (2016).
- Jung, P., et al. Isolation and in vitro expansion of human colonic stem cells. Nat Med. 17 (10), 1225-1227 (2011).
- Lancaster, M. A., Knoblich, J. A. Organogenesis in a dish: Modeling development and disease using organoid technologies. Science. 345 (6194), 1247125(2014).
- Huch, M., et al. In vitro expansion of single Lgr5+ liver stem cells induced by Wnt-driven regeneration. Nature. 494 (7436), 247-250 (2013).
- Greenlee, A. R., Kronenwetter-Koepel, T. A., Kaiser, S. J., Liu, K. Comparison of Matrigel and gelatin substrata for feeder-free culture of undifferentiated mouse embryonic stem cells for toxicity testing. Toxicol In Vitro. 19 (3), 389-397 (2005).
- Geltrex LDEV-Free, HESC-Qualified, Reduced Growth Factor Basement Membrane Matrix User Guide (Pub.No. MAN0007336 3.0. Fisher Scientific. , Available from: https://www.thermofisher.com/document-connect/document-connect.html?url=https://assets.thermofisher.cn/TFS-Assets%2FLSG%2Fmanuals%2FGeltrex_LDEV_Free_hESC_qualified_PI.pdf (2024).
- biotechne R&D Systems. Cultrex Stem Cell Qualified Reduced Growth Factor. biotechne R&D Systems. , (2024).
- VitroGel Organoid Protocol. TheWell Bioscience. , Available from: https://www.thewellbio.com/video-protocols (2024).
- Spence, J. R., et al. Directed differentiation of human pluripotent stem cells into intestinal tissue in vitro. Nature. 470 (7332), 105-110 (2011).
- Henderson, J. K., et al. Preimplantation human embryos and embryonic stem cells show comparable expression of stage-specific embryonic antigens. Stem Cells. 20 (4), 329-337 (2002).
- Haruna, N. F., Huang, J. Investigating the dynamic biophysical properties of a tunable hydrogel for 3D cell culture. J Cytol Tissue Biol. 7, 30(2020).
- Cherne, M. D., et al. A synthetic hydrogel, VitroGel ORGANOID-3, improves immune cell-epithelial interactions in a tissue chip co-culture model of human gastric organoids and dendritic cells. Front Pharmacol. 12, 707891(2021).
- Stewart, D. C., et al. Quantitative assessment of intestinal stiffness and associations with fibrosis in human inflammatory bowel disease. PLoS One. 13, e0200377(2018).
- Hernandez-Gordillo, V., et al. Fully synthetic matrices for in vitro culture of primary human intestinal enteroids and endometrial organoids. Biomaterials. 254, 120125(2020).
- Broguiere, N., et al. Growth of epithelial organoids in a defined hydrogel. Adv Mater. 30, 1801621(2018).
- Barthes, J., et al. Cell microenvironment engineering and monitoring for tissue engineering and regenerative medicine: The recent advances. BioMed Res Int. 2014, 921905(2014).
- Engler, A. J., Sen, S., Sweeney, H. L., Discher, D. E. Matrix elasticity directs stem cell lineage specification. Cell. 126 (4), 677-689 (2006).
- Aisenbrey, E. A., Murphy, W. L. Synthetic alternatives to Matrigel. Nat Rev Mater. 5 (7), 539-551 (2020).
Access restricted. Please log in or start a trial to view this content.
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados