JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
구슬에 갈렉틴-3 - U1 snRNP 복합체에 의한 접합 활동의 보완
요약
이 문서에서는 (a) 핵 추출물에서 U1 snRNP의 고갈에 대한 실험 절차에 대해 설명하며, 접합 활동의 수반되는 손실; (b) Galectin-3에 의한 U1 고갈 된 추출물에서 접합 활성의 재구성 - U1 snRNP 입자는 항 갈류 -3 항체와 함께 공동으로 구슬에 결합된다.
초록
고갈-재헌법 실험은 galectin-3이 핵 추출물에 필요한 접합 인자임을 나타냅니다. 접합 경로에 galectin-3을 통합하는 메커니즘은이 논문에서 해결된다. 12%-32% 글리세롤 그라데이션에 HeLa 세포 핵 추출물의 침전물은 galectin-3 및 U1 snRNP를 포함하는 내생 ~10S 입자에 농축된 분획을 산출합니다. 우리는 지금 접합 활동의 수반되는 손실로 U1 snRNP의 핵 추출물을 고갈시키는 프로토콜을 기술합니다. U1 고갈된 추출물의 접합 활성은 항갈류-3 항체와 함께 아가로스 비드에 갇힌 U1 snRNP 입자에 의해 재구성될 수 있다. 결과는 galectin-3 - U1 snRNP - 사전 mRNA 대동맥 복합체가 접합 반응의 중간 및 제품으로 이어지는 기능적인 E 복합체이고 galectin-3는 U1 snRNP와의 협회를 통해 접합 통로를 입력한다는 것을 표시합니다. 특정 접합계의 고갈된 추출물에서 접합 활성을 재구성하기 위해 비드에서 복잡한 친화성 또는 면역 선택을 사용하는 방식은 일반적으로 다른 시스템에 적용될 수 있다.
서문
대부분의 진핵 메신저 RNA (mRNA)의 생산은 핵 프로세스에서 인트론의 제거및 엑손의 결찰을 포함 1. RNA 단백질 복합체(RNP)의 2부류는 스플리세이세탈 복합체를 통해 숙사 RNA를 성숙한 mRNA로 직접 처리한다. 1부, 초기 사전 메신저 RNP는, 이질적인 핵 RNP 단백질 및 SR 제품군의 일부 구성원을 포함한 다른 RNA 결합 단백질의 결합에 의해 공동 전사적으로 형성되어 hnRNP 복합체2를 산출한다. 두 번째 클래스, 우라실이 풍부한 소형 핵 RNP(U1, U2, U4, U5 및 U6 snRNAs를 가진 U snRNP)는 U-특이적 및 핵심 단백질3,4와 관련이 있습니다. U snRNP는 인트론이 절제되고 성숙한 mpRNPs5를 생성하기 위해 기근이 계게됨에 따라 동적 리모델링 경로에서 사전 메신저 RNP의 특정 영역과 정렬 된 방식으로 상호 작용합니다. 많은 추가 핵 단백질이 이러한 처리 이벤트에 참여6.
Galectin-1 (Gal1) 및 galectin-3 (Gal3)는 고갈 재구성 연구에 의해 표시된 바와 같이 접합 경로에 필요한 두 가지 단백질입니다7,8. 유능한 핵 추출물(NE)을 접합하는 데 서 두 갈대린을 제거하는 것은 초기 단계에서 화려한 조립과 접합 활동을 폐지합니다. 이러한 이중 고갈 된 NE에 둘 다 갈대린의 추가는 두 활동을 복원. Gal1 및 Gal3는 Gal1 또는 Gal39에 특이적인 항혈제에 의해 사전 mRNA, 접합 중기 및 성숙한 mRNA의 특정 면역 침전에 의해 입증된 활성 스pliceosomes의 성분입니다. 중요한 것은, Gal3는 항갈3 항세라10에 의한 스니프의 침전에 의해 도시된 바와 같이 접합 통로 외부에 NE내 입자를 함유하는 내인성 U snRNA와 연관된다. 마지막으로, HeLa 세포에 있는 Gal3의 침묵은 수많은 유전자11의 접합 패턴을 변경합니다.
미리 배양된 NE에서 미리 배양되어 미리 형성된 스플리세솜12, snRNPs는 7S에서 60S 이상으로 글리세롤 그라데이션에서 퇴적하는 여러 복합체에서 발견된다. 글리세롤 그라데이션 분획은 스플리케소말 복합체 및 성분의 분리를 위한 일반적인 기술이지만(예를 들어 참조13,14,15 참조), 항체 면역 침전을 사용하여 특정 분수를 특성화하여 이 방법을 확장했습니다. 10S에서 snRNP 퇴적물은 Gal3와 함께 U1 snRNA만 포함합니다. 갈3 또는 U1 snRNP에 특이적 항세라를 가진 10S 분획의 면역 침전은 U1과 Gal3 의 일부를 나타내는 U1 과 Gal3 모두 의 일부를 나타내는 Gal310에 결합된다. U1 snRNP는 화려한 어셈블리1,5에서 사전 mRNP에 결합하는 첫 번째 복합체이므로 이 단계는 접합 경로에 Gal3의 잠재적 진입 지를 나타냅니다. 이를 바탕으로, 10S Gal3-U1 snRNP 단입자가 U1 snRNP고갈약된 NE에 접합 활성을 복원한 구슬을 함유한 항갈3에 결합된 단입자가 이 복합체를 spliceosopathway16에 모집되는 하나의 메커니즘으로 확립하는 것으로 나타났습니다. 이것은 접합 반응의 특정 단계에서 spliceosomes를 분리하고 관련 요인을 분류하는 시도와 대조17,18. 이러한 연구에서, 특정 요인의 존재는 어느 시점에서 확인 하지만 그들은 로드 된 메커니즘.
우리는 이전에 NE의 준비, 접합 기판, 접합 반응 혼합물의 조립 및 사전 mRNA 접합19에서 galectins의 역할에 대한 문서화에서 제품의 분석을 상세히 설명했습니다. 우리는 지금 Gal3 - U1 snRNP 복합체 및 U1 고갈된 핵 추출물에서 접합 활동을 재구성하기 위하여 후자 의 면역 선택을 위해, Gal3에 농축된 분획을 위한 실험 절차를 기술합니다.
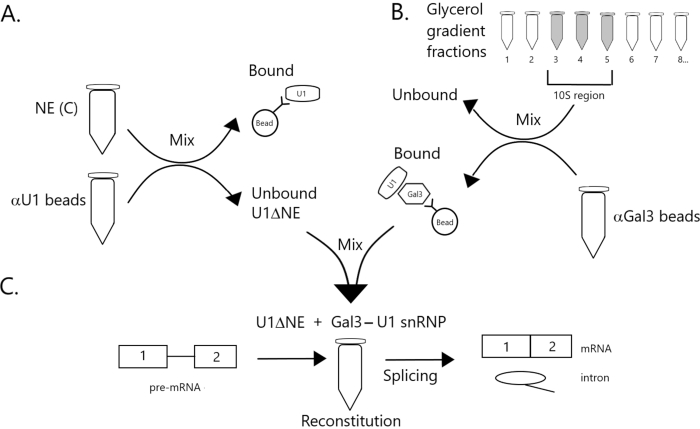
도 1: 구슬에 Gal3-U1 snRNP 복합체에 의해 U1 snRNP의 고갈된 핵 추출물에서 접합 활성의 보완을 보여주는 회로도 도표. (A) NE 버퍼 C (NE(C)) 단백질 A-Sepharose 비즈와 함께 배양된다.). 언바운드 분수는 U1 snRNP(U1ΔNE)의 고갈됩니다. (B) 완충D(NE)에서 NE은 초원심분리에 의한 12%-32% 글리세롤 그라데이션을 통해 분획된다. 10S 영역(분수 3-5)에 대응하는 분획은 항갈3 항체(αGal3 구슬)와 함께 구슬과 결합되고 혼합된다. 구슬에 바인딩된 재료에는 Gal3-U1 snRNP 단입자가 포함되어 있습니다. (C) 파트(B)로부터의 Gal3-U1 snRNP 복합체는 32P 표지된 MINX 프리mRNA 기판 및 접합 반응의 중간 및 제품을 사용하여 부품(A)으로부터 U1ΔNE과 혼합되어 겔 전기전구및 사방사선에 의해 분석된다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
Access restricted. Please log in or start a trial to view this content.
프로토콜
1. 일반 절차에 대한 참고 사항
- 모든 화학 물질(완충 성분, 효소 등)이 리보누칼리스(RNase)가 없는지 확인합니다. 격리는 일반 실험실 사용에서 상업적으로 구입 한 모든 시약 병. 실험 절차의 모든 단계에 장갑을 착용하십시오. 구운 유리 제품과 기구만 사용하십시오(아래 1.2단계 참조) 및 사전 처리된 솔루션(아래 단계 1.3 참조).
- 모든 유리제품(비커, 플라스크, 병, 파이펫 등)을 최소 4시간 177°C로 굽습니다. 같은 조건에서 베이킹하기 전에 다른 기구 (주걱, 볶음 바 등)를 알루미늄 호일에 싸서.
- 이중 증류수(ddH2O)에서 디틸피로카보네이트(DEPC)의 0.1%(vol/vol) 용액을 준비한다. 마그네틱 스터드 바를 사용하여 이 용액을 밤새 저어준 다음 자동 클라브를 사용합니다. 이 DEPC 처리 H2O를 사용하여 Tris를 포함하는 모든 솔루션을 만듭니다. 그런 다음, 필터는 병 상단 진공 필터를 사용하여 살균. 일반 ddH2O를 사용하여 트리스 없이 다른 모든 솔루션을 준비합니다. 그런 다음 DEPC (0.1 %, vol/vol) 및 autoclave로 치료하십시오.
참고: 다음 실험 절차 집합에 사용되는 버퍼는 표 1에 알파벳 순으로 나열됩니다.
| 버퍼 이름 | 구성 |
| 붕산 버퍼 | 0.2 M 산염, pH 9 |
| 버퍼 C | 20m HEPES, pH 7.9, 25% (vol/vol) 글리세롤, 0.42 M NaCl, 1.5 mM MgCl2, 0.2 mM EDTA, 0.5 mM 페닐메틸술포닐 플루오라이드 (PMSF), 0.5 mM 디티오트레이톨 (DTT) |
| 버퍼 D | 10mHEPES, pH 7.9, 20% (vol/vol) 글리세롤, 0.1 M KCl, 0.2 mM EDTA, 0.5 mM PMSF, 0.5 mMM DTT |
| 60%D | 60% 버퍼 D 및 40% H2O |
| 에타놀라민 | 0.2 M 에탄올라민, pH 8 |
| HEPES 바인딩 버퍼 | 20 mM 헤페, pH 7.9 |
| HEPES 세척 버퍼 | 20m HEPES, pH 7.9, 0.5 M NaCl |
| RNA 로딩 버퍼 | 90% 포르마미드, 20mM EDTA, pH 8, 0.05% (w/v) 브로모페놀 블루 |
| SDS 페이지 버퍼 | 25mM Tris, 169m 글리신, 0.1% 나트륨 도데딜 황산염(SDS), pH 8.8 |
| SDS 샘플 버퍼 | 62.5 mM Tris, pH 6.8, 2% SDS, 10% 글리세롤, 5% 2-메르카토에탄올, 0.1% (w/v) 브로모페놀 블루 |
| RNA 젤용 TBE 버퍼 | 89 mM 트리스, 89 mM 붕산, 2.5 mM EDTA, pH 8.3 |
| TE 버퍼 | 10 mM 트리스, pH 8, 1 mM EDTA |
| 전송 버퍼 | 25 mM 트리스, 1.92 M 글리신, 20% 메탄올, pH 8.3 |
| T-TBS 버퍼 | 10mM Tris, 0.5 M NaCl, 0.05% Tween 20, pH 7.5 |
| TX 세척 버퍼 | 0.05% 트리톤 X-100(TX) 60%D |
표 1: 버퍼의 이름 및 구성
2. U1 snRNP의 고갈 된 NE 준비 (U1 ΔNE)
- 면역 흡착을 위한 항 U1 구슬의 준비
- 사전 팽창 50 mg 단백질 A-Sepharose CL-4B 구슬을 초과 DEPC 처리 H2O에서 약 200 μL의 부은 구슬을 생산한 다음 HEPES 세척 버퍼에서 세척합니다.
- 이 세척 및 모든 후속 세척을 위해, 구슬은 원심분리(10-15초 동안 4°C의 스윙 버킷 로터에 1,000 x g )에 의해 펠릿을 제거하고 마이크로파이프를 사용하여 언바운드 세척을 제거하고 폐기한다.
- U1 snRNP에 특이적 인 인간 자가 면역 혈청의 150 μL과 세척 된 구슬 150 μL을 혼합합니다 (1:1의 비율로 구슬의 부피에 항체의 부피).
- 조정,의 총 부피 (위의 단계 2.1.3에서 ~300 μL), 혼합물에 20 mHEPES, pH 7.9, HEPES 바인딩 버퍼의 조건에 해당; 이 혼합물을 실온에서 60분 동안 연속 흔들면 배양합니다.
- 붕대 완충제(0.2M 화분, pH 9)로 항체로 묶인 구슬을 세척하고 동일한 붕산 완충제의 1mL로 재연한다.
- 단백질 A-Sepharose 구슬에 결합된 항체를 공동으로 결합하려면 20mMM의 최종 농도에 디메틸플리메이니데이트를 추가하고 60분 동안 흔들면 실온에서 배양합니다.
- 붕대 버퍼 1mL로 구슬을 씻으시다.
- 반응하지 않은 교차 연결 시약을 차단하려면 0.2 M 에탄올라민(pH 8)의 1mL을 추가하고 실온에서 60분 동안 흔들리면 배양합니다.
- 항체 결합 구슬을 세척하되, 그 후 항U1 구슬로 지정되어 TX 세척 버퍼0.5mL(60% D에서 0.05% 트리톤 X-100)로 두 번 세척한다.
- NE에서 U1 snRNP의 고갈 ( 그림 1A 참조)
참고: HeLa 세포에서 NE를 준비하기 위한 절차는 처음에 Dignam et al.20에 의해 개발되었습니다. 우리는 assays19 를 접합하기위한 NE의 준비를위한 재료와 자세한 방법을 설명했습니다 (해당 참조의 단계 2.1 및 3.1 을 참조하십시오). NE는 처음에 준비된 대로 버퍼 C에 있으며, 그 후 NE(C)로 지정됩니다. 완충D로 투약되고 평형된 NE(C)는 NE(D)로 지정됩니다.- 위의 단계 2.1.9에서 안티 U1 구슬의 100 μL와 NE (C)의 200 μL을 배양한다.
- 혼합물에 RNasin 5 μL을 추가합니다.
- 마이크로튜브 헤드 오버 테일을 4°C에서 1시간 동안 회전합니다.
- 10-15s의 4°C에서 스윙 버킷 로터에서 원심분리(1,000 x g )에 의한 혼합물을 펠렛하고 해밀턴 주사기를 사용하여 언바운드 재료(U1ΔNE)를 수집한다.
- U1ΔNE의 전체 부피와 함께 원래 비고갈이 없는 NE(C)의 별도의 50 μL 알리쿼트와 함께 마이크로디아리저의 별도의 구획에서 교반하여 8K 분자 중량 차단이 있는 투석 막을 사용하여 60% D에 대해 75분 동안 투석을 합니다.
- 투석 직후, 이러한 제제(U1ΔNE 및 NE를 60% D)로 나누어 20 μL 알리쿼트로 나눈다. 그런 다음 드라이 아이스 /에탄올 욕조에서 동결을 스냅하고 -80 ° C에 보관하십시오.
- U1ΔNE의 RNA 및 단백질 함량 분석 및 항 U1 구슬에 결합된 물질
- 언바운드 재료(U1ΔNE)(단계 2.2.4)를 제거한 후, TX 세척 버퍼의 0.5mL을 추가하여 U1 구슬에 묶인 재료를 세척한다. 원심분리에 의한 혼합물을 펠렛(10-15s에 대해 4°C의 스윙 버킷 로터에서 1,000 x g )하고 마이크로파이프를 사용하여 상류체를 제거하고 폐기한다.
- 워시 단계 2.3.1을 두 번 반복하십시오.
- 구슬의 100 μL에 2x SDS 샘플 버퍼의 100 μL을 추가하고 실온에서 10 분 동안 배양하여 안티 U1 구슬에 바인딩 된 재료를 제거합니다.
- 원심분리에 의한 혼합물을 펠렛(10-15s에 대해 4°C에서 스윙 버킷 로터에 1,000 x g ); 해밀턴 주사기의 상체를 제거하고 드라이 아이스/에탄올 욕조에서 얼립니다. -80 °C에 보관하십시오.
- 비고갈이 없는 NE, 고갈된 NE(U1ΔNE) 및 구슬에 바인딩된 물질을 비교합니다(위의 단계 2.3.3 및 2.3.4단계에 설명된 대로 SDS 샘플 버퍼에 의해 구슬에서 제거됨). RNA 분석을 위한 단계 2.3.6-2.3.8 또는 단백질 분석을 위한 단계 2.3.9-2.3.10을 따르십시오.
- 각 샘플에 대해 페놀 클로로폼의 200 μL로 RNA를 추출하십시오(50:50, v/v); 그런 다음 클로로폼 이소아밀 알코올의 180 μL로 다시 추출하십시오 (25:1, v/v). 추출 후, 감기 200-proof 에탄올의 300 μL을 추가하고, 혼합을 반전시키고, 침전된 RNA를 하룻밤 동안 -20°C에 저장한다.
- 원심분리기에 에탄올 침전 RNA(4°C에서 10분 동안 12,000 x g ). 차가운 70% 에탄올의 150 μL로 펠릿을 씻으시다. 원심분리기는 4°C에서 다시 (12,000 x g)를 15분 동안. 마이크로 파이프를 사용하여 상체를 제거하고 열없이 10-15 분 동안 속도 진공 청소기에서 펠릿을 건조하십시오.
- RNA 적재 버퍼의 10 μL에서 건조 된 RNA 펠릿을 다시 중단, 부드럽게 소용돌이, 90 s에 대한 75-85 ° C에 가열, 다음 2 분 동안 얼음에 배양. 젤 전기포고증(16mA에서 2시간)을 13% 폴리아크릴아미드-8.3M 우레아 젤로 분리한 다음 에티듐 브로마이드로 얼룩지거나 북부 블로팅10,16의 대상이 된다.
- 단백질 샘플을 적재하여, SDS 샘플 버퍼에서 2.3.5단계로부터, 12.5% 폴리아크릴아미드 젤및 전기파에 200V에서 SDS-PAGE(나트륨 도데딜 황파폴산염 폴리아크라이미드 젤 전기포프리시스) 완충제에 적재한다.
- 분리된 단백질을 400mA에서 니트로셀룰로오스 막으로 전달하여 2시간 동안 전달완충제한다. 전송 후, 10 % 비 지방 건조 우유를 포함하는 T-TBS에서 하룻밤 배양하여 멤브레인을 차단합니다. 이어서, 특정 단백질을 드러내기 위해 막을 면역플로트8,21.
3. 항갈3에 의한 글리세롤 그라데이션의 10S 분획의 면역 침전
- 면역 흡착을위한 항 갈3 구슬의 준비
참고: 토끼 #2421 및 토끼 #4910에 대한 Gal3에 대한 토끼 폴리클론 항세라의 파생 및 특성화는 이전에 설명되었습니다.- 토끼 #49에서 면역 세럼을 제어로 사용하십시오.
- 항갈3 구슬의 제조를 위해 이전에 안티 U1 구슬의 제조를 위해 이전에 설명된 절차를 따르며(단계 2.1), 2.1.3 단계에 해당하는 경우를 제외하고, 비드에 대한 항세럼의 비율(예를 들어, 항갈3, #49)의 비율은 3:1이다.
- 사용하기 직전에 항체 결합 구슬을 세척하여 항갈3 구슬로 지정하여 0.5mL의 TX 세척 버퍼로 두 번 세척하십시오. 먼저 마이크로 파이프로 액체의 대부분을 꺼내고 해밀턴 주사기를 사용하여 액체를 구슬에서 꺼내십시오. 버리다.
- 항갈3에 의한 글리세롤 그라데이션 분획의 면역전시전탄( 그림 1B 참조)
- 12%-32% 글리세롤 그라데이션10을 통해 NE(D)를 분수합니다. 그라데이션의 10S 영역 근처에 있는 글리세롤 그라데이션 분수 3, 4 및 5(그라데이션의 상단에서 번호가 매겨짐)를 결합하고 혼합합니다.
- 두 개의 샘플을 준비, 결합 된 그라데이션 분획의 150 μL 알리쿼트 각각 3-5 (단계 3.2.1), 안티 Gal3 구슬의 50 μL에 배치.
- 병렬로, 분획 1의 150 μL (U1 snRNP10복합체, 단계 3.2.1)로 각각 두 개의 샘플을 준비하고 항 갈3 구슬의 50 μL에 놓습니다.
- 대조군으로서, 50 μL 항갈3 구슬의 또 다른 마이크로튜브에 60% D의 150 μL을 놓습니다.
- 튜브를 눌러 부드럽게 섞은 다음 마이크로튜브 헤드 오버 테일을 4°C에서 1시간 동안 회전합니다.
- 부드러운 원심분리에 의해 혼합물을 펠렛 (10-15 s에 대한 4 ° C에서 스윙 버킷 로터에서 1,000 x g ).
- 해밀턴 주사기를 사용하여 상수(언바운드 재질)를 제거합니다. 구슬을 씻지 말고 접합 반응(섹션 4.2)을 추가하려면 즉시 사용하십시오.
- 10S 그라데이션 분획의 항 갈3 강수량으로부터 의 결합되지 않은 물질내 RNA 및 단백질 함량 분석
- 10S 그라데이션 분획의 항갈3 강수량으로부터 바운드 및 언바운드 물질의 성분을 분석하기 위해, 언바운드 물질(단계 3.2.6 후 의 상수화)을 수집하고, 신선한 마이크로튜브로 옮기고, -20°C에서 동결한다.
- TX 세척 버퍼0.5mL를 추가하여 침전된 구슬을 3.2.6단계(항갈3에 결합한 재료 포함)에서 세척합니다.
- 부드러운 원심분리에 의해 혼합물을 펠렛 (10-15 s에 대한 4 °C에서 스윙 버킷 로터에서 1,000 x g ); 마이크로 파이프를 사용하여 상체를 제거하고 폐기합니다. 세척 단계를 두 번 더 반복합니다.
- 세척 및 펠렛 안티 Gal3 구슬에 2X SDS 샘플 버퍼의 50 μL을 추가합니다.
- 구슬을 부드럽게 섞고 실온에서 10분 동안 배양합니다.
- 부드러운 원심분리(10-15s의 경우 4°C의 스윙 버킷 로터에서 1,000x g )로 혼합물을 펠릿, 해밀턴 주사기에 의해 상전자를 수집하고 -20°C에서 신선한 마이크로튜브에 보관한다.
- RNA 및 단백질 성분의 관점에서 항갈3 강수량의 언바운드 물질(3.3.1) 및 결합물질(Step 3.3.6)을 단계 2.3.6에 기재된 바와 같이 절차를 이용하여 비교한다. 각각 2.3.10으로.
4. 접합 반응 및 제품 분석 의 조립
- 접합 기판의 준비
참고: MINX로 지정된 사전 mRNA 기판에는 Adenovirus22의 2개의 엑손 서열과 1개의 인트론 서열이 포함되어 있습니다. 플라스미드내의 MINX DNA 서열은 T3, T7 또는 SP6 RNA 폴리머라제 프로모터의 통제하에 있다. BamHI 제한 엔도넬리스를 가진 MINX 플라스미드 DNA의 선형화를 위한 재료 및 상세한 방법, α-32P[GTP]의 존재에 SP6 RNA 폴리머라제에 의한 전사 및 접합 을 위한 32P 표지 MINX의 정제는 이전에 설명된다(그 참조의 단계 2.2 및 3.2 참조).- -20°C에서 방사성 라벨이 부착된 MINX를 에탄올 침전으로 저장; 4-6 주 이내에 표시 된 접합 기판을 사용 하 여 전사 후.
- 사용하기 직전에, 원심분리기 32P-라벨 MINX에서 12,000 x g 에서 4°C에서 10분 동안 원심분리; 마이크로 파이프로 상체를 제거하고 폐기하십시오.
- 4°C에서 15분 동안 12,000 x g 에서 70% 에탄올과 원심분리기의 150 μL을 추가합니다. 상체를 버리고 15 분 동안 열없이 속도 진공 청소기로 펠릿을 건조시킵니다.
- DEPC 물의 50 μL에서 펠릿을 다시 수분화합니다. 2개의 GF/C 필터 각각에 2 μL을 점; 필터를 차가운 5% 트리클로로아세트산(TCA)에 10분 동안 담가 두는 다. 차가운 5% TCA로 헹구고, 진공 플라스크에 180방지 에탄올이 있습니다. 공기는 필터를 건조하고 안전 해결의 4mL에서 신경화 카운트에 따라.
- 32P 라벨MINX를 60% D에서 104 cpm/μL로 희석하여 접합 분석에 대해 희석합니다.
- 접합 반응의 어셈블리 ( 그림 1C 참조)
- 조립, 얼음에, 24 μL의 총 부피에서 접합 반응 (8 μL U1ΔNE (단계 2.2.6에서), 3.5 mM MgCl2, 1.5m ATP, 20mM 크레아틴 인산염, 0.5mM DTT, 20대 RNasin, 4 μL 32P 라벨MINX 접합 기판(104cpm /μL), 60% D) 및 각 비드 튜브에 3.2.7. U1ΔNE없이 24 μL의 총 부피로 접합 반응의 동일한 세트를 조립하고 단계 3.2.7에서 구슬의 각 튜브에 추가합니다.
- 12 μL 총 부피 (4 μL NE (D), 3.5 mM MgCl2, 1.5 mM ATP, 20 mM 크레아틴 인산염, 0.5 mM DTT, 20 단위 RNasin, 2 μL 32P 라벨 MINX 염자 기판 (10 pm /D4)에서 제어 접합 반응을 준비합니다.
- 튜브를 30°C에서 90분 동안 탭하여 부드럽게 섞습니다. 10-15s의 4°C에서 스윙 버킷 로터에서 1,000 x g 의 부드러운 원심분리로 혼합물을 펠렛합니다.
- 비드를 함유하는 튜브에 24 μL의 24 μL을 추가하고, 2x SDS 샘플 버퍼의 12 μL을 NE하지만 구슬을 함유한 대조관에 첨가하여 비드에서 단백질을 억제한다. 튜브를 100°C에서 7분 동안 가열합니다.
- 10-15s의 경우 4°C의 스윙 버킷 로터에서 튜브를 1,000 x g 로 부드럽게 원심분리합니다.
- 수퍼나티(elutions)를 신선한 마이크로튜브로 전송: 비드 튜브에서 약 48μL, NE 대조관으로부터 24 μL.
- 단백질을 소화하고 용해시키기 위해 Proteinase K(20 mg/mL)를 추가합니다: 구슬에서 48 μL 용출에 5 μL을 추가하고 24 μL NE 제어에 2.5 μL을 추가합니다.
- 튜브를 37°C에서 40분 동안 배양합니다.
- 튜브를 1,000 x g 의 스윙 버킷 로터에서 10초 동안 부드럽게 원심분리합니다.
- 39.5 μL TE 및 10 μL 3 M 나트륨 아세테이트로 비드 에출을 희석시하십시오. 63.5 μL TE 및 10 μL 3 M 아세테이트 나트륨으로 NE 제어를 희석시.
- 아래에 설명된 바와 같이 RNA를 추출하고 분석한다(섹션 4.3).
- 접합 반응의 제품 분석
- 페놀 클로로폼을 가진 각 샘플에서 RNA를 추출하고 클로로폼 이소아밀 알코올을 추출합니다. 에탄올, 원심분리기로 RNA를 침전시키고, 펠릿을 세척하고, 상체를 제거하고 2.3.6 및 2.3.7 단계에 설명된 것과 동일한 절차에 따라 펠릿을 건조시한다.
- RNA 적재 버퍼의 10 μL에서 건조 된 RNA 펠릿을 다시 중단, 부드럽게 소용돌이, 90 s에 대한 75-85 ° C에 가열, 다음 2 분 동안 얼음에 배양.
- 8.3M 우레아에서 13% 폴리아크릴아미드(bisacrylamide: 아크릴라미드, 1.9:50 [wt/wt])를 함유한 용액의 20mL를 준비; 이 용액을 사용하여 길이 15cm의 젤을 주조합니다.
- 젤이 주전되면 TBE를 실행 버퍼로 사용하여 20 분 동안 400 V에서 (적재된 샘플없이) 전기 phorese. 이 단계를 마친 후 TBE 실행 버퍼로 우물을 세척합니다.
- RNA 샘플을 적재하고, RNA 적재 버퍼에, 및 TBE 실행 버퍼를 3.5 에서 4 h에 대한 400 V로 전기 phorese. 전기 전구 후, 10 분 동안 증류수에 젤을 몰입하고 회전하여 우레아를 제거하십시오.
- 3M 필터 용지에 젤을 진공 건조시키고, 먼저 80°C에서 2시간 15분, 열 없이 30분 동안 천천히 식힙니다. 방사성 성분의 이동 위치를 감지하기 위해 필름에 사인 방사선 촬영에 건조 된 젤을 피사체.
Access restricted. Please log in or start a trial to view this content.
결과
NE는 항갈3(단계 3.2.7)에 의해 접합된 글리세롤 그라데이션 면역침전의 10S 영역에서 U1 snRNP(섹션 2.2.6에서 U1ΔNE) 및 Gal3-U1 snRNP 복합체를 접합 반응으로 혼합하였다. 이러한 반응 혼합물에는 U1 snRNA(도 2A, 레인 3)뿐만 아니라 U1 특이단백질, U1-70K(도 2B, 레인 3)가 함유되어 있었다. 예상대로, 항 갈3은 Gal3 (그림 2B
Access restricted. Please log in or start a trial to view this content.
토론
이 보고서는 항갈3 코팅 구슬에 갇힌 U1 snRNP 복합체가 사전 mRNA 기판에 결합할 수 있으며 이 삼차 복합체는 원래 갈라크토에 기초하여 분리된 단백질 의 가족 중 하나인 U1 snRNP 고갈된 NE. Gal3에 접합 활동을 복원할 수 있는 실험적 세부 사항을 제공합니다. . 초기 면역 형광 및 세포 전형 연구는 접합 기계의 구성 요소와 Gal3의 협회의 초기 힌트를 제공: 스네RNPs의 Sm 코어 폴리 펩티?...
Access restricted. Please log in or start a trial to view this content.
공개
저자는 공개 할 것이 없습니다.
감사의 말
이 작품은 국립 과학 재단 그랜트 MCB-0092919 및 미시간 주립 대학 교교 연구 보조금 09-CDFP-2001 (RJP) 및 건강 보조금 GM-38740 및 미시간 AgBioResearch 프로젝트 MICL02455 (JLW)에 의해 지원되었습니다.
접합 표식에 사용된 MINX 사전 mRNA 기판은 닥터 수잔 버겟 (미국 휴스턴, 텍사스, 텍사스) 박사수잔 버겟에게서 친절한 선물이었습니다.
Access restricted. Please log in or start a trial to view this content.
자료
| Name | Company | Catalog Number | Comments |
| anti-U1 snRNP | The Binding Site | Hu ENA-RNP #33471 | human autoimmune serum specific for U1 snRNP |
| bottle top vacuum filter | Fisher Scientific | Corning 431153 (0.22 μm; PES 150 ml) | for filtering solutions containing Tris |
| centrifuge | International Equipment Company | IEC Model PR-6 | for pelletting Sepharose beads in immunoprecipitation |
| diethylpyrocarbonate (DEPC) | Sigma-Aldrich | 159220-5G | for treatment of water used in preparation of all solutions |
| dimethylpimelimidate (DMP) | Sigma-Aldrich | 80490-5G | for cross-linking antibody to Sepharose beads |
| electrophoresis cell | BioRad Laboratories, Inc | Mini-Protean II | for SDS-PAGE separation of proteins |
| ethanolamine | Sigma-Aldrich | 411000-100ml | for blocking after the cross-linking reaction |
| gel electrophoresis system | Hoefer, Inc | HSI SE 500 Series | for separating snRNAs by gel electrophoresis |
| gel slab dryer | BioRad | Model 224 | for drying gel slabs for autoradiography |
| Hybond ECL membrane | GE Healthcare | RPN3032D (0.2 μm; 30 cm x 3 m) | for immunoblotting of proteins on membrane |
| microdialyzer (12 x 100 μl sample capacity) | Pierce | Microdialyzer System 100 | for exchanging the buffer of nuclear extract |
| microdialyzer membranes (8K cutoff) | Pierce | 66310 | for exchanging the buffer of nuclear extract |
| non-fat dry milk | Spartan Stores | Spartan Instant Non-fat Dry Milk | |
| Protein A Sepharose CL-4B | Millipore-Sigma | GE 17-0780-01 | for coupling antibody to beads |
| Proteinase K | Millipore-Sigma | P2308-5mg | for stopping the splicing reaction to isolate the RNAs |
| RNasin | Promega | N2111 | for inhibiting ribonuclease activity |
| rocker/rotator | Lab Industries, Inc | Labquake Shaker 400-110 | for mixing protein solutions in coupling reactions and in immunoprecipitation |
| Safety-Solve | Research Products International Corp. | No. 111177 | scintillation counting cocktail for determination of radioactivity in splicing substrate |
| scintillation counter | Beckman Instruments | LS6000SC | scintillation counter for determination of radioactivity |
| speed vaccum concentrator | Savant | SVC 100H | for drying ethanol-precipitated RNA pellets |
| Transphor electrophoresis unit | Hoefer, Inc | Hoefer TE Series Transphor | for protein transfer from SDS-PAGE to blotting membrane |
참고문헌
- Hoskins, A. A., Moore, M. J. The spliceosome: a flexible, reversible macromolecular machine. Trends In Biochemical Sciences. 37, 179-188 (2012).
- Choi, Y. D., Grabowski, P., Sharp, P. A., Dreyfuss, G. Heterogeneous nuclear ribonucleoproteins: role in RNA splicing. Science. 231, 1534-1539 (1986).
- Lerner, M., Steitz, J. A. Snurps and scyrps. Cell. 25, 298-300 (1981).
- Maniatis, T., Reed, R. The role of small nuclear ribonucleoprotein particles in pre-mRNA splicing. Nature. 325, 673-678 (1987).
- Hoskins, A. A., et al. Ordered and dynamic assembly of single spliceosomes. Science. 331, 1289-1295 (2011).
- Coppin, L., Leclerc, J., Vincent, A., Porchet, N., Pigny, P. Messenger RNA life-cycle in cancer: emerging role of conventional and non-conventional RNA-binding proteins. International Journal of Molecular Sciences. 19, 650-676 (2018).
- Dagher, S. F., Wang, J. L., Patterson, R. J. Identification of galectin-3 as a factor in pre-mRNA splicing. Proceedings of the National Academy of Sciences of the United States of America. 92, 1213-1217 (1995).
- Vyakarnam, A., Dagher, S. F., Wang, J. L., Patterson, R. J. Evidence for a role for galectin-1 in pre-mRNA splicing. Molecular and Cellular Biology. 17, 4730-4737 (1997).
- Wang, W., Park, J. W., Wang, J. L., Patterson, R. J. Immunoprecipitation of spliceosomal RNAs by antisera to galectin-1 and galectin-3. Nucleic Acids Research. 34, 5166-5174 (2006).
- Haudek, K. C., Voss, P. G., Locascio, L. E., Wang, J. L., Patterson, R. J. A mechanism for incorporation of galectin-3 into the spliceosome through its association with U1 snRNP. Biochemistry. 48, 7705-7712 (2009).
- Fritsch, K., et al. Galectin-3 interacts with components of the nuclear ribonucleoprotein complex. BMC Cancer. 16, 502-511 (2016).
- Conway, G. C., Krainer, A. R., Spector, D. L., Roberts, R. J. Multiple splicing factors are released from endogenous complexes during in vitro pre-mRNA splicing. Molecular and Cellular Biology. 9, 5273-5280 (1989).
- Dery, K. J., Yean, S. L., Lin, R. J. Assembly and glycerol gradient isolation of yeast spliceosomes containing transcribed or synthetic U6 snRNA. Methods in Molecular Biology. 488, 41-63 (2008).
- Yoshimoto, R., Kataoka, N., Okawa, K., Ohno, M. Isolation and characterization of post-splicing lariat-intron complexes. Nucleic Acids Research. 37, 891-902 (2009).
- Malca, H., Shomron, N., Ast, G. The U1 snRNP base pairs with the 5' splice site within a penta-snRNP complex. Molecular and Cellular Biology. 23, 3442-3455 (2003).
- Haudek, K. C., Voss, P. G., Wang, J. L., Patterson, R. J. A 10S galectin-3 - snRNP complex assembles into active spliceosomes. Nucleic Acids Research. 44, 6391-6397 (2016).
- Rappsilber, J., Ryder, U., Lamond, A. I., Mann, M. Large-scale proteomic analysis of the human spliceosome. Genome Research. 12, 1231-1245 (2002).
- Jurica, M. S., Moore, M. J. Capturing splicing complexes to study structure and mechanism. Methods. 28, 336-345 (2002).
- Patterson, R. J., Haudek, K. C., Voss, P. G., Wang, J. L. Examination of the role of galectins in pre-mRNA splicing. Methods in Molecular Biology. 1207, 431-449 (2015).
- Dignam, J. D., Lebovitz, R. M., Roeder, R. G. Accurate transcription initiation by RNA polymerase II in a soluble extract from isolated mammalian nuclei. Nucleic Acids Research. 11, 1475-1489 (1983).
- Agarwal, N., Sun, Q., Wang, S. Y., Wang, J. L. Carbohydrate-binding protein 35. I. Properties of the recombinant polypeptide and the individuality of the domains. Journal of Biological Chemistry. 268, 14932(1993).
- Zillmann, M., Zapp, M. I., Berget, S. M. Gel electrophoretic isolation of splicing complexes containing U1 small nuclear ribonucleoprotein particles. Molecular and Cellular Biology. 8, 814-821 (1988).
- Barondes, S. H., et al. Galectins: a family of animal β-galactoside-binding proteins. Cell. 76, 597-598 (1994).
- Laing, J. G., Wang, J. L. Identification of carbohydrate binding protein 35 in heterogeneous nuclear ribonucleoprotein complex. Biochemistry. 27, 5329-5334 (1988).
- Vyakarnam, A., Lenneman, A. J., Lakkides, K. M., Patterson, R. J., Wang, J. L. A comparative nuclear localization study of galectin-1 with other splicing components. Experimental Cell Research. 242, 419-428 (1998).
- Michaud, S., Reed, R. An ATP-independent complex commits pre-mRNA to the mammalian spliceosome assembly pathway. Genes & Development. 5, 2534-2546 (1991).
- Chiu, Y. -F., et al. Cwc25 is a novel splicing factor required after Prp2 and Yju2 to facilitate the first catalytic reaction. Molecular and Cellular Biology. 29, 5671-5678 (2009).
- Krishnan, R., et al. Biased Brownian ratcheting leads to pre-mRNA remodeling and capture prior to first-step splicing. Nature Structural and Molecular Biology. 20, 1450-1457 (2013).
- Gray, R. M., et al. Distinct effects on splicing of two monoclonal antibodies directed against the amino-terminal domain of galectin-3. Archives of Biochemistry and Biophysics. 475, 100-108 (2008).
Access restricted. Please log in or start a trial to view this content.
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유